自2019年12月8日起,在中国武汉爆发了以呼吸道为主要症状的肺炎,经二代测序、病毒分离和鉴定等手段,确定为一种新型的冠状病毒[1-2],世界卫生组织于2020年1月19日将其暂命名为2019新型冠状病毒(2019 Novel Coronavrius, 2019 nCoV)[3],于2020年2月11日正式命名为SARS-CoV-2病毒,引起的疾病称为2019冠状病毒病(COVID-19)[4]。至2020年2月13日,中国新型冠状病毒肺炎确诊59 895例,疑似病例16 067例,死亡1367例;中国以外492例,波及日本、泰国等24个国家[5];随着诊断试剂的足量应用和疫情的进展,确诊病例数量可能更进一步增加,严重威胁到人们的生活和生命健康[6]。因此,在2020年1月30日,世界卫生组织突发事件委员会经过讨论决定:将此次新型冠状病毒感染疫情确认为“国际关注的突发公共卫生事件”[7],以保护其他国家,尤其是为了保护一些医疗系统比较脆弱的国家,以便这些国家对此肺炎疫情进行更好的预防和控制。该病成为2009年H1N1、2014年脊髓灰质炎、2014年埃博拉、2016年寨卡、2019年埃博拉疫情之后[8],第6起国际关注的突发公共卫生事件。由此可见,此次新型冠状病毒疫情的严重性。
在疫情的进行中,中国的科学家对病毒特征、疾病的特征进行了很好的解析和描述[9-10],为疾病的防控打下了坚实的基础。病毒的溯源,推测是由蝙蝠作为病毒库传播而来[11],至于有无中间宿主、中间可能的宿主是什么还待于进一步研究确定。在病毒疫情进行中,有一项重要的研究内容是:病毒进化过程起源时间,疫情进展中病毒的变异情况如何;这个内容对病毒疫情防控也具有重要的意义。本文根据GISAID中提交的序列(截止到2020年1月29日)分析病毒进化的相关情况。
1 材料和方法 1.1 病毒株序列从https://www.gisaid.org/上下载关于SARS-CoV-2序列,共计45条全长基因组序列。其中39条序列用于病毒最近共同祖先时间的计算(tMRCA)。其它冠状病毒序列从NCBI序列数据库中下载。
1.2 进化树构建、序列相似性分析软件MEGA-X下载于https://www.megasoftware.net/;tMRCA相关分析软件组合BEAUti、BEAST、TreeAnnotator下载于http://www.beast2.org/, 从http://tree.bio.ed.ac.uk/software/下载进化树数据显示软件Tracer v1.6、进化树显示软件FigTree、进化分子钟检测软件TempEst。
1.3 进化树构建方法将序列导入MEGA-X中,应用软件找到最佳核苷酸替代模型参数,并应用此参数构建进化树和tMRCA的计算,进化树的可靠性检验采用自展法(Bootstrap= 1000次重复);病毒采样时间导入BEAUti中,以2020年1月23日作为0时间,以天为单位,以病毒株BetaCoV/ Wuhan/IPBCAMS-WH-02/2019|EPI_ISL_403931作为局外群(outgroup,树根),进行推算tMRCA。
2 结果 2.1 基因组序列基本信息病毒序列用二代测序或结合三代测序测定,测序长度为29 688~29 899 bp之间[12],全部涵盖了病毒的编码区。病毒编码的结构蛋白S、E、M和N蛋白,非结构蛋白ORF1a、ORF1ab、ORF3等在文献和数据库中有详细的解析[13-14]。
39株SARS-CoV-2全长基因组核苷酸序列经比对后,与SARS冠状病毒(SARS-CoV)、中东呼吸综合征(MERS CoV)的序列相似性分别平均为78.7%和48.7%,与来自于蝙蝠冠状病毒株bat-SL-CoVZC45 (简写CoVZC45)和bat-SL-CoVZXC21 (简写CoVZXC21)较为接近,相似性为87.5%和87.3%,与BetaCoV/bat/ Yunnan/RaTG13/2013 (简写RaTG13)最为接近,相似性为95.9%。其他冠状病毒之间差异较大,在48.0%~ 87.4%之间,也决定了它们在进化上分属于不同的病毒属(表 1)。
| 表 1 39株SARS-CoV-2病毒与其他冠状病毒核苷酸的相似性比较 Tab.1 Comparison of nucleotide similarities between 39 SARS-CoV-2 isolates and other coronaviruses (%) |
各冠状病毒编码蛋白质ORF1ab、S、E、M和N的氨基酸相似性性如表 2~6所示。选用序列MN908947作为参考株进行比较,结果与基因组的核苷酸相似性一致。ORF1ab、S、M和N蛋白与RaTG13、CoVZC45、CoVZXC21相似性最高,其次是SARS-CoV,与MERS CoV相似最低,结合后面进化树分析的结果,在同一亚属内有着高的相似性,不同亚属之间的相似性较低。值得注意的是E蛋白高度保守,SARS-CoV-2与RaTG13、CoVZC45、CoVZXC21完全一致,与SARS-CoV仅有4个氨基酸的差别。
| 表 2 冠状病毒ORF1ab的氨基酸相似性 Tab.2 Amino acid similarities of ORF1ab among different coronaviruses (%) |
| 表 3 冠状病毒S蛋白的氨基酸相似性 Tab.3 Amino acid similarities of the S protein among different coronaviruses (%) |
| 表 4 冠状病毒E蛋白的氨基酸相似性 Tab.4 Amino acid similarities of the E protein among different coronaviruses (%) |
| 表 5 冠状病毒M蛋白的氨基酸相似性 Tab.5 Amino acid similarities of the M protein among different coronaviruses (%) |
| 表 6 冠状病毒N蛋白的氨基酸相似性 Tab.6 Amino acid similarities of the N protein among different coronaviruses (%) |
应用MEGA-X软件寻找最佳核苷酸替代模型模块,以BIC (Bayesian Information Criterion)最小值为最佳核苷酸替代模型参数,显示TN93模型为最优。以TN93替代模型、NJ (Neighbor joint)法构建全基因组进化树,结果如下图 1A所示。根据国际病毒分类委员会的分类,目前冠状病毒的分类是Alpha、Beta、Gamma和Delta[15]。从图 1中,我们可以看到SARS-CoV-2与BetaCoV/bat/Yunnan/RaTG13/2013、bat-SL-CoVZC45和bat-SL-CoVZXC21形成了sarbecovirus属的一个分支,而SARS-CoV等病毒构成了sarbecovirus属的另一个分支。
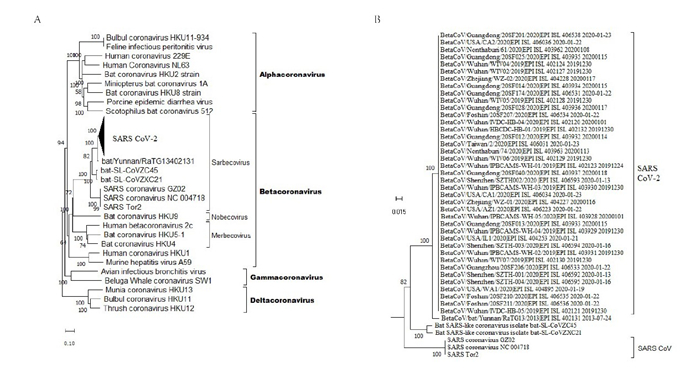
|
图 1 SARS-CoV-2的全基因组进化分析 Fig.1 Phylogenetic analysis of the full-length genome of SRAS-CoV-2. A: Whole genome evolutionary tree of SARS-CoV-2 virus strains(compressed in triangle); B: Unfolded evolutionary tree of 38 isolates of SARS-CoV-2 virus. |
应用MEGA-X软件构建进化树,导入进化时间信号分析软件TempEst中,分析是否具有进化上的时间信息。如图 2A所示,显示有微弱正向进化信号,粗估时钟速率是2.24×10-6substitution/site/day,tMRCA为-131 d,R2=0.08。在此基础上,我们应用BEAST软件进行精细评估tMRCA。如图 2B所示,tMRCA平均时间是73.0 d (2019年11月10日),95%可信区间是38.9~119.3 d,后验概率是100%。结果提示病毒的出现日期在2019年9月23日~2019年12月15日之间。这与文献中报道的第1例病例在2019年12月1日出现也是相吻合的。
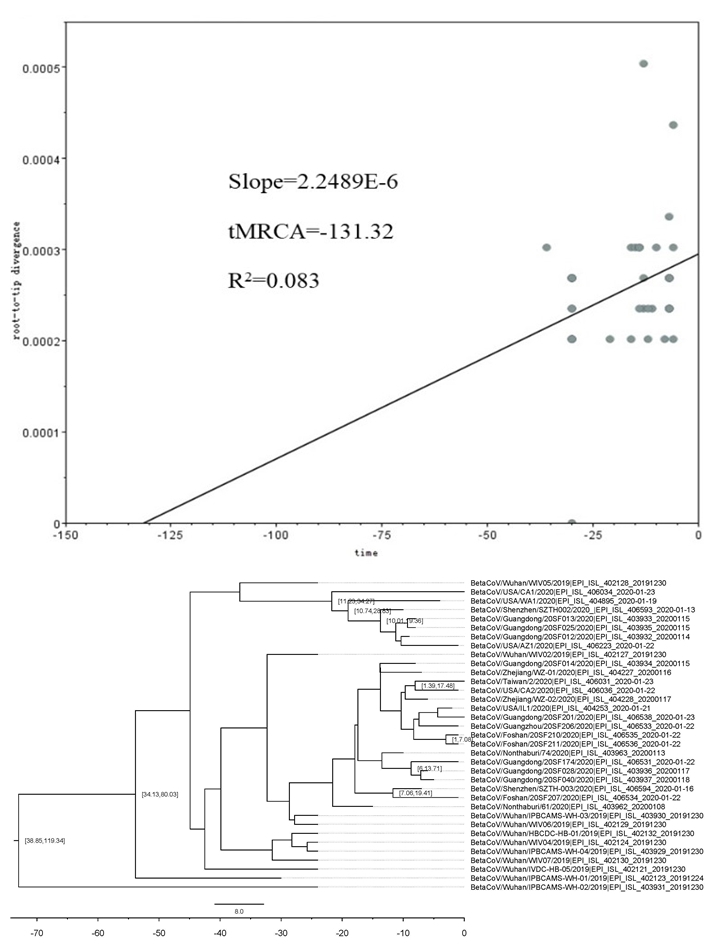
|
图 2 SARS-CoV-2病毒的tMRCA计算 Fig.2 tMRCA of SARS-CoV-2. A: Calculation of evolutionary time signals of SARS-CoV-2 virus. The point indicates the genetic distance of each isolate of the virus from the reference strain (outgroup, tree root) on the time scale; B: Analysis of tMRCA of SARSCoV-2 virus using BEAST. |
应用进化时间信号分析软件TempEst检测SARSCoV- 2与BetaCoV/bat/Yunnan/RaTG13/2013、bat- SL-CoVZC45、SARS-CoV之间是否有进化上的时间关系。结果显示:SARS-CoV-2与BetaCoV/bat/Yunnan/ RaTG13/2013不存在时间进化关系,斜率为-1.8×10- 5 substitution/site/day,R2=0.998 (图 3A),说明在自然界自然进化中是很难实现的,然而与CoVZC45、SARS-CoV确存在着明显的正性时间进化信号(图 3B、C)。
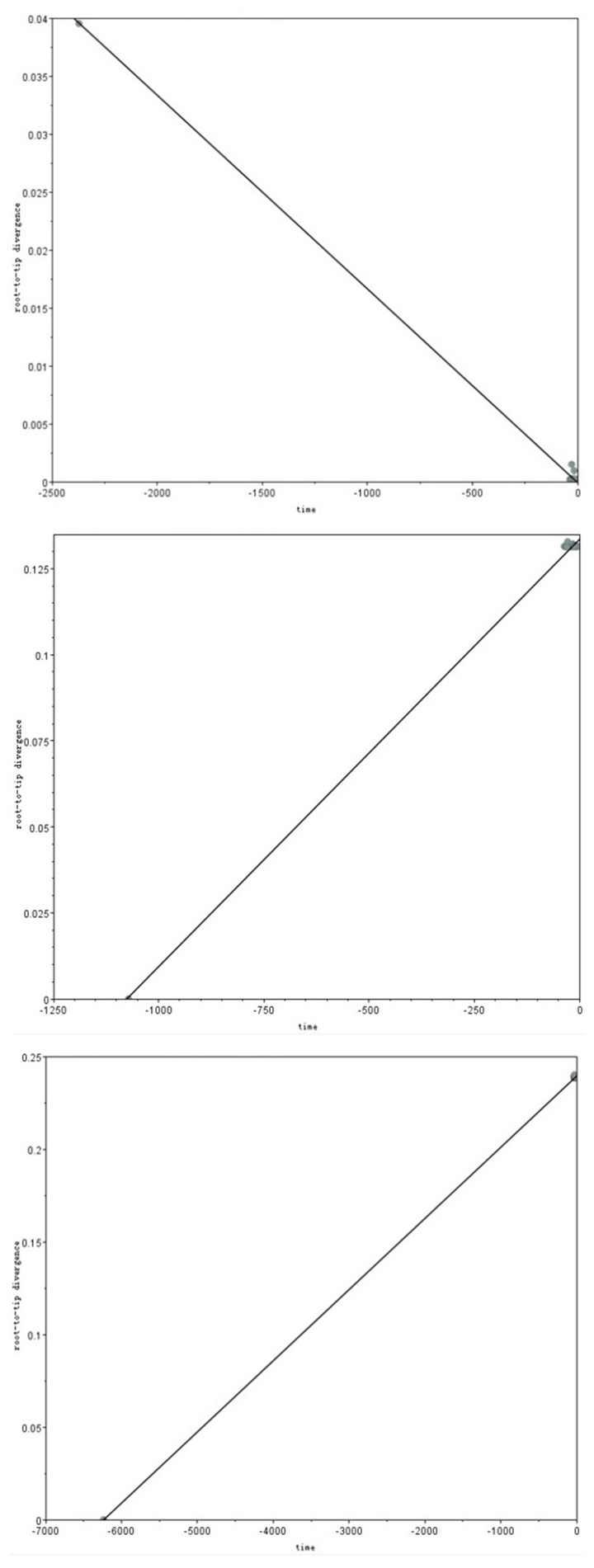
|
图 3 SARS-CoV-2病毒与其他冠状病毒进化时间信号检测 Fig.3 Temporal signal test of molecular phylogenies of SARS-CoV-2 together with other coronaviruses. A, B, and C: Evolution time signal detection results of SARS-CoV-2 virus strain and RaTG13, CoVZC45, and SARS-CoV viruses, respectively. |
我们将38株序列进行比较,核苷酸之间变化差异非常小,以最早测定的序列BetaCoV/Wuhan/ IPBCAMS- WH- 01/2019|EPI_ISL_402123为参考序列,共计有117处变异,如图 4所示,变异位点没有聚集性,相对均匀地分布整个基因组。从中也可以看出,有3个位点的变异(发生在ORF1ab上),稳定地表达在其它36株病毒中,同时有多个位点在病毒株序列中聚集。
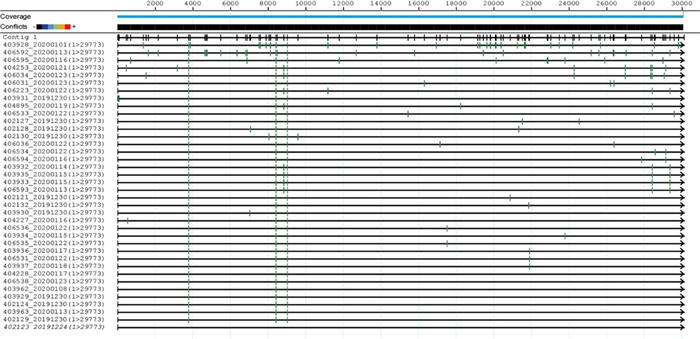
|
图 4 38株病毒序列变异 Fig.4 Variation of 38 SARS-CoV-2 strains. Each green vertical line represents the the location of variation in the genome, using the earliest BetaCoV/Wuhan/IPBCAMS-WH-01/2019|EPI_ISL_402123 20191224 as the reference sequence. |
我们将变异序列导入http://www.datamonkey.org/中,分析其变异是否存在选择压力。如表 7所示,参与病毒复制相关蛋白有48处变异位点,采用固定效应似然比检验模型(FEL)发现有4处存在净化选择,而采用相对保守的方法单一似然祖先计数(SLAC)法只能检出一个位点。在S蛋白、M蛋白和N蛋白中只能用FEL法检出具有统计学意义的2~3种净化选择,在N蛋白中检出1种正向选择进化。
| 表 7 SARS-CoV-2病毒主要蛋白的选择进化压力分析 Tab.7 Analysis of selection pressure of the main proteins of SARS-CoV-2 |
自2019年12月初起,从武汉流行的新现的冠状病毒[8],至2020年2月,引发了至目前为止全中国6万人的感染,病死人数1100余人,对人们健康危害已经远超2003年前后的SARS流行[16-17]。该病毒由于与SARSCoV病毒有较高的相似性,与SARS-CoV同属于β冠状病毒属的Sarbecovirus亚属(图 1),以及对人们的健康产生严重影响,国际病毒命名委员会将其命名为SARSCoV-2,与SARS-CoV是姊妹病毒[18-19]。世界卫生组织将该病毒引起的疾病称为COVID-19。这也是目前发现第7种能使人致病的冠状病毒[20]。
该病毒具有比SARS-CoV更强的传播力、更长的潜伏期,这是病毒广泛感染人的基础[21-22]。尽管病毒的基因组在疾病流行期间很快被解析[2],有多篇文献报道了其基因组特征[23-24],但这些特征还难以解释病毒的强传播力[8]。影响病毒播散的另外一些重要因素是病毒的溯源,包括:病毒出现的时间,是否有中间宿主,传染源是什么,以及传染源是否一直存在。科学家根据现存数据库中病毒序列信息,以及当前的溯源研究,推测是来源于菊头蝙蝠[25]。
关于病毒出现的可能时间,我们应用进化分析软件TempEst分析当前基因组数据,结果提示已经出现了分子钟事件信号,这是后面进行tMRCA计算前提。在此基础上,我们应用BEAST软件推算病毒可能出现的时间为38.9~119.3 d (从1月23日往前推),也就是在2019年9月23日~2019年12月15日之间。这也提示目前武汉市高感染原因之一。我们注意到,本文中关于tMRCA的推算因为时间跨度太短(只有30 d),可能结果并不是很准确,但也提供了一定的信息。
关于病毒的来源,目前与SARS-CoV-2相似性比较高从蝙蝠中分离到的病毒是RaTG13 (云南,2013年)、bat- SL- CoVZC45和bat- SL- CoVZXC21(2017年,浙江),其中相似性最高的是RaTG13病毒株。如果SARS-CoV-2是来自于RaTG13病毒株,那么RaTG13病毒就应与SARS-CoV-2具有时间进化信号。我们检测了两种病毒时间进化上的信息,发现呈负相关(进化速率是负的)(图 3A);如果SARS-CoV-2演化于RaTG13病毒,应该是正性进化速率,因此我们推测:SARS-CoV-2来源于RaTG13病毒的可能性不大,尽管它们之间有较高的相似性。我们随后演算了SARS-CoV-2与bat-SLCoVZC45和SARS-CoV之间的进化关系,发现它们具有很明显的正性相关关系(图 3B、C)。提示SARS-CoV-2的产生与bat-SL-CoVZC45等冠状病毒有一定的关系。这一点仍需更多的实验进行支持。
病毒在流行过程中,是否产生对抗外界压力的适应力,也是影响病毒传播能力一个重要因素[26],从临床的数据显示,病毒的传播确是不断增加[27]。我们通过全基因组数据选择进化压力分析,结果显示病毒表现一种进化选择,在高度保守的基因内,发生多频次的同义替代,这种替代远高于非同义替代,这些替代的位置可能对病毒的生命活力具有重要的影响。继续加强这些位点的监测,将有助于解析SARS-CoV-2强传播能力,对后期的防控具有一定的指导作用。
致谢: 衷心感谢众多研究者将研究数据提交到GISAID中,为本文奠定了基础!| [1] |
Wu F, Zhao S, Yu B, et al. A new coronavirus associated with human respiratory disease in China[J]. Nature, 2020. DOI:10.1038/s41586-020-2008-3 |
| [2] |
Zhou P, Yang XL, Wang XG, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin[J]. Nature, 2020, 10. |
| [3] |
Rubin EJ, Baden LR, Morrissey S, et al. Medical Journals and the 2019-nCoV Outbreak[J]. N Engl J Med, 2020, 10. |
| [4] |
Guarner J. Three emerging coronaviruses in two decades[J]. Am J Clin Pathol, 2020, aqaa029. |
| [5] |
Stein RA. The 2019 Coronavirus: Learning Curves, Lessons, and the Weakest Link[J]. Int J Clin Pract, 2020, e13488. |
| [6] |
Velavan TP, Meyer CG. The COVID-19 epidemic[J]. Trop Med Int Health, 2020, 10. |
| [7] |
World Health Organization. Statement on the second meeting of the international health regulations (2005) emergency committee regarding the outbreak of novel coronavirus (2019-nCoV). Retrieved from https://www.who.int/news-room/detail/30-01-2020-statementon-the-second-meeting-of-the-international-health-regulations-(2005)-emergency-committee-regarding-the-outbreak-of-novel-coronavirus-(2019-ncov).
|
| [8] |
Wang FS, Zhang C. What to do next to control the 2019-nCoV epidemic?[J]. Lancet, 2020, 395(10222): 391-3. DOI:10.1016/S0140-6736(20)30300-7 |
| [9] |
Chen Y, Liu Q, Guo D. Emerging coronaviruses: Genome structure, replication, and pathogenesis[J]. J Med Virol, 2020, 10. |
| [10] |
Chen ZM, Fu JF, Shu Q, et al. Diagnosis and treatment recommendations for pediatric respiratory infection caused by the 2019 novel coronavirus[J]. World J Pediatr, 2020, 10. |
| [11] |
Benvenuto D, Giovanetti M, Ciccozzi A, et al. The 2019-new coronavirus epidemic: Evidence for virus evolution[J]. J Med Virol, 2020, 10. |
| [12] |
Li X, Wang W, Zhao X, et al. Transmission dynamics and evolutionary history of 2019-nCoV[J]. J Med Virol, 2020, 10. |
| [13] |
Wu A, Peng Y, Huang B, et al. Genome composition and divergence of the novel coronavirus (2019-nCoV) originating in China[J]. Cell Host Microbe, 2020, S1931-3128(20)30072-X. |
| [14] |
Ceraolo C, Giorgi FM. Genomic variance of the 2019-nCoV coronavirus[J]. J Med Virol, 2020, 10. |
| [15] |
Phan T. Novel coronavirus: From discovery to clinical diagnostics[J]. Infect Genet Evol, 2020, 79: 104211. DOI:10.1016/j.meegid.2020.104211 |
| [16] |
Riou J, Althaus CL. Pattern of early human-to-human transmission of Wuhan 2019 novel coronavirus (2019-nCoV), December 2019 to January 2020[J]. Euro Surveill, 2020, 25(4): 2000058. |
| [17] |
Cowling BJ, Leung GM. Epidemiological research priorities for public health control of the ongoing global novel coronavirus (2019-nCoV) outbreak[J]. Euro Surveill, 2020, 10. |
| [18] |
Chen L, Liu W, Zhang Q, et al. RNA based mNGS approach identifies a novel human coronavirus from two individual pneumonia cases in 2019 Wuhan outbreak[J]. Emerg Microbes Infect, 2020, 9(1): 313-9. DOI:10.1080/22221751.2020.1725399 |
| [19] |
Malik YS, Sircar S, Bhat S, et al. Emerging novel Coronavirus (2019-nCoV) - Current scenario, evolutionary perspective based on genome analysis and recent developments[J]. Vet Q, 2020, 1-12. |
| [20] |
Zhu N, Zhang D, Wang W, et al. A Novel Coronavirus from Patients with Pneumonia in China, 2019[J]. N Engl J Med, 2020, 10. |
| [21] |
Wu JT, Leung K, Leung GM. Nowcasting and forecasting the potential domestic and international spread of the 2019-nCoV outbreak originating in Wuhan, China: a modelling study[J]. Lancet, 2020, S0140-6736(20): 30260-9. |
| [22] |
Chen N, Zhou M, Dong X, et al. Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study[J]. Lancet, 2020, 395(10223): 507-13. DOI:10.1016/S0140-6736(20)30211-7 |
| [23] |
Chan JF, Kok KH, Zhu Z, et al. Genomic characterization of the 2019 novel human-pathogenic coronavirus isolated from a patient with atypical pneumonia after visiting Wuhan[J]. Emerg Microbes Infect, 2020, 9(1): 221-36. DOI:10.1080/22221751.2020.1719902 |
| [24] |
Paraskevis D, Kostaki EG, Magiorkinis G, et al. Full-genome evolutionary analysis of the novel corona virus (2019-nCoV) rejects the hypothesis of emergence as a result of a recent recombination event[J]. Infect Genet Evol, 2020, 79: 104212. DOI:10.1016/j.meegid.2020.104212 |
| [25] |
Lu R, Zhao X, Li J, et al. Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding[J]. Lancet, 2020, S0140-6736(20): 30251-8. |
| [26] |
Wilder-Smith A, Freedman DO. Isolation, quarantine, social distancing and community containment: pivotal role for old-style public health measures in the novel coronavirus (2019-nCoV) outbreak[J]. J Travel Med, 2020, taaa020. DOI:10.1093/jtm/taaa020 |
| [27] |
Liu Y, Gayle AA, Wilder-Smith A, Rocklöv J. The reproductive number of COVID-19 is higher compared to SARS coronavirus[J]. J Travel Med, 2020, taaa021. DOI:10.1093/jtm/taaa021 |
 2020, Vol. 40
2020, Vol. 40

