3. 南方医科大学公共卫生学院,广东 广州 510515;
2. 华南理工大学食品科学与工程学院,广东 广 州 510640;
4. 广州市三兴生物技术有限公司,广东 广州 510000;
5. 中山大学附属第一医院,广东 广州 510080
3. School of Public Health, Southern Medical University, Guangzhou 510515, China;
2. College of Light Industry and Food Sciences, South China University of Technology, Guangzhou 510640, China;
4. Guangzhou Sanxing Biotechnology Co., Ltd., Guangzhou 510000, China;
5. First Affiliated Hospital, Sun Yat- sen University, Guangzhou 510080, China
自噬是指一个细胞吞噬自身蛋白或细胞器并使其包裹进入囊泡,溶酶体再将这些物质吞噬、降解,从而实现细胞代谢和细胞器更新的一个过程。自噬有利于维持细胞内环境稳态及维持细胞存活,是一种细胞面临营养缺乏或氧化应激状态时的保护机制[1-3]。自噬可分为三类:巨自噬、微自噬以及分子伴侣介导自噬(CAM)[4]。自噬主要包括起始、延伸、成熟、自噬体与溶酶体融合和降解等步骤(图 1)[5]。在自噬过程的第一步中,Atg1/ ULK1复合物(包括Atg1、Atg13、Atg17等)参与调控自噬体的形成;细胞收到诱导自噬信号后,在Atg12-Atg5- Atg16L复合体等物质的调控下,在胞浆中形成分离膜,即前自噬体。前自噬体继续在Atg12-Atg5-Atg16L复合体和微管相关蛋白1A/1B轻链3(LC3;Atg8)的作用下,不断拓展膜的长度,从而形成成熟的闭合自噬泡;接着,自噬体被运输到溶酶体,两者的生物膜相互融合,形成自噬溶酶体;自噬溶酶体中的酸性水解酶可以降解囊泡中的水解物,成为降解性自噬体[6-10]。
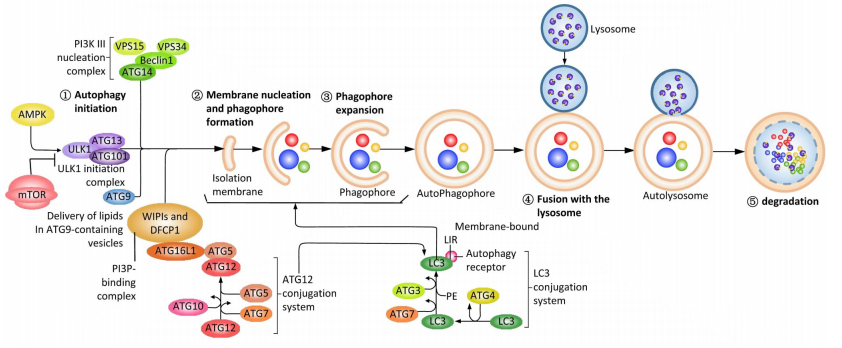
|
图 1 自噬的形成过程[11] Fig.1 The biological process of autophagy. |
在过去的10年中,大量研究发现,为了调节脂质储存的异常,机体会发生一种选择性的自噬形式——脂噬,这种自噬专门针对一种独特的中性脂质储存细胞器——脂滴(LDs)。在肝脏中,这种特殊的自噬途径可能与传统的脂肪酶驱动的脂质分解作用在脂质循环中发挥着同样重要的作用。进一步研究发现,脂噬作用不仅存在于肝脏中,在胰腺和脂肪组织中也同样观察到了这种特殊的自噬作用。这篇综述中,我们重点介绍了脂噬与主要脏器(肝脏、胰腺、脂肪组织)脂质代谢的关系,并归纳总结调控脂噬作用的分子机制,为脂肪肝、肥胖等疾病的防治提供理论基础。
1 脂噬与脂质代谢的关系脂肪酸在细胞内可酯化成甘油三酯和胆固醇酯,减轻游离脂肪酸对细胞的毒性[12-13]。这一合成代谢反应的发生场所为内质网,它会导致在内质网双层膜之间的一个透镜状的微域内不断沉积中性脂质[13-14],最终导致内质网双层的外叶扩张,形成一种独特的球形细胞器,即脂滴。LDs是一种复杂的亚细胞器,它以前被认为是脂肪固定的惰性位点,现在已知它具有独特的、动态的蛋白质组。这些蛋白质组随着细胞代谢状态的变化而波动,这可能对细胞器内中性脂质的组成有一定的决定作用[15-21]。
自噬对各种细胞器降解作用在上世纪就已经广为人知,但其对LDs的降解作用在近些年才深入研究。在饥饿导致机体营养缺乏的时候,细胞内储存在LDs的脂质以甘油三酯的形式水解成脂肪酸从而产生能量。与此同时,饥饿还会引起一个细胞反应——诱导自噬。LDs可以与自噬小体结合,使其中的游离脂肪酸的释放,这些脂肪酸还可以作为线粒体β型氧化的燃料[22-24](图 2)。细胞内的脂质通过自噬体转运至溶酶体分解的过程,称为“脂噬作用”,常简称为脂噬,这为调节细胞脂质水平提供另一种潜在的途径。
|
图 2 脂噬和脂质代谢之间的关系[25] Fig.2 The relationship between autophagy and lipid metabolism |
肝脏是人体脂肪酸包装、补充以及加工的中心场所。因此,肝脏出现功能障碍会导致脂肪的含量异常。其中,非酒精性脂肪肝(NAFLD)就是个典型事例。NAFLD是一系列的疾病,它可以导致脂肪变性到脂肪肝甚至肝硬化[26]。自噬对于脂肪肝的影响在前后两个时期有不同的体现。在前期,自噬主要通过降解细胞内LDs的途径来缓解脂肪变性。Zhang等[27]发现在支链氨基酸和高脂饲养的小鼠模型中,虽然支链氨基酸饲养后能使高脂饲养饲养小鼠的体质量明显降低,并且肝内的甘油三酯含量也有所降低,但支链氨基酸激活了mTOR使得肝脏的自噬作用受到抑制,使NAFLD恶化。在后期,自噬过度的激活,也可以诱发细胞死亡,加重病情[28]。因此在NAFLD发病的不同阶段对自噬采取相应的手段更有利NAFLD的治疗。
在最近的10年间,吞噬肝脏LDs的调节机制已经逐渐成型(图 3)[29-33]。Singh等[22]为自噬和肝脂代谢之前的关系提供了第1个具体的实验证据。他们用溶酶体抑制剂3-甲基腺嘌呤和小干扰RNA(siRNA)介导的自噬调节基因Atg5的下调处理肝细胞,其结果为肝细胞中TAG含量增加,且对接下来线粒体的β型氧化有一定的干扰。此外,免疫荧光成像显示的自噬机制的关键成分与LDs密切相关。而在电镜下观察发现,禁食6 h的小鼠肝细胞的双膜自噬小体内含有可检测的脂质含量,这是发生巨自噬的一种标志。为了证实小鼠体内确实发生了巨自噬,又检测了一只肝细胞特异性敲除Atg7基因的小鼠,发现与对照组小鼠相比,实验组的小鼠肝脏肿大,TAG随着饥饿程度逐渐积累[22]。
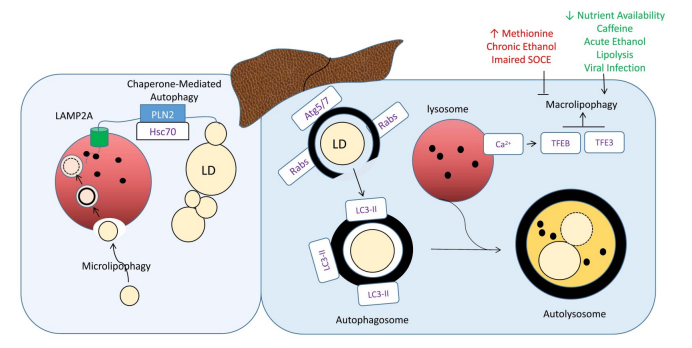
|
图 3 肝脏中的脂噬作用[45] Fig.3 Lipophagy in the liver |
为了进一步验证Singh等人的研究,一份报告揭示了分子伴侣介导自噬(CMA)和巨自噬在清除肝脏LDs方面有潜力[34]。CMA是一种直接内化具有典型KFERQ基序的特定蛋白底物的机制[35-37]。在肝细胞中,LDs所处的围磷脂蛋白PLIN2和PLIN3 [38-40]被检测含有这些基序,并显示与热休克同源蛋白70(Hsc70)直接相互作用。Hsc70是CMA的特异性伴侣,用于介导蛋白质传递到溶酶体[34, 41]。PLIN2中这一五肽基序的突变导致了LDs的积累,这表明去除LDs周围的包被是脂解作用和经典脂噬作用的先决条件。
目前已证实脂解作用和经典脂噬作用之间存在协同和交叉作用。例如,脂肪酶驱动的LDs分解被证明其部分过程需要依赖于自噬作用[42]。由于观察到关键的细胞质脂肪酶ATGL和激素敏感型脂肪酶都含有能与LC3直接相互作用的基序,推测由此导致的肝脂吞噬的上调与脂解作用存在协同作用。同样的,ATGL活性也可能是调节脂质吞噬作用的一个重要检查位点,ATGL的过表达可能会通过Sirtuin-1的机制增加肝脏的脂肪吞噬作用[43-44]。因此,肝细胞脂质代谢的一个关键研究领域就是对脂噬作用的调控。
1.2 胰腺中的脂噬作用脂噬同样也存在于胰腺细胞中。脂噬对胰腺细胞内的细胞器具有调控功能,保证其内环境的稳定和分泌功能的正常运行[46],同时还在各种细胞器(内质网、酶原颗粒、溶酶体、线粒体等)的协调与帮助下,共同维持胰腺细胞蛋白的高水平合成与分泌[47]。
β细胞为胰腺内分泌部分(胰岛)中能分泌胰岛素的细胞,高脂刺激诱导β细胞自噬活性的改变可以用来研究胰腺中的脂噬作用。β细胞暴露在游离脂肪酸(FFA)中,会引起细胞脂质沉积和胰岛素分泌受损,同时还会激活它的自噬功能,这揭示了自噬活性与胰腺脂质积累之间的相关性[48-49]。有小鼠模型实验证实,高脂饮食喂养的老鼠体内β细胞的自噬水平较对照组高[50-51]。高脂诱导胰腺自噬的机制可能是细胞高脂应激或脂肪酸氧化水平降低时,大量FFA进入胰腺,β细胞无法及时将所有的FFA氧化,过量的FFA则酯化为TG,导致产生大量LDs并沉积于胰腺组织中。临床数据显示,当糖尿病组患者的胰腺β细胞暴露于FFA中,其溶酶体相关膜蛋白2A(LAMP2)和组织蛋白酶B、D的转录水平较对照组有所下降,这表明了糖尿患者胰腺β细胞的自噬水平升高,但同时该细胞的死亡数目增加[52]。上述研究表明,升高自噬水平是胰腺脂代谢异常时的一种代偿反应。在一定程度上激活自噬的功能对增强胰岛素敏感性和抑制胰腺脂质沉积有重要意义,但其具体机制还需要进一步研究。
1.3 脂肪组织中的脂噬作用脂肪组织是由大量的脂肪细胞聚集而成,根据脂肪细胞结构和功能的不同,体内的脂肪组织可以分为白色脂肪(WAT)和棕色脂肪(BAT)两种:WAT能够将体内多余的能量以中性脂肪的形式储存,在机体急需时释放;BAT细胞中含有大量的线粒体,它们主要以β型氧化降解脂肪酸为细胞供能。
脂噬作用具有调控脂肪细胞的分化的功能。Hahm等[53]发现在小鼠3T3-L1前脂肪细胞和鼠胚胎成纤维细胞的正常分化过程中,存在LC3-Ⅱ的表达、AMPK的活性以及酸性囊泡的形成。实验中,在抑制小鼠细胞自噬后发现,小鼠前脂肪细胞的LDs聚集受阻,从而导致前脂肪细胞分化成为脂肪细胞的过程被抑制。除此之外,在特异性敲除自噬基因Atg7、对其使用沉默自噬基因或者自噬抑制剂的小鼠体内,可以观察到它们的WAT均表现出BAT的典型特征(增强BAT功能,提高胰岛素敏感性),但是脂噬是否能调节棕色脂肪组织的早期发育还是未知的[22, 53]。敲除Atg7的小鼠体内棕色脂肪分化和功能出现障碍,小鼠体温升高,脂肪酸氧化增强,肌肉量与葡萄糖耐受性降低[54]。这些现象表明了脂噬是白色脂肪细胞分化的必备条件,抑制脂噬就可以抑制WAT的分化。
除此之外,还有实验发现,盐皮质激素受体(MR)有调控脂肪细胞的功能,Armani等[55]发现,使用MR拮抗剂和DRSP处理高脂肪食物喂养的小鼠90 d,可以使它们的葡萄糖耐受性得到提高,从而阻止了体质量的增加和WAT的扩张,并且内脏和腹股沟处的BAT标志因子显著提升,解偶联蛋白(UCPI)水平显著提升,WAT中出现棕色脂肪细胞。这一实验现象表明MR可以通过脂噬调控WAT中棕色脂肪的重塑。已经有研究发现,脂肪组织中脂噬可以通过调节脂肪细胞的分化来调节脂质平衡[56],自噬激活可以诱导前脂肪细胞朝白色脂肪细胞方向分化,而脂噬被抑制则会使前脂肪细胞朝棕色脂肪细胞方向分化[57]。以上研究表明脂噬与脂肪组织的分化密切相关,但脂噬调控前脂肪细胞分化的机制目前尚不明确,有待进一步研究。
2 调节脂噬作用的分子机制尽管LDs在脂质组成、大小和细胞位置方面有所不同,但可能最能突出其特征的是它们的蛋白质组。LDs的表面包含数百个影响LDs代谢和信号传导的蛋白。这些蛋白具有调节LDs的特异性功能,包括脂噬。
2.1 Rab GTPase小型Rab GTPase是脂溶性调节蛋白中一个很前沿的例子。它作为Ras超家族单体G蛋白的一部分,以在膜转运活动中担任主配位体而被人熟知[58]。Rab GTPase作为分子开关,在鸟嘌呤交换因子(GEFs)和GTPase活化蛋白的帮助下,在GTP(活性)结合物和鸟嘌呤二磷酸结合物(非活性)状态之间进行转换。所有Rab蛋白都会在C端半胱氨酸残基处发生烯基化反应,这是将Rab蛋白插入目标膜的必要准备。活性Rab蛋白一旦到达作用位点,就会与特异性效应蛋白相互作用,从而调节囊泡的形成、运动、栓系和融合[58-59]。由于脂噬是一个依赖于配位膜转运活动的过程,因此有许多的Rab蛋白被确定为LDs蛋白的一部分[17-18, 60-61]。但是,有很多与LDs相关的Rab GTPase所扮演的角色仍未明确。下面,我们主要讨论Rab7、Rab10、Rab18和Rab32在脂噬过程中发挥的作用。
(1)Rab7:Rab7的主要功能是介导多泡体和溶酶体在脂质吞噬过程中向LDs的补充。Rab7主要存在于多泡体和晚期核内体上,它参与了晚期内吞室的物质运输以及溶酶体的生物发生过程[62]。重要的是,Rab7还参与了自噬小泡的成熟过程,这使它成为促进LDs与降解细胞器相互作用从而促进LDs降解的有力候选者[63]。最近研究发现Rab7在介导肝细胞的脂噬中起着中心作用,他们发现营养缺乏会刺激LDs上的Rab7蛋白以及饥饿时与LDs相关的囊泡结构[64]。活跃的Rab7可能会介导多泡体和溶酶体与LDs的相关反应。
(2)Rab10:Rab10也是GTPase的一种,在LDs蛋白组学筛选中多次出现,其主要功能为介导LDs在脂噬过程中的自噬行为。Rab10已经被证实参与的反应有:基底外侧内吞循环过程、将胰岛素依赖型葡萄糖转运蛋白4型运输到质膜以及对内质网小管动力学和形态学上的调节[65-67]。Li等[68]发表过关于Rab10在脂质吞噬过程中调节LDs降解的研究,他们的研究表明在营养缺乏或自噬诱导的条件下,Rab10会被激活,然后促进其对LDs表面那些对LC3、Atg16L1和溶酶体相关膜蛋白1表现为阳性的膜状结构的补充。一旦Rab10在自噬膜上被激活,它就会与其效应蛋白(内吞适配物Eps15同源性[EH]域结合蛋白1)以及含两个蛋白的膜重构腺苷三磷酸酶EH域结合。上述反应形成的三聚体复合物将自噬膜延伸至LDs表面,最终导致LDs完全被自噬体吞噬。因此可以得知,Rab10有促进LDs在脂溶性进展过程中的自噬功能。除此之外,还观察到siRNA介导的使该蛋白的基因沉默或让显性阴性突变体表达对Rab10产生的破坏会导致LDs的积累,这一发现又证实了上述观点的正确性。有趣的是,Rab7的缺失会对Rab10向LDs周围自噬膜下游补充有显著的影响,而Rab7的定位不受Rab10缺失的影响,这说明了这两种脂质调节因子之间不存在重叠作用[68]。
(3)Rab18:Rab18是一种LDs专一型的Rab GTPase。Rab7和Rab10的作用范围除LDs外还包括溶酶体膜和自噬膜,而Rab18是一种只存在于LDs上的蛋白。曾经有两个独立的研究小组同时报道,在脂肪细胞和非脂肪细胞模型中,Rab18与LDs单层直接相关[69-70]。在免疫荧光和免疫金电镜下观察发现,荧光标记的野生型或组成活性(但非显性阴性)的Rab18突变体直接位于LDs表面。并且在表达Rab18的细胞中,围绕LDs的内质网网络很近,这表明这种Rab GTPase可能调节了LDs与内质网间的联系。虽然Rab18早在十多年前就被发现存在于LDs上,但它的作用尚未完全确定。在观察Rab18是否将LDs与内质网联系在一起的过程中产生了一个问题,即Rab18是否参与调控了内质网中LDs的形成或生长。用胰岛素或类β-肾上腺素能药物刺激脂肪细胞,结果显示这种刺激会增加Rab18向LDs表面补充,表明其可能在介导脂肪细胞的脂肪生成和分解方面发挥着作用[69, 71]。关于Rab18在LDs稳态调节中作用的研究上,最具有代表性的证据可能来源于对Warburg微综合征患者的研究,这是一种由Rab18突变引起的神经系统疾病[72]。对从这些患者体内分离出的成纤维细胞与对照组细胞进行18或24 h的油酸治疗,发现实验组会导致更多的LDs积累[73]。近年来,实验发现转运蛋白颗粒Ⅱ在脂质负载后被补充到LDs表面,作为与LDs相关的GEF为Rab18发挥作用[74]。有趣的是,转运蛋白颗粒ⅡGEF活性丧失,其导致的最终结果与抑制Rab18观察到的结果类似,即脂质分解受损以及大型LDs的积累。
(4)Rab32:Rab32位于线粒体,它发挥着作为A激酶锚定蛋白作用的同时,不仅能调节线粒体的活动[75],还能通过调节黑素酶在高尔基体转运的方式来参与黑素体的生物发生过程[76]。在调节LDs储存方面,第1个发现Rab32有该作用的证据来自对果蝇幼虫脂肪组织的研究。Wang等[77]发现Rab32的显性阴性表达或功能缺失突变体的表达会导致LDs的缩小和总TAG含量的降低。Rab32功能的缺失会导致自噬功能受损,这表明Rab32可能通过影响脂溶性LDs的分解来调节LDs的储存。在哺乳动物模型的实验与前面在果蝇模型的实验显示结果一致——Rab32的缺失会导致细胞内LDs水平降低[78]。
2.2 酶甘氨酸-N-甲基转移酶(GNMT)是一种可能间接影响肝脂噬的酶,在小鼠体内,GNMT的表达不足会导致循环蛋氨酸的水平升高[79],血清蛋氨酸的升高则导致自噬通量降低,而在敲除GNMT基因的小鼠体内则检测到了较高的蛋氨酸水平。这是由于哺乳动物体内雷帕霉素相互作用导致蛋白磷酸酶2A的靶蛋白甲基化异常,从而对自噬产生了抑制作用。鉴于在敲除GNMT的肝细胞中,对溶酶体的抑制并没有导致细胞TAG水平继续升高,这说明GNMT可能在肝脂吞噬中发挥重要的作用。因此,脂肪肝和GNMT缺失的患者体内高水平的LDs可能会因为无法通过噬脂作用动员LDs而使肝脏变性恶化。
在对肝脏特异性敲除超氧化物歧化酶1(SOD1)的禁食小鼠肝脏中也可以观察到类似的现象[80]。SOD1的作用机理是将超氧化物转化为过氧化氢,作为减轻氧化应激反应的一种手段。实验还发现敲除SOD1小鼠的肝细胞出现了膨胀的表型[80]。除此之外,在禁食48 h后,还观察到PLIN2水平升高,这意味着LDs水平也在提升。
2.3 离子通道根据钙稳态受损是导致肥胖或糖尿病背后代谢功能障碍的因素之一[81],钙信号传导也被认为可能是调节肝脏脂肪吞噬的一种机制。最近有研究发现,钙离子流入细胞过程中的储存可运转的钙离子入口(SOCE)损失,会导致肝细胞内脂质的积累[82]。SOCE是线粒体正常工作的所必需的,尤其在基因表达的水平上,它能够控制某些参与脂肪酸氧化的关键因素以及电子传递链的组成部分。SOCE缺陷的细胞内包浆脂肪酶表达会受损,因此在这些小鼠体内的钙离子信号传导过程中,通过传统脂解法从LDs释放脂肪酸的过程会受到损害。然而,细胞却能够自动地通过促进脂质吞噬的方式来解决细胞内的脂质负荷。此外,在SOCE缺陷的小鼠骨骼肌中也观察到自噬液泡的形成有所增加,这一发现巩固了钙离子信号在调控肝脏和其他细胞中噬脂作用的地位。
2.4 转录因子脂质吞噬的转录调控可能由类似转录因子TFEB等因子参与介导,TFEB的作用机理是通过与溶酶体的生物发生过程中关键启动子相结合来对转录产生影响[83]。例如,有研究发现,在肝癌细胞中,慢性病毒介导的TFEB因子过表达导致了LC3与LDs的相关性增加,细胞内总脂质含量降低[84]。相似的,如果特异性敲除了TFEB因子(另一种溶酶体生物发生的转录调控因子)则会导致肝细胞中LDs的积累[85]。但是值得注意的是,TFEB的过表达能够挽救小鼠的肥胖和代谢综合征,这一现象支撑了一个观点,即这些钙离子控制的转录调控因子对肝脏中LDs过度积累产生的负面影响有一定的治疗效果。
除此之外,转录因子叉头框蛋白O1(FOXO1)在营养缺乏的条件下,可激活脂肪细胞中的脂噬[86]。而特异性敲除肝脏中FOXO1/3/4,小鼠发生肝脏脂肪变性和高甘油三酯血症,自噬流减少[87]。
2.5 小分子物质近年来,许多科学家研究发现小分子物质对脂噬有调节作用(表 1)。
| 表 1 小分子物质对脂噬的调节 Tab.1 Regulation of lipophagy by small molecules |
脂噬对于调节细胞稳态具有重要的作用。在过去的10年内,脂噬研究领域有了重大突破,但对这一复杂过程相关影响因素和具体途径的研究还仍在进行中,就像NAFLD、肥胖、糖尿病等疾病的治疗与干预方法还尚未成熟。其中最突出的问题无疑是脂肪酶与核心脂噬机制之间的交叉作用。随着年龄的增长,脂质逐渐堆积,自噬活动减弱,这表明脂质堆积可能是导致自噬随年龄增长而下降的原因之一。另外,目前的研究多关注单一器官或细胞内脂噬对脂质代谢的影响,而从整个机体来看,脂噬可作用于肝脏、脂肪组织、胰岛、心血管等多个靶器官,脂噬水平的改变引起的不仅仅是单一器官代谢的改变,而是伴随着整个机体的相关改变。对此,有待于进一步探索脂噬与整个机体的动态关系。
| [1] |
Watson AS, Mortensen M, Simon AK. Autophagy in the pathogenesis of myelodysplastic syndrome and acute myeloid leukemia[J]. Cell Cycle, 2011, 10(11): 1719-25. DOI:10.4161/cc.10.11.15673 |
| [2] |
Bernard A, Klionsky DJ. Autophagosome formation: tracing the source[J]. Dev Cell, 2013, 25(2): 116-7. DOI:10.1016/j.devcel.2013.04.004 |
| [3] |
Pan HM, Chen LX, Xu YH, et al. Autophagy-associated immune responses and cancer immunotherapy[J]. Oncotarget, 2016, 7(16): 21235-46. |
| [4] |
Thapalia BA, Zhou Z, Lin X. Autophagy, a process within reperfusion injury: an update[J]. Int J Clin Exp Pathol, 2014, 7(12): 8322-41. |
| [5] |
Tanida I. Autophagosome formation and molecular mechanism of autophagy[J]. Antioxid Redox Signal, 2011, 14(11): 2201-14. DOI:10.1089/ars.2010.3482 |
| [6] |
Bento CF, Renna M, Ghislat G, et al. Mammalian autophagy: how does it work?[J]. Annu Rev Biochem, 2016, 85(3): 685-713. |
| [7] |
Glick D, Barth S, Macleod KF. Autophagy: cellular and molecular mechanisms[J]. J Pathol, 2010, 221(1): 3-12. |
| [8] |
He CC, Klionsky DJ. Regulation mechanisms and signaling pathways of autophagy[J]. Annu Rev Genet, 2009, 43(1): 67-93. DOI:10.1146/annurev-genet-102808-114910 |
| [9] |
Kaur J, Debnath J. Autophagy at the crossroads of catabolism and anabolism[J]. Nat Rev Mol Cell Biol, 2015, 16(8): 461-72. DOI:10.1038/nrm4024 |
| [10] |
Noda NN, Inagaki F. Mechanisms of autophagy[J]. Annu Rev Biophys, 2015, 44(1): 101-22. DOI:10.1146/annurev-biophys-060414-034248 |
| [11] |
Hansen M, Rubinsztein DC, Walker DW. Autophagy as a promoter of longevity: insights from model organisms[J]. Nat Rev Mol Cell Biol, 2018, 19(9): 579-93. DOI:10.1038/s41580-018-0033-y |
| [12] |
Walther TC, Farese RJ. Lipid droplets and cellular lipid metabolism[J]. Annu Rev Biochem, 2012, 81(12): 687-714. |
| [13] |
Wilfling F, Haas JT, Walther TC, et al. Lipid droplet biogenesis[J]. Curr Opin Cell Biol, 2014, 29(11): 39-45. |
| [14] |
Khandelia H, Duelund L, Pakkanen KI, et al. Triglyceride blisters in lipid bilayers: implications for lipid droplet biogenesis and the Mobile lipid signal in cancer cell membranes[J]. PLoS One, 2010, 5(9): e12811. DOI:10.1371/journal.pone.0012811 |
| [15] |
Brasaemle DL, Dolios G, Shapiro L, et al. Proteomic analysis of proteins associated with lipid droplets of basal and lipolytically stimulated 3T3-L1 adipocytes[J]. J Biol Chem, 2004, 279(45): 46835-42. DOI:10.1074/jbc.M409340200 |
| [16] |
Cermelli S, Guo Y, Gross SP, et al. The lipid-droplet proteome reveals that droplets are a protein-storage depot[J]. Curr Biol, 2006, 16(18): 1783-95. DOI:10.1016/j.cub.2006.07.062 |
| [17] |
Hodges BD, Wu CC. Proteomic insights into an expanded cellular role for cytoplasmic lipid droplets[J]. J Lipid Res, 2010, 51(2): 262-73. DOI:10.1194/jlr.R003582 |
| [18] |
Yang L, Ding YF, Chen Y, et al. The proteomics of lipid droplets: structure, dynamics, and functions of the organelle conserved from bacteria to humans[J]. J Lipid Res, 2012, 53(7): 1245-53. DOI:10.1194/jlr.R024117 |
| [19] |
Krahmer N, Hilger M, Kory N, et al. Protein correlation profiles identify lipid droplet proteins with high confidence[J]. Mol Cell Proteomics, 2013, 12(5): 1115-26. DOI:10.1074/mcp.M112.020230 |
| [20] |
Currie E, Guo XL, Christiano R, et al. High confidence proteomic analysis of yeast LDs identifies additional droplet proteins and reveals connections to dolichol synthesis and sterol acetylation[J]. JLipid Res, 2014, 55(7): 1465-77. |
| [21] |
Hsieh K, Lee YK, Londos C, et al. Perilipin family members preferentially sequester to either triacylglycerol-specific or cholesteryl-ester-specific intracellular lipid storage droplets[J]. J Cell Sci, 2012, 125(17): 4067-76. DOI:10.1242/jcs.104943 |
| [22] |
Singh R, Kaushik S, Wang Y, et al. Autophagy regulates lipid metabolism[J]. Nature, 2009, 458(7242): 1131-5. DOI:10.1038/nature07976 |
| [23] |
Komatsu M, Waguri S, Ueno T, et al. Impairment of starvationinduced and constitutive autophagy in Atg7-deficient mice[J]. J Cell Biol, 2005, 169(3): 425-34. DOI:10.1083/jcb.200412022 |
| [24] |
Mizushima N, Levine B, Cuervo AM, et al. Autophagy fights disease through cellular self-digestion[J]. Nature, 2008, 451(7182): 1069-75. DOI:10.1038/nature06639 |
| [25] |
Zhang XY, Evans TD, Jeong SJ, et al. Classical and alternative roles for autophagy in lipid metabolism[J]. Curr Opin Lipidol, 2018, 29(3): 203-11. DOI:10.1097/MOL.0000000000000509 |
| [26] |
Rinella ME. Nonalcoholic fatty liver disease: a systematic review[J]. JAMA, 2015, 313(22): 2263-73. DOI:10.1001/jama.2015.5370 |
| [27] |
Zhang FY, Zhao SH, Yan WJ, et al. Branched chain amino acids cause liver injury in obese/diabetic mice by promoting adipocyte lipolysis and inhibiting hepatic autophagy[J]. EBioMedicine, 2016, 13(11): 157-67. |
| [28] |
Arab JP, Arrese M, Trauner M. Recent insights into the pathogenesis of nonalcoholic fatty liver disease[J]. Annu Rev Pathol, 2018, 13(6): 321-50. |
| [29] |
Johansen T, Lamark T. Selective autophagy mediated by autophagic adapter proteins[J]. Autophagy, 2011, 7(3): 279-96. DOI:10.4161/auto.7.3.14487 |
| [30] |
Liu K, Czaja MJ. Regulation of lipid stores and metabolism by lipophagy[J]. Cell Death Differ, 2013, 20(1): 3-11. DOI:10.1038/cdd.2012.63 |
| [31] |
droplets WL, lipophagy. And beyond[J]. Biochim Biophys Acta, 2016, 1861(8 Pt B): 793-805. |
| [32] |
Singh R, Cuervo AM. Lipophagy: connecting autophagy and lipid metabolism[J]. Int J Cell Biol, 2012, 28(3): 282041. |
| [33] |
Weidberg H, Shvets E, Elazar Z. Lipophagy: selective catabolism designed for lipids[J]. Dev Cell, 2009, 16(5): 628-30. DOI:10.1016/j.devcel.2009.05.001 |
| [34] |
Kaushik S, Cuervo AM. Degradation of lipid droplet-associated proteins by chaperone- mediated autophagy facilitates lipolysis[J]. Nat Cell Biol, 2015, 17(6): 759. DOI:10.1038/ncb3166 |
| [35] |
Arias E, Cuervo AM. Chaperone-mediated autophagy in protein quality control[J]. Curr Opin Cell Biol, 2011, 23(2): 184-9. DOI:10.1016/j.ceb.2010.10.009 |
| [36] |
Cuervo AM, Dice JF. A receptor for the selective uptake and degradation of proteins by lysosomes[J]. Science, 1996, 273(5274): 501-3. DOI:10.1126/science.273.5274.501 |
| [37] |
Dice JF. Peptide sequences that target cytosolic proteins for lysosomal proteolysis[J]. Trends Biochem Sci, 1990, 15(8): 305-9. DOI:10.1016/0968-0004(90)90019-8 |
| [38] |
Hickenbottom SJ, Kimmel AR, Londos C, et al. Structure of a lipid droplet protein: The PAT family member TIP47[J]. Structure, 2004, 12(7): 1199-207. DOI:10.1016/j.str.2004.04.021 |
| [39] |
Jiang HP, Harris SE, Serrero G. Molecular cloning of a differentiationrelated mRNA in the adipogenic cell line 1246[J]. Cell Growth Differ, 1992, 3(1): 21-30. |
| [40] |
Kimmel AR, Brasaemle DL, Mcandrews-Hill MA, et al. Adoption of PERILIPIN as a unifying nomenclature for the mammalian PATfamily of intracellular lipid storage droplet proteins[J]. J Lipid Res, 2010, 51(3): 468-71. DOI:10.1194/jlr.R000034 |
| [41] |
Chiang HL, Terlecky SR, Plant CP, et al. A role for a 70-kilodalton heat shock protein in lysosomal degradation of intracellular proteins[J]. Science, 1989, 246(4928): 382-5. DOI:10.1126/science.2799391 |
| [42] |
Martinez-Lopez N, Garcia-Macia M, Sahu SA, et al. Autophagy in the CNS and periphery coordinate lipophagy and lipolysis in the brown adipose tissue and liver[J]. Cell Metab, 2016, 23(1): 113-27. DOI:10.1016/j.cmet.2015.10.008 |
| [43] |
Khan SA, Sathyanarayan A, Mashek MT, et al. ATGL-Catalyzed lipolysis regulates SIRT1 to control PGC-1 alpha/PPAR-alpha signaling[J]. Diabetes, 2015, 64(2): 418-26. DOI:10.2337/db14-0325 |
| [44] |
Sathyanarayan A, Mashek MT, Mashek DG. ATGL promotes autophagy/lipophagy via SIRT1 to control hepatic lipid droplet catabolism[J]. Cell Rep, 2017, 19(1): 1-9. |
| [45] |
Schulze RJ, Drizyte K, Casey CA, et al. Hepatic lipophagy: new insights into autophagic catabolism of lipid droplets in the liver[J]. Hepatol Commun, 2017, 1(5): 359-69. DOI:10.1002/hep4.v1.5 |
| [46] |
Gukovskaya AS, Pandol SJ, Gukovsky I. New insights into the pathways initiating and driving pancreatitis[J]. Curr Opin Gastroenterol, 2016, 32(5): 429-35. DOI:10.1097/MOG.0000000000000301 |
| [47] |
Gukovsky I, Gukovskaya AS. Impaired autophagy triggers chronic pancreatitis: lessons from pancreas-specific Atg5 knockout mice[J]. Gastroenterology, 2015, 148(3): 501-5. DOI:10.1053/j.gastro.2015.01.012 |
| [48] |
Wang J, Wu J, Wu H, et al. Liraglutide protects pancreatic beta-cells against free fatty acids in vitro and affects glucolipid metabolism in apolipoprotein E-/- mice by activating autophagy[J]. Mol Med Rep, 2015, 12(3): 4210-8. DOI:10.3892/mmr.2015.3944 |
| [49] |
Komiya K, Uchida T, Ueno T, et al. Free fatty acids stimulate autophagy in pancreatic beta-cells via JNK pathway[J]. Biochem Biophys Res Commun, 2010, 401(4): 561-7. DOI:10.1016/j.bbrc.2010.09.101 |
| [50] |
Sun QQ, Nie SS, Wang LX, et al. Factors that affect pancreatic islet cell autophagy in adult rats: evaluation of a calorie-restricted diet and a high-fat diet[J]. PLoS One, 2016, 11(3): e151104. |
| [51] |
Chu KY, O'reilly L, Ramm G, et al. High-fat diet increases autophagic flux in pancreatic beta cells in vivo and ex vivo in mice[J]. Diabetologia, 2015, 58(9): 2074-8. DOI:10.1007/s00125-015-3665-x |
| [52] |
Masini M, Bugliani M, Lupi R, et al. Autophagy in human type 2 diabetes pancreatic beta cells[J]. Diabetologia, 2009, 52(6): 1083-6. DOI:10.1007/s00125-009-1347-2 |
| [53] |
Hahm JR, Noh HS, Ha JH, et al. Alpha-lipoic acid attenuates adipocyte differentiation and lipid accumulation in 3T3-L1 cells via AMPK-dependent autophagy[J]. Life Sci, 2014, 100(2): 125-32. DOI:10.1016/j.lfs.2014.02.001 |
| [54] |
Martinez-Lopez N, Athonvarangkul D, Sahu SA, et al. Autophagy in Myf5+ progenitors regulates energy and glucose homeostasis through control of brown fat and skeletal muscle development[J]. EMBO Rep, 2013, 14(9): 795-803. DOI:10.1038/embor.2013.111 |
| [55] |
Armani A, Cinti F, Marzolla V, et al. Mineralocorticoid receptor antagonism induces browning of white adipose tissue through impairment of autophagy and prevents adipocyte dysfunction in high-fat-diet-fed mice[J]. FASEB J, 2014, 28(8): 3745-57. DOI:10.1096/fj.13-245415 |
| [56] |
Singh R, Xiang Y, Wang Y, et al. Autophagy regulates adipose mass and differentiation in mice[J]. J Clin Invest, 2009, 119(11): 3329-39. |
| [57] |
Baerga R, Zhang Y, Chen PH, et al. Targeted deletion of autophogyrelated 5(atg5) impairs adipogenesis in a cellular model and in mice[J]. Autophagy, 2009, 5(8): 1118-30. DOI:10.4161/auto.5.8.9991 |
| [58] |
Stenmark H. Rab GTPases as coordinators of vesicle traffic[J]. Nat Rev Mol Cell Biol, 2009, 10(8): 513-25. DOI:10.1038/nrm2728 |
| [59] |
Hutagalung AH, Novick PJ. Role of Rab GTPases in membrane traffic and cell physiology[J]. Physiol Rev, 2011, 91(1): 119-49. DOI:10.1152/physrev.00059.2009 |
| [60] |
Khan SA, Wollaston-Hayden EE, Markowski TW, et al. Quantitative analysis of the murine lipid droplet-associated proteome during dietinduced hepatic steatosis[J]. J Lipid Res, 2015, 56(12): 2260-72. DOI:10.1194/jlr.M056812 |
| [61] |
Fujimoto Y, Itabe H, Sakai J, et al. Identification of major proteins in the lipid droplet-enriched fraction isolated from the human hepatocyte cell line HuH7[J]. Biochim Biophys Acta, 2004, 1644(1): 47-59. DOI:10.1016/j.bbamcr.2003.10.018 |
| [62] |
Bucci C, Thomsen P, Nicoziani P, et al. Rab7: A key to lysosome biogenesis[J]. Mol Biol Cell, 2000, 11(2): 467-80. DOI:10.1091/mbc.11.2.467 |
| [63] |
Jager S, Bucci C, Tanida I, et al. Role for Rab7 in maturation of late autophagic vacuoles[J]. J Cell Sci, 2004, 117(20): 4837-48. DOI:10.1242/jcs.01370 |
| [64] |
Schroeder B, Schulze RJ, Weller S, et al. The small GTPase Rab7 as a central regulator of hepatocellular lipophagy[J]. Hepatology, 2014, 60: 809A. DOI:10.1016/j.jhep.2013.11.018 |
| [65] |
English AR, Voeltz GK. Rab10 GTPase regulates ER dynamics and morphology[J]. Nat Cell Biol, 2013, 15(2): 169-78. DOI:10.1038/ncb2647 |
| [66] |
Sano H, Roach WG, Peck GR, et al. Rab10 in insulin-stimulated GLUT4 translocation[J]. Biochem J, 2008, 411(1): 89-95. DOI:10.1042/BJ20071318 |
| [67] |
Chen CC, Schweinsberg PJ, Vashist S, et al. RAB-10 is required for endocytic recycling in the Caenorhabditis elegans intestine[J]. Mol Biol Cell, 2006, 17(3): 1286-97. DOI:10.1091/mbc.e05-08-0787 |
| [68] |
Li ZP, Schulze RJ, Weller SG, et al. A novel Rab10-EHBP1-EHD2 complex essential for the autophagic engulfment of lipid droplets[J]. SciAdv, 2016, 2(12): e1601470. |
| [69] |
Martin S, Driessen K, Nixon SJ, et al. Regulated localization of rab18 to lipid droplets-effects of lipolytic stimulation and inhibition of lipid droplet catabolism[J]. J Biol Chem, 2005, 280(51): 42325-35. DOI:10.1074/jbc.M506651200 |
| [70] |
Ozeki S, Cheng J, Tauchi-Sato K, et al. Rab18 localizes to lipid droplets and induces their close apposition to the endoplasmic reticulumderived membrane[J]. J Cell Sci, 2005, 118(Pt 12): 2601-11. |
| [71] |
Pulido MR, Diaz-Ruiz A, Jimenez-Gomez YA, et al. Rab18 dynamics in adipocytes in relation to lipogenesis, lipolysis and obesity[J]. PLoS One, 2011, 6(7): e22931. DOI:10.1371/journal.pone.0022931 |
| [72] |
Bem D, Yoshimura S, Nunes-Bastos R, et al. Loss-of-function mutations in RAB18 cause warburg micro syndrome[J]. Am J Hum Genet, 2011, 88(4): 499-507. DOI:10.1016/j.ajhg.2011.03.012 |
| [73] |
Liegel RP, Handley MT, Ronchetti A, et al. Loss-of-function mutations in TBC1 D20 cause cataracts and male infertility in blind sterile mice and warburg micro syndrome in humans[J]. Am J Hum Genet, 2013, 93(6): 1001-14. DOI:10.1016/j.ajhg.2013.10.011 |
| [74] |
Li C, Luo X, Zhao S, et al. COPI-TRAPPII activates Rab18 and regulates its lipid droplet association[J]. EMBO J, 2017, 36(4): 441-57. DOI:10.15252/embj.201694866 |
| [75] |
Alto NM, Soderling J, Scott JD. Rab32 is an A-kinase anchoring protein and participates in mitochondrial dynamics[J]. J Cell Biol, 2002, 158(4): 659-68. DOI:10.1083/jcb.200204081 |
| [76] |
Wasmeier C, Romao M, Plowright L, et al. Rab38 and Rab32 control post-Golgi trafficking of melanogenic enzymes[J]. J Cell Biol, 2006, 175(2): 271-81. DOI:10.1083/jcb.200606050 |
| [77] |
Wang C, Liu ZH, Huang X. Rab32 is important for autophagy and lipid storage in drosophila[J]. PLoS One, 2012, 7(2): e32086. DOI:10.1371/journal.pone.0032086 |
| [78] |
Li Q, Wang J, Wan Y, et al. Depletion of Rab32 decreases intracellular lipid accumulation and induces lipolysis through enhancing ATGL expression in hepatocytes[J]. Biochem Biophys Res Commun, 2016, 471(4): 492-6. DOI:10.1016/j.bbrc.2016.02.047 |
| [79] |
Zubiete-Franco I, Garcia-Rodriguez JL, Martinez-Una M, et al. Methionine and S-adenosylmethionine levels are critical regulators of PP2A activity modulating lipophagy during steatosis[J]. J Hepatol, 2016, 64(2): 409-18. DOI:10.1016/j.jhep.2015.08.037 |
| [80] |
Kurahashi T, Hamashima S, Shirato T, et al. An SOD1 deficiency enhances lipid droplet accumulation in the fasted mouse liver by aborting lipophagy[J]. Biochem Biophys Res Commun, 2015, 467(4): 866-71. DOI:10.1016/j.bbrc.2015.10.052 |
| [81] |
Arruda AP, Hotamisligil GS. Calcium homeostasis and organelle function in the pathogenesis of obesity and diabetes[J]. Cell Metab, 2015, 22(3): 381-97. DOI:10.1016/j.cmet.2015.06.010 |
| [82] |
Maus M, Cuk M, Patel B, et al. Store-Operated Ca(2+) entry controls induction of lipolysis and the transcriptional reprogramming to lipid metabolism[J]. Cell Metab, 2017, 25(3): 698-712. DOI:10.1016/j.cmet.2016.12.021 |
| [83] |
Martina JA, Diab HI, Lishu L, et al. The nutrient-responsive transcription factor TFE3 promotes autophagy, lysosomal biogenesis, and clearance of cellular debris[J]. Sci Signal, 2014, 7(39): a9. |
| [84] |
Xiong J, Wang KZ, He JP, et al. TFE3 alleviates hepatic steatosis through autophagy-induced lipophagy and PGC1 alpha-Mediated fatty acid beta-Oxidation[J]. Int J Mol Sci, 2016, 17(3): 387. DOI:10.3390/ijms17030387 |
| [85] |
Settembre C, De Cegli R, Mansueto GA, et al. TFEB controls cellular lipid metabolism through a starvation- induced autoregulatory loop[J]. Nat Cell Biol, 2013, 15(6): 647. DOI:10.1038/ncb2718 |
| [86] |
Lettieri BD, Tatulli G, Aquilano K, et al. FoxO1 controls lysosomal acid lipase in adipocytes: implication of lipophagy during nutrient restriction and metformin treatment[J]. Cell Death Dis, 2013, 17(3): e861. |
| [87] |
Xiong X, Tao R, Depinho RA, et al. The autophagy-related gene 14(Atg14) is regulated by forkhead box O transcription factors and circadian rhythms and plays a critical role in hepatic autophagy and lipid metabolism[J]. J Biol Chem, 2012, 287(46): 39107-14. DOI:10.1074/jbc.M112.412569 |
| [88] |
Ding WX. Drinking coffee burns hepatic fat by inducing lipophagy coupled with mitochondrial beta-Oxidation[J]. Hepatology, 2014, 59(4): 1235-8. DOI:10.1002/hep.26736 |
| [89] |
Sinha RA, Farah BL, Singh BK, et al. Caffeine stimulates hepatic lipid metabolism by the autophagy-lysosomal pathway in mice[J]. Hepatology, 2014, 59(4): 1366-80. DOI:10.1002/hep.26667 |
| [90] |
Thomes PG, Trambly CS, Fox HS, et al. Acute and chronic ethanol administration differentially modulate hepatic autophagy and transcription factor EB[J]. Alcohol Clin Exp Res, 2015, 39(12): 2354-63. DOI:10.1111/acer.12904 |
| [91] |
Ding WX, Ni HM. Critical role of foxo3a in alcohol-induced autophagy and hepatotoxicity[J]. Am J Pathol, 2012, 36(2, SI): 55A. |
| [92] |
Schulze RJ, Rasineni K, Weller SG, et al. Ethanol exposure inhibits hepatocyte lipophagy by inactivating the small guanosine triphosphatase Rab7[J]. Hepatol Commun, 2017, 1(2): 140-52. DOI:10.1002/hep4.1021 |
| [93] |
Hernández-Gea V, Ghiassi-Nejad Z, Rozenfeld R, et al. Autophagy releases lipid that promotes fibrogenesis by activated hepatic stellate cells in mice and in human tissues[J]. Gastroenterology, 2012, 142(4): 938-46. DOI:10.1053/j.gastro.2011.12.044 |
| [94] |
Fraulob JC, Ogg-Diamantino R, Fernandes-Santos C, et al. A mouse model of metabolic syndrome: insulin resistance, fatty liver and NonAlcoholic fatty pancreas disease (NAFPD) in C57BL/6 mice fed a high fat Diet[J]. J Clin Biochem Nutr, 2010, 46(3): 212-23. DOI:10.3164/jcbn.09-83 |
| [95] |
Zhou HY, Huang SL. Current development of the second Generation of mTOR inhibitors as anticancer agents[J]. Chin J Cancer, 2012, 31(1): 8-18. |
 2019, Vol. 39
2019, Vol. 39