2. 蚌埠医学院 生物科学系,安徽 蚌埠 233003
2. First Affiliated Hospital, Department of Biological Sciences, Bengbu Medical College, Bengbu 233003, China
最新世界癌症报告显示:胃癌是全球第4种最常见的癌症类型,也是癌症相关死亡的第二大原因[1-2]。已确认的几种胃癌危险因素包括:性别、幽门螺旋菌感染、吸烟史、慢性萎缩性胃炎以及胃部手术等[3]。目前,治疗胃癌最有效的方法仍然是手术。然而,近一半的胃癌患者在手术切除后复发并发展成为晚期疾病,致使胃癌患者的五年生存率相对较差[4]。因此迫切需要对胃癌患者实行有效的预防、治疗以及预后监测,本研究旨在为新的胃癌预后标志物和潜在的治疗靶点提供策略。
外切核酸酶3'-5'结构域3(EXD3)基因属于mut-7家族,位于人类9号染色体长臂上,具有编码蛋白的功能。然而,EXD3在人类已知肿瘤中,包括在胃癌中的表达及功能目前尚无相关文献报道。在此项研究中,通过生物信息学分析技术,利用Morpheus在线软件分析基因芯片GSE100935的数据,获得在胃癌与正常胃粘膜组织中存在差异表达的基因EXD3;利用UALCAN在线分析获得EXD3基因在胃癌与正常胃粘膜组织中表达上调;Kaplan-Meier生存分析显示EXD3高表达的胃癌患者预后较差;最后采用Fun Rich软件进行GO功能富集分析,网站STRING构建目的基因蛋白互作网络。结果提示:EXD3可能是胃癌中潜在的癌基因,可能与DNA损伤修复有关,其表达上调在胃癌的发生发展乃至预后的过程中发挥重要作用,并将可能成为判断胃癌患者预后的重要的生物学指标。
1 资料和方法 1.1 基因芯片数据GEO数据库是一个主要由微阵列研究产生的公共数据库,来自美国国立生物技术信息中心(National Institutes of Health, Bethesda, MD, USA)[5]。从GEO上下载基因芯片GSE100935的数据,该基因芯片含有66个临床标本的基因表达谱数据,其中包括11个正常胃粘膜、18个胃癌组织以及来自新加坡的37个胃癌组织。
1.2 识别差异表达基因在线基因芯片数据分析软件Morpheus(http://software.broadinstitute.org/morpheus)用于分析11个正常胃粘膜和18个胃癌组织中差异表达的基因。将下载的27例样本数据整理后导入Morpheus,随后对正常胃粘膜和胃癌组织间差异表达基因进行筛选。点击Marker Selection选择信噪比(SNR)作为常用筛选依据,设置类型为正常组和肿瘤组以及筛选后的基因数量设为100。生成的100个基因按照信噪比从高到低依次排列,获得排在前100的差异表调基因,成聚类分析热图。EXD3基因居于首位,相比于正常胃粘膜组织,EXD3基因在胃癌组织中表达上调。
1.3 EXD3基因在人胃癌组织及正常胃粘膜组织中的表达分析通过在线分析软件UALCAN(http://ualcan.path.uab.edu/analysis.html)分析EXD3基因在人胃癌组织及正常胃粘膜中的表达。在网站主页输入目的基因EXD3,选择胃癌组织,获得目的基因在胃癌组织和胃粘膜组织中的差异表达,本研究将从原发胃癌、胃癌分级、组织亚型以及感染幽门螺杆菌等不同角度分析EXD3的表达差异。
1.4 生存分析利用在线分析网站Kaplan- Meier Plotter分析EXD3基因分别在高表达和低表达胃癌患者中的生存情况。进入网站(http://www.kmplot.com/),输入目的基因,选择在胃癌中进行分析。根据胃癌患者的治疗方式以及HER2的表达情况分别选取EXD3基因高表达和低表达的临床样本进行总生存分析。
1.5 GO功能富集分析并构建蛋白互作(PPI)网络利用软件Fun Rich对表达上调的16637个差异表达基因进行GO功能分析,从分子功能,生物过程和细胞组成的角度对其富集分析(通过P值统计学筛选表达前6的条目)。STRING(http://string-db.org/)是基于网络的生物信息学分析工具,包含已知和预测的蛋白质相互作用的数据库,包括从基因组分析,高通量实验和共表达研究中获得的物理和功能关联,用于检索目的基因蛋白相互作用网络[6]。将目的基因“EXD3”输入STRING数据库进行分析,并选择来自人(Homo sapiens)的蛋白质相互作用网络。
2 结果 2.1 差异表达基因及其热图使用在线分析工具Morpheus分析基因芯片GSE100935,得到前100个在正常胃粘膜和胃癌组织间出现明显表达差异的基因(图 1);通过信噪比SNG大小进行排序,获得信噪比最高的差异表达基因EXD3,用Spearman相关分析生成聚类分析热图。在基因芯片数据分析中,相比于正常胃粘膜组织,EXD3基因在胃癌组织中表达上调。
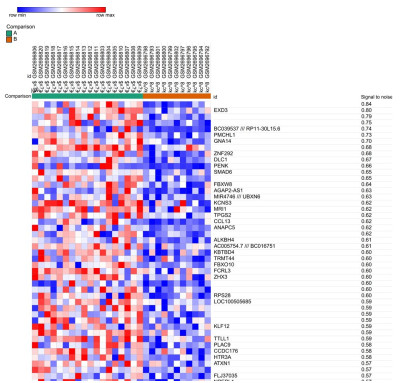
|
图 1 差异表达基因及其热图(部分结果) Fig.1 Differentially expressed genes in gastric cancer and heat map (partial results). Red: Up-regulated expression; Blue: Down-regulated expression. |
利用在线数据库UALCAN分析来自TCGA数据库的不同的胃癌组织和正常胃粘膜组织,获得EXD3基因在原发组织、不同分级和组织亚型以及感染幽门螺菌属的胃癌和正常胃粘膜中的表达情况(图 2)。结果显示:与正常胃粘膜相比,EXD3基因在胃癌组织中表达上调,这与在线分析工具Morpheus预测的结果相一致。值得注意的是,在没有感染幽门螺菌属和胃癌1级的患者中,EXD3基因上调更明显。
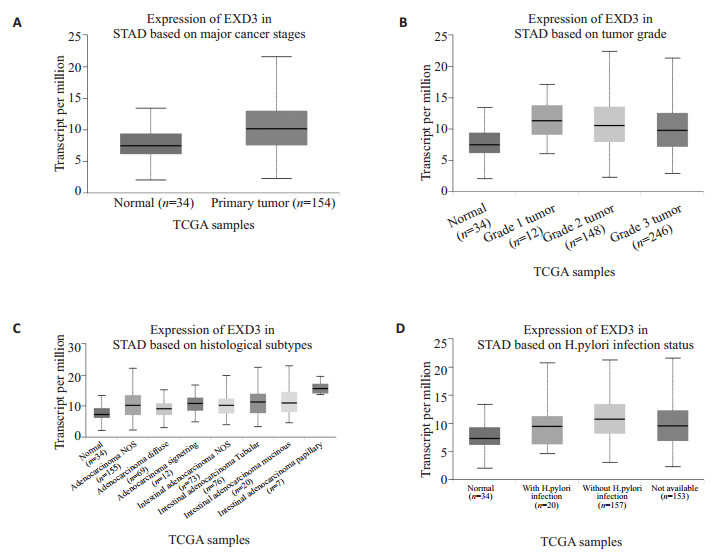
|
图 2 EXD3在正常胃粘膜与原发胃癌(A)、不同分级的胃癌(B)、不同组织亚型的胃癌(C)以及是否感染幽门螺菌属的胃癌(D)中的差异性表达 Fig.2 Differential expressions of EXD3 in normal gastric mucosa and primary gastric cancer (A), gastric cancer of different grades (B), gastric cancer of different tissue subtypes (C), and gastric cancer with Helicobacter pylori infection (D), showing up-regulation of EXD3 in the tumor tissues (P < 0.05 vs normol). |
通过在线数据库Kaplan- Meier Plotter分别获得EXD3在不同胃癌患者中的表达数据,根据表达差异分为低表达组和高表达组,对治疗方式和HER2基因表达不同的胃癌患者进行生存率分析(图 3),结果显示:与EXD3低表达组相比,EXD3高表达患者的生存率相对较差,差异有统计学意义(P< 0.05),Kaplan-Meier生存分析说明了EXD3高表达的胃癌患者生存率较低,预后相对较差。其中5-氟尿嘧啶(5-FU)治疗后EXD3基因高表达的胃癌患者生存率较高,预后相对较好,这提示EXD3可能参与了5-FU的抑制癌细胞核苷酸合成的过程。
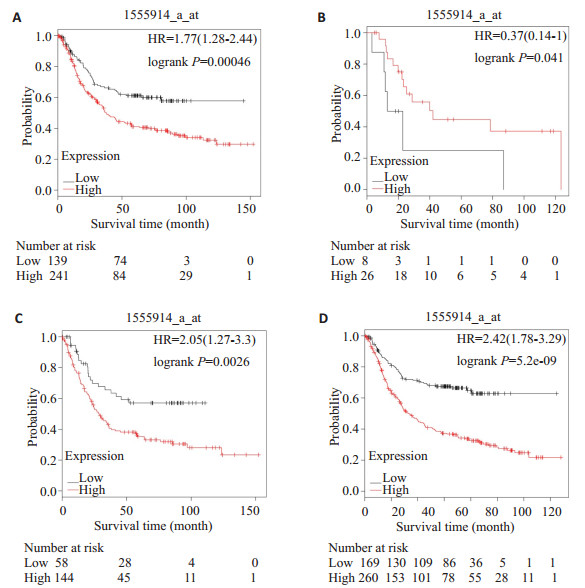
|
图 3 EXD3表达在手术治疗后(A)、5-FU治疗后(B)、HER2表达阳性(C)以及HER2表达阴性(D)的胃癌患者中的预后价值 Fig.3 Prognostic value of EXD3 expression in gastric cancer patients after surgery (A), after 5- FU treatment (B), with HER2 positivity (C), and with HER2 negativity (D). The patients with gastric cancer showing a high expression of EXD3 had poorer prognosis compared with the patients with low expression EXD3 (P < 0.05 vs low expression group). |
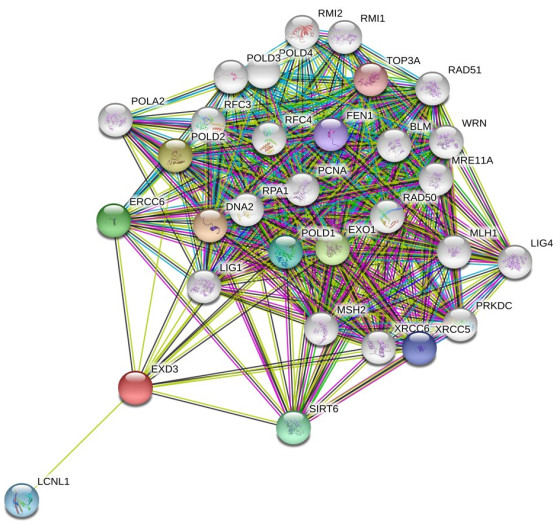
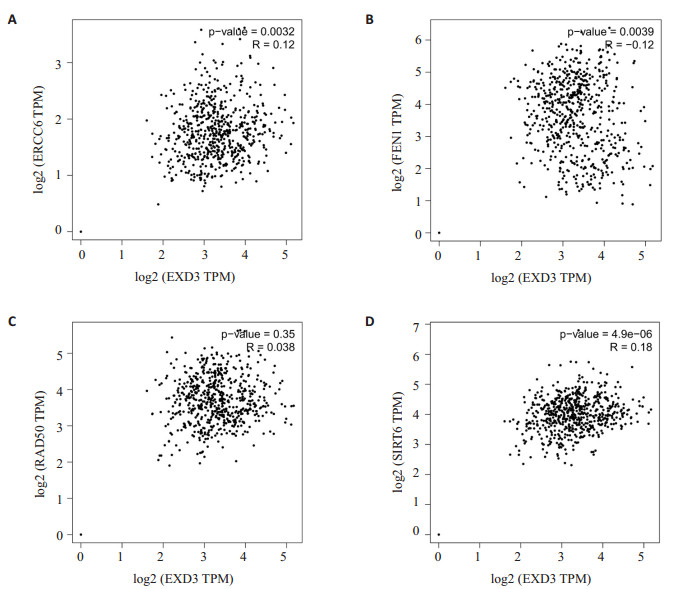
差异表达基因GO功能分析结果显示为:生物过程:16.2%基因涉及核苷酸新陈代谢的调节;细胞组成:40.8%基因定位在细胞质中;分子功能:4.95%基因调节转录因子的活动(P<0.001,图 4)。同时为了更好地了解EXD3的功能作用机制,使用STRING数据库用于构建EXD3的蛋白互作网络。据报道,共有31个预测的功能蛋白与EXD3相互作用(图 5)。蛋白质之间的连接线越多,相互关联性越强。在通过STRING鉴定的31种预测的蛋白互作网络中,有4种相关蛋白引起了注意,这些蛋白基因被证实与胃癌的发生发展和预后有关。如:沉默信息调节因子6(SIRT6)通过JAK2/STAT3信号通路抑制胃癌的发生发展[7-9],修复交叉互补6(ERCC6)的高表达可以表明胃癌患者的总生存率较差[10-13],高表达DNA修复蛋白(RAD50)的胃癌患者的预后较差[14-15],敲低片状核酸内切酶1(FEN1)可以导致胃癌细胞顺铂耐药性的增加[16],提示EXD3基因可能也与胃癌患者的预后和耐药性有关。为了进一步确定他们之间的关系,通过在线网站GEPIA(gepia.cancer-pku.cn)分析这些蛋白基因(图 6),结果发现EXD3基因与ERCC6、SIRT6的表达成正相关,与FEN1成负相关,进一步提示EXD3作为一种癌基因与胃癌患者预后不良相关,并且可能通过借助正向调节ERCC6参与DNA修复。
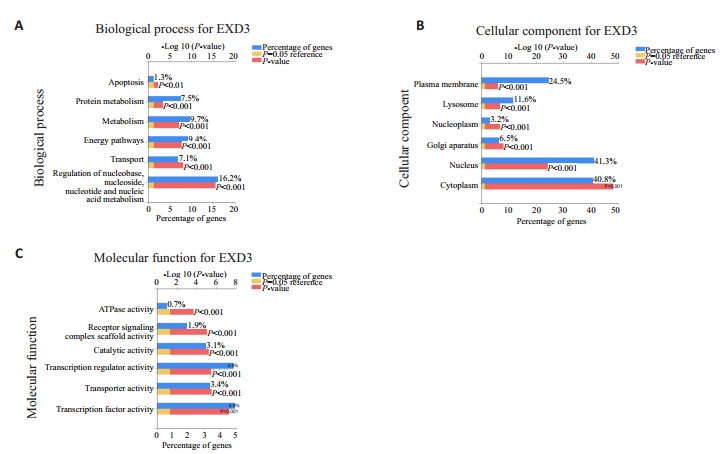
|
图 4 差异基因相关GO功能富集分析(前6位条,P < 0.001) Fig.4 GO functional enrichment analysis of the differentially expressed genes (first 6 bars, P < 0.001). |
|
图 5 构建EXD3基因相关的PPI网络(共有31个预测的功能蛋白) Fig.5 EXD3-associated PPI network constructed in this study (including 31 predicted functional proteins). |
|
图 6 EXD3基因与ERCC6(A)、SIRT6(D)的表达成正相关,与FEN1(B)成负相关(P < 0.05) Fig.6 EXD3 gene expression was positively correlated with the expression of ERCC6 (A) and SIRT6 (D) and negatively correlated with FEN1 (B) (P < 0.05). |
胃癌作为全球最常见的癌症之一,在我国各种恶性肿瘤中发病率居首位,胃癌发病率存在很高的地域差异,约70%的病例发生在发展中国家(其中约一半是在中国),男性的发病率约为女性的两倍[17]。由于人口增长,老龄化,环境污染以及传统生活方式的改变,未来几十年胃癌发病率将显着上升[18]。胃癌发生的主要危险因素是幽门螺杆菌的感染,近80%的胃癌患者感染幽门螺杆菌后出现免疫细胞的炎症反应和异常激活[19],胃癌的发生也是多因子,多步骤相互作用的复杂生物学过程,了解胃癌的发生发展对于其诊断和治疗非常重要[20]。近年来随着高通量测序及基因芯片技术的发展,越来越多的胃癌潜在基因治疗靶点被研究发现,这也为诊断和治疗胃癌提供了一个新的方向。本研究通过对基因表达谱芯片GSE100935数据进行生物信息学分析,获得在正常胃粘膜和胃癌组织间的差异表达基因,在选取的100个差异表达基因中,有TPCN1[21-24]、ATXN2[25-30]、END1[31-35]、DDB2[36-40]等多个基因的功能及相关作用机制已经得到研究报道。
然而,目前关于EXD3基因的功能作用尚无相关报道,尤其EXD3在胃癌中的功能机制未见任何研究。通过在线分析数据库UALCAN来获取EXD3在胃癌中的表达数据,结果显示:相比于正常胃粘膜,EXD3在胃癌组织中表达上调,这与基因芯片表达谱GSE100935的数据分析结果相一致,提示EXD3可能是胃癌中潜在的癌基因;然后从TGCA数据库获取了不同类型胃癌患者EXD3的表达数据,并根据目的基因的表达水平分相应地分为高表达组和低表达组,通过在线分析网站Kaplan-Meier Plotter对胃癌患者总体生存率进行分析,结果发现EXD3高表达的患者总体生存率相对较差,提示预后不良;利用在线网站STRING试图构建EXD3蛋白互作网络,PPI可以更好地理解EXD3与其他蛋白质之间的功能关联,为研究目的基因的功能提供线索,结果发现了共有31个预测的功能蛋白与EXD3相互作用,其中的四种蛋白基因被证实与胃癌的发生发展有关,于是再次利用在线数据库GEPIA对EXD3与这四种基因蛋白进行Spearman相关分析,得出EXD3基因与ERCC6、SIRT6的表达成正相关,与FEN1成负相关,这提示EXD3极有可能与DNA损伤修复有关,并且EXD3可能通过借助正向调节ERCC6发挥作用。GO功能富集分析则发现差异表达基因主要与胃癌细胞核苷酸的新陈代谢调节以及转录因子的活动有关。综上所述,本研究通过利用生物信息分析的方法最终认定EXD3可能是胃癌中的潜在的癌基因,同时生存分析也说明EXD3可能成为监测胃癌患者预后的一个重要的生物学指标,Spearman相关分析和GO功能富集分析说明EXD3可能参与了核苷酸的代谢调节与DNA的损伤修复有关。这一发现能为胃癌患者临床治疗发现新的靶点提供一定的理论依据。
本研究通过基因芯片数据分析寻找胃癌与正常胃粘膜组织中的差异表达基因,并结合生物信息学分析网站对差异表达基因在临床样本中的表达水平进行分析验证,最后进行胃癌患者的分组及生存分析,提出EXD3可能是胃癌中的潜在的促癌基因,并进行GO功能分析与构建EXD3蛋白互作网络,结果提示EXD3极有可能与DNA损伤修复有关。生物信息学分析方法利用了网络芯片数据和在线数据分析网站,缺乏相应的实验数据作为支持验证,存在一定的局限性。因此可以从临床的角度进行后续验证EXD3与肿瘤增殖状态、病理分级、病理类型手术生存状态的关系;也可以从基础的角度验证敲低EXD3后胃癌细胞系的增殖凋亡情况,EXD3在胃癌发生发展中的具体功能及其作用机制仍有待进一步研究证实。
| [1] |
Bray F, Ferlay J, Soerjomataram I, et al. Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J].
CA Cancer J Clin, 2018, 68(6): 394-424.
DOI: 10.3322/caac.v68.6. |
| [2] |
Aoyama T, Huctchins G, Arai T, et al. Identification of a high-risk subtype of intestinal-type Japanese gastric Cancer by quantitative measurement of the luminal tumor proportion[J].
Cancer Med, 2018, 7(10): 4914-23.
DOI: 10.1002/cam4.2018.7.issue-10. |
| [3] |
Arco DD, Estrada ML, Molina RE, et al. Immunohistochemical classification of gastric cancer based on new molecular biomarkers:a potential predictor of survival[J].
Virchows Arch, 2018, 473(6): 687-95.
DOI: 10.1007/s00428-018-2443-9. |
| [4] |
Gao Y, Qi WW, Sun LB, et al. FOXO3 inhibits human gastric adenocarcinoma (AGS) cell growth by promoting autophagy in an acidic microenvironment[J].
Cell Physiol Biochem, 2018, 49(1): 335-48.
DOI: 10.1159/000492884. |
| [5] |
Hao L, Leng J, Xiao RJ, et al. Bioinformatics analysis of the prognostic value of Tripartite Motif 28 in breast cancer[J].
Oncol Lett, 2017, 13(4): 2670-8.
DOI: 10.3892/ol.2017.5764. |
| [6] |
von Mering C, Huynen M, Jaeggi D, et al. STRING:a database of predicted functional associations between proteins[J].
Nucleic Acids Res, 2003, 31(1): 258-61.
DOI: 10.1093/nar/gkg034. |
| [7] |
Shen XB, Li PF, Xu YC, et al. Association of sirtuins with clinicopathological parameters and overall survival in gastric cancer[J].
Oncotarget, 2017, 8(43): 74359-70.
|
| [8] |
He XX, Xie BS. Autophagy inhibition enhanced 5FUinduced cell death in human gastric carcinoma BGC823 cells[J].
Mol Med Rep, 2018, 17(5): 6768-76.
|
| [9] |
Zhou JM, Wu A, Yu XT, et al. SIRT6 inhibits growth of gastric cancer by inhibiting JAK2/STAT3 pathway[J].
Oncol Rep, 2017, 38(2): 1059-66.
DOI: 10.3892/or.2017.5753. |
| [10] |
Liu JW, He CY, Sun LP, et al. The DNA repair gene ERCC6 rs1917799 polymorphism is associated with gastric cancer risk in Chinese[J].
Asian Pac J Cancer Prev, 2013, 14(10): 6103-8.
DOI: 10.7314/APJCP.2013.14.10.6103. |
| [11] |
Jing JJ, Lu YZ, Sun LP, et al. Epistatic SNP interaction of ERCC6 with ERCC8 and their joint protein expression contribute to gastric cancer/atrophic gastritis risk[J].
Oncotarget, 2017, 8(26): 43140-52.
|
| [12] |
Xu Q, Liu JW, He CY, et al. The interaction effects of pri-let-7a-1 rs10739971 with PGC and ERCC6 gene polymorphisms in gastric cancer and atrophic gastritis[J].
PLoS One, 2014, 9(2): e89203.
DOI: 10.1371/journal.pone.0089203. |
| [13] |
Liu JW, Deng N, Xu Q, et al. Polymorphisms of multiple genes involved in NER pathway predict prognosis of gastric cancer[J].
Oncotarget, 2016, 7(30): 48130-42.
|
| [14] |
Matsutani, N. Expression of MRE11 complex (MRE11, RAD50, NBS1) and hRap1 and its relation with telomere regulation, telomerase activity in human gastric carcinomas[J].
Pathobiology, 2001, 69(4): 219-24.
DOI: 10.1159/000055946. |
| [15] |
Altan B, Yokobori T, Ide M, et al. High expression of MRE11-RAD50-NBS1 is associated with poor prognosis and chemoresistance in gastric cancer[J].
Anticancer Res, 2016, 36(10): 5237-47.
DOI: 10.21873/anticanres. |
| [16] |
Wang KX, Chen D. Flap endonuclease 1 is a promising candidate biomarker in gastric cancer and is involved in cell proliferation and apoptosis[J].
Int J Mol Med, 2014, 33(5): 1268-74.
DOI: 10.3892/ijmm.2014.1682. |
| [17] |
Lazar DC, Avram MF, Romoan IA, et al. Prognostic significance of tumor immune microenvironment and immunotherapy:Novel insights and future perspectives in gastric cancer[J].
World J Gastroenterol, 2018, 24(32): 3583-616.
DOI: 10.3748/wjg.v24.i32.3583. |
| [18] |
Asadi-Lari MH, Esmaeili-Hesari A.Changes in health-related quality of Life in oesophagogastric cancer patients participating in palliative and curative therapies[J], 2018, 32(4): 31-2.
|
| [19] |
Che, Y.Helicobacter pylori-induced exosomal Met educates tumourassociated macrophages to promote gastric cancer progression[J], 2018, 22(11): 5708-19.
|
| [20] |
欧玉荣, 康敏, 周蕾, 等. 胃癌中幽门螺杆菌L型感染与MIF、MMP9、VEGF表达的关系[J].
南方医科大学学报, 2014, 34(2): 180-7.
|
| [21] |
Garcia-Rua V, Feijoo-Bandin S, Garcia-Vence M, et al. Metabolic alterations derived from absence of Two-Pore Channel 1 at cardiac level[J].
J Biosci, 2016, 41(4): 643-58.
DOI: 10.1007/s12038-016-9647-4. |
| [22] |
Arndt L, Castonguay J, Arlt E, et al. NAADP and the two-pore channel protein 1 participate in the acrosome reaction in mammalian spermatozoa[J].
Mol Biol Cell, 2014, 25(6): 948-64.
DOI: 10.1091/mbc.e13-09-0523. |
| [23] |
Patel SC, E Brailoiu N. (2+) signals through two-pore channel-1 require arginine residues in the first S4-S5 linker[J].
Cell Calcium, 2017, 68(12): 1-4.
|
| [24] |
Horton JS, Wakano CT, Speck M, et al. Two-pore Channel 1 interacts with citron kinase, regulating completion of cytokinesis[J].
Channels (Austin), 2015, 9(1): 21-9.
DOI: 10.4161/19336950.2014.978676. |
| [25] |
Auburger, G. 12q24 locus association with type 1 diabetes:SH2B3 or ATXN2?[J].
World J Diabetes, 2014, 5(3): 316-27.
DOI: 10.4239/wjd.v5.i3.316. |
| [26] |
Borghero G, Pugliatti M, Marrosu FA, et al. ATXN2 is a modifier of phenotype in ALS patients of Sardinian ancestry[J].
Neurobiol Aging, 2015, 36(10): e1-5.
|
| [27] |
Di ng, D. ATXN2 polymorphism modulates age at onset in Machado-Joseph disease[J].
Brain, 2016, 139(10): e59.
|
| [28] |
Chio A, Calvo A, Moglia C, et al. ATXN2 polyQ intermediate repeats are a modifier of ALS survival[J].
Neurology, 2015, 84(3): 251-8.
DOI: 10.1212/WNL.0000000000001159. |
| [29] |
Alekseev S, Kool H, Rebel H, et al. Enhanced DDB2 expression protects mice from carcinogenic effects of chronic UV-B irradiation[J].
Cancer Res, 2005, 65(22): 10298-306.
DOI: 10.1158/0008-5472.CAN-05-2295. |
| [30] |
Almaguer-Mederos LE, Aguilera-Rodriguez RA, Almaguer-Gotay D, et al. Normal ATXN2 alleles influences on the age at onset in spinocerebellar ataxia type 2[J].
Mov Disord, 2017, 32(9): 1329-30.
DOI: 10.1002/mds.27073. |
| [31] |
Fa ng, Z. Association of endothelin-1 gene polymorphisms with essential hypertension in a Chinese population[J].
Genet Mol Res, 2017, 16(3): gmr16037446.
|
| [32] |
Becraft PW. Cell fate specification in the cereal endosperm[J].
Semin Cell Dev Biol, 2001, 12(5): 387-94.
DOI: 10.1006/scdb.2001.0268. |
| [33] |
Suzuki A, Niimi Y, Shinmyozu K, et al. Dead end1 is an essential partner of NANOS2 for selective binding of target RNAs in male germ cell development[J].
EMBO Rep, 2016, 17(1): 37-46.
DOI: 10.15252/embr.201540828. |
| [34] |
Li M, Lopato S, Hrmova M, et al. Expression patterns and protein structure of a lipid transfer protein END1 from Arabidopsis[J].
Planta, 2014, 240(6): 1319-34.
DOI: 10.1007/s00425-014-2155-6. |
| [35] |
Altmae S, Reimand J, Hovatta O, et al. Research resource:interactome of human embryo implantation:identification of gene expression pathways, regulation, and integrated regulatory networks[J].
Mol Endocrinol, 2012, 26(1): 203-17.
DOI: 10.1210/me.2011-1196. |
| [36] |
Alekseev, S. Cellular concentrations of DDB2 regulate dynamic binding of DDB1 at UV-induced DNA damage[J].
Mol Cell Biol, 2008, 28(24): 7402-13.
DOI: 10.1128/MCB.01108-08. |
| [37] |
Bagchi S, Raychaudhuri P. Damaged-DNA binding protein-2 drives apoptosis following DNA damage[J].
Cell Div, 2010, 5(1): 3.
DOI: 10.1186/1747-1028-5-3. |
| [38] |
Barbieux C, Bacharouche J, Soussen C, et al. DDB2(damagedDNA binding 2) protein:a new modulator of nanomechanical properties and cell adhesion of breast cancer cells[J].
Nanoscale, 2016, 8(9): 5268-79.
DOI: 10.1039/C5NR09126H. |
| [39] |
Cui TT, Srivastava AK, Han CH, et al. DDB2 represses ovarian cancer cell dedifferentiation by suppressing ALDH1A1[J].
Cell Death Dis, 2018, 9(5): 561-82.
DOI: 10.1038/s41419-018-0585-y. |
| [40] |
Barakat BM, Wang QE, Han CH, et al. Overexpression of DDB2 enhances the sensitivity of human ovarian cancer cells to cisplatin by augmenting cellular apoptosis[J].
Int J Cancer, 2010, 127(4): 977-88.
|
 2019, Vol. 39
2019, Vol. 39

