2. 南方医科大学南方医院心血管外科,广东 广州 510515
2. Department of Cardiovascular Surgery, Nanfang Hospital, Southern Medical University, Guangzhou 510515, China
食管癌是我国常见恶性肿瘤之一,虽然根治性食管癌切除术联合化疗的治疗方案已经普及,但因为食管癌的早期诊断困难,其5年总体生存率仍较低[1-2]。因此,针对食管癌高危人群,寻找有效分子标志物用于筛选、早期诊断以及预后判断,是临床关注热点。近年来,分子生物学研究已阐明食管癌发生过程中大量异常表达的分子事件,并由此筛选出一系列的特异性治疗靶点和生物学标志[5-7]。食管癌本身为多基因、多环节、多因素介导的恶性肿瘤,采取单一肿瘤标志物用于诊断的准确度不高,而通过选取多种肿瘤标志物进行联合测定可避免单一肿瘤标志物测定中的局限性,其中利用肿瘤特异表达的受体及其配体进行肿瘤诊断预测,是一种联合测定的方法之一[8-10]。
转化生长因子激活激酶1(TAK1),又名MAP3K7或NR2C2,在功能与序列上与丝裂原激活蛋白激酶激酶激酶(MAPKKK)相关,属于MAP3Ks家族成员之一[11]。TAK1可以被细胞内多种细胞因子包括TGF-β、TNF、IL-1、LPS等激活,进而参与NF-κB等包括多种信号通路的调节,它与多种人类疾病的发生发展密切相关[12-13]。TAK1在多种肠癌、乳腺癌等多种肿瘤的形成、浸润与转移有密切联系,可能作为肿瘤的特异性分子标记物之一[14-15]。转化生长因子β活化激酶结合蛋白1(TAB1)为TAK1的特异性配体[16],但目前TAK1、TAB1在食管癌患者中的表达特点及临床意义尚无相关文献报道。本研究通过免疫组织化学染色检测食管癌组织中TAK1、TAB1的表达水平,并分析它们与食管癌临床病理参数及预后的相关性,有助于食管癌患者获得精准的靶向治疗,并更加精确地预测食管癌患者的预后。
1 资料和方法 1.1 标本来源组织标本来自2009~2013年间在广东省广州市南方医院胸外科行食管癌根治性切除术的84名患者,患者均通过病理组织活检确诊,术前未进行化疗、放疗等特殊治疗,且无糖尿病、甲亢、类风湿关节炎等其它系统疾病,均于术后开始全程跟踪随访。实验所取癌组织为距病灶边缘 < 1 cm组织,术后经病理学检查证实为食管癌,癌旁组织为超过肿瘤边缘 > 2 cm组织。患者临床病理分期采用第8版UICC/AJCC食管癌手术病理分期(pTNM分期)[17]。
1.2 主要试剂与仪器兔抗人TAK1、TAB1多克隆抗体(Proteintech);HRP标记山羊抗兔二抗(武汉谷歌);DakoEnVision Plus过氧化物酶染色试剂盒(丹麦Dako);正置相差显微镜(Olympus)。
1.3 免疫组织化学染色分析新鲜标本取材后用4%多聚甲醛固定,脱水,包埋成石蜡标本,切成约4 μm切片,经常规烤片,脱蜡,水化,微波炉法抗原修复15 min,3%过氧化氢阻断内源性过氧化物酶15 min,山羊血清白蛋白孵育30 min。其后分别孵育TAK1一抗(anti-TAK1,浓度为1:100)及TAB1一抗(anti-TAB1,浓度为1:100),在4 ℃条件下孵育过夜,次日孵育二抗1 h,二氨基联苯胺(DAB)镜下显色,苏木精复染后,梯度酒精脱水,二甲苯透明,中性树脂封片。
染色半定量分析:由病理科室具有10年以上且经验丰富的2名高年资医师对染色结果进行双盲评价,每例切片均应分别对超过200个观察细胞数的高倍镜视野进行独立评价,且这样的高倍镜视野不能少于5个。结果判定标准:凡可见胞浆着色均判定为阳性,结果则由以下两项评分的乘积来判定:(1)以切片中细胞着色深浅评分:细胞无着色为0分;浅黄色为1分;棕黄色为2分;棕褐色为3分;(2)以阳性细胞数在同类细胞数中所占百分比评分:无阳性细胞为0分;阳性细胞占比 < M10%为1分;10%~50%为2分;> 50%为3分。两项乘积为最终的得分。定义得分 < 3分定义为低表达,≥4分定义为高表达。
1.4 生物信息学分析采用GEPIA数据库(http://gepia.cancer-pku.cn/detail.php?clicktag=correlation)评价TAK1和TAB1基因表达水平的相关性[18]。
1.5 统计学分析所有数据均采用IBM SPSS 20.0处理。计量资料采用均数±标准差进行统计描述。计数资料比较则采用χ2检验。采用Kaplan-Meier法进行生存分析,并用Log-rank检验比较组间差异。以α=0.05为检验标准,对以上结果均进行双侧检验。
2 结果 2.1 TAK1与TAB1蛋白在食管癌组织中高表达免疫组化实验表明,TAK1与TAB1蛋白均表达于细胞质,细胞核基本无表达(图 1)。其中,TAK1在癌组织中的高表达率为65.5%(55/84),显著高于癌旁组织(25.0%,21/84;P < 0.01);而TAB1在癌组织的高表达率为52.4%(44/84),也明显高于癌旁组织(27.4%,23/84;P < 0.05)。
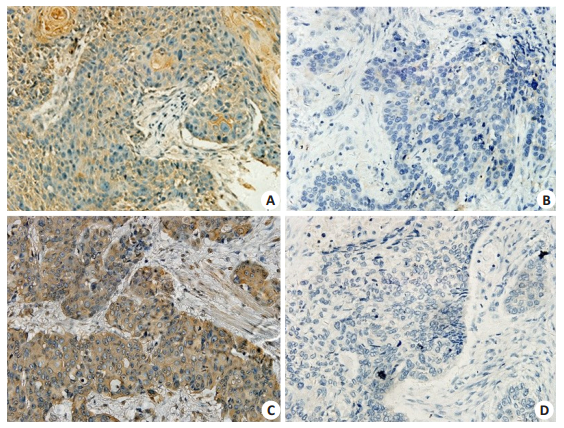
|
图 1 TAK1、TAB1蛋白在食管癌组织中的表达 Figure 1 Expression of TAK1 and TAB1 proteins in esophageal cancer tissues (Original magnification: × 400). A: TAK1 high expression; B: TAK1 low expression; C: TAB1 high expression; D: TAB1 low expression. |
统计学分析TAK1与TAB1表达水平与食管癌患者临床病理参数的相关性发现,TAK1蛋白的表达与肿瘤大小(P < 0.05)、肿瘤浸润深度(P < 0.01)、淋巴结转移(P < 0.01)、病理TNM分期(P < 0.001)等显著相关,而与患者性别、年龄、肿瘤位置、病理分化程度等参数无关(表 1);而TAB1蛋白的表达与肿瘤浸润深度(P < 0.05)、淋巴结转移(P < 0.05)、病理TNM分期(P < 0.05)等显著相关,与患者性别、年龄、肿瘤大小、肿瘤位置、病理分化程度等参数无关(表 1)。
| 表 1 TAK1、TAB1表达水平与食管癌患者临床病理参数的相关性分析 Table 1 Correlations of TAB1 and TAK1 protein expressions and the clinicopathological parameters of the patients with esophageal cancer (n=84) |
采用GEPIA数据库(http://gepia.cancer-pku.cn/detail.php?gene=)评价TAK1(即NR2C2)与TAB1基因的相关性发现,无论在正常食道粘膜组织(r=0.91,P < 0.001,图 2A),还是在食管癌组织(r=0.39,P < 0.001,图 2B),两者均存在显著正相关;本研究的免疫组化定性分组的实验结果也证实,TAK1与TAB1蛋白存在正相关表达(r=0.360,P < 0.01,表 2)。
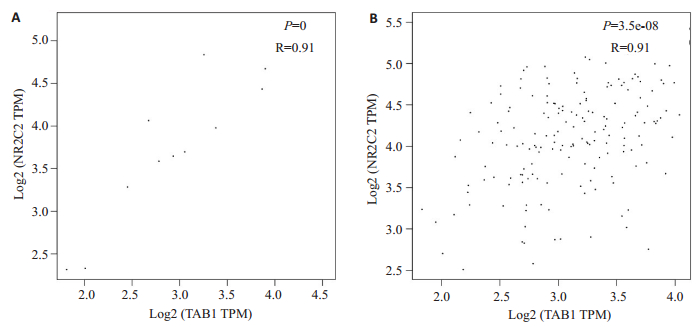
|
图 2 食管癌组织TAK1、TAB1基因表达水平间的相关性分析(GEPIA数据库) Figure 2 Correlation between TAK1 and TAB1 gene expression (GEPAI dataset). A: Normal group; B: Esophageal cancer group. |
| 表 2 食管癌组织TAK1、TAB1蛋白表达水平间的相关性分析 Table 2 Correlation between TAK1 and TAB1 protein expression |
基于免疫组化结果,将患者分为共表达组(同时高表达TAK1与TAB1)与非共表达组(单独高表达TAK1、TAB1、同时低表达TAB1与TAK1),统计分析表明,共表达TAK1与TAB1蛋白与肿瘤大小(P < 0.01)、肿瘤浸润深度(P < 0.05)、淋巴结转移(P < 0.01)、病理TNM分期(P < 0.01)等参数显著相关,而与患者性别、年龄、肿瘤位置、病理分化程度等参数无关(表 1)。
2.5 TAK1与TAB1的表达与食管癌患者无瘤生存时间的关系采用Kaplan-Meier法分析食管癌组织TAK1、TAB1蛋白表达水平与患者无瘤生存时间的相关性。结果显示,食管癌组织TAK1高表达组患者术后无瘤生存期显著短于低表达组(18.0±2.65月vs 44.0± 5.36月,P < 0.05,图 3A);食管癌组织TAB1的表达与患者术后无瘤生存期未见显著差异(P > 0.05,图 3B)。此外,共表达TAK1/TAB1的患者术后无瘤生存期显著短于非共表达组(18.0±0.63月vs 44.0±8.27月,P < 0.05,图 3C)。
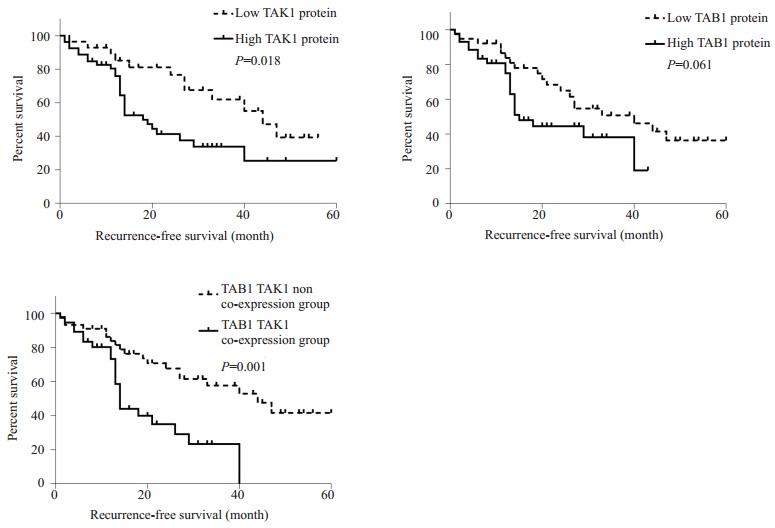
|
图 3 TAK1、TAB1表达及两者共表达和食管癌患者无瘤生存时间的关系 Figure 3 Correlation of different TAK1 and TAB1 expression patterns with the recurrence-free survival of esophageal cancer patients. A: TAK1 protein; B: TAB1 protein; C: TBA1-TAK1. |
本研究中,我们采用免疫组织化学染色检测了食管癌患者癌、癌旁组织TAK1与TAB1蛋白的表达,发现两者均在癌组织中表达上调,且癌组织中TAK1、TAB1蛋白的表达水平与肿瘤大小(仅TAK1)、肿瘤浸润深度、淋巴结转移、病理TNM分期等参数显著相关。生存分析显示癌组织高表达TAK1及共表达TAK1/TAB1蛋白的患者术后无瘤生存期均显著短于低表达TAK1及非共表达组。
肿瘤的发生发展与炎症反应密切相关,尤其在食管癌中,慢性食管炎、Barrett食管炎等已是公认的癌前病变。NF-κB和MAPK信号通路在炎症反应介导的肿瘤发生发展中发挥了重要的作用[19-20]。而TAK1是MAP3K家族的成员之一,它可以被众多炎症调节因子(如TGF-β、BMP、TNF-α、IL-1β、TCR、BCR等)所激活[21-23]。细胞通过TGF-β受体或BMP受体接收细胞外信号后,激活E3泛素连接酶XIAP,后者直接作用于TAB1,诱使TAB1结合于TAK1并激活其功能,导致下游NF-κB和MAPK(JUN和p38)信号通路活化,进而上调转录因子NF-κB和AP-1下游靶基因的表达[24]。因此,TAK1、TAB1是肿瘤细胞内汇集NF-κB和MAPK通路信号的关键分子,它们的异常表达与激活可能参与介导了食管癌的发生和发展。
既往有研究报道,TAK1在胃癌[25]、胰腺癌[26]中表达上调,并且其表达水平与胃癌、胰腺癌有无淋巴结转移、TNM分期等恶性行为高度相关;体内外实验亦表明,选择性抑制TAK1后,胃癌与胰腺癌细胞的增殖、迁移与侵袭能力显著降低;此外,敲低胰腺癌细胞株中TAK1的表达,不仅诱导肿瘤细胞发生凋亡,还导致细胞产生G1/S期阻滞,而这一效应正是通过抑制NF-κB和MAPK通路的活化而实现的。同时研究亦发现抑制TAK1蛋白可促进结肠癌细胞凋亡[27]。在本研究中,我们采用免疫组织化学染色检测TAK1在食管癌中的表达,发现TAK1在癌组织中较癌旁组织呈显著高表达,它的表达水平与食管癌的肿瘤大小、浸润深度、有无淋巴结转移、病理TNM分期等临床病理参数显著相关,提示高表达TAK1可能使食管癌细胞获得了更强的侵袭转移能力。
虽然TAK1可被众多细胞外炎症调节因子所激活,但TAB家族蛋白与TAK1的结合是激活TAK1必备的环节。TAB家族共有TAB1、TAB2、TAB3成员,TAK1的自我磷酸化激活有赖于TAK1/TAB1/TAB2或TAK1/TAB1/TAB3复合物的形成[24]。TAB1是两种复合物的共有组分,研究显示,TAK1蛋白的自我磷酸化激活首先发生在Ser192位点,而这一过程在TAB1存在的情况下才得以完成,因此,TAB1是TAK1最重要的激活配体,TAK1/TAB1的共表达可能显著影响癌细胞的恶性生物学功能,进而影响肿瘤患者的预后[28]。研究发现,同一队列非小细胞肺癌患者癌组织中,TAK1与TAB1的表达与患者有无淋巴结转移和TNM分期均相关,且癌组织TAK1、TAB1表达阳性组较阴性组术后五年生存率更低[29]。然而,上述研究未进一步分析TAK1与TAB1表达水平间的相关性,以及TAK1/TAB1共表达的临床意义。而在本研究中,我们通过生物信息学分析和免疫组化染色证实,TAK1与TAB1的表达存在显著的正相关。此外,食管癌组织是否共表达TAK1/TAB1与肿瘤大小、肿瘤浸润深度、淋巴结转移、病理TNM分期等参数显著相关,虽然TAB1的表达与患者术后无瘤生存期之间尚未见统计学差异(P=0.061;可能源于样本选择的偶然性和样本量的限制),但是,共表达TAK1/ TAB1患者术后无瘤生存期较非共表达者明显缩短,一方面提示TAK1-TAB1轴调控的下游效应可能在食管癌的转移复发中发挥关键作用,另一方面,可能也提示综合评估TAK1和TAB1的表达与无瘤生存期的关系可能比单独评价单一指标更具有临床意义。
总之,本研究发现TAK1/TAB1在食管癌组织中表达上调,且两者的高表达与食管癌的多种恶性生物学特性显著相关,食道癌组织高表达TAK1或共表达TAK1/ TAB1提示患者无瘤生存期缩短,说明TAK1、TAB1的异常表达与食管癌的疾病进展密切相关,而且联合检测二者的表达可作为更为理想的肿瘤标志物辅助食管癌的诊断并指导临床治疗,然而TAK1-TAB1轴发挥生物学效应的具体分子机制仍待进一步研究。
| [1] |
Zhang N, Wen D, Shan B, et al. Clustering and geographic variation of upper gastrointestinal cancers in a high-risk region of esophageal cancer in northern China[J].
Asian Pac J Cancer Prev, 2011, 12(1): 193-8.
|
| [2] |
Lin Y, Totsuka Y, Shan B, et al. Esophageal cancer in high-risk areas of China: research progress and challenges[J].
Ann Epidemiol, 2017, 27(3): 215-21.
DOI: 10.1016/j.annepidem.2016.11.004. |
| [3] |
Yamanouchi S, Sako Y, Suemitsu S, et al. Early esophageal cancer with epidermization diagnosed and treated with endoscopic resection[J].
Clin J Gastroenterol, 2018, 11(1): 29-33.
DOI: 10.1007/s12328-017-0792-6. |
| [4] |
Guo LW, Huang HY, Shi JF, et al. Medical expenditure for esophageal cancer in China: a 10-year multicenter retrospective survey (2002- 2011[J].
Chin J Cancer, 2017, 36(1): 73.
DOI: 10.1186/s40880-017-0242-3. |
| [5] |
Uemura N, Kondo T. Current status of proteomics of esophageal carcinoma[J].
Expert Rev Proteomics, 2016, 13(11): 1-12.
|
| [6] |
Wen Y, Hu X. Expression of esophageal carcinoma related gene 4 (ECRG4) and its clinical significance in prognosis of esophageal carcinoma[J].
Int J Clin Exp Pathol, 2015, 8(11): 14772-8.
|
| [7] |
Liu X, Wang Z, Zhang G, et al. High TRAF6 expression is associated with esophageal carcinoma recurrence and prompts cancer cell invasion[J].
Oncol Res, 2017, 25(4): 485-93.
DOI: 10.3727/096504016X14749340314441. |
| [8] |
Zhao CB, Bao JM, Lu YJ, et al. Co-expression of RAGE and HMGB1 is associated with cancer progression and poor patient outcome of prostate cancer[J].
Am J Cancer Res, 2014, 4(4): 369-77.
|
| [9] |
Sugiura K, Ozawa S, Kitagawa Y, et al. Co-expression of aFGF and FGFR-1 is predictive of a poor prognosis in patients with esophageal squamous cell carcinoma[J].
Oncol Rep, 2007, 17(3): 557-64.
|
| [10] |
Hao Y, Wu W, Shi F, et al. Prediction of long noncoding RNA functions with co-expression network in esophageal squamous cell carcinoma[J].
BMC Cancer, 2015, 15(1): 168.
DOI: 10.1186/s12885-015-1179-z. |
| [11] |
Geuking P, Narasimamurthy R, Lemaitre B, et al. A non-redundant role for Drosophila Mkk4 and hemipterous/Mkk7 in TAK1-mediated activation of JNK[J].
PLoS One, 2009, 4(11): e7709.
DOI: 10.1371/journal.pone.0007709. |
| [12] |
Mihaly SR, Ninomiya-Tsuji J, Morioka S. TAK1 control of cell death[J].
Cell Death Differ, 2014, 21(11): 1667-76.
DOI: 10.1038/cdd.2014.123. |
| [13] |
Roh YS, Song J, Seki E. TAK1 regulates hepatic cell survival and carcinogenesis[J].
J Gastroenterol, 2014, 49(2): 185-94.
DOI: 10.1007/s00535-013-0931-x. |
| [14] |
Yuan G, Zhao Y, Wu D, et al. miRNA-20a upregulates TAK1 and increases proliferation in osteosarcoma cells[J].
Future Oncol, 2018, 14(5): 461-9.
DOI: 10.2217/fon-2017-0490. |
| [15] |
Totzke J, Gurbani D, Raphemot R, et al. Takinib, a selective TAK1 inhibitor, broadens the therapeutic efficacy of TNF-α inhibition for cancer and autoimmune disease[J].
Cell Chem Biol, 2017, 24(8): 1029-1039.
DOI: 10.1016/j.chembiol.2017.07.011. |
| [16] |
Bao SY, Sun QX, Yao CL. The interaction of TAK1 and TAB1 enhances LPS-induced cytokine release via modulating NF-κB activation (Larimichthys crocea)[J].
Fish Shellfish Immunol, 2018, 74: 450-8.
DOI: 10.1016/j.fsi.2018.01.005. |
| [17] |
Donohoe CL, Phillips AW. Cancer of the esophagus and esophagogastric junction: an 8th edition staging primer[J].
J Thorac Dis, 2017, 9(3).
|
| [18] |
Tang Z, Li C, Kang B, et al. GEPIA: a web server for cancer and normal gene expression profiling and interactive analyses[J].
NucleicAcids Res, 2017, 45(W1): W98-102.
DOI: 10.1093/nar/gkx247. |
| [19] |
Hughes PE, Caenepeel S, Wu LC. Targeted therapy and checkpoint immunotherapy combinations for the treatment of cancer[J].
Trends Immunol, 2016, 37(7): 462-76.
DOI: 10.1016/j.it.2016.04.010. |
| [20] |
Taniguchi K, Karin M. NF-kappaB, inflammation, immunity and cancer: coming of age[J].
Nat Rev Immunol, 2018, 18(5): 309-24.
DOI: 10.1038/nri.2017.142. |
| [21] |
Yamaguchi K, Shirakabe K, Shibuya H, et al. Identification of a member of the MAPKKK family as a potential mediator of TGF-beta signal transduction[J].
Science, 1995, 270(5244): 2008-11.
DOI: 10.1126/science.270.5244.2008. |
| [22] |
Dai L, Aye Thu C, Liu XY, et al. TAK1, more than just innate immunity[J].
IUBMB Life, 2012, 64(10): 825-34.
DOI: 10.1002/iub.v64.10. |
| [23] |
Ajibade AA, Wang HY, Wang RF. Cell type-specific function of TAK1 in innate immune signaling[J].
Trends Immunol, 2013, 34(7): 307-16.
DOI: 10.1016/j.it.2013.03.007. |
| [24] |
Hirata Y, Takahashi M, Morishita T, et al. Post-Translational modifications of the TAK1-TAB complex[J].
Int J Mol Sci, 2017, 18(1): 205.
DOI: 10.3390/ijms18010205. |
| [25] |
Yang YE, Qiu YD, Tang MB, et al. Expression and function of transforming growth factor-beta-activated protein kinase 1 in gastric cancer[J].
Mol Med Rep, 2017, 16(3): 3103-10.
DOI: 10.3892/mmr.2017.6998. |
| [26] |
Huang FT, Peng JF, Cheng WJ, et al. MiR-143 targeting TAK1 attenuates pancreatic ductal adenocarcinoma progression via MAPK and NF-κB pathway in vitro[J].
Dig Dis Sci, 2017, 62(4): 944-57.
DOI: 10.1007/s10620-017-4472-7. |
| [27] |
Singh A, Sweeney MF, Yu M, et al. TAK1 inhibition promotes apoptosis in KRAS-dependent colon cancers[J].
Cell, 2012, 148(4): 639-50.
DOI: 10.1016/j.cell.2011.12.033. |
| [28] |
Scholz R, Sidler CL, Thali RF, et al. Autoactivation of transforming growth factor beta-activated kinase 1 is a sequential bimolecular process[J].
J Biol Chem, 2010, 285(33): 25753-66.
DOI: 10.1074/jbc.M109.093468. |
| [29] |
Zhu J, Li Q, He JT, et al. Expression of TAK1/TAB1 expression in non-small cell lung carcinoma and adjacent normal tissues and their clinical significance[J].
Int J Clin Exp Pathol, 2015, 8(12): 15801-7.
|
 2018, Vol. 38
2018, Vol. 38

