2. 南方医科大学 医学图像处理重点实验室,广东 广州 510515
2. Key Lab for Medical Imaging of Southern Medical University, Guangzhou, 510515, China
丘脑、海马体、伏隔核、尾状核等关键脑结构的位置、体积、形态等的变化与多种脑部疾病息息相关,对其精准分割是进行定量分析的前提[1]。脑部磁共振图像中不同的脑组织相互混迭,结构复杂,同时,由于磁共振图像受到伪影、磁场的不均匀性及噪声的影响,使得在磁共振图像中这些结构边缘模糊、边界难以界定,因此传统的分割方法储如阈值法、区域生长法、水平集的方法等多种方法由于各种原因难以准确分割,例如阈值法[2] 只采用了图像中像素的灰度信息,没有使用像素的空间信息,因此对噪声和图像的非均匀性敏感。区域生长法[3] 对每一个需要提取的目标都必须手动指定一个种子点,并且由于受噪声和偏移场的影响,会导致区域内有空洞甚至是根本不连续的分割结果。而水平集方法[4]需要通过周期性地初始化水平集函数,来获得稳定的曲线演化,而初始化水平集函数计算量大,并且很难确定何时和如何重新初始化水平集函数。
而基于图谱配准的分割方法利用手动分割精度高的优势,将分割问题转化为配准问题。Baillard等[5]利用基于单图谱的分割方法分割出脑部结构。Heckemann 等[6]利用29 组图谱分割出了67 个脑部结构。Klein 等[7] 应用弹性配准的方式分割3D MR 图像中的前列腺。Isgum 等[8]使用前向搜索法来减少图谱数以提高分割速度。Jia 等[9]提出把基于迭代多图谱方法和基于树形结构的方法相结合以充分考虑图谱间及待分割图像间相互关系,从而提高了图像的分割精确。以上基于图像配准的分割方法各有优势,但得到的结果表面不光滑,在边界处会出现阶梯状,离临床应用还存在一定差距。本文将多图谱配准与引导滤波相结合,提出了一种基于引导滤波的多图谱医学图像分割方法。
1 材料与方法 1.1 实验数据为了便于和目前国际上主流的分割方法进行比较,我们采用MICCAI 2013 竞赛中的公开脑部数据进行了左、右海马的分割实验,该数据的训练集中共有35 组脑部T1 加权磁共振三维数据,包含灰度图像及对应的专家手动勾画的标号图像。我们随机的选择20 组作为图谱,其余15 组作为测试。计算重叠率Dice 和Hausdorff 距离用于验证分割方法的精度,其定义分别为:
| $\begin{align} & \text{Dice}\left( \text{A},\text{B} \right)=\frac{2V\left( A\bigcap B \right)}{V\left( A \right)+V\left( B \right)} \\ & \text{HD=max}\left( {{\text{H}}_{1}},{{\text{H}}_{2}} \right) \\ \end{align}$ |
其中分别表示自动分割结果和专家手动勾画结果的体积,V(A∩B)表示自动分割结果和专家手动勾画结果重叠部分的体积;H1=maxa∈A(minb∈Bd(a,b)),H2=maxb∈B(mina∈Ad(a,b)),d(a,b)表示a,b两个点的欧氏距离。
1.2 算法流程本文方法由图像预处理、多图谱配准、标号融合、引导滤波、阈值处理五部分组成。本文方法系统流程图如图 1所示。
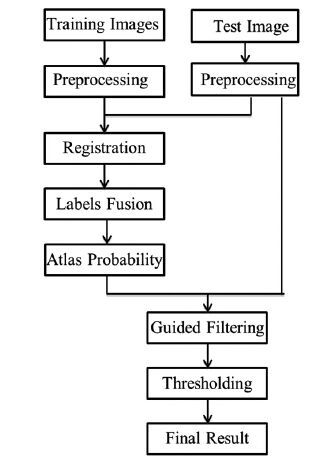
|
图 1 系统流程图 Figure 1 Flowchart of the proposed method. |
图像预处理是图像分割的第一步,在前期的工作基础中,我们发现脑壳和偏移场会对图像的分割有一定的影响,我们用BET 算法[10]去除脑壳;用N4 算法[11]去除磁共振图像中的偏移场;进行灰度归一化完成图像的预处理。下图 2 为预处理后的脑部磁共振图像。
|
图 2 预处理后的脑部磁共振图像 Figure 2 Preprocessed brain MR image. |
在基于图谱配准的分割方法中,图谱的选择对分割结果有一定的影响,为了降低图谱选择的不确定性,我们选取多张图谱,图谱由两部分组成:图谱灰度图像Ii 和影像专家手动分割好的标号图像Li,Li 是一个二值图像。首先,将图谱灰度图像Ii配准到待分割图像T 得到变形场。然后用变形场对图谱标号图像Li变形,得到变形后的标号图像L'i,最后融合变形后的标号图像L'i即可得到最终的分割结果。
1.2.3 概率图谱传统方法在融合时一般需要重新计算变形后的图谱与待分割图像的相似性,如互信息量、互相关系数、归一化的互相关或者其它的相似性测度,这种方法额外增加了计算量,比较耗时,而且这种整体的相似性测度并不能很好的度量每个像素点在配准、分割时的作用。ANTS 配准方法利用局部相似性作为配准的相似性测度,并且对变形场施加了有效的约束及平滑可以达到比较好的配准效果[12]。我们以ANTS 配准的局部相似性作为融合时的权重,相似性测度高的像素点,说明配准更加准确,对最后的分割结果影响就越大。局部权重概率图谱的计算公式为:
| $\text{L=}\frac{\sum\nolimits_{i=1}^{N}{{{\omega }_{i}}}\times L{{'}_{i}}}{\sum\nolimits_{i=1}^{N}{{{\omega }_{i}}}}$ | (1) |
其中ωi为ANTS配准的局部相似性测度,L'i为变形后的标号图像。
1.2.4 引导滤波引导滤波[13]于2013 年提出,它是一种边缘保持滤波,可以实现图像边缘的平滑、细节增强、以及图像融合去噪等功能。主要原理是通过一幅引导图像对输入图像进行滤波处理,输出图像在保留输入图像整体特征的时候还能充分获得引导图像的边缘特征。该算法涉及一个引导图像I,输入图像P,输出图像O。
下图显示了引导滤波的结果,图 3A 为133×138 大小的脑部MR 图像。将图 3A 作为引导滤波的引导图像和输入图像,图 3B 为滤波输出图像。对比图 3A 和图 3B 可以看出,引导滤波会保留图像的边缘特征,但会弱化图像的细节。改变输入图像,图A 仍为引导图像,将图 3C作为输入图像,而图 3D为引导滤波后的结果。观察图 3C,可以看出图 3C受噪声影响,并且在图像边缘处有孤立点,对比图 3C图 3D可以看出,引导滤波后,图像的噪声被消除,并且那些孤立的点和图像边缘连接到一起。如前所述,输出图像在保留输入图像整体特征的时候还能充分获得引导图像的边缘特征。
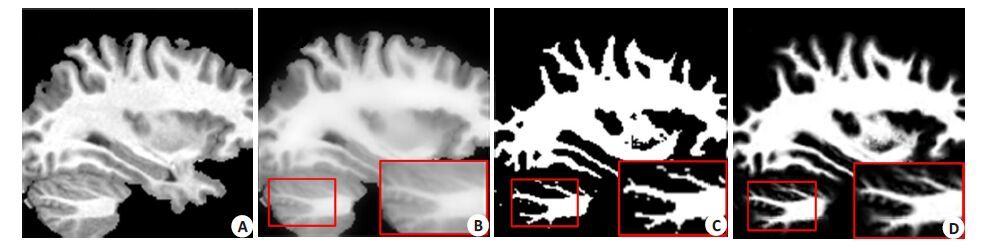
|
图 3 引导滤波的结果 Figure 3 Two examples of guided filtering. A,C: Two input images; B,D: The output images of the former two images independently. |
在本文中,由公式(1)得到局部权重概率图谱,将得到的概率图谱L 进行引导滤波,引导图像为待分割图像I。通过这种局部线性模型,我们将引导图像(待分割图像)中的结构信息引入到输出图像,滤波后的概率图谱(输出图像)记作FL,则最终的分割结果R为:
| $\text{R=}\left\{ \begin{align} & 1,FL\ge M \\ & 0,FL<M \\ \end{align} \right.$ | (5) |
为验证本文方法的有效性,我们与目前国际上主流的几种方法进行了比较,分别为STAPLE[14],Spatial STAPLE [15],Major Voting [16],Weight Voting [17] 和SIMPLE[18]。操作平台为Windows 7 Service Pack 1,Inte(l R)Core(TM)i5-3330 CPU,8.0 G内存。
左、右海马的分割结果:表 1 给出了不同分割方法对左、右海马分割结果的重叠率。STAPLE,Spatial STAPLE,Major Voting,Weight Voting 和SIMPLE 五种方法对左、右海马分割的重叠率分别为0.84 及0.85 左右,而本文方法可以达到0.86 及0.87,提高了2.4%。图 4 给出了重叠率盒状图(第一行)和Hausdorff 距离盒状图(第二行),红线为中位数。从盒状图中可以看出本文方法重叠率和距离值都比较集中,而其它方法的结果则比较分散,说明本文方法具有较好的一致性。重叠率越高说明分割精度越高,本文方法的重叠率中位数在盒状图的偏上方,说明对于大部分测试图像都可以达到较好的分割。Hausdorff 距离值越小说明分割精度越高,本文方法的Hausdorff 距离中位数在盒状图的偏下方,同样可以说明对于大部分测试图像本文方法可以达到较好的分割,红点为出格点,即偏离平均值较远的点,本文方法没有出格点,而其它方法都存在出格点,同样说明了本文方法具有较好的一致性。
| 表 1 不同方法左、右海马分割结果与手动分割结果的重叠率 Table 1 Dice similarity for the various segmetation as compared with manual segmentations |
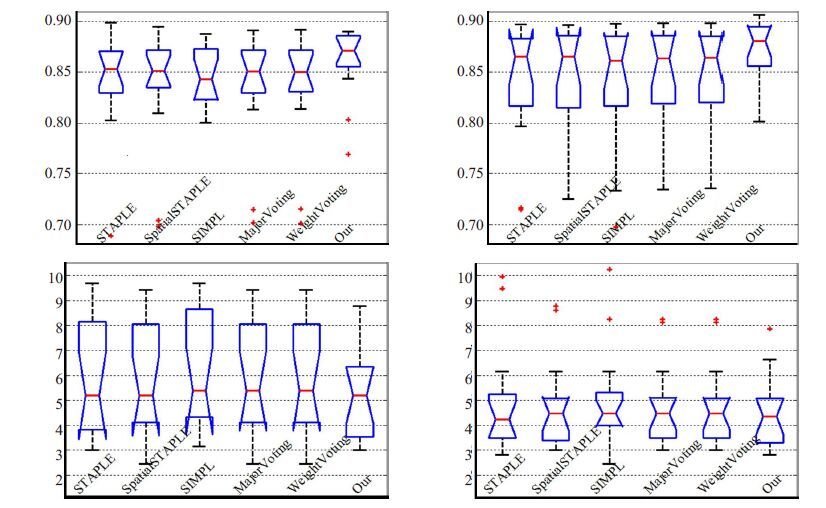
|
图 4 不同方法分割结果重叠率(第一行)和Hausdorff距离(第二行)的箱线图 Figure 4 Box-plot of performance metric for segmentation methods,as compared against manual segmeatation . The top row contains the dice similarity,and the bottom row contains the Hausdorff distance. The left column is the left hippocampus result and the right column is the right hippocampus. |
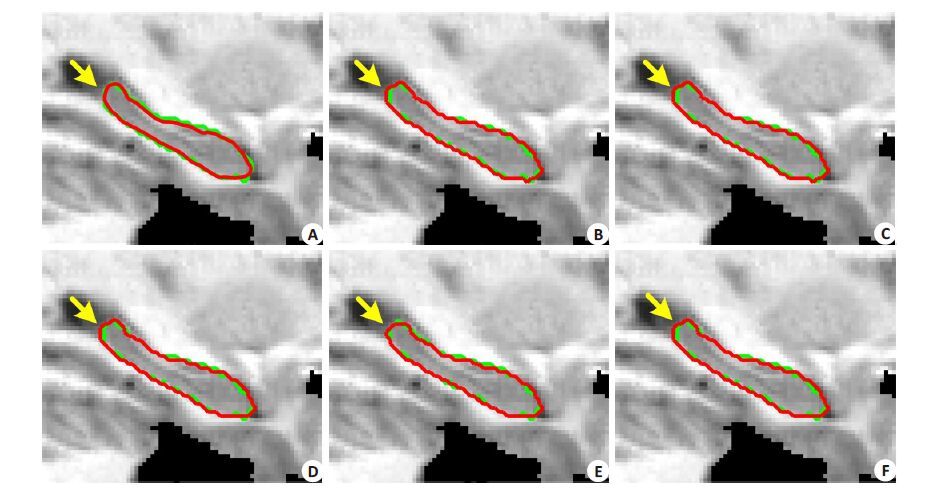
图 5A~F 分别为本文方法及STAPLE,Spatial STAPLE,Major Voting,Weight Voting,SIMPLE 对左、 右海马分割的对编号为1006 的数据的分割结果。绿色为专家手动分割结果,红色为不同方法的分割结果。从A 中可以看出本文方法与专家手动分割结果更加接近,特别在脑室附近(黄色箭头所示),其实方法都出现了错误的分割,而本文方法在通过引导滤波引入待分割图像的信息,成功的将演化曲线吸引到了真实的海马边界。
|
图 5 不同方法对编号为1006的数据的分割结果 Figure 5 Sagital view of the various segmetation results of MICCAI image. A-F: STAPLE,Spatial STAPLE,Major Voting,Weight Voting and SIMPLE. Automatic segmentation results are illustrated in red contour,the manually-delineated the hippocampus boundary shown by the green contour. |
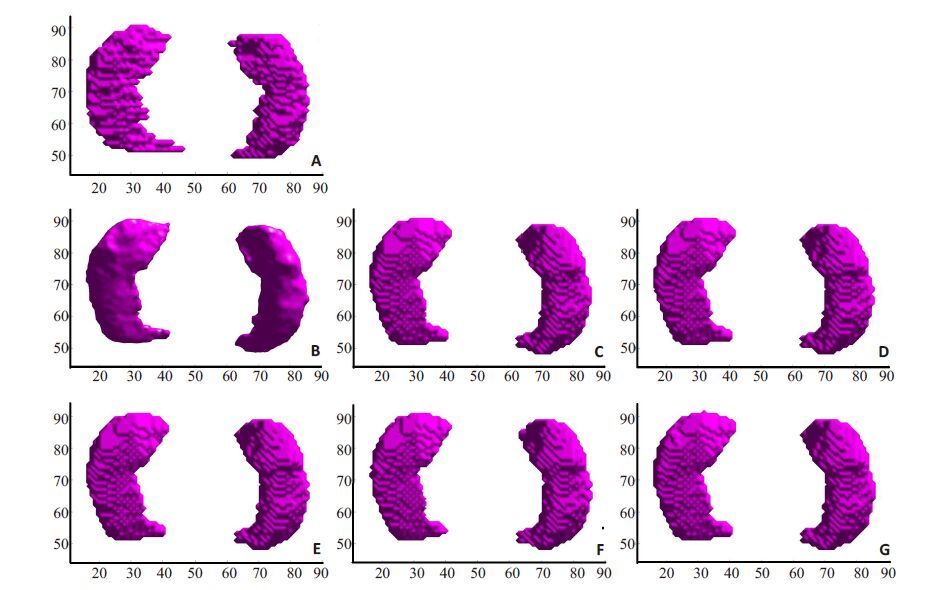
比较各方法三维重建的结果(如图 6),用其它方法所得到的结果存在明显的阶梯状效果,在边界出会出现一些毛刺或凹陷现象,而新方法利用引导滤波可以得到光滑、连续的边界,与脑部结构的真实情况更加接近,便于医生观察体积与形态的变化。
|
图 6 左、右海马分割后三维重建结果 Figure 6 Surface rendering of ground truth and segmentation results. Three-dimensional hippocampus segmentations obtained by six methods. A is the manual segmentation; B-G: the proposed method,STAPLE,Spatial STAPLE,Major Voting,Weight Voting,SIMPLE. |
大脑内部结构的位置、体积与形态变化与多种疾病息息相关,而要确定和分析这些变化首先需要对这些脑结构进行精确的分割。我们对目前脑内结构分割方法进行了详细分析,并针对这些方法存在的缺点和不足,提出了一种新的基于引导滤波的多图谱医学图像分割方法。新方法中包含两个重要部分:多图谱配准和引导滤波。概率图谱的准确性将直接影响到分割的精度,利用配准的局部相似性作为权重计算局部权重概率图谱,保证了概率图谱的准确性;利用引导滤波,引入待分割图像的结果信息,保证了信息的完整性,从而较正配准引起的误差,得到光滑、准确的分割结果。
| [1] | Alivisatos AP, Chun M, Church GM, et al. The brain activity map[J]. Science,2013, 339 (6125) : 1284-5. DOI: 10.1126/science.1236939. |
| [2] | Manikandan S, Ramar K, Willjuice Iruthayarajan M, et al. Multilevel thresholding for segmentation of medical brain images using real coded genetic algorithm[J]. Measurement,2014, 47 (47) : 558-68. |
| [3] | Lu XQ, Wu JS, Ren XY, et al. The study and application of the improved region growing algorithm for liver segmentation[J]. Optik,2014, 125 (9) : 2142-7. DOI: 10.1016/j.ijleo.2013.10.049. |
| [4] | Bai PR, Liu QY, Li L, et al. A novel region-based level set method initialized with mean shift clustering for automated medical image segmentation[J]. Comput Biol Med,2013, 43 (11) : 1827-32. DOI: 10.1016/j.compbiomed.2013.08.024. |
| [5] | Baillard C, Hellier P, Barillot C. Segmentation of brain 3D Mr images using level sets and dense registration[J]. Med Image Anal,2001, 5 (3) : 185-94. DOI: 10.1016/S1361-8415(01)00039-1. |
| [6] | Heckemann RA, Hajnal JV, Aljabar P, et al. Automatic anatomical brain MRI segmentation combining label propagation and decision fusion[J]. Neuroimage,2006, 33 (1) : 115-26. DOI: 10.1016/j.neuroimage.2006.05.061. |
| [7] | Klein S, Van Der Heide UA, Lips IM, et al. Automatic segmentation of the prostate in 3D Mr images by Atlas matching using localized mutual information[J]. Med Phys,2008, 35 (4) : 1407-17. DOI: 10.1118/1.2842076. |
| [8] | Isgum I, Staring M, Rutten A, et al. Multi-Atlas-Based segmentation with local decision Fusion-Application to cardiac and aortic segmentation in CT scans[J]. IEEE Trans Med Imaging,2009, 28 (7) : 1000-10. DOI: 10.1109/TMI.2008.2011480. |
| [9] | Jia HJ, Yap PT, Shen DG. Iterative multi-atlas-based multi-image segmentation with tree-based registration[J]. Neuroimage,2012, 59 (1, SI) : 422-30. DOI: 10.1016/j.neuroimage.2011.07.036. |
| [10] | Popescu V, Battaglini M, Hoogstrate WS, et al. Optimizing parameter choice for FSL-Brain Extraction Tool (BET) on 3D T1 images in multiple sclerosis[J]. Neuroimage,2012, 61 (4) : 1484-94. DOI: 10.1016/j.neuroimage.2012.03.074. |
| [11] | Tustison NJ, Avants BB, Cook PA, et al. N4ITK: improved N3 bias correction[J]. IEEE Trans Med Imaging,2010, 29 (6) : 1310-20. DOI: 10.1109/TMI.2010.2046908. |
| [12] | Avants BB, Tustison NJ, Song G, et al. A reproducible evaluation of ANTs similarity metric performance in brain image registration[J]. Neuroimage,2011, 54 (3) : 2033-44. DOI: 10.1016/j.neuroimage.2010.09.025. |
| [13] | He KM, Sun J, Tang XO. Guided image filtering[J]. IEEE Trans Pattern Anal Mach Intell,2013, 35 (6) : 1397-409. DOI: 10.1109/TPAMI.2012.213. |
| [14] | Warfield SK, Zou KH, Wells WM. Simultaneous truth and performance level estimation (STAPLE): An algorithm for the validation of image segmentation[J]. IEEE Trans Med Imaging,2004, 23 (7) : 903-21. DOI: 10.1109/TMI.2004.828354. |
| [15] | Commowick O, Akhondi-Asl A, Warfield SK. Estimating a reference standard segmentation with spatially varying performance parameters: local MAP STAPLE[J]. IEEE Trans Med Imaging,2012, 31 (8) : 1593-606. DOI: 10.1109/TMI.2012.2197406. |
| [16] | Aljabar P, Heckemann RA, Hammers A, et al. Multi-atlas based segmentation of brain images: Atlas selection and its effect on accuracy[J]. Neuroimage,2009, 46 (3) : 726-38. DOI: 10.1016/j.neuroimage.2009.02.018. |
| [17] | Sjöberg C, Ahnesjö A. Multi-atlas based segmentation using probabilistic label fusion with adaptive weighting of image similarity measures[J]. Comput Methods Programs Biomed,2013, 110 (3) : 308-19. DOI: 10.1016/j.cmpb.2012.12.006. |
| [18] | Langerak TR, Van Der Heide UA, Kotte A, et al. Label fusion in atlas-based segmentation using a selective and iterative method for performance level estimation(SIMPLE)[J]. IEEE Trans Med Imaging,2010, 29 (12) : 2000-8. DOI: 10.1109/TMI.2010.2057442. |
 2015, Vol. 35
2015, Vol. 35
