2. 南方医科大学南方医院 血管外科,广东 广州 510515 ;
3. 南方医科大学华瑞医院健康管理科,广东 广州 510630 ;
4. 佛山市顺德区桂洲医院超声科,广东 佛山 528305
2. Department of Vascular Surgery, Nanfang Hospital of Southern Medical University, Guangzhou 510515, China ;
3. Department of Health Management, Huarui Hospital of Southern Medical University, Guangzhou 510630, China ;
4. Department of Ultrasound, Gui Zhou hospital of Shunde District, Foshan 528305, China
在全球范围内,心血管疾病是危害人类健康的主要疾病。在致残致死的心血管疾病中,75%以上是由动脉粥样硬化(atherosclerosis, AS)引起,故AS是心脑血管疾病发生的重要病理基础[1]。目前普遍认为AS是一种慢性、进展性、炎症性疾病,而明确AS的发病机制是防治心脑血管病的根本措施。越来越多的研究表明,急性心脑血管事件的发生不仅取决于动脉管腔的狭窄程度,更重要的决定因素是动脉粥样硬化不稳定斑块的形成[2]。不稳定斑块形成的病理生理学机制主要包括斑块内炎症反应的增加、新生血管形成和细胞大量凋亡,而诱导这些病理生理过程改变的分子机制并不完全清楚[3]。
基因表达芯片技术可同时比较成千上万个基因的表达变化,全面筛选某一表型或某一疾病的所有相关基因,还可揭示不同基因表达变化之间的相互关系,从而为研究基因与基因之间的内在联系提供线索[4]。在本次研究中,我们通过基因表达谱分析获取不稳定斑块和稳定斑块间差异表达基因,并运用生物信息学方法分别在基因本体(gene ontology, GO)、通路和蛋白互作网络中分析不稳定斑块形成的分子机制,旨在寻找该疾病相关生物学过程、通路和风险模块。这些为深入研究不稳定斑块发生机制、筛选心血管事件早期预警指标和寻找不稳定斑块治疗靶点奠定一定的理论基础。
1 材料和方法 1.1 实验试剂与仪器TRIGene提取试剂,批号P118-01,购自北京康润诚业生物科技有限公司;PrimeScriptTM RT reagent Kit反转录试剂盒,批号RR037A,购自Takara;SYBR® Premix Ex Taq(Tli RNaseH Plus)-QPCR试剂盒,批号RR420A,购自Takara。PCR引物由生工生物工程(上海)股份有限公司提供。本研究所使用仪器主要有普通标准PCR仪(Biometra)和荧光定量PCR仪(LightCycler 480 System)等。
1.2 样本数据描述与处理本次研究使用的E-MTAB-2055数据集来源于ArrayExpress数据库(http://www.ebi.ac.uk/arrayexpress/),采用的芯片是A-MEXP-931-Illumina HumanRef-8 v2 Expression BeadChip。该芯片数据的样本包含24例稳定斑块组织和24例不稳定斑块组织,均来源于经颈动脉内膜剥脱术后病人的病变血管,包含血管内膜和中膜[5]。
下载E-MTAB-2055的CEL文件后,分别对探针水平的信号值进行质控、变异稳定性分析、背景矫正、数据归一化和基因注释,最终转化为基因水平的表达值[6]。稳定斑块和不稳定斑块组织间差异基因采取T检验和倍比法(fold-change, FC)获取,筛选阈值为Pvalue≤0.01且|logFC| >1。采用DAVID数据库[7](Database for Annotation, Visualization and Integrated Discovery, http://david.abcc.ncifcrf.gov/home.jsp)进行后续的GO功能注释及Pathway分析。通过string [8](版本号9.1)网站和Cytoscape [9](版本号2.8.2)实现蛋白互作网络构建和可视化。
1.3 组织标本RNA的提取及浓度、纯度的检测5对动脉粥样硬化斑块和斑块旁组织标本从超低温冰箱取出后,按照Trizol法操作步骤提取总RNA,用琼脂糖凝胶电泳判断RNA无降解,并用紫外分光光度法检测所提取RNA的浓度、纯度,RNA的A260/280吸光值比率在1.8~2.1范围内者方可进行后续实验。
1.4 组织标本mRNA表达水平分析反转录合成cDNA反应根据反转录试剂盒(PrimeScriptTM RT reagent Kit, Takara)说明书进行实验操作。实时荧光定量PCR引物见表 1。荧光定量PCR反应使用SYBR法Q-PCR试剂盒(SYBR® Premix Ex Taq, Takara)在荧光定量PCR(LightCycler 480 System)上操作,PCR反应条件:95 ℃预变性30 s,95 ℃变性10 s,60 ℃退火30 s共45个循环,60 ℃~95 ℃融解曲线分析。引物序列从引物数据库[10]下载并由生工生物工程(上海)股份有限公司合成。采用GAPDH作为内参基因,采用2-ΔΔCt方法[11]计算基因相对表达值。各个基因的表达值测量均重复3次。
| 表 1 实时荧光定量PCR引物 Table 1 Primers for Real Time PCR |
本实验结果均采用SPSS 19.0统计软件包进行处理,不稳定斑块与稳定斑块组织之间各个基因表达情况差异的比较采用Mann-whitney U检验统计分析。检验水准:α=0.05,P < 0.05有统计学意义。
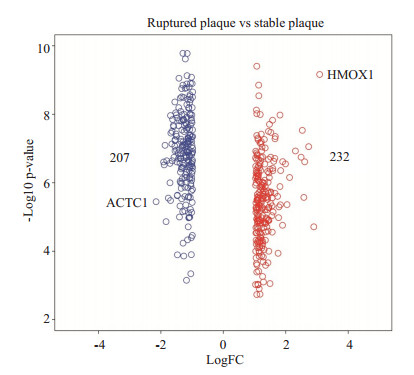
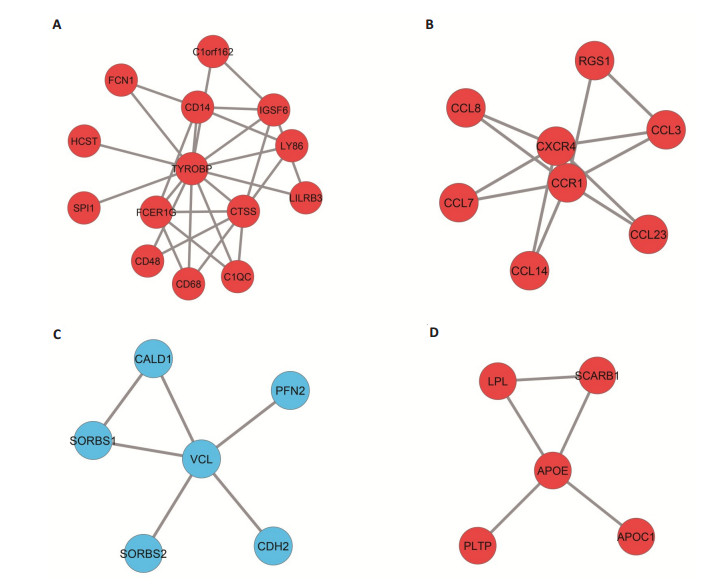
2 结果 2.1 基因芯片分析结果通过表达谱差异基因分析,本研究共获取了439个差异基因,其中上调基因232个,下调基因207个(图 1)。GO富集结果显示差异基因在免疫应答、免疫防御、炎症应答、细胞因子富集显著性最高,这些生物过程可能参与不稳定斑块形成。通路分析结果显示9个通路满足富集阈值要求(表 2)。在蛋白互作网络层面,本研究共获取了4个与不稳定斑块相关的风险模块(图 2),所有模块中互作基因差异表达方向均一致,同时TYROBP、CXCR4、CCR1、VCL和APOE分别是4个模块中的枢纽基因。此外,TYROBP、CXCR4和VCL在蛋白互作网络和疾病模块中均位于核心位置,可能参与不稳定斑块形成,为本次研究的重点对象。
|
图 1 不稳定斑块和稳定斑块间差异表达基因火山图 Figure 1 Volcano plots for all differentially expressed genes (DEGs) in unstable plaques. The red and blue dots indicate that up-and down-regulated DEGs, respectively, which were selected with the cutoff value; HMOX1 is the most up-regulated gene, and ACTC1 the most down-regulated gene. |
| 表 2 差异表达基因富集KEGG通路结果 Table 2 Results of KEGG pathway enrichment analysis for DEGs |
|
图 2 蛋白互作网络中获取的4个疾病相关模块 Figure 2 Four disease-related modules obtained from the PPI network Red color indicates up-regulated genes annotated in the PPI network; Blue color indicates the down-regulated genes in the PPI network. |
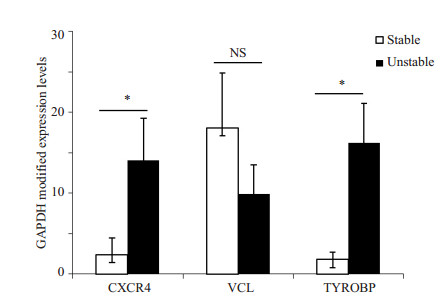
通过荧光定量PCR方法,在5对颈动脉粥样硬化不稳定斑块与稳定斑块标本中分析CXCR4、VCL和TYROBP基因的表达改变。结果显示,2个差异表达基因与分析结果完全一致。其中,CXCR4和TYROBP在不稳定斑块组织中表达上调。VCL在不稳定斑块和稳定斑块组织间差异无统计学意义(图 3)。
|
图 3 3个差异表达基因的荧光定量PCR结果 Figure 3 Results of real-time PCR of 3 DEGs between stable and unstable plaques (2-ΔCt, Mean±SD). *P < 0.05, NS: P > 0.05. |
基因表达芯片技术可同时比较成千上万个基因的表达变化,而生物信息学的快速发展,使急剧增加的基因芯片表达谱数据得到了更好的阐释,也使得更多蕴藏在数据中的生物信息能够被发现。本次研究利用人颈动脉粥样硬化基因芯片数据进行初步分析,共获取了439个DEGs,其中上调基因232个,下调基因207个,它们共同构成了不稳定斑块差异基因表达谱,与不稳定斑块的形成密切相关。GO和通路富集分析结果显示主要与炎症、免疫应答、溶酶体通路、细胞因子信号通路和PPAR信号通路等有关。蛋白互作网络和模块分析结果显示CXCR4、VCL和TYROBP在生物网络和疾病风险模块均处于核心位置,在促进不稳定斑块形成过程中发挥重要作用。
CXCR4位于染色体2q21,属于G蛋白耦联趋化因子受体,可以与基质细胞衍生因子1(stromal-derived factor 1, SDF-1)和巨噬细胞游走抑制因子(macrophage migration inhibitory factor, MIF)发生特异性结合,参与趋化、细胞阻滞、血管生成和细胞生存等过程,促进HIV病毒转染宿主细胞、自身免疫性疾病发生和肿瘤细胞增殖、迁移及侵袭[12]。Melchionna等[13]通过不同强度剪切力处理人脐静脉内皮细胞发现低流速剪切力可以引起血管内皮损伤,损伤的血管内皮细胞较正常血管内皮细胞高表达CXCR4蛋白从而诱导内皮细胞凋亡,研究者推测CXCR4可能参与早期动脉粥样硬化斑块形成,而下调CXCR4对血管起保护作用。国内研究人员通过使用冠心康干预小鼠主动脉粥样硬化进程发现,伴随主动脉粥样硬化程度减轻,冠心康组小鼠主动脉组织中SDF-1和CXCR4表达水平明显低于未干预组,研究者推测SDF-1/CXCR4轴可能是药物干预动脉粥样硬化进程的作用靶点[14]。Bernhagen等[15]研究指出,对ApoE (-/-)小鼠使用MIF单克隆抗体,其通过抑制MIF/CXCR4轴活性可引起主动脉斑块体积减少,促使晚期斑块转归为稳定斑块,研究者推测MIF/CXCR4轴激活可能参与不稳定斑块形成。另有研究表明,ApoE (-/-)小鼠上使用共轭亚油酸抑制CXCR4表达可以诱导早期动脉粥样斑块消退,降低斑块局部白细胞粘附能力和数量[16]。此外,通路富集分析结果显示CXCR4主要参与细胞因子信号通路、细胞因子-细胞因子受体互作和白细胞跨膜迁移。本次研究从基因角度验证了CXCR4基因在不稳定斑块中表达上调,该基因异常表达可能与不稳定斑块形成有关,其相关机制有待进一步研究。VCL,又称为黏着斑蛋白,位于染色体10q22.2,是一种细胞骨架蛋白,参与细胞-细胞或细胞-基质之间连接。Constantinescu等[17]研究发现,高血脂血清刺激鼠主动脉内皮细胞会引起VCL表达和分布发生异常改变,研究者推断VCL的异常变化可能促进动脉粥样硬化发生发展。另一项研究通过激光纤维切割联合二维差异凝胶电泳技术分析冠状动脉粥样硬化斑块和早期病变血管中膜组织中差异蛋白,该研究发现VCL蛋白在斑块组织中表达下调,而后续免疫组化实验也证实了VCL表达水平的降低。研究者推断,在动脉粥样硬化进程中,VCL的表达异常促进动脉粥样硬化进展[18]。Rocchiccioli等[19]运用高效液相色谱法和质谱技术分析14对颈动脉斑块组织和斑块旁组织发现,与斑块旁组织相比,斑块组织低表达VCL蛋白,随后免疫组化和蛋白印记也证实了VCL在斑块组织中表达水平的下调,并推测VCL下调可能参与斑块发生发展。而VCL富集到的通路主要是白细胞跨膜迁移和肌动蛋白细胞骨架调节。本次研究中,VCL表达呈现下调趋势,但无统计学差异,可能与样本数量有限有一定关系,需要进一步扩大样本量进行分析验证。TYROBP,别名DAP12,位于染色体19q13.1,编码一种跨膜信号分子多肽,其编码的蛋白主要参与骨塑形、大脑髓鞘形成、信号传导和炎症反应[20]。本研究显示TYROBP在不稳定斑块中差异上调,可能参与不稳定斑块形成,其相关机制有待进一步研究。值得一提的是,TYROBP基因在动脉硬化斑块的表达情况尚未见报道,通过我们的分析验证,TYROBP基因可能是不稳定斑块发生过程中新的诊断或预后相关基因,其功能将是我们下一步研究的重点。
综上可知,研究不稳定斑块发生发展过程中基因表达谱的改变,对全面、系统、综合认识不稳定斑块形成机制具有重要意义,可以为动脉粥样硬化发病机制研究提供新的思路。本研究初步筛选并验证了CXCR4和TYROBP基因与不稳定斑块相关,初步描述了不稳定斑块形成有关的病理过程及信号通路。CXCR4基因可能是不稳定斑块的特殊致病基因,TYROBP基因可能是新的不稳定斑块形成相关基因,其机制有待深入研究。
| [1] | Lewis SJ. Prevention and treatment of atherosclerosis: a practitioner's guide for 2008[J]. Am J Med,2009, 122 (1 Suppl) : S38-50. |
| [2] | 李公信, 王露朝, 刘芃, 等. 超小超顺磁性氧化铁增强MRA联合炎症因子检测兔动脉粥样硬化易损斑块[J]. 南方医科大学学报,2014, 34 (9) : 1324-8. |
| [3] | Hennerici MG. The unstable plaque[J]. Cerebrovasc Dis,2004, 17 (3) : 17-22. DOI: 10.1159/000075300. |
| [4] | Marshall A, Hodgson J. DNA chips: an array of possibilities[J]. Nat Biotechnol,1998, 16 (1) : 27-31. DOI: 10.1038/nbt0198-27. |
| [5] | Eijgelaar WJ, Manca M, Herías MV, et al. Multi-platform approach for plaque-derived biomarkers for the vulnerable plaque[J]. Challenges and Promises,2014, 9 (13) : 43-9. |
| [6] | 邓祯祥, 王文辉, 李金明. 整合卵巢癌染色体拷贝数变异与差异表达基因的生物信息学分析[J]. 南方医科大学学报,2014 (6) : 813-7. |
| [7] | Dennis GJ, Sherman BT, Hosack DA, et al. David: database for annotation, visualization, and integrated discovery[J]. Genome Biol,2003, 4 (9) : P3. |
| [8] | Franceschini A, Szklarczyk D, Frankild S, et al. STRING v9.1: protein-protein interaction networks, with increased coverage and integration[J]. Nucleic Acids Res,2013, 41 (Database issue) : D808-15. |
| [9] | Shannon P, Markiel A, Ozier O, et al. Cytoscape: a software environment for integrated models of biomolecular interaction networks[J]. Genome Res,2003, 13 (11) : 2498-504. DOI: 10.1101/gr.1239303. |
| [10] | Wang X, Seed B. A PCR primer bank for quantitative gene expression analysis[J]. Nucleic Acids Res,2003, 31 (24) : e154. DOI: 10.1093/nar/gng154. |
| [11] | Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C (T)) Method[J]. Methods,2001, 25 (4) : 402-8. DOI: 10.1006/meth.2001.1262. |
| [12] | Döring Y, Pawig L, Weber C, et al. The CXCL12/CXCR4 chemokine ligand/receptor axis in cardiovascular disease[J]. Front Physiol,2014, 5 (2) : 212. |
| [13] | Melchionna R, Porcelli D, Mangoni A, et al. Laminar shear stress inhibits CXCR4 expression on endothelial cells: functional consequences for atherogenesis[J]. FASEB J,2005, 19 (6) : 629-31. |
| [14] | 秦合伟, 刘萍. 冠心康抗动脉粥样硬化作用机制与SDF-1/CXCR4生物轴失衡相关性的研究[J]. 中华中医药杂志,2014, 33 (5) : 1558-62. |
| [15] | Bernhagen J, Krohn R, Lue H, et al. MIF is a noncognate ligand of CXC chemokine receptors in inflammatory and atherogenic cell recruitment[J]. Nat Med,2007, 13 (5) : 587-96. DOI: 10.1038/nm1567. |
| [16] | de Gaetano M, Dempsey E, Marcone S, et al. Conjugated linoleic acid targets β2 integrin expression to suppress monocyte adhesion[J]. J Immunol,2013, 191 (8) : 4326-36. DOI: 10.4049/jimmunol.1300990. |
| [17] | Constantinescu E, Alexandru D, Raicu M, et al. Exposure to hypercholesterolemic serum modifies the expression of cytoskeletal proteins in cultured endothelia[J]. J Submicrosc Cytol Pathol,1997, 29 (4) : 543-51. |
| [18] | de la Cuesta F, Zubiri I, Maroto AS, et al. Deregulation of smooth muscle cell cytoskeleton within the human atherosclerotic coronary media layer[J]. J Proteomics,2013, 82 (3) : 155-65. |
| [19] | Rocchiccioli S, Pelosi G, Rosini S, et al. Secreted proteins from carotid endarterectomy: an untargeted approach to disclose molecular clues of plaque progression[J]. J Transl Med,2013, 11 (3) : 260. |
| [20] | Takaki R, Watson SR, Lanier LL. DAP12: an adapter protein with dual functionality[J]. Immunol Rev,2006, 214 (6) : 118-29. |
 2015, Vol. 35
2015, Vol. 35
