2. 深圳市龙岗中心医院病理科,广东 深圳 518116 ;
3. 郑州市儿童医 院病理科,河南 郑州 450053 ;
4. 南方医科大学基础医学院病理学系,广东 广州 510515
2. Department of Pathology, Longgang Central Hospital, Shenzhen 518116, China ;
3. Department of Pathology, Zhengzhou Children's Hospital, Zhengzhou 450053, China ;
4. Department of Pathology, School of Basic Medical Science, Southern Medical University, Guangzhou 510515, China
结直肠癌(colorectal carcinnoma, CRC)是我国最常见的恶性肿瘤之一, 近年来其发病率在我国有所增加。目前研究表明, CRC的发生、发展是一个多因素、多步骤的复杂生物学行为过程。亦有研究显示, miRNA (microRNA)与CRC存在紧密的关系[1-4]。miR-200家族(包括miR-200a, miR-200b, miR-200c和miR-141)已经被证实在多种肿瘤中通过调控上皮-间质转化(epithelial-mesenchymal transition, EMT)影响肿瘤的进展。然而, miR-200a在CRC组织中的表达报道不一致, 有报道称其在CRC组织中表达减低[5], 但其他研究则显示了相反的结果[6-8]。虽然miR-200家族表达的减弱或缺失被认为是EMT的基本特征[9], 且有研究在CRC细胞系HCT116中诱导miR-200家族的表达后出现EMT受到抑制[10], 但miR-200a在具有高转移能力的CRC细胞系Lovo中的作用未见研究报道。本研究应用实时荧光定量PCR筛查人CRC的6种细胞系中miR-200a表达差异情况, 在高转移细胞系Lovo中瞬时转染miR-200a mimics, 检测高表达miR-200a后Lovo细胞增殖、迁移、细胞同质黏附能力和凋亡等情况的变化, 为进一步研究miR-200a在CRC中的作用打下基础。
1 材料和方法 1.1 细胞人类CRC细胞系HCT116、HT29, LS174T、SW480、SW620和Lovo由南方医科大学基础医学院病理学系购自中国科学院上海生命科学研究院并在实验室液氮中保存, 使用时复苏, 置于37℃、5% CO2培养箱内, 用含10%胎牛血清的RPMI 1640完全培养基进行培养, 取生长状态良好的细胞用于实验。
1.2 试剂RPMI 1640培养基购自美国Hyclone, 胎牛血清及转染所用阳离子脂质体LipofectamineTM 2000及Opti-MEM购自美国Invitrogen, 总RNA提取试剂盒-RNAisoTM PLUS购自日本TaKaRa; miRNA qRTPCR检测试剂盒购自美国GeneCopoeiaTM; miR-200a的引物及内参U6的引物由杰特伟公司设计合成; miR-200a的类似物(miR-200a mimics, sense序列: 5'-UAACACUGUCUGGUAACGAUGU-3';anti-sense序列:5'-AUCGUUACCAGACAGUGUUAUU-3)'及对应的阴性对照(miR-200a negative control, sense序列: 5'-UUCUCCGAACGUGUCACGUTT-3';anti-sense序列:5'-ACGUGACACGUUCGGAGAATT-3)'由上海吉玛公司设计并合成; 细胞原位凋亡检测试剂盒(TUNEL assay)购自瑞士Roche Diagnostics; Cell Counting Kit-8试剂盒购自碧云天生物技术, Transwell小室购自美国Corning。
1.3 检测miR-200a在6种CRC细胞中的表达取对数生长期的细胞, 按照试剂盒说明提取总RNA, 紫外分光光度计法测定RNA浓度, -20℃保存备用。检测时取2 μg总RNA逆转为cDNA, 以合成的cDNA为模板链进行PCR扩增, 同时扩增U6为内参, 反应体系置入ABI 7500荧光定量PCR仪中进行qRT-PCR, 以Folds=2-ΔΔCt表示目的miRNA在实验组与对照组中表达的倍比关系, 采用如下计算公式:ΔΔCt=[Ct (miRNA))-Ct (U6)]实验组-[Ct (miRNA)-Ct (U6)]对照组, 重复3次实验。HCT116细胞为对照组, 其余细胞与之比较, 检测不同细胞系中miR-200a的表达。
1.4 Lovo细胞瞬时转染取生长状态良好的Lovo细胞, 1.5×105/孔的细胞数铺六孔板, 铺板后20 h, 通过瞬时转染方法向Lovo细胞转染miR-200a mimics (miR-200a组)和negative control (NC组), 同时以加入等量阳离子脂质体LipofectamineTM 2000的细胞为blank组, 转染方法和用量严格按照说明书进行, qRT-PCR检测转染后miR-200a的表达。
1.5 增殖实验mimics组、NC组和blank组的细胞转染48 h后, 每孔加10 μl的CCK-8, 置于37℃, 50 ml/L CO2中培养箱中避光孵育2 h, 后置于酶联免疫检测仪450 nm处测定吸光度, 计算6个复孔平均值。
1.6 Transwell迁移实验细胞转染约18 h后, 消化制备成细胞悬液, 调整细胞浓度为2×104/ml, 每个transwell内室加入200 μl细胞悬液, 下室加入500 μl含10%胎牛血清的完全培养基, 于细胞培养箱中30 h后取出, 甲醇固定, 苏木素染膜, 显微镜下观察膜的下室面, 随机选取10个400倍视野计数穿过膜的细胞数。
1.7 TUNEL法检测细胞凋亡将3组细胞在转染48 h后离心制作成细胞蜡块, 细胞蜡块3 μm厚切片, 操作步骤参照细胞原位凋亡检测试剂盒说明书进行。切片染色完成后于显微镜下观察, 随机选取5个高倍视野计数凋亡细胞并拍照。计算各组细胞凋亡指数, 凋亡指数=凋亡细胞数/细胞总数× 100%。
1.8 同质性肿瘤细胞黏附实验NC组和miR-200a组细胞转染24 h后, 消化制成1×105/ml的单细胞悬液, 相对应地吸取100 μl悬液到已经完全长满96孔板的各组细胞, 每组重复5个复孔, 置于培养箱培养60 min, 轻轻晃动96孔板, 将未黏附的细胞吸出, PBS轻轻洗两次, 尽量将未黏附的细胞完全吸出。光镜下计数, 以加入和吸出肿瘤细胞数量之差计算肿瘤细胞粘附数值, 取平均值。
1.9 统计学分析采用SPSS13.0统计软件, miR-200a的表达、CCK-8法增殖实验、划痕实验和TUNEL凋亡实验的数据均进行方差齐性检验和单向方差分析, 同质性肿瘤细胞黏附实验比较采用两独立样本t检验; 双侧检验P < 0.05有统计学意义。
2 结果 2.1 miR-200a在人CRC细胞系中的表达以HCT116细胞为对照, qRT-PCR结果显示miR-200a在CRC细胞中的表达量分别为HT29(0.275±0.013)倍、LS174T (0.108±0.009)倍、SW480(0.010±0.001)倍、SW620(0.005±0.00013)倍、Lovo (0.0004±0.00007)倍(P < 0.05)。结果表明miR-200a在高转移能力的Lovo中的表达水平均最低, 成瘤但不转移的细胞系HCT116表达最高。
2.2 瞬时转染对Lovo细胞的miR-200a表达影响转染48 h, 以NC为对照, qRT-PCR检测结果显示, miR-200a组的细胞miR-200a表达升高了2053.500±55.506倍(P < 0.05), 与blank组的表达量(0.976±0.650)倍无显著差异(P > 0.05), 表明瞬时转染成功。
2.3 miR-200a对Lovo细胞增殖能力的影响在Lovo过表达miR-200a后, miR-200a组的OD值是1.286±0.070, 比NC组和blank组的1.697±0.071和1.743±0.034均显著减低(F=101.928, P=0.000)。而后两组增殖能力无显著差异(P=0.214, 图 1)。说明过表达miR-200a能显著抑制Lovo细胞增殖。
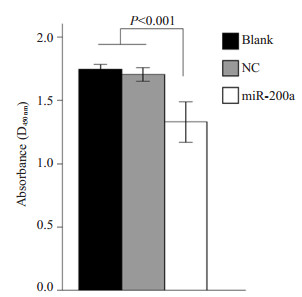
|
图 1 miR-200a对Lovo细胞增殖的影响 Figure 1 Effect of miR-200a overexpression on LoVo cell proliferation. |
Transwell迁移实验结果显示miR-200a组的细胞穿孔数是47.673±9.155, 显著低于NC组和blank组的89.602±7.890和85.671±7.480(F=119.464, P=0.000)。NC组和blank组穿孔数无显著差异(P=0.196, 图 2)。
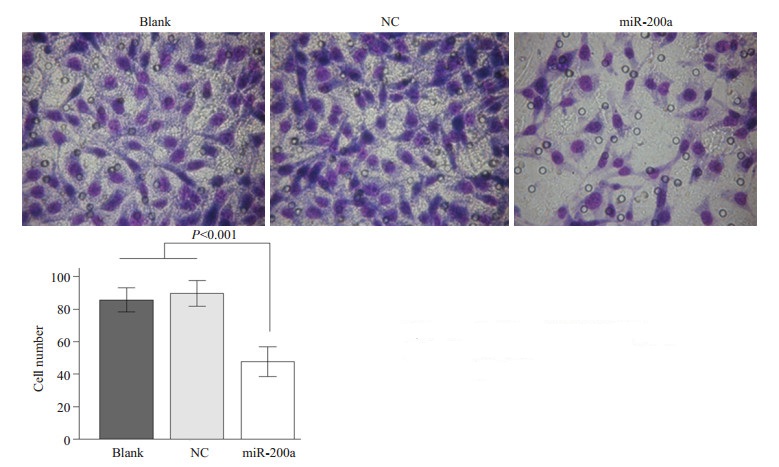
|
图 2 miR-200a对Lovo细胞迁移的影响 Figure 2 Effect of miR-200a overexpression on LoVo cell migration. |
TUNEL染色将凋亡细胞的核染成棕褐色, 未发生凋亡的细胞核经过苏木素染成蓝色, 检测结果显示miR-200a组的细胞凋亡率是36.51%, 显著低于NC组和blank组的12.13%和9.32%(F=119.464, P=0.000), NC组和blank组凋亡率无显著差异(P=0.424, 图 3)。
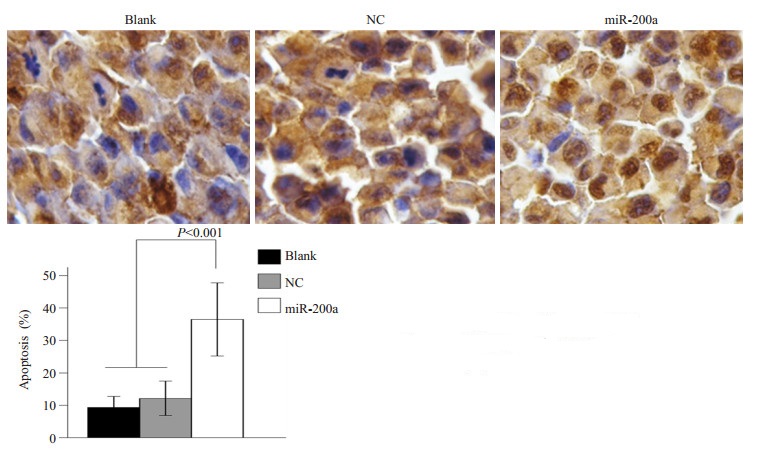
|
图 3 miR-200a对Lovo细胞凋亡的影响 Figure 3 Effect of miR-200a overexpression on LoVo cell apoptosis. |
在Lovo过表达miR-200a后, 肿瘤细胞同质性黏附实验检测转染48 h各组细胞-细胞之间黏附变化情况, 显示miR-200a组细胞的同质性黏附细胞数是1238.75±62.612, NC组的是1043.25±67.401, 统计发现miR-200a组的细胞同质性粘附能力显著增强(t=-4.250, P=0.005)。
3 讨论近年来, miR-200家族在调节肿瘤进展方面受到广泛关注, 此家族被认为能通过抑制EMT而影响肿瘤进展, 它们在某些肿瘤中表达的缺失或下调能促进肿瘤侵袭和转移。miR-200在大部分肿瘤中表达下调, 如在脑膜瘤[11]、鼻咽癌[12]、甲状腺未分化癌[13]、肝癌[14-15]、EB病毒相关胃癌[16]、肾透明细胞癌[17]、卵巢癌[18]、浸润性宫颈鳞状细胞癌[19]等多种肿瘤中表达明显低于相应的正常组织。但在一些肿瘤中则表达上调, 如在胰腺癌[20], 黑素瘤[21], 食管腺癌[22]等肿瘤中表达明显高于相应的正常组织, 也有报道卵巢癌中其表达上调[23-25]。作为miR-200家族的一员, miR-200a在CRC组织中的表达报道不一致, 有报道称其在CRC组织中表达减低[5], 另一些研究却显示相反结果[6-8]。本研究检测6种结直肠癌细胞系LoVo, SW620, SW480, LS174T, HT29, HCT116中miR-200a表达, 发现miR-200a在HCT116表达最高, LoVo表达最低, 在这6种细胞中, LoVo和SW620属于高转移潜能细胞, HT29和HCT116属于惰性转移潜能细胞, 而SW480和LS174T转移潜能居于两者之间, 本研究结果提示miR-200a表达下调与CRC侵袭、转移潜能密切有关。
为了进一步研究miR-200a在CRC中的作用, 本研究选择表达miR-200a最低的LoVo细胞进行过表达实验, 然后进行细胞生物学行为改变的检测包括细胞增殖、迁移、同质性黏附能力及凋亡的变化。结果显示在Lovo细胞过表达miR-200a后, 细胞增殖和迁移能力明显减弱, 同质性黏附能力增强, 细胞凋亡指数增加。细胞增殖和细胞凋亡与肿瘤生长速度和转移密切相关[26]。同质性黏附是指癌细胞与癌细胞间的黏附, 黏附能力越强, 则癌细胞间细胞连接越紧密, 不利于癌细胞迁移、游走, 从而阻碍癌细胞侵袭转移的发生。迁移能力是肿瘤细胞离开原发灶和向远处侵袭、转移的前提[26]。本研究结果进一步证实了miR-200a于人结直肠癌侵袭、转移密切相关的可能。有研究在CRC细胞系HCT116中诱导miR-200家族的表达后出现EMT受到抑制[10], 而在其他肿瘤中, miR-200家族被证实能调控EMT和细胞迁移, 在Boyden transwell小室中证实miR-200a, -200c和-141促进E-cadherin介导的黏附形成和抑制细胞迁移[27-30]; 并且能影响细胞生长、凋亡[31]和黏附[32]。据此并结合本研究结果, 推测miR-200a通过调控EMT而影响结直肠癌的生物学行为, 但确切分子机制有待进一步研究。
综上所述, 本研究发现miR-200a在人结直肠癌细胞系中表达失常, 在转移潜能最高的Lovo细胞表达最低, 并成功通过瞬时转染方法在Lovo细胞高表达miR-200a, 初步确定miR-200a能抑制Lovo细胞的增殖和迁移能力, 增强细胞间黏附力和促进细胞发生凋亡, 为明确miR-200a在结直肠癌发展过程中的基因调控和相应的作用机制奠定了研究基础, 为结直肠癌的预防和靶向性治疗提供了初步实验依据。
| [1] | Liu L, Chen L, Xu Y, et al. microRNA-195 promotes apoptosis and suppresses tumorigenicity of human colorectal Cancer cells[J]. Biochem Biophys Res Commun,2010, 400 (2) : 236-40. DOI: 10.1016/j.bbrc.2010.08.046. |
| [2] | Yamakuchi M, Ferlito M, Lowenstein CJ. miR-34a repression of SIRT1 regulates apoptosis[J]. Proc Natl Acad Sci U S A,2008, 105 (36) : 13421-6. DOI: 10.1073/pnas.0801613105. |
| [3] | Schimanski CC, Frerichs K, Rahman FA, et al. High miR-196a levels promote the oncogenic phenotype of colorectal Cancer cells[J]. World J Gastroenterol,2009, 15 (17) : 2089-96. DOI: 10.3748/wjg.15.2089. |
| [4] | Hu MH, Xia MG, Chen XB, et al. MicroRNA-141 regulates Smad interacting protein 1 (SIP1) and inhibits migration and invasion of colorectal Cancer cells[J]. Dig Dis Sci,2010, 55 (8) : 2365-72. DOI: 10.1007/s10620-009-1008-9. |
| [5] | Cummins JM, He YP, Leary RJ, et al. The colorectal microRNAome[J]. Proc Natl Acad Sci U S A,2006, 103 (10) : 3687-92. DOI: 10.1073/pnas.0511155103. |
| [6] | Yang L, Belaguli N, Berger DH. MicroRNA and colorectal cancer[J]. World J Surg,2009, 33 (4) : 638-46. DOI: 10.1007/s00268-008-9865-5. |
| [7] | Xi Y, Formentini A, Chien M, et al. Prognostic values of microRNAs in colorectal Cancer[J]. Biomark Insights,2006, 2 : 113-21. |
| [8] | Monzo M, Navarro A, Bandres E, et al. Overlapping expression of microRNAs in human embryonic colon and colorectal Cancer[J]. Cell Res,2008, 18 (8) : 823-33. DOI: 10.1038/cr.2008.81. |
| [9] | Zhang H, Li Y, Lai M. The microRNA network and tumor metastasis[J]. Oncogene,2010, 29 (7) : 937-48. DOI: 10.1038/onc.2009.406. |
| [10] | Park SM, Gaur AB, Lengyel E, et al. The miR-200 family determines the epithelial phenotype of cancer cells by targeting the E-cadherin repressors ZEB1 and ZEB2[J]. Genes Dev,2008, 22 (7) : 894-907. DOI: 10.1101/gad.1640608. |
| [11] | Saydam O, Shen YP, Wuerdinger T, et al. DownregulatedMicroRNA-200a in meningiomas promotes tumor growth by reducing E-Cadherin and activating the Wnt/beta-Catenin signaling pathway[J]. Mol Cell Biol,2009, 29 (21) : 5923-40. DOI: 10.1128/MCB.00332-09. |
| [12] | Xia H, Ng SS, Jiang S, et al. miR-200a-mediated downregulation of ZEB2 and CTNNB1 differentially inhibits nasopharyngeal carcinoma cell growth, migration and invasion[J]. Biochem Biophys Res Commun,2010, 391 (1) : 535-41. DOI: 10.1016/j.bbrc.2009.11.093. |
| [13] | Braun J, Hoang-Vu C, Dralle H, et al. Downregulation of microRNAs directs the EMT and invasive potential of anaplastic thyroid carcinomas[J]. Oncogene,2010, 29 (29) : 4237-44. DOI: 10.1038/onc.2010.169. |
| [14] | Murakami Y, Yasuda T, Saigo K, et al. Comprehensive analysis of microRNA expression patterns in hepatocellular carcinoma and non-tumorous tissues[J]. Oncogene,2006, 25 (17) : 2537-45. DOI: 10.1038/sj.onc.1209283. |
| [15] | Huang XH, Wang Q, Chen JS, et al. Bead-based microarray analysis of microRNA expression in hepatocellular carcinoma: miR-338 is downregulated[J]. Hepatol Res,2009, 39 (8) : 786-94. DOI: 10.1111/hep.2009.39.issue-8. |
| [16] | Shinozaki A, Sakatani T, Ushiku T, et al. Downregulation of MicroRNA-200 in EBV-Associated gastric carcinoma[J]. Cancer Res,2010, 70 (11) : 4719-27. DOI: 10.1158/0008-5472.CAN-09-4620. |
| [17] | Liu HQ, Brannon AR, Reddy AR, et al. Identifying mRNA targets of microRNA dysregulated in cancer: with application to clear cell renal cell carcinoma[J]. BMC Syst Biol,2010, 4 : 51. DOI: 10.1186/1752-0509-4-51. |
| [18] | Hu XX, Macdonald DM, Huettner PC, et al. A miR-200 microRNA cluster as prognostic marker in advanced ovarian cancer[J]. Gynecol Oncol,2009, 114 (3) : 457-64. DOI: 10.1016/j.ygyno.2009.05.022. |
| [19] | Lee JW, Choi CH, Choi JJ, et al. Altered MicroRNA expression in cervical carcinomas[J]. Clin Cancer Res,2008, 14 (9) : 2535-42. DOI: 10.1158/1078-0432.CCR-07-1231. |
| [20] | Li A, Omura N, Hong SM, et al. Pancreatic cancers epigenetically silence SIP1 and hypomethylate and overexpress miR-200a/200b in association with elevated circulating miR-200a and miR-200b levels[J]. Cancer Res,2010, 70 (13) : 5226-37. DOI: 10.1158/0008-5472.CAN-09-4227. |
| [21] | Elson-Schwab I, Lorentzen A, Marshall CJ. MicroRNA-200 family members differentially regulate morphological plasticity and mode of melanoma cell invasion[J]. PLoS One,2010, 5 (10) : 125. |
| [22] | Hu YX, Correa AM, Hoque A, et al. Prognostic significance of differentially expressed miRNAs in esophageal cancer[J]. Int J Cancer,2011, 128 (1) : 132-43. DOI: 10.1002/ijc.v128:1. |
| [23] | Bendoraite A, Knouf EC, Garg KS, et al. Regulation of miR-200 family microRNAs and ZEB transcription factors in ovarian Cancer: Evidence supporting a mesothelial-to-epithelial transition[J]. Gynecol Oncol,2010, 116 (1) : 117-25. DOI: 10.1016/j.ygyno.2009.08.009. |
| [24] | Iorio MV, Visone R, Di Leva G, et al. MicroRNA signatures in human ovarian cancer[J]. Cancer Res,2007, 67 (18) : 8699-707. DOI: 10.1158/0008-5472.CAN-07-1936. |
| [25] | Nam EJ, Yoon H, Kim SW, et al. MicroRNA expression profiles in serous ovarian carcinoma[J]. Clin Cancer Res,2008, 14 (9) : 2690-5. DOI: 10.1158/1078-0432.CCR-07-1731. |
| [26] | Hanahan D, Weinberg RA. Hallmarks of cancer: the next Generation[J]. Cell,2011, 144 (5) : 646-74. DOI: 10.1016/j.cell.2011.02.013. |
| [27] | Burk U, Schubert J, Wellner U, et al. A reciprocal repression between ZEB1 and members of the miR-200 family promotes EMT and invasion in cancer cells[J]. EMBO Rep,2008, 9 (6) : 582-9. DOI: 10.1038/embor.2008.74. |
| [28] | Gregory PA, Bert AG, Paterson EL, et al. The mir-200 family and mir-205 regulate epithelial to mesenchymal transition by targeting ZEB1 and SIP1[J]. Nat Cell Biol,2008, 10 (5) : 593-601. DOI: 10.1038/ncb1722. |
| [29] | Korpal M, Lee ES, Hu GH, et al. The miR-200 family inhibits epithelial-mesenchymal transition and cancer cell migration by direct targeting of E-cadherin transcriptional repressors ZEB1 and ZEB2[J]. J Biol Chem,2008, 283 (22) : 14910-4. DOI: 10.1074/jbc.C800074200. |
| [30] | Park SM, Gaur AB, Lengyel E, et al. The miR-200 family determines the epithelial phenotype of cancer cells by targeting the E-cadherin repressors ZEB1 and ZEB2[J]. Genes Dev,2008, 22 (7) : 894-907. DOI: 10.1101/gad.1640608. |
| [31] | Hyun S, Lee JH, Jin H, et al. Conserved MicroRNA miR-8/miR-200 and its target USH/FOG2 control growth by regulating PI3K[J]. Cell,2009, 139 (6) : 1096-108. DOI: 10.1016/j.cell.2009.11.020. |
| [32] | Kim YK, Yeo J, Ha M, et al. Cell adhesion-dependent control of microRNA decay[J]. Mol Cell,2011, 43 (6) : 1005-14. DOI: 10.1016/j.molcel.2011.07.031. |
 2015, Vol. 35
2015, Vol. 35
