2. 慢性疾病免疫学基础与临床安徽省重点实验室,安徽 蚌埠 233004
2. Anhui Province Key Laboratory of Immunology in Chronic Diseases, Bengbu 233004, China
原发性干燥综合征(pSS)是一种病因不明的全身性自身免疫性疾病,其特征是外分泌腺的局灶性淋巴细胞浸润,导致腺体功能障碍而出现干眼和口干,此外pSS患者还会出现关节炎,肺部受累,肾脏疾病,血管炎和神经系统病变等腺外表现[1]。在干燥综合征的疾病进程中,消化道症状也是临床常见表现,pSS中常常会遇到食管动力障碍,胃轻瘫,萎缩性胃炎和胰腺功能不全,尤以肠易激综合征和肠道运动功能障碍最常见[2]。
近年来,肠道菌群与疾病的关系越来越受到重视。肠道菌群有助于免疫系统的发展,涉及先天和适应性免疫的免疫防御过程,特定的肠道细菌系统型与免疫耐受过程有关,包括通过产生短链脂肪酸(SCFA)激活T调节细胞[3]。肠道健康微生物群的复原力保护我们免受与营养不良相关的疾病的困扰,例如炎症性肠病(IBD)或代谢紊乱,而不良生物群增加可能会引起疾病[4]。研究发现,在原发性干燥综合征患者体内,肠道微生物、上皮和胃肠道粘膜的免疫细胞之间均有着密切的相互作用;由于外分泌腺的功能异常,肠道粘液的分泌量明显减少,胃肠道上皮的保护和更新功能受到影响,可能导致微生物群—宿主免疫系统紊乱,影响上皮细胞及其屏障功能,从而出现消化道症状[5]。
微生物群组合的改变,通常被称为生态失调。新近的研究表明,肠道菌群的失衡通过影响促炎和抗炎免疫反应之间的平衡,促成一系列风湿性疾病的发生或发展,包括风湿性关节炎,系统性红斑狼疮,强直性脊柱炎,系统性硬化症和干燥综合征[6]。在系统性硬化症,近期的研究发现,患者肠道菌群较健康对照组有明显差异,特征是减少了产生保护性丁酸盐的细菌,并增加了促炎性有害菌属[7]。近年来,菌群生态失衡与pSS的关系也越来越受到关注。研究证明肠道菌群失衡可能会使pSS小鼠模型的病情恶化[8]。近年来国外已有研究发现pSS患者和健康对照相比,存在口腔菌群结构紊乱[9],但针对pSS患者肠道菌群与疾病的联系方面研究尚较缺乏。1项在对42名干燥综合征和35名健康对照进行的研究中,发现患者的肠道生态失衡比对照组高,而且程度与疾病严重程度相关,患者肠道内双歧杆菌属和Alistipes菌属群的数量明显降低[10]。上述研究均提示肠道微生物紊乱可能在pSS的发生发展中发挥某些关键作用,但具体的菌群变化和对疾病的影响尚待进一步研究。本研究拟应用16SrDNA测序技术,来明确干燥综合征患者与正常对照人相比,肠道微生物出现何种改变,为进一步明确肠道菌群失调在干燥综合征发生发展中的作用机制做初步探讨,本研究特别关注了菌群结构紊乱与疾病活动度之间的关系,为干燥综合征的治疗提供新的思路,为患者合理应用益生菌提供新的参考。
1 资料和方法 1.1 一般资料我科2018年11月~2019年11月住院pSS患者18例,因干燥综合征以女性多见,男女比例为1:9~1:10,结合收集标本时本科室实际情况,本研究纳入患者均为女性[11];年龄31~71岁,均符合2002年美国风湿病学学会-欧洲风湿病联盟共同修订的干燥综合征分类标准[12],也符合2016年美国风湿病学会-欧洲风湿病联盟(ACR/EULAR)联合制定的原发性干燥综合征国际统一分类标准[13]。排除标准:有其他自身免疫性疾病如SLE、RA、SSc、AS等,近3月内使用任何抗生素、益生菌或抗微生物制剂;曾有消化道手术或患有其他消化道器质性疾病。采用干燥综合征疾病活动指数[14](SSDAI)评价疾病活动度对患者进行分组。病情活动组SSDAI评分≥5,病情不活动或低活动组SSDAI评分 < 5分为病情不活动或低活动。所有患者留样前均为初诊初治,未曾应用激素类药物治疗。健康对照组10名,系患者健康直系家属,年龄26~71岁。近3月内未使用任何抗生素、益生菌或抗微生物制剂;消化道未进行过手术;消化道无其他器质性疾病。本实验获得本单位伦理委员会批准,所有受试对象均知情同意。
1.2 主要试剂QIAamp Fast DNA Stool Mini kit(QIAGEN),Gene JET凝胶回收试剂盒(Thermo Scientific),Running buffer(北京诺德莱科技有限公司),琼脂糖凝胶(Thermo Fisher),Omega粪便试剂盒(Omega),Hi Seq PE Rapid Cluster Kit v2 PE-402-4002(illumina),Hi Seq Rapid SBS Kit v2(500 cycles)FC-402-4023(illumina)。
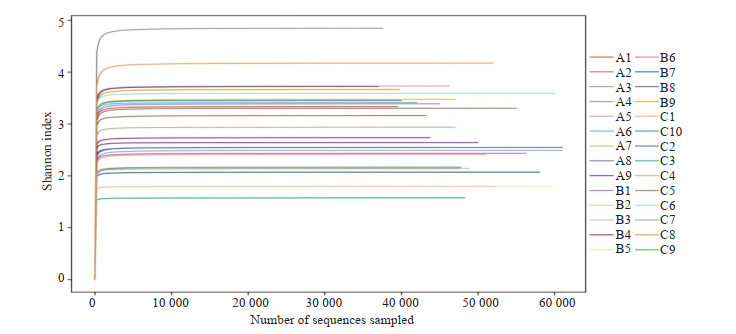
1.3 研究方法所有健康人及pSS患者分别于清晨6:00~9:00用粪便采集管收集中间段新鲜(没有接触空气)的粪便标本3~5 g。尽快将装有样品的采集管置于-80 ℃冰箱保存,达到较好地保持样品原始状态的完整性。采用QIAamp®DNA Stool Mini®方法具体抽提程序按试剂盒要求严格操作。具体为:(1)将需要测序的粪便与ASL液混合,混匀后的样品进行细菌裂解(100 ℃×10 min);(2)加入药物Inhibit Ex(QIAGEN),将孵育离心后的样品进行清除粪便抑制剂处理;(3)使用醇沉法得到DNA,将多糖、蛋白质舍弃;4)用缓冲液过柱极性洗脱,回收洗脱液并溶解于AE液中,获取粪便细菌总DNA。采用illumina hiseq平台对细菌16 s rDNA V3~V4区进行高通量测序,其结果进行生物信息学分析。首先去除标签序列(barcode标记)以及去除标签序列后 < 50 bp的序列,筛选出有效序列,并统计有效序列的分布情况包括其序列大小和序列长度。再对有效序列除杂后得到优化序列。利用OTUshannon指数等绘制稀释曲线分析3组样本的复杂度,显示所测数据量较完整,能够反映研究对象中所有微生物的多样性信息。再对样本进行OTU分析、样品多样性分析、物种注释、样品组间显著性差异分析及LEfSe分析。
1.4 统计学方法Venn图、物种profiling面积图和柱状图、Alpha指数的稀释曲线以及Alpha多样性指数盒形图的差异分析与作图均通过R软件(v3.1.1)进行。生物信息学分析中的数据采用R软件中的秩和检验方法。n=2,采用两样品比较的Wilcoxon Rank-Sum Test(R语言中的wilcox.test);n > 2,采用多样品比较的Kruskal-Wallis Test(R语言中的kruskal.test)。P < 0.05表示差异具有统计学意义,LEfSe分析使用软件Lefse实现。
2 结果 2.1 研究对象的一般特征本实验主要针对9例为病情活动组(A组)、9例为病情不活动或低活动组(B组),10例健康对照组(C组)进行OTU分析、样品多样性分析、样品组间显著性差异分析及LEfSe分析。
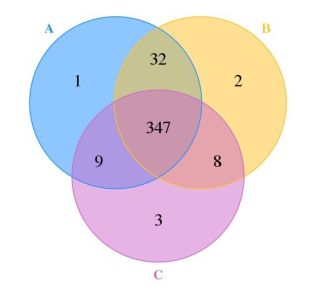
2.2 OTU分析不同样本(组)之间OTU数量及Venn图分析28个样品共产生402个OTU,其中A组9个样品测出OTU个数分别为176、177、199、334、199、129、182、154、114,平均每个样品含185个OTU;B组为176、74、84、142、94、82、114、201、345,平均每个样品含146个OTU;C组179、163、161、61、142、190、182、253、202、117,平均每个样品含165个OUT。从以上数据可知:3组OTU的平均数量为A > C > B。探究不同样本和组之间共有OTU个数及特有的OTU个数,同样在给定相似度下,聚类所得到的每个样品OTU个数绘制Venn图(图 1)。
|
图 1 图中交叠部分为两个样品或组别之间共有的OTU数 Fig.1 Number of OUTs shared between two samples or groups. |
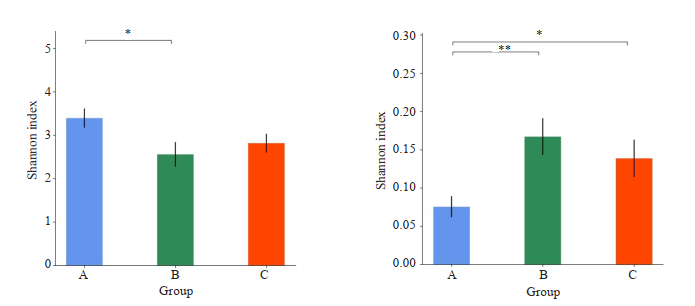
对3组α多样性(Alpha diversity)指数(包括Shannon指数、Simpson指数、ACE指数、Chao1指数)进行统计分析,两两组间比较,Shannon指数在A组和B组间、Simpson指数在A组和B组间及A组和C组差异存在统计学意义(P < 0.05,表 1、图 2),其余Alpha多样性指数组间不存在显著差异(P > 0.05,表 1)。根据shannon指数的期望值绘制Shannon指数曲线(Shannon Index)(图 3)。
| 表 1 各组α多样性指数组间差异比较 Tab.1 Comparison of the index of dataset diversity among the 3 groups (Mean±SD) |
|
图 2 组间Alpha柱形图 Fig.2 Alpha bar graph of the 3 groups (*P < 0.05, **P < 0.01). |
|
图 3 样品Shannon Index曲线 Fig.3 Shannon Index curves of the samples. As the amount of sequencing increased, more species were found, and no new OTU was found by increasing the number of sampling strips until the species was saturated. |
将所得到的所有OTU进行物种注释分析,在门、纲、目、科、属、种几个分类等级对各个样品作物种profiling面积图和柱状图,并针对门和属两个等级进行生物信息多样性分析。将物种丰度在所有样品均低于0.5%的物种全部合并成Others。
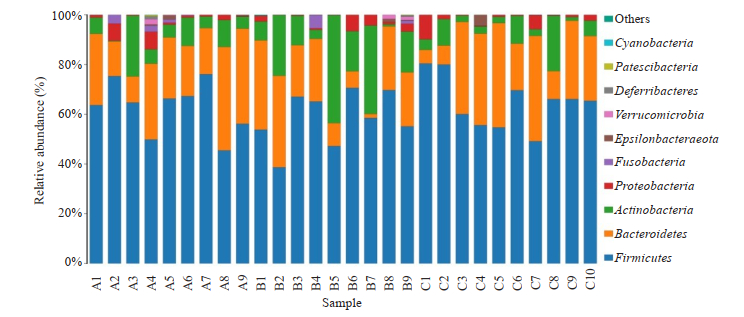
2.4.1 在门水平上的差异将A组、B组和C组进行序列分析,在门水平上3组肠道菌群共分属12个菌门,其中A组发现10个菌门,B组发现11个菌门,C组发现10个菌门,结果如下(图 4)。3组均主要有4个菌门组成,分别是:厚壁菌门(Firmicutes),拟杆菌门(Bacteroidetes),放线菌门(Actinobacteria),变形菌门(Proteobacteria),这4种主要菌门在这3组之间两两比较无显著性差异(P > 0.05),但各组间的含量波动范围差异明显。
|
图 4 样品在门分类水平中物种profiling柱状图 Fig.4 Species profiling histogram of the samples at gate classification level. |
3组样本共得到138个属,且3组间均发现100个属,结果如下(图 5),3组均主要有4个菌属组成,分别是:大芬戈尔德菌属(Finegoldia),普雷沃菌属(Prevotella),链球菌属(Streptococcus),棒状杆菌属(Corynebacterium_1),这4种主要菌属在这3组之间两两比较差异无统计学意义(P > 0.05),但Streptococcus属在3组之间比较,差异具有统计学意义(P < 0.05)。
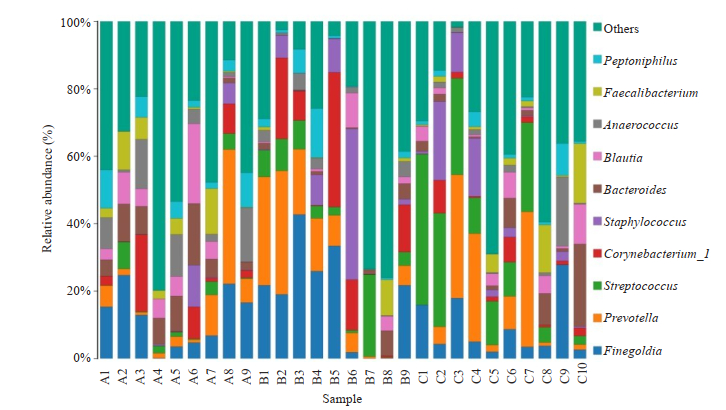
|
图 5 样品在属分类水平中物种profiling柱状图 Fig.5 Species profiling histogram of the samples at genus classification level. |
在属水平上,3组间相比,在Alloscardovia属、Bacteroides属、Barnesiella属、Butyricicoccus属、Facklamia属、Faecalibacterium属、Lachnospiraceae_FCS020_group属、Lachnospiraceae_ND3007_group属、Lachnospiraceae_UCG-001属、Lachnospiraceae_UCG-004属、Lachnospiraceae_UCG-008属、Ruminococcaceae_UCG-002属、Streptococcus属之间差异具有统计学意义P < 0.05);在Coprococcus_1属差异具有统计学意义(P < 0.01,图 6、表 2)。
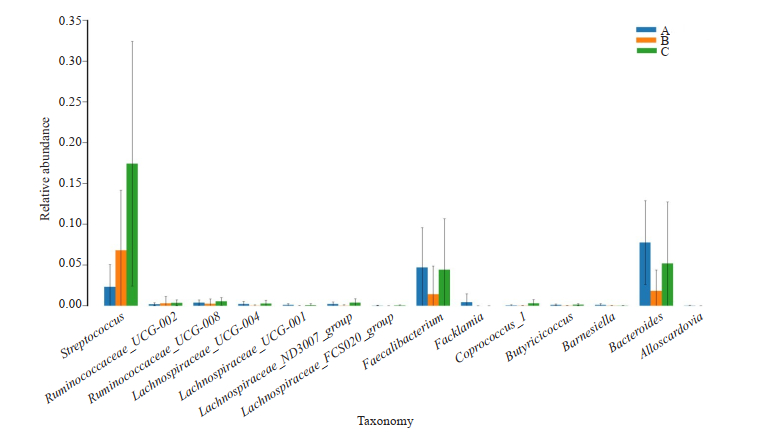
|
图 6 细菌在属水平上3组间显著性差异柱状图 Fig.6 Bar chart of the significant differences among the 3 groups at the genus level. |
| 表 2 细菌在属水平上3组间显著性差异结果 Tab.2 Significant differences among the 3 groups at the genus level (Mean±SD) |
应用LEFSE软件进行多级物种判别,对3组间进行肠道微生物进行高维生物标识和揭示基因组特征的分析,差异存在统计学意义(P < 0.05)。根据LDA SCORE (effect size)大小,A、B、C 3组相比较,A组微生物群落的核心菌群是Barnesiellaccae、Aerococcaceae、Family-XⅢ科、Bacteroidaceae、Lachnospiraceae_UCG-001属、Barnesiella属、Facklamia属、Alloscardovia属、Faecalibacterium属和Bacteroides属,C组微生物的核心菌群是Streptococcaceae、Streptococcus属、Lachnospiraceae_FCS020_group属、Coprococcus_1属、Ruminococcaceae_UCG-002属、Lachnospiraceae_UCG-004属、Lachnospiraceae_ND3007_group属、Lachnospiraceae_UCG-008属和Butyricicoccus属,B组未发现其核心菌群(图 7)。
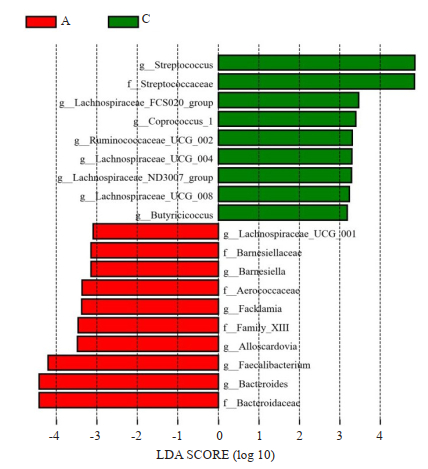
|
图 7 LDA值分布柱状图 Fig.7 Histogram of LDASCORE distribution. |
众所周知,肠道微生物,尤其是细菌,在先天性和适应性免疫应答的发展中起着至关重要的作用[15]。通过宿主与微生物之间的相互作用,菌群失衡会损害肠道粘膜屏障,导致肠道粘膜免疫功能紊乱,促炎性细胞因子增加,包括IL-1,IL-6,IL-17和肿瘤坏死因子-α(TNF-α),随后诱发慢性炎症[16]。持续性慢性炎症是风湿性疾病的主要原因,后者是由环境和遗传因素的复杂相互作用触发的,这些因素可能在没有明确原因的情况下涉及任何器官或组织[17]。越来越多的实验和临床证据表明,菌群失调引起的免疫应答是诱发自身免疫疾病发展的关键免疫病理机制[18-19],包括类风湿关节炎[20-21],系统性红斑狼疮[22-23],系统性硬化症[24-25],强直性脊柱炎[26-27],自身免疫性胰腺炎[28]和克罗恩病[29]。
本实验通过对18例pSS患者和10例健康对照进行高通量测序后,利用OTUshannon指数等绘制稀释曲线分析3组样本的复杂度,测序结果表明:样本检测覆盖深度均在97%以上,即所测数据量较完整,本次测序几乎能够完整反映研究对象中所有微生物的多样性信息。菌群多样性方面,α多样性分析多样性指数,两两组间比较,Shannon指数在A组和B组间、Simpson指数在A组和B组间及A组和C组差异存在统计学意义(P < 0.05),说明病例组及对照组组间物种多样性存在一定的差异。这一结果与de Paiva等[8]在干燥鼠模型上的研究一致,干燥鼠模型研究显示SS眼和全身疾病的严重程度与微生物多样性成反比;SS鼠表现出共生细菌的较低相对丰度和潜在致病属的较高相对丰度。在对干燥鼠模型进行脱水和抗生素处理后,与对照组相比,试验组表现出明显更差的干眼表型,更低的杯状细胞密度,更低的梭菌水平和更高的肠杆菌(大肠杆菌)水平。该研究表明菌群紊乱与SS小鼠模型和SS患者的较严重的眼粘膜疾病有关[8]。研究提示pSS患者普遍存在肠道菌群失衡,且菌群失调的程度与疾病的病情活动有关[10]。
菌群结构组成方面,3组在4种主要菌门和4个主要菌属之间两两比较差异均无统计学意义(P > 0.05),但各组间的含量波动范围差异明显,说明3组之间肠道菌群的构成比不同。进一步分析我们发现,A组与C组相比在Alloscardovia属、Barnesiella属、Bacteroides属、Coprococcus_1属、Streptococcus属、Facklamia属、Lachnospiraceae_UCG-001属、Faecalibacterium属丰度增加,而在Butyricicoccus属、Lachnospiraceae_FCS020_group属、Lachnospiraceae_ND3007_group属、Lachnospiraceae_UCG-004属、Lachnospiraceae_UCG-008属和Ruminococcaceae_UCG-002属丰度减少。随后我们应用LEFSE软件查找3组优势菌群,着力找出各组之间有显著差异丰度的物种即各自的优势菌群(即biomaker)。该分析在合成数据中,假阳性发生率非常低[30]。分析结果显示A、C两组存在优势菌群。在健康人体内,拟杆菌门和厚壁菌门的成员在肠道微生物组中占主导地位,Bacteroides属是体内最丰富的拟杆菌门菌群,与此同时,Faecalibacterium属是健康人粪便微生物组中的另一种丰富微生物,它是梭状芽胞杆菌IV(厚壁菌门)的成员,并产生SCFA(包括丁酸盐)。我们的研究发现,与健康对照相比,pSS肠道内Faecalibacterium属较健康人明显减少,该菌属是丁酸盐主要生产者之一。此结果和de Paiva等研究结果一致,后者在pSS鼠模型和pSS患者均发现粪便微生物组中Faecalibacterium属明显减少,在SS患者的粪便中Faecalibacterium属减少了约50%[8]。在活动组的pSS肠道内Butyricicoccus属细菌、疣微菌科Ruminococcaceae_UCG-002属细菌明显减少,两者也均与体内丁酸盐浓度有关,其中Butyricicoccus是一种定植于结肠粘膜产丁酸的梭状芽胞杆菌属[31-32]。而丁酸盐在机体的免疫中有重要作用,可促进Treg细胞的活化增殖、促进IL-10的生成、从而增强肠黏膜的免疫屏障功能;同时还能抑制IL-2、IL-8和TNF-α等促炎因子的, 释放,起到有效的抗炎作用[33]。pSS患者肠道内上述菌群减少,导致体内丁酸炎浓度下降,致炎因子和抗炎因子之间的平衡被破坏,影响机体免疫平衡而呈现出炎症状态。
除了上述菌群外,我们还发现,与健康对照相比,病情活动组SS粪便中,除Lachnospiraceae_UCG-001属外厚壁菌门毛螺菌科(Lachnospiraceae)菌的丰度下降,目前研究提示该类菌种可能为一种潜在的有益菌[34-35],参与多种碳水化合物的代谢,发酵葡萄糖的产物包括甲酸、乳酸、乙酸等。毛螺菌科菌也参与丁酸盐的产生,其在肠易激综合征大鼠模型[36]和慢性胰腺炎小鼠模型[37]肠道内丰度下降;有研究发现增加Gammaproteobacteria Erwinia和Pseudomonas菌属,同时减少毛螺菌属可诱导出反应性关节炎[38]。提示毛螺菌科也可能参与疾病的发生发展。其Lachnospiraceae_UCG-001属在病情稳定组下降,但在活动组轻度上升(P=0.42),考虑可能与本研究样本量较小有关。另外,我们发现,pSS患者肠道内链球菌(Streptococcus)数量增多,增加的原因尚不清楚,在IgG4相关性胰腺炎和慢性胰腺炎的研究中也发现患者粪便中此类细菌数量增多,推测可能反映吸收不良和/或胰腺酶减少有关[28]。
肠道微生物对人体健康具有重要影响。本研究发现pSS的发生和发展主要与其肠道菌群中一些产生丁酸盐的菌群数量减少有关,菌群结构紊乱可能参与pSS的发生发展过程中,这与de Luca等研究结果相吻合,后者发现pSS患者口腔和肠道菌群结构紊乱始终存在,其共生菌多肽可能通过激活Ro60反应性T细胞引起免疫应答[39]。重建肠道菌群对pSS患者有潜在的治疗作用,特别是提高产丁酸菌群的浓度。肠道茵群与pSS的关系值得进一步研究,对菌群的治疗策略可能用于缓解疾病症状、提高患者生存质量,做到精准治疗和达标治疗。
| [1] |
Bowman SJ. Primary Sjögren's syndrome[J]. Lupus, 2018, 27(suppl1): 32-5. |
| [2] |
Popov Y, Salomon-Escoto K. Gastrointestinal and hepatic disease in sjogren syndrome[J]. Rheum Dis Clin North Am, 2018, 44(1): 143-51. DOI:10.1016/j.rdc.2017.09.010 |
| [3] |
Bellocchi C, Fernández-Ochoa Á, Montanelli G, et al. Identification of a shared microbiomic and metabolomic profile in systemic autoimmune diseases[J]. J Clin Med, 2019, 8(9): E1291. DOI:10.3390/jcm8091291 |
| [4] |
Sommer F, Anderson JM, Bharti R, et al. The resilience of the intestinal microbiota influences health and disease[J]. Nat Rev Microbiol, 2017, 15(10): 630-8. DOI:10.1038/nrmicro.2017.58 |
| [5] |
Tsigalou C, Stavropoulou E, Bezirtzoglou E. Current insights in microbiome shifts in sjogren's syndrome and possible therapeutic interventions[J]. Front Immunol, 2018, 9: 1106. DOI:10.3389/fimmu.2018.01106 |
| [6] |
Zhong DL, Wu CY, Zeng XF, et al. The role of gut microbiota in the pathogenesis of rheumatic diseases[J]. Clin Rheumatol, 2018, 37(1): 25-34. DOI:10.1007/s10067-017-3821-4 |
| [7] |
Bellocchi C, Fernández-Ochoa Á, Montanelli G, et al. Microbial and metabolic multiomic correlations in systemic sclerosis patients[J]. Ann N YAcad Sci, 2018, 1421(1): 97-109. DOI:10.1111/nyas.13736 |
| [8] |
de Paiva CS, Jones DB, Stern ME, et al. Altered mucosal microbiome diversity and disease severity in sjögren syndrome[J]. Sci Rep, 2016, 6: 23561. DOI:10.1038/srep23561 |
| [9] |
Sharma D, Sandhya P, Vellarikkal SK, et al. Saliva microbiome in primary Sjögren's syndrome reveals distinct set of diseaseassociated microbes[J]. Oral Dis, 2020, 26(2): 295-301. |
| [10] |
葛均波, 徐永健. 内科学[M]. 北京: 人民卫生出版社, 2013.
|
| [11] |
Mandl T, Marsal J, Olsson P, et al. Severe intestinal dysbiosis is prevalent in primary Sjögren's syndrome and is associated with systemic disease activity[J]. Arthritis Res Ther, 2017, 19(1): 237. DOI:10.1186/s13075-017-1446-2 |
| [12] |
Vitali C, Bombardieri S, Jonsson R, et al. Classification criteria for Sjögren's syndrome: a revised version of the european criteria proposed by the american-european consensus group[J]. Ann Rheum Dis, 2002, 61(6): 554-8. DOI:10.1136/ard.61.6.554 |
| [13] |
Shiboski CH, Shiboski SC, Seror R, et al. 2016 american college of rheumatology/european league against rheumatism classification criteria for primary Sjögren's syndrome: a consensus and data-driven methodology involving three international patient cohorts[J]. Ann Rheum Dis, 2017, 76(1): 9-16. DOI:10.1136/annrheumdis-2016-210571 |
| [14] |
Vitali C, Palombi G, Baldini C, et al. Sjögren's Syndrome Disease Damage Index and disease activity index: scoring systems for the assessment of disease damage and disease activity in Sjögren's syndrome, derived from an analysis of a cohort of Italian patients[J]. Arthritis Rheum, 2007, 56(7): 2223-31. DOI:10.1002/art.22658 |
| [15] |
Kamada N, Seo SU, Chen GY, et al. Role of the gut microbiota in immunity and inflammatory disease[J]. Nat Rev Immunol, 2013, 13(5): 321-35. DOI:10.1038/nri3430 |
| [16] |
Koboziev I, Reinoso Webb C, Furr KL, et al. Role of the enteric microbiota in intestinal homeostasis and inflammation[J]. Free Radic Biol Med, 2014, 68: 122-33. DOI:10.1016/j.freeradbiomed.2013.11.008 |
| [17] |
Sparks JA, Costenbader KH. Genetics, environment, and geneenvironment interactions in the development of systemic rheumatic diseases[J]. Rheum Dis Clin NorthAm, 2014, 40(4): 637-57. DOI:10.1016/j.rdc.2014.07.005 |
| [18] |
van der Meulen TA, Harmsen H, Bootsma H, et al. The microbiomesystemic diseases connection[J]. Oral Dis, 2016, 22(8): 719-34. DOI:10.1111/odi.12472 |
| [19] |
Nikitakis NG, Papaioannou W, Sakkas LI, et al. The autoimmunityoral microbiome connection[J]. Oral Dis, 2017, 23(7): 828-39. DOI:10.1111/odi.12589 |
| [20] |
Chen J, Wright K, Davis JM, et al. An expansion of rare lineage intestinal microbes characterizes rheumatoid arthritis[J]. Genome Med, 2016, 8(1): 43. |
| [21] |
Liu XF, Zeng BH, Zhang J, et al. Role of the gut microbiome in modulating arthritis progression in mice[J]. Sci Rep, 2016, 6: 30594. DOI:10.1038/srep30594 |
| [22] |
He ZX, Shao TJ, Li HC, et al. Alterations of the gut microbiome in Chinese patients with systemic lupus erythematosus[J]. Gut Pathog, 2016, 8: 64. DOI:10.1186/s13099-016-0146-9 |
| [23] |
Hevia A, Milani C, López P, et al. Intestinal dysbiosis associated with systemic lupus erythematosus[J]. mBio, 2014, 5(5): e01548-14. |
| [24] |
Andréasson K, Alrawi Z, Persson A, et al. Intestinal dysbiosis is common in systemic sclerosis and associated with gastrointestinal and extraintestinal features of disease[J]. Arthritis Res Ther, 2016, 18(1): 278. DOI:10.1186/s13075-016-1182-z |
| [25] |
Volkmann ER, Chang YL, Barroso N, et al. Association of systemic sclerosis with a unique colonic microbial consortium[J]. Arthritis & Rheumatology (Hoboken, N.J.), 2016, 68(6): 1483-92. |
| [26] |
Costello ME, Ciccia F, Willner D, et al. Brief report: intestinal dysbiosis in ankylosing spondylitis[J]. Arthrits Rheumat, 2015, 67(3): 686-91. DOI:10.1002/art.38967 |
| [27] |
Klingberg E, Magnusson MK, Strid H, et al. A distinct gut microbiota composition in patients with ankylosing spondylitis is associated with increased levels of fecal calprotectin[J]. Arthritis Res Ther, 2019, 21(1): 248. |
| [28] |
Hamada S, Masamune A, Nabeshima T, et al. Differences in gut microbiota profiles between autoimmune pancreatitis and chronic pancreatitis[J]. Tohoku J Exp Med, 2018, 244(2): 113-7. DOI:10.1620/tjem.244.113 |
| [29] |
Matsuoka K, Kanai T. The gut microbiota and inflammatory bowel disease[J]. Semin Immunopathol, 2015, 37(1): 47-55. DOI:10.1007/s00281-014-0454-4 |
| [30] |
Segata N, Izard J, Waldron L, et al. Metagenomic biomarker discovery and explanation[J]. Genome Biol, 2011, 12(6): R60. DOI:10.1186/gb-2011-12-6-r60 |
| [31] |
Nava GM, Stappenbeck TS. Diversity of the autochthonous colonic microbiota[J]. Gut Microbes, 2011, 2(2): 99-104. DOI:10.4161/gmic.2.2.15416 |
| [32] |
Zvanych R, Lukenda N, Kim JJ, et al. Small molecule immunomodulins from cultures of the human microbiome member Lactobacillus plantarum[J]. JAntibiot, 2014, 67(1): 85-8. |
| [33] |
Sarigul M, Yazisiz V, Bassorgun CI, et al. The numbers of Foxp3 + Treg cells are positively correlated with higher grade of infiltration at the salivary glands in primary Sjogren's syndrome[J]. Lupus, 2010, 19(2): 138-45. DOI:10.1177/0961203309348234 |
| [34] |
Meehan CJ, Beiko RG. A phylogenomic view of ecological specialization in the Lachnospiraceae, a family of digestive tractassociated bacteria[J]. Genome Biol Evol, 2014, 6(3): 703-13. DOI:10.1093/gbe/evu050 |
| [35] |
Huang G, Zhang S, Zhou C, et al. Influence of Eimeria falciformis infection on gut microbiota and metabolic pathways in mice[J]. Infect Immun, 2018, 86(5): e00073-18. |
| [36] |
Zhang J, Song L, Wang Y, et al. Beneficial effect of butyrateproducing Lachnospiraceae on stress-induced visceral hypersensitivity in rats[J]. J Gastroenterol Hepatol, 2019, 34(8): 1368-76. |
| [37] |
Han MM, Zhu XY, Peng YF, et al. The alterations of gut microbiota in mice with chronic pancreatitis[J]. Ann Transl Med, 2019, 7(18): 464. DOI:10.21037/atm.2019.08.18 |
| [38] |
Manasson J, Shen N, Garcia Ferrer HR. Gut microbiota perturbations in reactive arthritis and postinfectious spondyloarthritis[J]. Arthritis Rheumatol, 2018, 70(2): 242-54. DOI:10.1002/art.40359 |
| [39] |
de Luca F, Shoenfeld Y. The microbiome in autoimmune diseases[J]. Clin Exp Immunol, 2019, 195(1): 74-85. DOI:10.1111/cei.13158 |
 2020, Vol. 40
2020, Vol. 40

