2. 南方医科大学南方医院感染内科,广东 广州 510515;
3. 南方医科大学公共卫生学 院流行病学系,广东 广州 510515
2. Department of Infectious Disease, Nanfang Hospital, Guangzhou 510515, China;
3. Department of Epidemiology, School of Public Health, Southern Medical University, Guangzhou 510515, China
耳念珠菌是于2009年在一名日本患者的外耳道分泌物中首次发现并得名[1],该真菌主要引起持续性、侵袭性感染,对氟康唑等常用抗真菌药物呈多重耐药性,感染患者的病死率高达30%~60%[2],可快速播散并能引起医院感染的暴发流行[3-4],故被称为“超级真菌”。目前,全球五大洲至少30个国家已有耳念珠菌感染病例的报道,2018年,我国的3个研究团队陆续报道了北京和辽宁省发现累计有18例耳念珠菌感染的散发病例[5-7],证实了耳念珠菌在我国本土的存在。
耳念珠菌作为一种新发现的具有多重耐药特性的医院感染病原真菌,目前对其生物学和病原学特性等方面的认识尚不够充分,其毒性特征也是值得关注的重要问题。由于不同地区分离株的菌株特性可能存在一定程度的差异,如日本和韩国的耳念珠菌分离株与印度和巴西的菌株在利用N-乙酰葡萄糖胺上有明显差异[8],我国第一例耳念珠菌分离株在生长特性方面存在很强的菌株特异性[7]。目前,有研究通过大蜡螟和小鼠感染模型证实了耳念珠菌毒力低于白色念珠菌[9-10]。但其感染方式均为静脉感染,并未探讨其可能导致血流感染的途径及方式。Biswal等[11]的研究表明耳念珠菌可以在人体胃肠道内长期定植,但尚未有足够的研究证明其是否可以通过肠粘膜入血,以及其入血后导致的病理变化。因此,本研究测定了3株耳念珠菌临床分离株与1株白色念珠菌在不同条件下的生长特性及生化表型,同时建立了裸鼠耳念珠菌胃肠道定植模型,初步探讨了其在体内的分布情况及造成的器质性病理改变特征,以期为了解耳念珠菌的生长规律、代谢特点和体内毒性特征提供基础数据。
1 材料和方法 1.1 菌株来源和菌株鉴定3株耳念珠菌和1株白色念珠菌用于生长和生化特性的测定。3株耳念珠菌均为感染患者血液标本分离株,1株为2012年于印度74岁男性患者分离株(CBS12766)[12](菌株购自广东省菌种保藏中心),该患者诊断为多器官功能障碍综合征、糖尿病、腹膜炎和败血症,于就诊后14 d死亡,其余2株(Ca1,Ca2)为印度某医院馈赠。白色念珠菌ATCC14053为美国马里兰州感染患者血液标本分离,购自北纳生物技术有限公司。所有菌株经分离培养后,采用PCR方法扩增大核糖体亚单位内转录间隔区(ITS)并测序进行鉴定[13-14]。扩增耳念珠菌和白色念珠菌核糖体亚单位ITS区域使用的引物为ITS-1(5'-TCCGTAGG TGAACCTTGCGG-3)'和ITS-4(5'-TCCTCCGCTTAT TGATATGC-3)('引物由上海生工生物工程股份有限公司合成),扩增产物经华大基因公司测序后在GenBank上进行BLAST比对鉴定。
1.2 菌株培养将冻存的4株实验菌株接种到沙氏葡萄糖琼脂培养基(SDA)(真菌蛋白胨10 g/L;葡萄糖40 g/L;琼脂15 g/L,pH 5.6±0.2)于37 ℃孵育48 h后,挑取单个菌落再次接种于SDA,37 ℃孵育48 h后,挑取单个菌落接种到5 mL沙氏液体培养基中,于恒温振荡培养箱中37 ℃、170 r/min下培养过夜。用磷酸盐缓冲溶液洗涤培养过夜的念珠菌,重复3次后用紫外分光光度计将各菌液调整至0.5麦氏浊度(A600 nm=0.1),连续稀释使所有菌株初始浓度为5×104 cfu/mL。
1.3 生长特性测定 1.3.1 生长曲线测定取上述培养菌液0.5 mL加入到含有100 mL沙氏液体培养基的锥形瓶中,于37 ℃振荡培养。每间隔4 h取菌液2 mL,用紫外分光光度计测定菌液于波长600 nm处的吸光光度值A600 nm。以时间为横坐标,A600 nm值为纵坐标作图绘制念珠菌生长曲线。
1.3.2 不同pH值菌株生长试验以pH值1为间隔,用1 mol/L Na0H和1 mol/L HCL配制pH值梯度为1~14的沙氏液体培养基,并高压灭菌。用移液器吸取0.1 mL上述菌液于已调好pH值的沙氏液体培养基,摇匀,置37 ℃恒温振荡培养箱中培养24 h。24 h后,以液体培养基作空白对照,用分光光度计测定菌液于波长600 nm处的吸光光度值A600 nm(比浊法)。
1.3.3 不同盐含量菌株生长试验分别称取NaCl 0.5、2.5、5、10 g加入SDA液体培养基,定容至100 mL,慢慢摇晃使其溶解。高压灭菌后将其分装于试管内,每种浓度盐溶液分装2管,5 mL/管。用移液器吸取0.1 mL上述菌液加入试管中,混匀,置37 ℃恒温摇床振荡培养24 h。以无菌沙氏液体培养基作空白对照,用分光光度计测定菌液于波长600 nm处的吸光光度值A600 nm(比浊法),并记录数值。
1.4 生化测定试验葡萄糖、麦芽糖、蔗糖、乳糖、半乳糖、木糖、肌醇、棉子糖、海藻糖、山梨醇和木糖醇发酵管检验菌种糖醇发酵情况,所有发酵管均购自青岛海博生物技术有限公司。分别挑取SDA上各念珠菌单个菌落接种生化管,37 ℃ 48 h后观察结果,对阴性或弱阳性管连续观察7-10 d,生化结果按试剂盒说明书判断结果。
1.5 体内毒性 1.5.1 菌株和实验动物耳念珠菌分离株CBS12766和白色念珠菌分离株ATCC14053用于动物实验。实验动物选择4周龄SPF级雄性裸鼠,体质量11~13 g,购自南方医科大学动物实验中心,健康饲养3 d后经口灌胃染毒。灌胃前将实验小鼠随机分为耳念珠菌感染组、白色念珠菌感染组和生理盐水对照组共3组,5只/组。
1.5.2 建立耳念珠菌裸鼠胃肠道感染模型小鼠灌胃前禁食6 h,用磷酸盐缓冲溶液洗涤实验菌株,重复3次后,采用血细胞计数板计数,将念珠菌细胞浓度调整至5× 109 cfu/mL。感染组灌胃剂量为0.3 mL菌液,即含有1.5×108CFU念珠菌细胞,对照组用0.3mL生理盐水灌胃。
1.5.3 实验动物剖检及组织病理学变化于灌胃后14 d心脏采血后处死所有裸鼠,取结肠组织、肝脏组织和肾脏组织于10%福尔马林缓冲液中固定后,进行常规病理切片,行HE和PAS染色,观察病理组织变化。
通过心脏穿刺获得小鼠血液,并将其置于血液增菌培养液中。通过手动按压小鼠腹部获得粪便,并将粪便、肝脏和肾脏组织分别置于1 mL磷酸盐缓冲溶液中,用无菌研磨棒研磨均匀后,进行10倍梯度稀释,共设置6个浓度梯度,振荡混匀后分别取0.2 mL接种于SDA平板。将剩余的组织研磨液分别置于含有8 mL营养肉汤的试管中。将血液增菌培养液、粪便或各组织营养肉汤于37 ℃静置增菌24 h,后接种于SDA。将SDA平板于37 ℃恒温培养箱培养48 h后进行菌落计数,并计算肝脏和肾脏组织的平均载菌量。
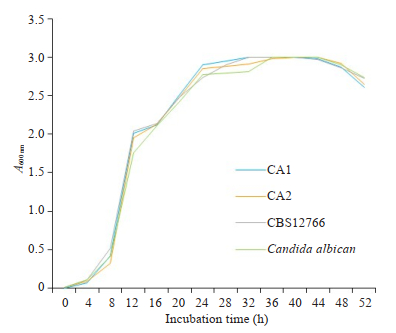
2 结果 2.1 菌株生长条件分析 2.1.1 生长曲线分析3株耳念珠菌及1株白色念珠菌在37 ℃的生长特性随时间变化如图 1所示,前8 h内4株念珠菌均处于生长迟缓期;8~24 h内念珠菌菌量增长迅速,处于对数生长期;24~48 h处于平台期,48 h后进入衰退期。耳念珠菌和白色念珠菌生长模式基本相同。
|
图 1 耳念珠菌和白色念珠菌生长曲线(比浊法) Fig.1 Growth curve of Candida auris and Candida albicans (turbidimetric method). |
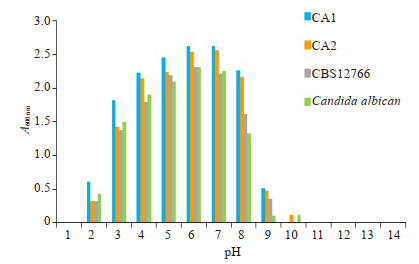
不同pH值条件下两种念珠菌在37 ℃的生长情况如图 2所示,菌株在pH值2.0~10.0均可生长,但略有差异。当pH值在5.0~ 7.0时,两种念珠菌均生长旺盛,为其最适生长pH值范围;在培养基pH值为8.0和9.0时,白色念珠菌A600 nm分别为1.33和0.1,明显低于耳念珠菌A600 nm均值(分别为2.02和0.45)。pH值为10.0时,未观察到CA1和CBS12766两株耳念珠菌生长。
|
图 2 耳念珠菌和白色念珠菌不同pH值生长情况(比浊法) Fig.2 Growth of Candida auris and Candida albicans at different pH values (turbidimetric method). |
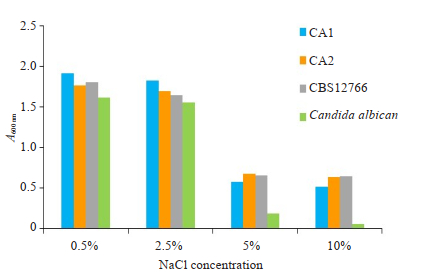
在37 ℃不同NaCl浓度的培养条件下,3株耳念珠菌生长情况接近。在NaCl浓度较低时(0.5%和2.5%),耳念珠菌和白色念珠菌均表现出较强的生长能力。随着NaCl浓度升高,两种念珠菌均出现了生长抑制,但在培养基NaCl浓度为5%和10%时,耳念珠菌仍可生长,A600 nm分别为0.635和0.599,而白色念珠菌菌液A600 nm分别为0.185和0.057,生长状况明显低于耳念珠菌(图 3)。
|
图 3 耳念珠菌和白色念珠菌不同盐含量培养生长情况(比浊法) Fig.3 Growth of Candida auris and Candida albicans in media with different salt contents (turbidimetric method). |
3株耳念珠菌均发酵葡萄糖、蔗糖、海藻糖和山梨醇,不发酵乳糖、半乳糖、麦芽糖、木糖、棉子糖、肌醇和木糖醇。白色念珠菌发酵葡萄糖、半乳糖和麦芽糖,但不发酵其他糖醇类(表 1)。
| 表 1 耳念珠菌与白色念珠菌糖、醇发酵结果 Tab.1 Fermentation of sugars (alcohols) by Candida auris and Candida albicans |
剖检耳念珠菌感染组、白色念珠菌感染组和对照组3个实验组裸鼠后,对其粪便、血液、以及研磨后的肝脏和肾脏培养,耳念珠菌感染组和白色念珠菌感染组均可分离出念珠菌,分别随机挑选念珠菌分离株经分子鉴定方法进行确认,均与所感染念珠菌种类一致。对照组的各类标本均未分离到念珠菌。耳念珠菌感染组和白色念珠菌感染组均以肝脏载菌量最高,分别约为9.3 log10 cfu/g和5.7 log10 cfu/g,肾脏组织的载菌量分别约为4.8 log10 cfu/g和2.3 log10 cfu/g。
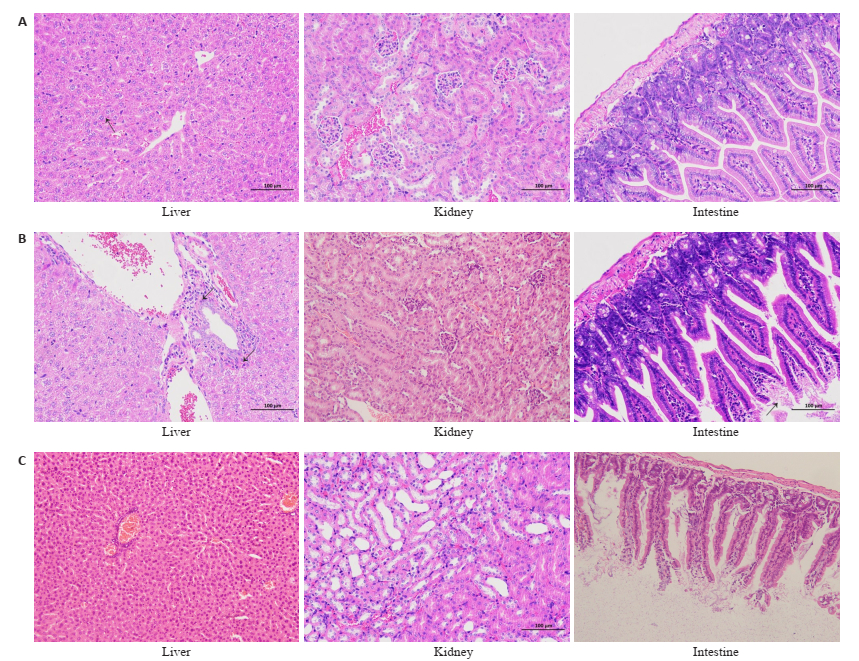
3.2.2 组织病理学变化耳念珠菌感染组和白色念珠菌感染组裸鼠的肝脏和肠组织均出现病理学变化(图 4A~B)。耳念珠菌感染组裸鼠肝组织可见肝细胞广泛肿胀,胞质疏松淡染,肠组织淋巴细胞浸润;白色念珠菌感染组裸鼠肝组织内肝细胞广泛肿胀,胞质疏松淡染,肝窦内偶见髓外造血灶,胆管周围少量炎性细胞浸润,肾脏间质充血,肠组织淋巴细胞浸润,绒毛顶端自溶;对照组病理结果未见变化(图 4C)。
|
图 4 各感染组裸鼠各器官病理变化 Fig.4 Pathological changes of organs in nude mice after infection. A: Candida auris infection; Liver: Clear structure of hepatic cord in tissue, extensive swelling of hepatocytes, loose and light staining of cytoplasm (black arrow); Kidney: No significant change; Intestine: lymphocyte infiltration; B: Candida albicans infection; Liver: Extensive swelling of hepatocytes in tissue, loose and light staining of cytoplasm (yellow arrow), occasional extramedullary hematopoietic focus in hepatic sinusoid (red arrow), a small amount of inflammatory cell infiltration around bile duct (black arrow); Kidney: Interstitial congestion; Intestine: Lymphocyte infiltration, villus tip autolysis (black arrow); C: Control group; No pathological changes were observed in liver, kidney and intestine. |
多重耐药耳念珠菌短时间内在全球范围的突然出现,引起了世界医疗卫生机构的高度关注,并迅速成为国际研究的热点真菌,但目前我国关于耳念珠菌的相关研究却极为有限,本研究对耳念珠菌不同条件下生长特性及体内毒性进行了初步探讨。
念珠菌的对数生长期在其增殖过程中尤为重要,该期菌株生长繁殖迅速,活力良好,生理活动和致病力能力强。Sherrington等[15]发现,pH值是影响念珠菌在机体内定植的重要因素之一。本研究发现耳念珠菌在37 ℃对数生长期为8~24 h,可在pH 2~10的培养环境中生长,最适生长pH范围为5.0~7.0。本研究所绘制的耳念珠菌生长曲线与已报道的念珠菌种属在全自动血培养仪中的生长曲线线型基本一致[16]。目前已有从人体多个部位分离出耳念珠菌的研究报道,如阴道(pH值4.0~5.0)[2]、皮肤表面(pH值4.0~7.0)[17]和小肠(pH值5.0~8.0)等[18-19]。虽然人体不同部位的pH值波动较大,但均处于耳念珠菌适应生长的PH值范围,而耳念珠菌对pH值的变化适应性较强的特性,可能是其可在机体不同部位广泛定植的原因之一,在机体免疫功能低下时导致机会性感染,此外,定植在人体皮肤表面的耳念珠菌有可能通过脱落的皮屑而导致医院内病原体的传播与暴发。
致病性念珠菌大多属于耐渗透酵母菌,并且可以在相对较高的NaCl浓度下生长。Krauke等[20]的研究表明,白色念珠菌与近平滑念珠菌具有较高的耐盐性。黄光华等[7]发现中国第一株耳念珠菌分离株可以在NaCl浓度为10%的培养基中生长。本研究探究了不同NaCl浓度对耳念珠菌和白色念珠菌生长的影响,结果表明,高盐环境(NaCl浓度5%和10%)虽对两种念珠菌均产生了生长抑制作用,但耳念珠菌仍能生长良好,表现出比白色念珠菌更强的耐盐能力。由于高盐和干燥环境均可使念珠菌细胞内水分流失导致细胞生长停滞,菌株的生长需通过产生相应的溶质来平衡细胞内外的渗透压[21]。与其他念珠菌相比,耳念珠菌表现出对环境更强的适应能力,已被证实可以在医院干燥的环境表面存活至少7 d[11],可能与其较强的的耐盐性有关。念珠菌的耐盐能力还取决于维持细胞内K+的浓度的能力,K+的浓度已被证明可以通过影响细胞的形态转换进而影响念珠菌耐药性[22]。本实验所使用的3株耳念珠菌均对氟康唑耐药,且目前大部分耳念珠菌临床分离株多具有多重耐药性[2],其耐盐机制与其多重耐药性是否存在关联有待进一步研究。
美国感染病学会2016年念珠菌临床实践指南显示[23],超过90%侵袭性感染通常由5种常见的念珠菌所致,即白色念珠菌、光滑念珠菌、热带念珠菌、近平滑念珠菌和克柔念珠菌。以糖醇发酵试验为主的生化试验可有效反应不同念珠菌的生物学特性。本研究结果表明,耳念珠菌可以发酵葡萄糖、蔗糖、海藻糖和山梨醇,而白色念珠菌只可发酵葡萄糖、半乳糖和麦芽糖。该结果与日本首例耳念珠菌分离株报道一致[1]。以往研究表明[24-25],克柔念珠菌与近平滑念珠菌可发酵葡萄糖;热带念珠菌可发酵葡萄糖、蔗糖、麦芽糖、半乳糖和海藻糖;光滑念珠菌可发酵葡萄糖和海藻糖。目前,临床上对念珠菌的鉴定以念珠菌显色培养基为主,并不能准确的鉴定耳念珠菌。本研究推荐将海藻糖和蔗糖发酵试验与念珠菌显色培养基相结合,可用于耳念珠菌与常见念珠菌种类的初步鉴定(表 2)。值得注意的是,耳念珠菌对于海藻糖和蔗糖的发酵属弱发酵,不同厂家生产的糖发酵微量生化管颜色变化程度不同,还应通过分子生物学方法进一步鉴定。
| 表 2 耳念珠菌与常见念珠菌生化鉴定 Tab.2 Biochemical identification of Candida auris and other Candida species |
侵袭性念珠菌感染是目前耳念珠菌感染的主要形式,病死率较高,其主要感染途径为静脉感染、胃肠道感染和呼吸道感染[26-27],具有糖尿病、艾滋病等基础疾病的免疫功能低下的患者更易发生侵袭性耳念珠菌感染[2, 28]。另有研究表明[11],耳念珠菌可以在人体胃肠道内长期定植,免疫系统损害是耳念珠菌有效感染小鼠的必要条件[9-10]。本研究首次以裸鼠作为实验动物,建立了免疫缺陷型小鼠耳念珠菌胃肠道感染模型。有研究表明白色念珠菌可在胃肠道感染后第8天突破肠屏障入血,并于感染后的第7天可从肝脏内分离出白色念珠菌[27]。由于目前认为耳念珠菌毒力低于白色念珠菌[9],为此本实验根据预实验的结果适当增加了小鼠胃肠道感染模型的观察时间,最终将观察时间定为14 d。真菌培养结果表明,感染14 d后仍可从裸鼠粪便、血液和肝肾组织中分离出耳念珠菌,提示经胃肠道感染的耳念珠菌在机体免疫抑制情况下,可能会突破肠道屏障入血引起菌血症,并能随血流播散到其他组织。肠粘膜屏障主要由机械屏障、化学屏障、免疫屏障和生物屏障构成。目前认为胃肠道感染念珠菌入血的3个主要机制如下:(1)分解细胞间的连接,使肠上皮通透性增加[29];(2)破坏肠道内微生物菌落平衡,使病原体过量繁殖并发育为毒力更强、更易粘附并穿透进入组织细胞的形态[30-31];(3)宿主免疫缺陷。本研究的病理组织学结果显示,耳念珠菌感染可导致肠淋巴细胞浸润,表明其可造成肠屏障的炎症损伤。有研究表明,以胃肠道感染的念珠菌造成肠粘膜屏障破坏后,可通过门静脉循环或胆道树转移至肝脏,再由肝脏传播至体内其他器官[32]。本研究也观察到小鼠肝脏内肝细胞广泛肿胀,胞质疏松淡染等病理变化。两组中肝脏载菌量远高于肾脏与其病理改变相符,同样支持该观点。值得注意的是,本研究使用的耳念珠菌CBS12766分离于患者血液标本,该患者被诊断为多器官衰竭综合征、败血症和腹膜炎等,其临床表现与耳念珠菌侵袭性感染所造成的多器官和组织损伤相符。虽然本研究观察到的耳念珠菌造成的组织病变严重程度低于白色念珠菌,但其造成侵袭性感染以及组织病理损伤的机制仍值得深入研究。
| [1] |
Satoh K, Makimura K, Hasumi Y, et al. Candida auris sp. nov., a novel ascomycetous yeast isolated from the external ear canal of an inpatient in a Japanese hospital[J]. Microbiol Immunol, 2009, 53(1): 41-4. |
| [2] |
Osei Sekyere J. Candida auris: a systematic review and metaanalysis of current updates on an emerging multidrug-resistant pathogen[J]. Microbiologyopen, 2019, 8(8): e00901. DOI:10.1002/mbo3.901 |
| [3] |
Calvo B, Melo AS, Perozo-Mena A, et al. First report of Candida auris in America: Clinical and microbiological aspects of 18 episodes of candidemia[J]. J Infect, 2016, 73(4): 369-74. DOI:10.1016/j.jinf.2016.07.008 |
| [4] |
Schelenz S, Hagen F, Rhodes JL, et al. First hospital outbreak of the globally emerging Candida auris in a European hospital[J]. Antimicrob Resist Infect Control, 2016, 5: 35. DOI:10.1186/s13756-016-0132-5 |
| [5] |
Chen Y, Zhao JY, Han L, et al. Emergency of fungemia cases caused by fluconazole-resistant Candida auris in Beijing, China[J]. J Infect, 2018, 77(6): 561-71. |
| [6] |
Tian SF, Rong C, Nian H, et al. First cases and risk factors of super yeast Candida auris infection or colonization from Shenyang, China[J]. Emerg Microbes Infect, 2018, 7(1): 128. |
| [7] |
Wang XJ, Bing J, Zheng QS, et al. The first isolate of Candida auris in China: clinical and biological aspects[J]. Emerg Microbes Infect, 2018, 7(1): 93. |
| [8] |
Prakash A, Sharma C, Singh A, et al. Evidence of genotypic diversity among Candida auris isolates by multilocus sequence typing, matrix-assisted laser desorption ionization time-of-flight mass spectrometry and amplified fragment length polymorphism[J]. Clin Microbiol Infect, 2016, 22(3): 277.e1-9. DOI:10.1016/j.cmi.2015.10.022 |
| [9] |
Borman AM, Szekely A, Johnson EM. Comparative pathogenicity of united kingdom isolates of the emerging pathogen Candida auris and other key pathogenic Candida species[J]. mSphere, 2016, 1(4): e00189-16. |
| [10] |
Larkin E, Hager C, Chandra J, et al. The emerging pathogen candida auris: Growth phenotype, virulence factors, activity of antifungals, and effect of SCY-078, a novel glucan synthesis inhibitor, on growth morphology and biofilm formation[J]. Antimicrob Agents Chemother, 2017, 24, 61(5): e02396-16. |
| [11] |
Biswal M, Rudramurthy SM, Jain N, et al. Controlling a possible outbreak of Candida auris infection: lessons learnt from multiple interventions[J]. J Hosp Infect, 2017, 97(4): 363-70. DOI:10.1016/j.jhin.2017.09.009 |
| [12] |
Sharma C, Kumar N, Pandey R, et al. Whole genome sequencing of emerging multidrug resistant Candida auris isolates in India demonstrates low genetic variation[J]. New Microbes New Infect, 2016, 13: 77-82. DOI:10.1016/j.nmni.2016.07.003 |
| [13] |
Ferrer C, Colom F, Frases S, et al. Detection and identification of fungal pathogens by PCR and by ITS2 and 5.8S ribosomal DNA typing in ocular infections[J]. J Clin Microbiol, 2001, 39(8): 2873-9. DOI:10.1128/JCM.39.8.2873-2879.2001 |
| [14] |
Kathuria S, Singh PK, Sharma C, et al. Multidrug-resistant Candida auris misidentified as Candida haemulonii: characterization by matrix-assisted laser desorption ionization-time of flight mass spectrometry and DNA sequencing and its antifungal susceptibility profile variability by vitek 2, CLSI broth microdilution, and etest method[J]. J Clin Microbiol, 2015, 53(6): 1823-30. |
| [15] |
Sherrington SL, Sorsby E, Mahtey N, et al. Adaptation of Candida albicans to environmental pH induces cell wall remodelling and enhances innate immune recognition[J]. PLoS Pathog, 2017, 13(5): e1006403. DOI:10.1371/journal.ppat.1006403 |
| [16] |
Klingspor L, Muhammed SA, Ozenci V. Comparison of the two blood culture systems, Bactec 9240 and BacT/Alert 3D, in the detection of Candida SPP. and bacteria with polymicrobial Sepsis[J]. Eur J Clin Microbiol Infect Dis, 2012, 31(11): 2983-7. DOI:10.1007/s10096-012-1650-6 |
| [17] |
Farage MA, Hood W, Berardesca E, et al. Intrinsic and extrinsic factors affecting skin surface pH[J]. Curr Probl Dermatol, 2018, 54: 33-47. |
| [18] |
Maurer JM, Schellekens RC, van Rieke HM, et al. Gastrointestinal pH and transit time profiling in healthy volunteers using the IntelliCap system confirms ileo-colonic release of ColoPulse tablets[J]. PLoS One, 2015, 10(7): e0129076. DOI:10.1371/journal.pone.0129076 |
| [19] |
Koziolek M, Grimm M, Becker D, et al. Investigation of pH and temperature profiles in the GI tract of fasted human subjects using the intellicap(®) system[J]. J Pharm Sci, 2015, 104(9): 2855-63. DOI:10.1002/jps.24274 |
| [20] |
Krauke Y, Sychrova H. Four pathogenic Candida species differ in salt tolerance[J]. Curr Microbiol, 2010, 61(4): 335-9. |
| [21] |
Parmar JH, Bhartiya S, Venkatesh KV. Characterization of the adaptive response and growth upon hyperosmotic shock in Saccharomyces cerevisiae[J]. Mol Biosyst, 2011, 7(4): 1138-48. |
| [22] |
Watanabe H, Azuma M, Igarashi K, et al. Relationship between cell morphology and intracellular potassium concentration in Candida albicans[J]. JAntibiot, 2006, 59(5): 281-7. |
| [23] |
Pappas PG, Kauffman CA, Andes DR, et al. Clinical practice guideline for the management of candidiasis: 2016 update by the Infectious diseases society of america[J]. Clin Infect Dis, 2016, 15; 62(4): e1-50. |
| [24] |
Silva S, Negri M, Henriques M, et al. Candida glabrata, Candida parapsilosis and Candida tropicalis: biology, epidemiology, pathogenicity and antifungal resistance[J]. FEMS Microbiol Rev, 2012, 36(2): 288-305. DOI:10.1111/j.1574-6976.2011.00278.x |
| [25] |
Samaranayake YH, Samaranayake LP. Candida krusei: biology, epidemiology, pathogenicity and clinical manifestations of an emerging pathogen[J]. J Med Microbiol, 1994, 41(5): 295-310. DOI:10.1099/00222615-41-5-295 |
| [26] |
Kataoka S, Kashiwa M, Saku K, et al. Candidemia in nonneutropenic patients with an intravenous hyperalimentation catheter: good prognosis of Candida parapsilosis infection[J]. Kansenshogaku Zasshi, 1995, 69(9): 969-74. DOI:10.11150/kansenshogakuzasshi1970.69.969 |
| [27] |
Takahashi K, Kita E, Konishi M, et al. Translocation model of Candida albicans in DBA-2/J mice with protein calorie malnutrition mimics hematogenous candidiasis in humans[J]. Microb Pathog, 2003, 35(5): 179-87. DOI:10.1016/j.micpath.2003.06.001 |
| [28] |
Rudramurthy SM, Chakrabarti A, Paul RA, et al. Candida auris candidaemia in Indian ICUs: analysis of risk factors[J]. J Antimicrob Chemother, 2017, 72(6): 1794-801. DOI:10.1093/jac/dkx034 |
| [29] |
Zhu WD, Filler SG. Interactions of Candida albicans with epithelial cells[J]. Cell Microbiol, 2010, 12(3): 273-82. DOI:10.1111/j.1462-5822.2009.01412.x |
| [30] |
Pierce JV, Kumamoto CA. Variation in Candida albicans EFG1 expression enables host-dependent changes in colonizing fungal populations[J]. mBio, 2012, 3(4): e00117-12. |
| [31] |
Sardi JC, Duque C, Mariano FS, et al. Candida SPP. in periodontal disease: a brief review[J]. J Oral Sci, 2010, 52(2): 177-85. DOI:10.2334/josnusd.52.177 |
| [32] |
Koh AY. Murine models of Candida gastrointestinal colonization and dissemination[J]. Eukaryotic Cell, 2013, 12(11): 1416-22. DOI:10.1128/EC.00196-13 |
 2020, Vol. 40
2020, Vol. 40

