2. 成都医学院第一附属医院 呼吸与危重症医学科, 四川 成都 610500
2. Department of Respiratory and Critical Care Medicine, First Affiliated Hospital of Chengdu Medical College, Chengdu 610500, China
胃癌(GC)是一种高发病率和高死亡率的消化道恶性肿瘤, 具有侵袭性强、恶性程度高、复发率转移率高以及生存期短等临床特点[1-3]。胃癌早期发现、早期诊断对于胃癌患者的综合治疗是非常重要, 可以延长胃癌患者的生存期[4]。研究发现多种基因的差异表达与胃癌的发生发展及其预后有着一定关联[5]。BUB1是一种纺锤体检测点蛋白, 在有丝分裂维持染色体正确分离、减少异倍体等生物学功能起着重要作用[6]。有研究证实BUB1基因表达的改变可影响非整倍体传代及染色体不稳定性, 从而促进肾癌、乳腺癌、前列腺癌等多种肿瘤的发生发展, 尤其在肝细胞肝癌组织中, BUB1呈现出显著高表达水平, 并参与了免疫细胞肿瘤内浸润, 影响机体的微环境和血管生成[6]。但目前关于BUB1基因在胃癌中表达水平与多种免疫细胞、免疫细胞浸润的关系, 以及是否与胃癌患者预后相关的研究较少。
Oncomine数据库是生信分析最热门的数据库之一, 整合了全球最大的癌基因芯片信息和数据挖掘平台, 利用Oncomine数据库可以比较全面地挖掘常见癌症亚型和癌旁组织的差异基因的表达, 也可以探索各个系统肿瘤特殊临床表现与分子生物学的关系[1, 7-8]。用该数据库研究胃癌患者尚未见报道。本研究通过搜集Oncomine为主的多个公共数据集, 挖掘胃癌患者中BUB1基因的表达水平, 并分析BUB1与免疫细胞浸润在胃癌组织中发挥的作用以及对胃癌患者预后的影响, 为研究胃癌的诊治和评估胃癌患者预后提供一定的生物信息学证据。
1 资料和方法 1.1 生物信息学分析资料通过BioGPS数据库(http://biogps.org)在线分析BUB1基因在正常人体不同组织和细胞中的分布情况。利用GEPIA数据库(http://gepia.cancer-pku.org)在线分析BUB1基因在正常胃组织和胃癌组织中的表达情况。利用Oncomine数据库分析胃癌组织与正常胃组织的基因差异表达和BUB1高表达研究的分析。利用Kaplan-Meier Plotter数据库(http://kmplot.com)的胃癌数据集在线分析OS、PFS与BUB1表达的关系。利用癌细胞百科全书(CCLE)分析BUB1基因在胃癌组织T细胞和B细胞中的表达。采用String数据库(http://string-db.org)分析BUB1与相互关联的基因及基因本体(GO)功能注释, 并绘制蛋白交互网络图, 通过京都基因和基因组百科全书(KEGG)挖掘BUB1基因相关蛋白的富集分析。肿瘤免疫评估数据库(TIMER)用于分析BUB1在B细胞、CD4+T细胞、中性粒细胞等6种免疫浸润细胞中的表达和对胃癌患者生存预后的影响。
1.2 实验验证数据的一般资料为进一步验证Oncomine数据库基因芯片结果, 我们收集了成都医学院第一附属医院2018年3月~2019年7月收治的胃癌患者30例, 男性15例, 女性15例, 平均年龄58.56岁, 纳入标准:(1)经胃镜活检或者外科手术后病理确诊为胃腺癌; (2)手术前未接受过放、化疗、靶向药物治疗及免疫治疗。排除标准:(1)患者已患自身免疫性疾病及结核、肝炎、艾滋病等传染性疾病; (2)伴有其他部位肿瘤; (3)合并有严重的心肺功能不全、肝肾功能不全及凝血障碍。30份胃腺癌组织取自胃癌癌巢, 同时获取的癌旁正常组织30份作为对照组(距离肿瘤边缘5 cm以上, 经术后病理证实无癌组织侵犯)手术标本切下后, 一部分组织放于冻存管中置于液氮中速冻保存; 一部分组织进行石蜡包埋。本研究经成都医学院第一附属医院伦理委员会批准通过, 所有的组织样本采集过程均获得患者本人及家属同意并签署知情同意书。
1.3 BioGPS数据库检索方法利用BioGPS数据库检索目录, 检索条件为Gene: BUB1。
1.4 GEPIA数据库检索方法设定检索标准为:Gene:BUB1, Log2 FC ≥ 1, P < 0.01, Cancer Type:Gastric cancer。
1.5 Oncomine数据库检索方法Oncomine数据库检索条件为:(1)Cancer Type: Gastric cancer; (2) Gene:BUB1;(3) Data Type:mRNA; (4) Analysis Type:Cancer vs. Normal Analysis; (5)临界值设定条件(P value < 1E-4, fold change>2, gene rank= top 10%)。采用柱状图描述结果。
1.6 Kaplan-Meier Plotter数据库分析筛选条件如下:(1)Cancer:Gastric cancer; (2) "Gene:BUB1";(3) "Survival:OS, PFS"。
1.7 CCLE数据库检索方法利用CCLE对BUB1基因在胃肿瘤组织T细胞和B细胞中的表达进行分析。
1.8 String数据库检索方法先使用Ggencards网站(https://www.genecards.org)分析BUB1基因相关蛋白功能, 并在String数据库设定限定条件为:(1)Protein Name:BUB1;(2) Organism:Human; (3) Pathway:KEGG; (4) P < 0.05。进行GO功能注释和KEGG通路富集分析。
1.9 TIMER数据库检索方法设定条件:(1) "Cancer:Lung Adenocarcinoma"; (2) "Gene:BUB1";(3) Survival Time Between:0-12/36/ 60 Months; (4) Correlation。
1.10 基础实验研究验证Oncomine数据库中的基因芯片分析 1.10.1 标本来源选择成都医学院第一附属医院2018年3月~2019年7月收治的胃癌患者30例。
1.10.2 制剂Trizol、RT-qPCR所用试剂; RIPA裂解缓冲液(ThermoFish公司); 抗BUB1抗体(Abcam公司)。
1.10.3 引物的设计和合成根据GenBank中人BUB1 mRNA序列, 采用Primer Premier3.0软件设计特异性引物, 由杭州齐步生物工程(浙江)股份有限公司合成, 以GAPDH为内参照, 设计的引物序列见表 1。
| 表 1 RT-qPCR使用的特异性引物序列 Tab.1 Specific primer sequence |
实时定量PCR按照商品的使用说明书用Trizol试剂从胃癌组织与正常组织中分别提取总RNA, 用ND-1200核酸定量检测仪(美国Thermo公司)测定总RNA的浓度和吸光度值, 以1.8 ≤吸光度值≤ 2.0为合格。总RNA经过1%琼脂糖凝胶电泳鉴定, 18 S、28 S两条区带清晰可见, 所有样本总RNA均为1.8 ≤吸光度值≤ 2.0, 表明所提取的总RNA质量合格。根据PrimeScriptTM RT Master Mix商品使用说明合成cDNA, 按照说明书使用SYBR® Premix Ex TaqTM 10 μL进行RT-qPCR, 反应在ABI 8000实时定量PCR仪进行, 以GAPDH为内参, 上游引物(10 μmol/L) 0.4 μL, 下游引物也是(10 μmol/L)0.4 μL, cDNA 1 μL, RNase Free Water 8.4 μL。反应条件设定为:95℃预变性30 s; 95℃、5 s; 60℃、34 s; 共40个循环; 60℃退火30 s。选择GAPDH作为内参, 并采用计算2-△△CT值方法进行表达量相对定量分析。
1.10.5 免疫组化将胃癌组织进行脱蜡、水化后, 用PBS洗涤, 放入pH为6.0的柠檬酸盐缓冲液中, 在微波炉中加热10 min, 再次用PBS洗涤, 在3%的双氧水中浸泡10 min进行淬灭以阻断内源性过氧化物酶, 用2%的牛血清蛋白液封闭30 min, 加入BUB1单克隆抗体4℃孵育过夜, 加入通用型二抗室温下孵育30 min, 滴加DAB显色后用苏木精液体复染, 浓度梯度乙醇脱水, 中性树胶封片后再显微镜下观察。结果的判读标准:每张病理切片至少用5个高倍视野进行观察, 当染色细胞数目占总的细胞数目的5%以下, 判读结果为BUB1阴性, 反之, 染色细胞数目>5%为阳性。
1.10.6 统计学分析采用SPSS20.0软件进行统计学分析。使用配对样本t检验分析BUB1在胃癌及其对应的癌旁正常胃组织中的表达差异。
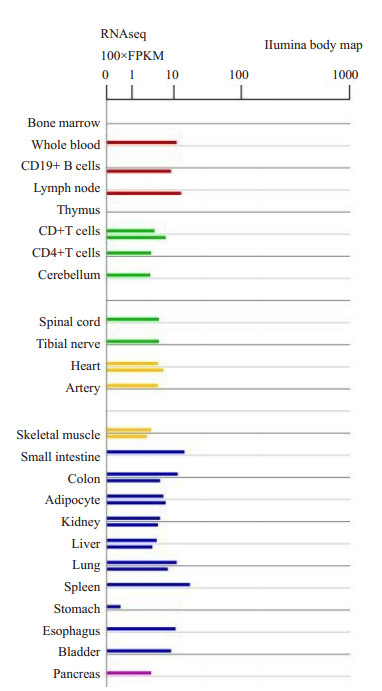
2 结果 2.1 BUB1 mRNA在正常胃组织、T细胞和B细胞中的表达分布BioGPS数据库分析结果显示, BUB1在正常胃组织、正常组织的T细胞和B细胞中均是低表达(图 1)。
|
图 1 BUB1在正常人体组织和细胞中的表达图 Fig.1 Expression of BUB1 in normal tissues and cells. |
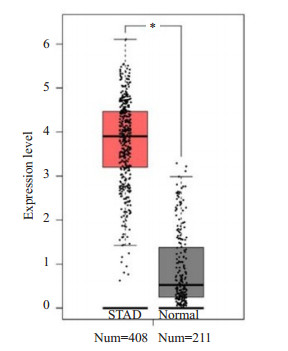
GEPIA数据库分析结果显示, 与胃正常组织相比, 胃癌组织中BUB1mRNA表达水平明显升高(P < 0.05, 图 2)。
|
图 2 BUB1在胃癌组织和正常胃组织中的不同表达情况 Fig.2 Different expression patterns of BUB1 in gastric cancer and normal gastric tissue. *P < 0.05 |
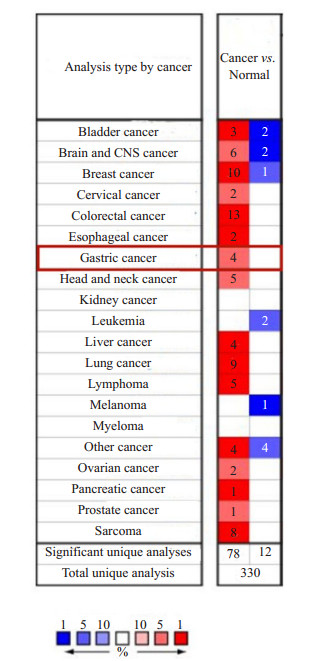
Oncomine数据库在线检索得到424项不同类型的研究, 其中BUB1表达有统计学差异的共得到90项, 包括BUB1表达增高的研究78项, 关于BUB1在胃癌中表达增高的研究有4项, 研究中表达降低的有12项。在肺癌、肝细胞肝癌、结直肠癌等肿瘤中BUB1表达明显升高; 而在血液系统肿瘤、黑色素瘤中呈现为低表达(图 3)。
|
图 3 BUB1 mRNA在不同肿瘤类型中的表达情况 Fig.3 Expression of BUB1 mRNAin different tumor types. |
Oncomine数据库检索结果显示, 在涉及到的4个关于BUB1基因与胃癌关系的研究中(图 4), 共有451例研究样本。此类研究分别发表于Mol Biol Cell[9], Clin Cancer Res[10], Nucleic Acids Res[11], Eur J Cancer[12]。我们通过Oncomine数据库在线分析这4项研究结果发现, 与正常组相比较, BUB1 mRNA在所有差异表达基因中其中位数值排名为445.0 (P < 0.05), 提示BUB1在胃癌组织中显著高表达(表 2)。

|
图 4 BUB1在胃癌各个研究中的结果分析 Fig.4 Analysis of BUB1 in different studies of gastric cancer. |
| 表 2 不同研究中BUB1 mRNA在胃癌组织中高表达情况 Tab.2 High expression of BUB1 mRNAin gastric cancer tissues in different studies |
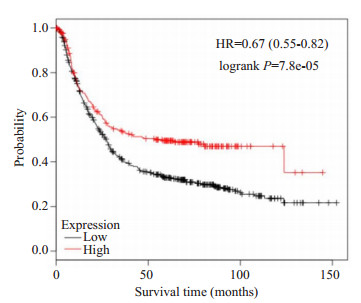
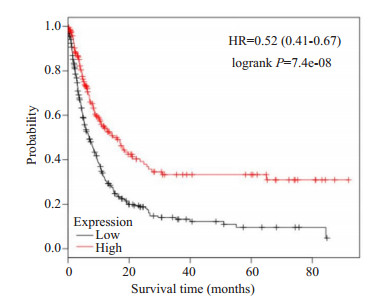
Kaplan-Meier Plotter数据库结果显示, BUB1 mRNA水平对胃癌患者的OS和PFS有显著影响(HR= 0.67, 95% CI=0.55~0.82, P < 0.05; HR=0.52, 95% CI= 0.41~0.67, P < 0.05), BUB1mRNA高表达组胃癌患者的OS和PFS均较低表达组显著延长(图 5、6)。
|
图 5 BUB1的表达和胃癌患者总生存期的关系 Fig.5 Relationship between the expression of BUB1 and the overall survival time of patients with gastric cancer. |
|
图 6 BUB1的表达和胃癌患者无进展生存期的关系 Fig.6 Relationship between expression of BUB1 and progression-free survival in patients with gastric cancer. |
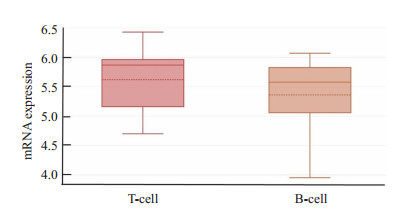
CCLE分析结果显示BUB1 mRNA在胃癌组织中的T细胞和B细胞中较正常胃组织显著高表达(P < 0.05, 图 7)。
|
图 7 BUB1 mRNA在胃癌组织中的T细胞和B细胞中的表达 Fig.7 Expression of BUB1 mRNA in T and B cells in gastric cancer tissues. |
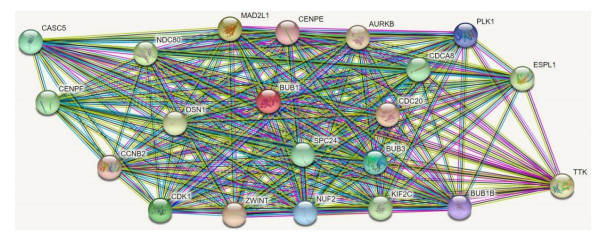
通过Genecards筛选出与BUB1相关的主要蛋白有CDCA8/CENPE/PLK1等20种, 利用String数据库构建BUB1相关蛋白网络图(图 8), 并分别进行功能注释和KEGG通路富集分析。蛋白网络主要涉及的KEGG信号通路包括细胞周期信号通路、P53相关信号通路、卵母细胞减数分裂信号通路以及卵母细胞成熟信号通路; 细胞组分主要集中在染色体着丝粒区域、纺锤体、微管细胞骨架、细胞溶质、细胞核等13类; 分子功能有蛋白丝氨酸/苏氨酸激酶活性、ATP结合、蛋白催化活性、蛋白复合绑定等4类; 生物学过程有染色体分离、细胞分裂、细胞循环、核分裂、防御反应、免疫系统的作用等12类过程(表 3)。
|
图 8 BUB1相互关联蛋白网络图 Fig.8 BUB1 interrelated protein network map. |
| 表 3 BUB1和相关基因的GO功能注释和KEGG通路富集 Tab.3 GO functional annotation and KEGG pathway enrichment of BUB1 and related genes |
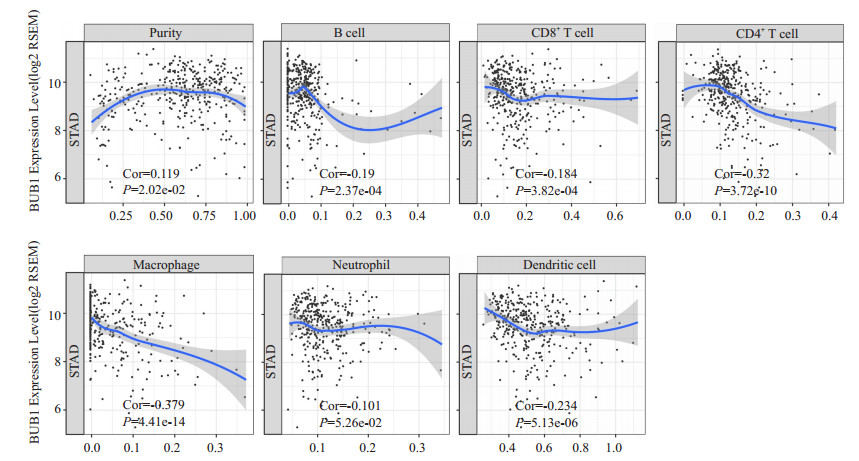
利用TIMER数据库研究发现, BUB1 mRNA在胃癌免疫微环境中与B细胞、肿瘤纯度、CD8+T细胞、巨噬细胞、CD4+T细胞和树突状细胞在胃癌组织的免疫微环境中的表达明显相关, 具有统计学意义(P < 0.05);而与中性粒细胞在胃癌组织免疫环境中的表达无确切关系, 差异无统计学意义(P>0.05)。其中BUB1 mRNA表达水平与肿瘤纯度呈正相关[偏相关系数(Partial.cor)= 0.12]表达量增加; 而与其余免疫细胞呈负相关(偏相关系数均为负值), 表达量下降(图 9)。CD4+T细胞和巨噬细胞中BUB1 mRNA高表达相对于低表达组的胃癌患者5年生存预后较好, 差异具有统计学意义(P < 0.05, 图 10);而在B细胞、CD8+T细胞和树突状细胞中, 高表达BUB1 mRNA的对胃癌患者的1年、3年、5年生存差异无统计学意义(P>0.05)。
|
图 9 BUB1与肿瘤免疫细胞间的相关性 Fig.9 Correlation between BUB1 and tumor immune cells. |
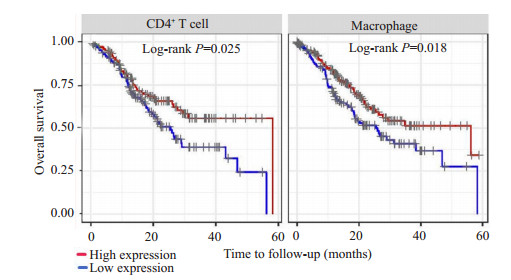
|
图 10 高表达BUB1 mRNA的CD4+T细胞和巨噬细胞对胃癌患者5年生存率的影响 Fig.10 Effects of CD4+ T cells and macrophages with high expression of BUB1 mRNA on 5-year survival rate of patients. |
30对组织样本中, 各样本的扩增曲线已达平台期, 溶解曲线均未检测到引物二聚体, 无非特异性荧光信号, 胃癌组织样本BUB1的表达水平(4.345±0.421)明显高于配对的癌旁正常胃粘膜组织(1.583±0.122)(t=34.63, P < 0.05, 图 11)。
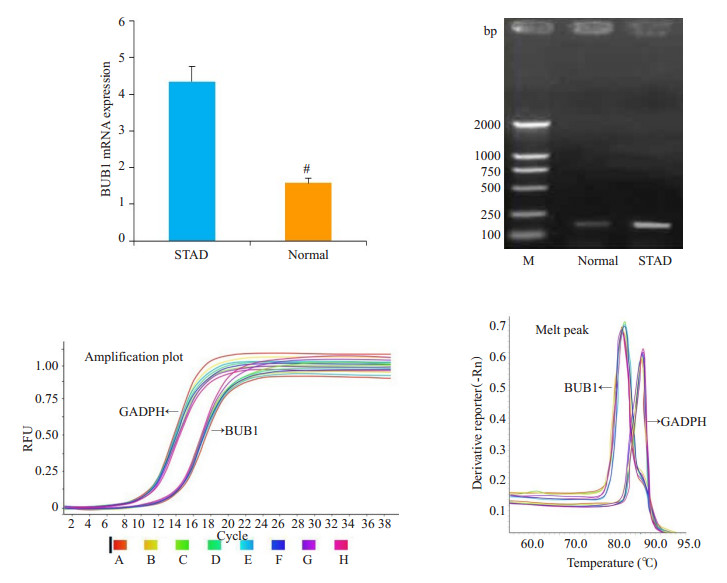
|
图 11 实验验证BUB1 mRNA在胃癌组织和正常组织的不同表达 Fig.11 Different expressions of BUB1 in surgical specimens of gastric cancer and normal gastric tissue. #P < 0.05 vs normal group. |
胃癌组织BUB1染色成阳性, 配对癌旁正常组织BUB1表达呈阴性(图 12)。
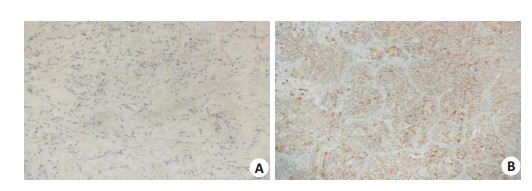
|
图 12 BUB1在胃癌及正常胃组织中表达的免疫组化染色 Fig.12 Immunohistochemical staining of BUB1 in gastric cancer and normal gastric tissues. A: Negative staining for BUB1 in normal gastric tissue (Original magnification: ×100); B: Positive staining of BUB1 in gastric cancer (×100). |
随着生活习惯、自然环境的变化以及饮食卫生条件的改变, 有数据显示胃癌的发病率呈逐年上升的趋势, 胃癌发病率占全世界恶性肿瘤发病率的第五位, 胃癌导致的死亡率位居恶性肿瘤的第三位, 已严重危害人类的生命健康。统计显示每年因胃癌导致死亡人数大约占全部恶性肿瘤死亡总人数的十分之一[13-14]。早期胃癌无明显症状, 部分患者一经诊断已处于肿瘤的中晚期, 失去了外科手术的机会[15]。虽然以铂类药物为基础联合氟尿嘧啶药物为主的化疗对胃癌患者有一定作用, 但铂类药物易产生耐药性, 难以长期维持, 患者总体生存期短, 生活质量差[16]。Pernot等[17]研究表明胃癌微环境中的各种免疫细胞的浸润与患者临床预后密切相关, 肿瘤中未分化树突状细胞、中性粒细胞、调节性T细胞等免疫细胞浸润被认为是可能成为肿瘤免疫治疗的靶标, 有助于对胃癌患者实施个体化治疗, 从而延长患者的总体生存期。因此, 胃癌微环境中的免疫机制及与预后相关免疫基因的研究, 将有助于临床医生对肿瘤的精准治疗, 现已成为近年来全世界医学研究的热点。
BUB1基因定位于人类染色体2q14上, 其编码的蛋白是纺锤体检查点的一种平台蛋白, 是纺锤体检查点其他细胞组分能精确定位于纺锤体的基础, 在有丝分裂期维持染色体正确分离、减少异倍体形成等方面起着不可或缺作用[6]。同时, BUB1也参与了有丝分裂期纺锤体组装和染色体与赤道板的排列, 在调控细胞周期、细胞程序性死亡、细胞衰老等生物学过程也起到重要作用[18]。近来有研究表明, BUB1可以直接通过磷酸化作用以形成BUB1-BUB3复合物的形式对其他重要的纺锤体检查点蛋白实施调控, 从而改变细胞周期的进程[19]。Shen等[20]报道, BUB1可能是子宫内膜癌中重要的一个枢纽基因, 在子宫内膜癌中具有高表达特性, 与子宫内膜癌的病理表型、肿瘤的分级、肿瘤的分期和预后密切相关, 且在非子宫内膜样癌组织中具有更高表达, 癌组织中的BUB1阳性表达率越低, 子宫内膜癌的分化程度越低, 预后越差。周洁等[21]研究证明鼻咽癌组织中BUB1的阳性率低于慢性鼻炎组织, 且BUB1 mRNA表达下降还与颈部淋巴结的转移相关。但也有学者提出不同结果, 如Baker等[22]报道, 在直结肠癌中, BUB1可以提高P53、DPC4、Rb等抑癌基因的突变率, 抑癌基因的突变将引起细胞分裂无法阻止在G1期, 从而促进了肿瘤的增殖。Ocaña等[23]也报道, 在卵巢癌中, 高表达BUB1基因的卵巢浆液性癌分化程度越低, 与肿瘤的侵袭深度和淋巴结转移呈正相关。上述研究提示BUB1基因在不同类型肿瘤中可能发挥着不同的作用。
目前关于BUB1基因在胃癌中的具体作用机制尚有争议, 且BUB1基因与胃癌免疫细胞浸润的关系尚未阐述[24-28], 因此, 基于目前研究背景, 本研究通过Oncomine数据库和Kaplan-Meier Plotter数据库深入挖掘BUB1基因表达的相关数据, 探寻BUB1基因在胃癌组织与正常胃组织中的不同表达及免疫细胞浸润与患者预后的关系。本研究结果发现, BUB1在多数肿瘤中呈现高表达, 表达增高的研究78项, 表达降低的研究12项, 胃癌在BUB1表达增高的研究中占了4项。BUB1在肺癌、肝细胞肝癌、结直肠癌等肿瘤组织中表达上调明显; 而在血液系统肿瘤、黑色素瘤中低表达, 通过基础实验验证, 结果与先前的研究基本一致。Kaplan-Meier Plotter数据库分析发现, BUB1基因表达水平对胃癌患者的OS、PFS存在明显的影响, BUB1mRNA高表达组胃癌患者的OS和PFS均显著延长, 表明BUB1基因高表达的胃癌患者预后可能会更好。在此基础上继续通过Genecards挖掘到和BUB1相关联的蛋白, 并对这一系列蛋白进行功能注释和KEGG通路富集分析, 发现主要涉及的KEGG信号通路有细胞周期信号通路、卵母细胞减数分裂信号通路、卵母细胞成熟信号通路、P53相关信号通路; 功能注释主要涉及多种免疫细胞功能, 如免疫系统作用、防御反应等。这与结果中CCLE分析显示BUB1 mRNA在胃癌组织中的T细胞和B细胞中较正常胃组织显著高表达相一致, 推测其原因是BUB1参与了多种免疫细胞的先天免疫防御功能, 减少肿瘤细胞对免疫细胞的抑制作用, 可提高胃癌患者机体自身的防御力[29-30], 从而延长胃癌患者总体生存期。而后, 本研究又通过TIMER数据对上述推论进行佐证, 发现高表达BUB1 mRNA的CD4+T细胞和巨噬细胞对胃癌患者5年生存预后较好, 说明高表达BUB1的肿瘤浸润CD4+T细胞和巨噬细胞对肿瘤细胞增殖具有抑制作用, 可能成为胃癌免疫治疗药物新的靶点。
综上所述, 我们通过对胃癌组织中BUB1相关基因芯片信息的深入挖掘, 提出BUB1在胃癌组织中高表达, 且与胃癌的预后及免疫细胞浸润密切相关。相对于传统的单个实验样本的研究, 肿瘤数据库具有样本量大, 可信度高的优点, 对疾病的诊治提供有力的生物学依据, 也为进一步探索BUB1基因与胃癌的关系奠定了前期的基础。
| [1] |
Melloni M, Bernardi D, Asti E, et al. Perforated gastric cancer:A systematic review[J]. J Laparoendosc Adv Surg Tech A, 2020, 30(2): 156-62. DOI:10.1089/lap.2019.0507 |
| [2] |
Rawicz-Pruszynski K, Mielko J, Pudlo K, et al. Yield of staging laparoscopy in gastric cancer is influenced by Lauren histologic subtype[J]. J Surg Oncol, 2019, 120(7): 1148-53. DOI:10.1002/jso.25711 |
| [3] |
Lee K, Kim K W, Lee J B, et al. Impact of remnant stomach volume and anastomosis on nutrition and body composition in gastric cancer patients[J]. Surg Oncol, 2019, 31: 75-82. DOI:10.1016/j.suronc.2019.09.008 |
| [4] |
Rosa F, Tortorelli AP, Quero G, et al. The impact of preoperative ASA-physical status on postoperative complications and long-term survival outcomes in gastric cancer patients[J]. Eur Rev Med Pharmacol Sci, 2019, 23(17): 7383-90. |
| [5] |
Boonyanugomol W, Rukseree K, Kongkasame W, et al. Genetic polymorphisms of CXCL8(-251)are associated with the susceptibility of helicobacter pylori infection increased the risk of inflammation and gastric cancer in Thai gastroduodenal patients[J]. Iran JAllergyAsthma Immunol, 2019, 18(4): 393-401. |
| [6] |
Leontiou I, London N, May KM, et al. The Bub1-TPR domain interacts directly with Mad3 to generate robust spindle checkpoint arrest[J]. Curr Biol, 2019, 29(14): 2407-14. DOI:10.1016/j.cub.2019.06.011 |
| [7] |
Zhang S, Yue W, Xie Y, et al. The fourmicroRNA signature identified by bioinformatics analysis predicts the prognosis of nasopharyngeal carcinoma patients[J]. Oncol Rep, 2019, 42(5): 1767-80. |
| [8] |
Vicente-Munuera P, Gomez-Galvez P, Tetley R J, et al. EpiGraph:an open-source platform to quantify epithelial organization[J]. Bioinformatics, 2019, 36(4): 1314-6. |
| [9] |
Chen X, Leung SY, Yuen ST, et al. Variation in gene expression patterns in human gastric cancers[J]. Mol Biol Cell, 2003, 14(8): 3208-15. DOI:10.1091/mbc.e02-12-0833 |
| [10] |
Cho JY, Lim JY, Cheong JH, et al. Gene expression signature-based prognostic risk score in gastric cancer[J]. Clin Cancer Res, 2011, 17(7): 1850-7. DOI:10.1158/1078-0432.CCR-10-2180 |
| [11] |
Cui J, Chen Y, Chou WC, et al. An integrated transcriptomic and computational analysis for biomarker identification in gastric cancer[J]. NucleicAcids Res, 2011, 39(4): 1197-207. DOI:10.1093/nar/gkq960 |
| [12] |
D'errico M, De Rinaldis E, Blasi MF, et al. Genome-wide expression profile of sporadic gastric cancers with microsatellite instability[J]. Eur J Cancer, 2009, 45(3): 461-9. DOI:10.1016/j.ejca.2008.10.032 |
| [13] |
任大钊, 曹生海. CA125, CEA, CRP联合检测对胃癌的诊断效果[J]. 实用癌症杂志, 2019, 34(9): 1432-4. |
| [14] |
Kang C, Dhillon S, Deeks ED. Correction to:Trifluridine/Tipiracil:A review in metastatic gastric cancer[J]. Drugs, 2019, 79(15): 1727. DOI:10.1007/s40265-019-01199-6 |
| [15] |
刘文博, 高敏, 刘婉婷, 等. 初步探究has-circ-0008797对胃癌细胞顺铂耐药性的影响[J]. 安徽医科大学学报, 2019, 34(11): 1702-7. |
| [16] |
张李琛, 张进萍, 邹云莲. 胃癌免疫治疗的研究进展[J]. 癌症进展, 2019, 17(16): 1875-8. |
| [17] |
Pernot S, Terme M, Radosevic-Robin N, et al. Infiltrating and peripheral immune cell analysis in advanced gastric cancer according to the Lauren classification and its prognostic significance[J]. GastricCancer, 2020, 23(1): 73-81. |
| [18] |
Pagotto S, VeroneseA, SorannoA, et al. HNRNPLrestrains miR-155 targeting of BUB1 to stabilize aberrant karyotypes of transformed cells in chronic lymphocytic leukemia[J]. Cancers (Basel), 2019, 11(4): pii:E575. DOI:10.3390/cancers11040575 |
| [19] |
殷复粉, 赵丽君, 李小平, 等. BUB1基因在妇科恶性肿瘤中的研究进展[J]. 中国妇产科临床杂志, 2019, 20(2): 186-7. |
| [20] |
Shen L, Liu M, Liu W, et al. Bioinformatics analysis of RNA sequencing data reveals multiple key genes in uterine corpus endometrialcarcinoma[J]. OncolLett, 2018, 15(1): 205-12. |
| [21] |
周洁, 陈应超, 王玮, 等. BUB1在鼻咽癌中的表达及临床意义[J]. 中华全科医学, 2014, 12(2): 212-4. |
| [22] |
Baker DJ, Jin F, Jeganathan KB, et al. Whole chromosome instability caused by Bub1 insufficiency drives tumorigenesis through tumor suppressor gene loss of heterozygosity[J]. Cancer Cell, 2009, 16(6): 475-86. DOI:10.1016/j.ccr.2009.10.023 |
| [23] |
Ocaña A, Pérez-Peña J, Alcaraz-Sanabria A, et al. In silico analyses identify gene-sets, associated with clinical outcome in ovarian cancer:role of mitotic kinases[J]. Oncotarget, 2016, 7(16): 22865-72. DOI:10.18632/oncotarget.8118 |
| [24] |
Kawakubo E, Matsumoto T, Yoshiya K, et al. BUBR1 insufficiency is correlated with eNOS reduction experimentally in vitro and in vivo, and in gastric cancer tissue[J]. Anticancer Res, 2018, 38(11): 6099-106. DOI:10.21873/anticanres.12960 |
| [25] |
Yuan W, Xie S, Wang M, et al. Bioinformatic analysis of prognostic value of ZW10 interacting protein in lung cancer[J]. Onco Targets Ther, 2018, 11: 1683-95. DOI:10.2147/OTT.S149012 |
| [26] |
Stahl D, Braun M, Gentles AJ, et al. Low BUB1 expression is an adverse prognostic marker in gastric adenocarcinoma[J]. Oncotarget, 2017, 8(44): 76329-39. DOI:10.18632/oncotarget.19357 |
| [27] |
Tong H, Wang J, Chen H. Transcriptomic analysis of gene expression profiles of stomach carcinoma reveal abnormal expression of mitoticcomponents[J]. LifeSci, 2017, 170: 41-9. DOI:10.1016/j.lfs.2016.12.001 |
| [28] |
Qu Z, Zou X, Zhang X, et al. Chelidonine induces mitotic slippage and apoptotic-like death in SGC-7901 human gastric carcinoma cells[J]. MolMedRep, 2016, 13(2): 1336-44. |
| [29] |
Pabla S, Conroy JM, Nesline MK, et al. Proliferative potential and resistance to immune checkpoint blockade in lung cancer patients[J]. JImmunotherCancer, 2019, 7(1): 27. |
| [30] |
Yang S, Yu J, Fan Z. Bub1 facilitates virus entry through endocytosis in a model of drosophila pathogenesis[J]. J Virol, 2018, 92(18): 218-54. |
 2020, Vol. 40
2020, Vol. 40

