食管癌是我国常见的恶性肿瘤之一,其中最为常见的病理亚型是食管鳞癌,其病理类型主要是食管鳞状细胞癌(ESCC)和食管腺癌(EAC)两大类,其中大部分为ESCC[1-2]。大多数的食管鳞癌在初次诊断时就已出现晚期转移,这导致食管鳞癌患者5年存活率不足10%[3-4]。因此对食管鳞癌的早期或预后转移预测显得特别重要。
长链非编码RNA(IncRNA)是长度超过200 bp的核苷酸分子,不编码或很少编码蛋白。随着高通量测序技术和IncRNA芯片检测的大规模应用,大量lncRNA在肿瘤组织和体液中被发现,作为促癌基因或者抑癌基因对肿瘤下游基因蛋白的表达调控起关键作用[5]。近年来,越来越多的研究表明IncRNA参与多种恶性肿瘤包括肺癌[6],膀胱癌[7],胃癌[8],结直肠癌[9]及食管鳞癌[10]的侵袭和转移。在食管癌中,有多个表达上调的lncRNA,常见的如HOTAIR[11]和MALAT1[12]等,但也有表达下调的lncRNA,如TUSC7[13]和91H[14]。研究发现,lncRNA TUSC7在食管癌组织中表达下调,低表达患者对放化疗耐受性增加,无病生存率和总生存率降低[15-16]。TUSC7低表达会促进细胞凋亡,进一步研究发现TUSC7序列上同时存在与抑癌因子p53及mi R-211的结合位点,同时受p53及mi R-211的双向调节[17]。提示TUSC7可指导ESCC患者的个体化治疗,同时可作为ESCC患者的独立预后指标。但是食管鳞癌患者血清中TUSC7是否存在表达差异及其在食管鳞癌肿瘤转移过程中发挥何种生物学功能,尚未见明确的机制研究报告。
本研究采用荧光实时定量PCR检测食管鳞癌患者血清中TUSC7的表达水平,分析TUSC7的表达与ESCC患者临床病理特征的关系,并通过一系列体外实验探讨TUSC7对ESCC细胞转移、侵袭、增殖和EMT的影响,为进一步研究TUSC7在ESCC侵袭转移过程中的作用机制提供了理论依据。
1 资料和方法 1.1 标本一般资料选择2017年1月~2019年5月南阳市第二人民医院和南阳市中心医院收治的ESCC患者60例,男性40例,女性20例。另选健康受试者60例,作为对照组,男性35例、女25例,两组性别、年龄无统计学差异。由专业医护人员于治疗前收集空腹外周静脉血5 mL,室温静置30 min,离心后取上清至无RNA酶离心管,-80 ℃冻存待检,整个过程避免溶血。本研究经医院伦理委员会批准,所有患者均知情同意。纳入标准:(1)所有患者结合相关临床诊断确诊为食管鳞癌;(2)术前没有接受任何抗肿瘤治疗;排除标准:排除伴发器质性疾病以及合并其他肿瘤者。
1.2 细胞株4种ESCC细胞株(KYSE-30、EC-9706、KYSE-70 and ECa-109)和正常食管上皮细胞(Het-1A)购自上海生命科学研究院细胞库。
1.3 主要试剂血清RNA提取试剂盒(Qiagen)、Prime ScriptTM RT Reagent Kit(TaKaRa)、SYBR® Premix Ex TaqTM Ⅱ(TaKaRa)。引物、pLenti-TUSC7及对照plenti-NC(Invitrigen);siRNA-TUSC7及对照si-NC(锐博)、RPMI 1640培养基、胎牛血清、0.25%胰蛋白酶(Gibco);LipofectamineTM 3000转染试剂(Invitrogen);E-钙粘蛋白,N-钙粘蛋白和波形蛋白,基质金属蛋白酶9抗体(Abcam);噻唑蓝(MTT)(碧云天);Matrigel胶(BD)。
1.4 实验方法 1.4.1 细胞培养4种ESCC细胞株(KYSE-30,EC-9706,KYSE-150 and ECa-109)和正常食管上皮细胞(Het-1A)用含10%胎牛血清,1%双抗(100 μ/mL青霉素和100 μg/mL链霉素)的RPIM 1640完全培养液,置于37 ℃,含5%CO2的细胞培养箱中培养。
1.4.2 总RNA提取及RT-qPCR检测根据美国国立生物技术信息中心(NCBI)数据库中TUSC7基因序列,通过PrimerExpress2.0设计扩增引物。(1)RNA提取及检测:按照血清RNA提取试剂盒及TRIZOL法分别提取血清中和ESCC细胞系中总RNA,电泳法检测RNA完整度;使用核酸蛋白测定仪(Thermo)测定RNA浓度,当总RNA浓度 > 210 ng/μL,A260/280值为1.8~2.1,认为样本质量良好,并于-80 ℃保存;(2)RNA逆转录:将总RNA按Prime ScriptTM RT reagent Kit试剂盒说明书将RNA逆转录为cDNA,所得cDNA -20℃保存;(3)qPCR检测:TUSC7及其内参GAPDH引物由上海生工生物工程有限公司设计合成。按SYBR® Premix Ex TaqTMⅡ试剂盒说明书,以cDNA为模板,GAPDH为内参,进行qPCR扩增。反应条件:95 ℃预变性1 min,95 ℃,10 s;64 ℃,30 s;72 ℃,30 s;共40个循环,72 ℃延伸5 min。基因表达量采用RT-qPCR相对定量法(2-ΔΔCt法)计算TUSC7相对表达量。
1.4.3 细胞转染将处于对数生长期的细胞接种到富含完全培养基的6孔培养板中,待细胞生长至50%~60%融合度,根据说明书,使用Lipo-fectamineTM 3000将pLenti-TUSC7,pLenti-NC,si-TUSC7,si-NC分别转染至KYSE-30细胞,各组转染至KYSE-30细胞,24 h、48 h后收集转染的细胞,提取RNA,RT-qPCR以确定转染效率,行后续实验。
1.4.4 Trans-well检测细胞迁移侵袭能力在Boyden小室上室加入200 μL稀释的Matrigel胶,过夜干燥。将转染后的KYSE-30细胞制成单细胞悬液并用无血清培养基漂洗3遍,细胞计数后,接种在Trans-well小室上室,在小室的下室加入300 μL含10%胎牛血清的DMEM培养基作为趋化因子。37 ℃、5% CO2培养箱,24 h后取出小室,PBS浸泡洗涤3次。95%乙醇浸泡固定Tran-swell上室穿膜细胞,PBS洗涤3次,以新鲜配制的结晶紫染液,浸染5~10 min,然后用棉签擦除上室中未通过滤膜的细胞,显微镜下观察拍照、计数。取平均值,以迁移至微孔膜下层细胞数来评价其侵袭能力。
1.4.5 划痕试验检测细胞迁移能力将转染后的KYSE-30细胞以1×106/孔接种于6孔板中并培养至80%融合度。采用200 μL移液管尖端刮擦单层细胞,然后将细胞在不含FBS的培养基中继续培养。在倒置显微镜下观察伤口愈合情况并分别在0、24、48 h拍照,使用伤口愈合率ImageJ软件测量伤口宽度。伤口愈合率=(原始差表示;两组间比较采用独立样本t检验。P < 0.05认为伤口宽度-不同时间点的伤口宽度/原始伤口宽度× 100%。
1.4.6 Western blot检测EMT蛋白标记物的表达转染后24 h收集细胞,RIPA裂解液裂解细胞提取总蛋白,BCA法测定蛋白质浓度。10%SDS-PAGE胶电泳分离蛋白质,转膜,用5%脱脂奶粉室温封闭1 h,一抗(Ncadherin、Vimentin、E-cadherin抗体稀释1:1000;β- actin抗体稀释1 :3000)4 ℃过夜孵育,TBST洗膜3次,每次5 min,二抗(抗体1:2000稀释)室温孵育1 h,TBST洗膜3次,每次5 min,暗室加入ECL发光液后曝光显影。1.4.7 MTT检测细胞增殖KYSE-30细胞按1×103/孔接种至96孔板内,重复5孔。转染pLenti-TUSC7及pLenti-NC后,常规培养12、24 h后,弃培养基,每孔加入20 μL的MTT,继续培养3 h后,加入150 μL DMSO溶解,酶标仪570 nm处检测各孔A值,绘制细胞增殖图,实验重复3次。
1.5 统计学分析采用Graph Pad Prism 5软件和SPSS 22.0进行统计分析。血清中TUSC7的表达量采用KolmogorovSmirnov检验为计量资料的非正态分布,故两组间比较采用Mann-Whitney U检验;其他计量资料用均数±标准差表示;两组间比较采用独立样本t检验。P < 0.05认为差异具有统计学意义。
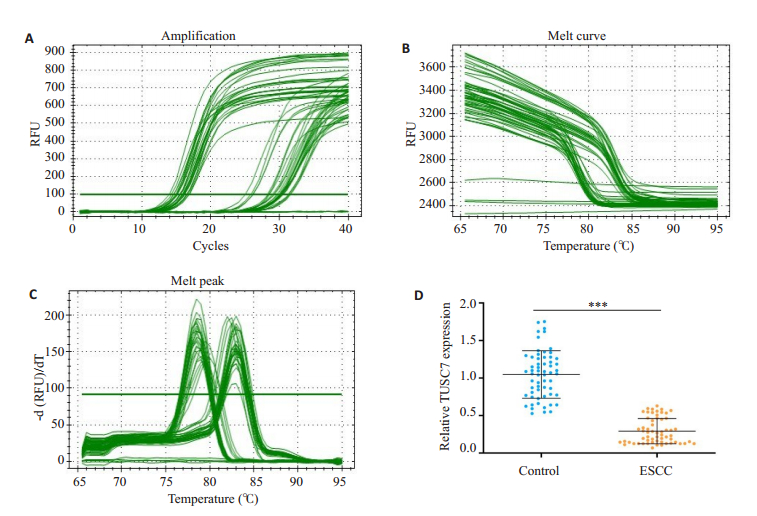
2 结果 2.1 TUSC7在食管鳞癌患者血清和正常人群血清中的表达通过RT-qPCR检测60例ESCC患者和60例健康对照组血清样本中TUSC7的表达情况,以GAPDH为内参,应用2-ΔΔCt公式计算食管鳞癌患者血清中TUSC7相对表达量。图 1A~C分别是120例样本中的30例样本(10例对照和20例患者血清样本)的qPCR检测TUSC7的扩增曲线,溶解曲线和溶解峰。因此,TUSC7在ESCC患者血清中的相对表达量显著低于健康对照组,差异具有统计学意义(图 1D)。
|
图 1 RT-qPCR检测ESCC患者和健康对照血清样本中TUSC7的相对表达量 Fig.1 Serum TUSC7 level in ESCC patients and normal control group detected by RT-qPCR. A-C: The amplification and melting curves and the melt peak of RT-qPCR for 30 clinical samples (10 control and 20 ESCC cases); D: Scatter plot showing relative serum TUSC7 level in ESCC patients and normal control group; 2-ΔΔCt method was used to measure the relative TUSC7 expression. Data are presented as Mean±SD (***P < 0.001) |
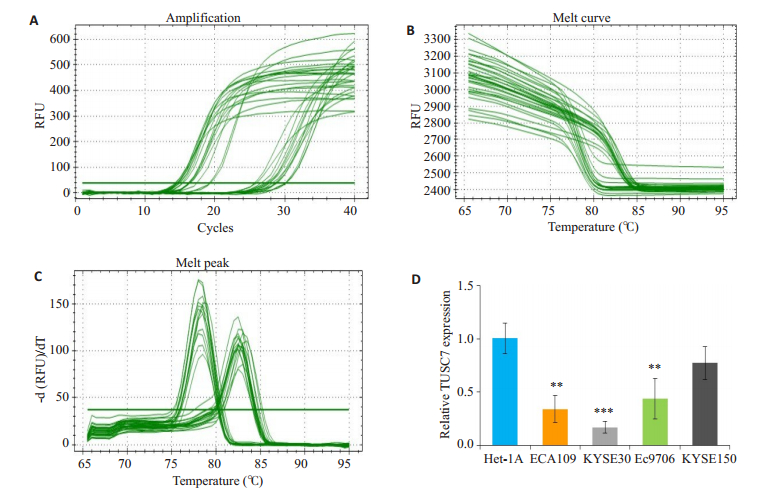
我们研究了4种ESCC细胞系(KYSE-30,EC-9706,KYSE-150,ECA109)和1种正常食管上皮细胞系中TUSC7的水平。图 2A~C分别为5株细胞PCR实验检测TUSC7表达量的扩增曲线,溶解曲线和溶解峰。结果显示,在4个肿瘤细胞株中,3株(ECA109,EC-9706,KYSE-30)与正常食管上皮细胞比较均下调,但KYSE150的差异无统计学意义(图 2D)。
|
图 2 RT-qPCR检测正常食管上皮细胞和ESCC细胞系中TUSC7的相对表达量 Fig.2 Relative expression of TUSC7 in normal esophageal epithelial cells and ESCC cell lines detected by RT-qPCR. A-C: The amplification and melting curves and the melting peak of RT-qPCR; D: Comparison of the relative expressions of TUSC7 in ESCC cell lines and het-1a cells. Data are presented as Mean±SD (**P < 0.01 vs Het-1A; ***P < 0.001 vs Het-1A; n=3) |
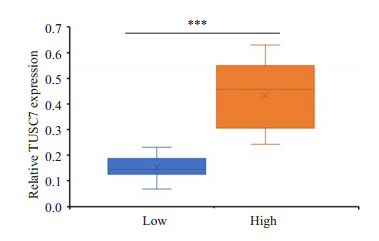
以TUSC7的中位表达水平为截止值,将60名患者分为TUSC7-高表达组(n=30)和低表达组(n=30),差异具有统计学意义(图 3,P < 0.001)。血清中TUSC7的表达与ESCC临床病理特征之间的关系详见表 1。两组相比,TUSC7低表达组与TNM分期和更多淋巴结转移和局部浸润程度相关;但TUSC7低表达与其他临床病理学数据(包括性别、年龄、吸烟状况、饮酒、肿瘤部位、和饮食习惯)之间不存在统计关联。
|
图 3 ESCC患者血清中TUSC7高表达组和TUSC7低表达组的相对表达水平比较 Fig.3 Comparison of serum TUSC7 level between ESCC patients with high (n=30) and low TUSC7 expression (n=30), with the median expression level as the cut-off (***P < 0.001) |
| 表 1 ESCC患者血清中lncRNA表达水平与临床病理特征之间的关系 Tab.1 Correlation between serum TUSC7 level and clinicopathological characteristics of ESCC patients |
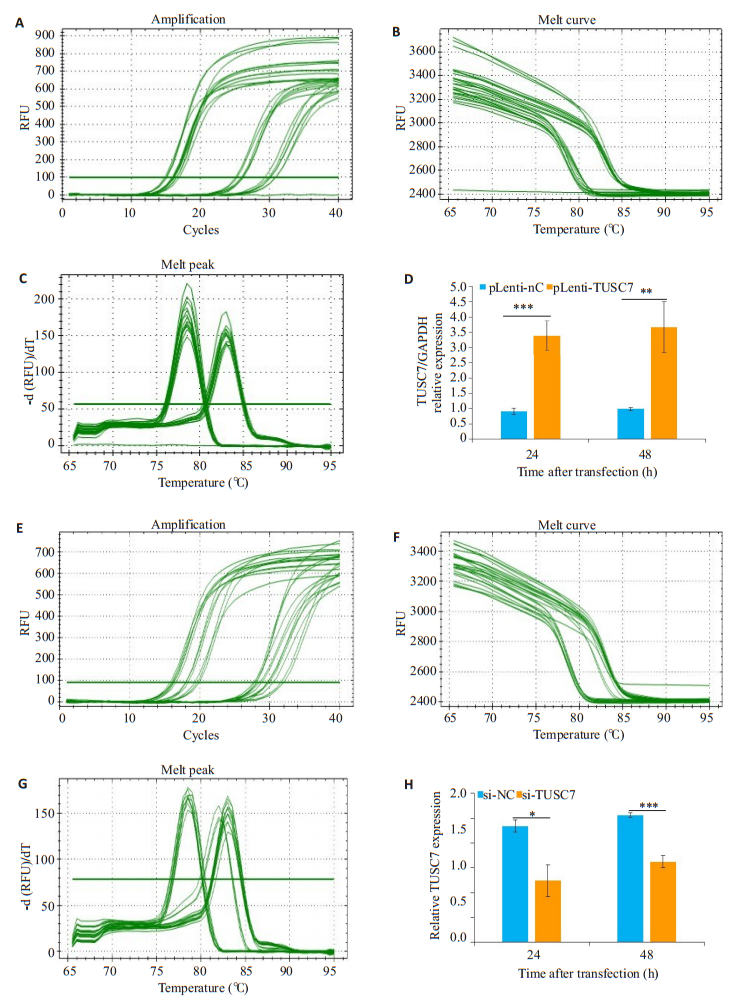
以GAPDH为内参,KYSE-30细胞分别转染pLenti-TUSC7、pLenti-NC,24、48 h后RT- qPCR检测TUSC7的相对表达量。图 4A~C分别为过表达TUSC7,qPCR实验检测TUSC7的扩增曲线,溶解曲线和溶解峰;图 4E~G分别为抑制TUSC7,qPCR实验检测TUSC7的扩增曲线,溶解曲线和溶解峰。结果显示,pLenti-TUSC7转染组TUSC7的相对表达量显著提高(图 4D);而分别转染si-TUSC7、si-NC后,24、48 h后RT-qPCR检测结果显示,si-TUSC7转染组TUSC7的相对表达量显著降低(图 4H)。
|
图 4 RT-qPCR检测过表达或抑制TUSC7后KYSE-30细胞中TUSC7的相对表达量 Fig.4 Relative expression of TUSC7 in KYSE-30 cells with TUSC7 overexpression or knockdown was detected by RT-qPCR. The amplification and melting curves and the melting peak are shown in A-C and E- G, D, H: Comparison of TUSC7 expression at 24 and 48 h after transfection of the cells with pLenti-TUSC7, pLenti-NC, si-NC or si-TUSC7 (*P < 0.05; **P < 0.01;***P < 0.001; n=3) |
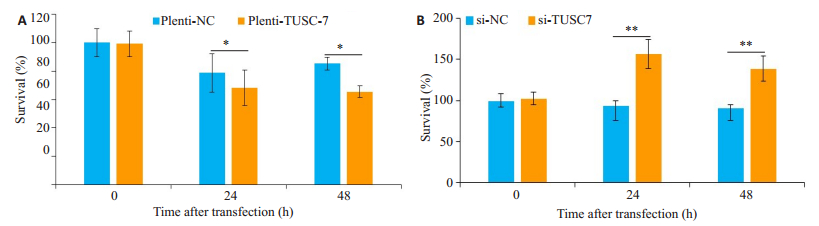
MTT方法检测结果显示,pLenti-TUSC7转染组细胞较对照组细胞在24、48 h增殖能力明显受抑制,差异具有统计学意义(P < 0.01,图 5A)。si-TUSC7转染组细胞对照组细胞在24、48 h增殖能力明显增强,差异具有统计学意义(P < 0.05,图 5B)。
|
图 5 过表达或下调TUSC7对KYSE-30细胞增殖的影响 Fig.5 Effects of TUSC7 overexpression or knockdown on the proliferation of KYSE-30 cells. (A) Cell viability assessed by MTT assay after overexpression of TUSC7 for 0, 24 and 48 h; (B) Cell viability assessed by MTT assay after knockdown of TUSC7 for 0, 24 and 48 h (*P < 0.05; **P < 0.01; n=3) |
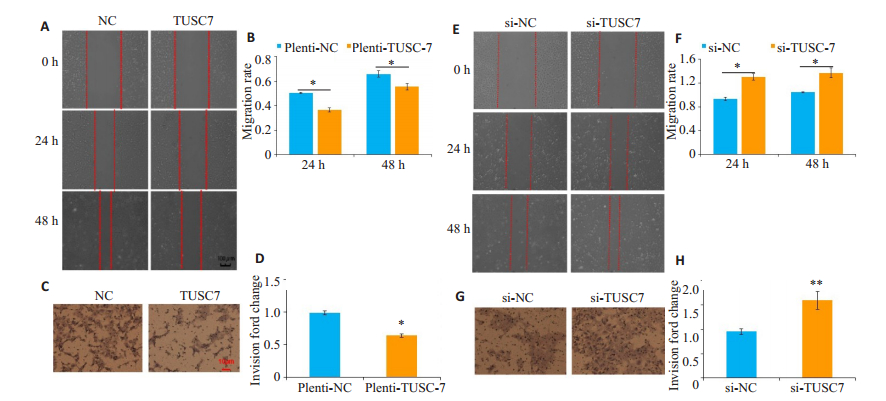
划痕实验结果表明:pLenti-TUSC7转染组细胞较对照组细胞在24、48 h迁移能力明显受抑制,划痕愈合率在24和48 h显著低于pLenti-NC组,组间差异有统计学意义(图 6A、B)。在Transwell小室侵袭实验中,pLenti-TUSC7转染组细胞从小室上层穿到下层的细胞明显少于pLenti-NC转染组细胞,侵袭能力较对照组显著降低(图 6C、D)。si-TUSC7转染组细胞在24、48 h迁移能力明显增强,划痕愈合率在高于显著低于si-NC组,组间差异有统计学意义(图 6E、F);在Transwell小室侵袭实验中,si-TUSC7组从小室上层穿到下层细胞数量明显多于si-NC组,侵袭能力较对照组显著增强(图 6G、H)。
|
图 6 过表达TUSC7抑制KYSE-30细胞的迁移和侵袭,下调TUSC7促进KYSE-30细胞的迁移和侵袭 Fig.6 Overexpression of TUSC7 decreases migration and invasion and TUSC7 knockdown promotes the migration and invasion of KYSE-30 cells. A, E: Migratory capacity were detected by wound-healing assay at 24 h and 48 h after transfection(×10); (C, G) Invasion cells were detected by Transwell assay (Original magnification: ×100); (B, D, F, H)Invasion rate and wound healing rate between pLenti-TUSC7 and pLenti-NC; si-NC and si-TUSC7 respectively. (*P < 0.05; **P < 0.01; n=3) |
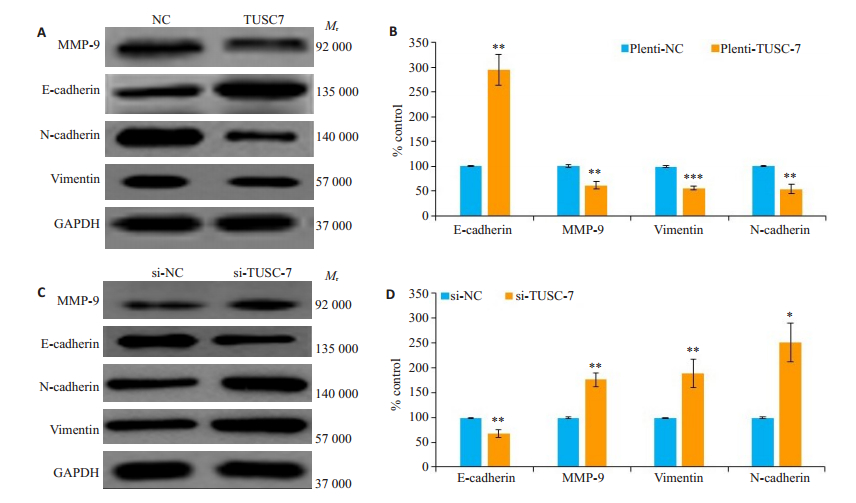
EMT与肿瘤的发生发展密切相关,为了进一步探讨TUSC7与食管鳞癌细胞EMT的相互关系,我们采用Western blot,检测了过表达或抑制TUSC7后,各组蛋白提取物EMT相关标志物蛋白表达水平,结果显示,pLenti-TUSC7转染组与对照组相比,E-cadherin蛋白表达水平显著上调,而MMP-9,N-cadherin和Vimentin蛋白表达水平显著下调(图 7A、B)。反之,si-TUSC7转染组与对照组相比,E-Cadherin蛋白表达水平显著下调,而使MMP-9,N-Cadherin和Vimentin蛋白表达量水平显著上调(图 7C、D)。
|
图 7 过表达或抑制TUSC7对KYSE-30细胞EMT相关蛋白相对表达量的影响 Fig.7 Effects of TUSC7 over-expression or knockdown on expression of EMT-related proteins in KYSE-30. A, C: Western blotting for MMP-9, E-cadherin, N-cadherin, and vimentin, with GAPDH as the loading control; B, D: Statistical analysis of the protein expressions (*P<0.05; **P<0.01; ***P<0.001; n=3) |
食管鳞癌是一种侵袭性恶性肿瘤, 虽然采用手术、新辅助化疗、放射性及生物治疗等多种手段结合的治疗方法进行治疗, 但由于其生长速度快, 容易发生区域或远处转移, 导致患者预后较差, 5年生存年率仍很低。肿瘤的侵袭和转移是癌症高死亡率的主要原因, 基质金属蛋白酶-9 (MMP-9)是1类重要的水解细胞外基质的蛋白水解酶, 参与到肿瘤的侵袭与转移过程。EMT与癌细胞的高侵袭性和转移密切相关, EMT过程中上皮细胞失去连接蛋白(如E-cadherin), 并获得间充质标志蛋白(Ncadherin和Vimentin) [20]。越来越多的证据表明lncRNAs通过调节EMT过程, 在某些肿瘤的发生发展过程中, 发挥促癌基因和抑癌基因的作用, 参与侵袭和转移过程[21-23]。近年来研究发现, LncRNAs与食管鳞癌也存在着密切的关联, 能够调控EMT进程, 参与食管鳞癌的侵袭和转移[18-20]。
LncRNA TUSC7:也称为边缘系统相关膜蛋白反RNA3(LSAMP antisense RNA 3;LOC285194; LINC00902; LSAMP-AS1; Gene ID: 285194), 由4个外显子组成, 位于染色体3q13.31, 属反义lncRNA。TUSC7于2010年首次发现于骨肉瘤中[15], 作为新的抑癌基因[24], 在多种恶性肿瘤组织中呈低表达, 与恶性肿瘤的临床病例特征及预后密切相关[25]。此前已有报道称TUSC7在胰腺导管腺癌[26]、骨肉瘤[27]、胃癌[28]和非小细胞肺癌(NSCLC) [24]等肿瘤组织中表达下调, 与淋巴转移和肿瘤体积及患者生存期负相关, 并参与调控化疗耐药[29]。另有研究显示TUSC7可作为ceRNA分别与miRNA (如miR-10a, miR-224, miR-23b, miR-211等)互作, 调控EMT进程, 参与肿瘤包括胃癌[28], 胶质瘤[30], 肝癌[31]的转移。在肝癌细胞中, 下调的TUSC7可促进肝癌细胞增殖和迁移, 进一步研究发现TUSC7/ miR-10a竞争性调控EPHA4, 影响EMT进程[31]。本课题组发现过表达TUSC7可显著下调MMP-9, N-cadherin和Vimentin蛋白表达量而上调E-cadherin蛋白表达量, 细胞迁移和侵袭率降低; 反之, 敲低TUSC7后, MMP-9, N-cadherin和Vimentin蛋白表达上调, E-cadherin蛋白表达量下调, 细胞迁移和侵袭率升高。以上研究提示lncRNA-TUSC7参与食管鳞癌细胞侵袭和转移, 过表达TUSC7对食管鳞癌细胞EMT有逆转作用。
随着非编码RNA作为肿瘤标志物的出现, 提示在肿瘤发生发展过程中, 非编码RNA比受体、蛋白等出现得更早, 而这对肿瘤的诊断尤为重要。相对肿瘤组织样本, 血清的来源及外周血生物标志物的检测方便且微创。多数lncRNA在肿瘤转移患者的血液中含量丰富, 能被检测并能稳定存在, 因此血清/血浆中的lncRNA作为肿瘤诊断指标具有明显的优势。目前, lncRNA作为循环肿瘤标志物在食管鳞癌的研究尚处于初级阶段。本研究采用RT-qPCR技术比较分析了食管鳞癌组与健康对照组血清中TUSC7的表达, 结果显示, 与健康对照组相比, 食管鳞癌患者血清中lncRNA-TUSC7表达水平显著下调。且其表达水平与临床病理参数中患者年龄、性别及吸烟饮酒史等无相关性, 而与患者局部浸润程度、淋巴结转移及TNM分期相关。总之, 血清中TUSC7可能参与食管鳞癌的侵润和转移, 有望成为ESCC新的循环肿瘤生物标记物并为ESCC的治疗提供新的靶点。但是下调TUSC7诱导EMT促进食管鳞癌转移和侵袭在ESCC发病过程中具体的分子机制和作用途径仍不是很清楚, 这有待进一步探索。
| [1] |
Codipilly DC, Qin Y, Dawsey SM, et al. Screening for esophageal squamous cell carcinoma: recent advances[J]. Gastrointest Endosc, 2018, 88(3): 413-26. DOI:10.1016/j.gie.2018.04.2352 |
| [2] |
Zeng H, Zheng R, Zhang S, et al. Esophageal cancer statistics in China, 2011: Estimates based on 177 cancer registries[J]. Thorac Cancer, 2016, 7(2): 232-7. |
| [3] |
Demeester SR. Epidemiology and biology of esophageal cancer[J]. Gastrointest Cancer Res, 2009, 3(2 Suppl): S2-5. |
| [4] |
常卫东, 王丽, 赵盼盼, 等. lncRNA在食管鳞癌中的研究进展[J]. 生物学杂志, 2017, 34(5): 89-92. DOI:10.3969/j.issn.2095-1736.2017.05.089 |
| [5] |
Zuckerwise L, Li J, Lu L, et al. H19 long noncoding RNA alters trophoblast cell migration and invasion by regulating TβR3 in placentae with fetal growth restriction[J]. Oncotarget, 2016, 7(25): 38398-407. DOI:10.18632/oncotarget.9534 |
| [6] |
Zhu H, Liyi Z, Shi Y, et al. LncRNA16 is a potential biomarker for diagnosis of early-stage lung cancer that promotes cell proliferation by regulating the cell cycle[J]. Oncotarget, 2016, 8(5): 7867-77. |
| [7] |
Liu D, Li Y, Luo G, et al. LncRNA SPRY4-IT1 sponges miR-101-3p to promote proliferation and metastasis of bladder cancer cells through up-regulating EZH2[J]. Cancer Lett, 388: 281-91.
|
| [8] |
Zhang JF, Sun ZS, Zhang QF, et al. Expression of long noncoding RNA STCAT3 in gastric cancer tissues and its effect on malignant phenotype of gastric cancer cells[J]. 中华医学杂志, 2016, 96(46): 3735-40. |
| [9] |
Qian W, Lei Y, Xin H, et al. Upregulated NNT-AS1, a long noncoding RNA, contributes to proliferation and migration of colorectal cancer cells in vitro and in vivo[J]. Oncotarget, 2016, 8(2): 3441-53. |
| [10] |
Lv D, Sun R, Yu Q, et al. The long non-coding RNA maternally expressed gene 3 activates p53 and is downregulated in esophageal squamous cell cancer[J]. Tumor Biol, 2016, 37. |
| [11] |
Tang Q, Hann S. HOTAIR: An oncogenic long non-coding RNA in human cancer[J]. Cell Physiol Biochem, 2018, 47(3): 893-913. DOI:10.1159/000490131 |
| [12] |
Yoshimoto R, Mayeda A, Yoshida M, et al. MALAT1 long noncoding RNA in cancer[J]. BBA-Gene Regulat Mechan, 2016, 1859(1): 192-9. |
| [13] |
Tong YS, Zhou XL, Wang XW, et al. Association of decreased expression of long non-coding RNA LOC285194 with chemoradiotherapy resistance and poor prognosis in esophageal squamous cell carcinoma[J]. J Transl Med, 2014, 12(1): 233. DOI:10.1186/s12967-014-0233-y |
| [14] |
Gao T, He B, Pan Y, et al. Long non-coding RNA 91H contributes to the occurrence and progression of esophageal squamous cell carcinoma by inhibiting IGF2 expression[J]. Chin J Clin Lab Sci, 2015, 54(5): 359-67. |
| [15] |
Ivan P, Adam S, Durbin AD, et al. Recurrent focal copy-number changes and loss of heterozygosity implicate two noncoding RNAs and one tumor suppressor gene at chromosome 3q13.31 in osteosarcoma[J]. Cancer Res, 2010, 70(1): 160-71.
|
| [16] |
Qi P, Xu MD, Ni SJ, et al. Low expression of LOC285194 is associated with poor prognosis in colorectal cancer[J]. J Transl Med, 2013, 11(1): 1-7. DOI:10.1186/1479-5876-11-1 |
| [17] |
Qian L, Jianguo H, Nanjiang Z, et al. LncRNA loc285194 is a p53- regulated tumor suppressor[J]. Nucleic Acids Res, 2013, 41(9): 4976-87. DOI:10.1093/nar/gkt182 |
| [18] |
Ren K, Li Y, Lu H, et al. Long noncoding RNA HOTAIR controls cell cycle by functioning as a competing endogenous RNA in esophageal squamous cell carcinoma[J]. Transl Oncol, 2016, 9(6): 489-97. DOI:10.1016/j.tranon.2016.09.005 |
| [19] |
Chen X, Han H, Li Y, et al. Upregulation of long noncoding RNA HOTTIP promotes metastasis of esophageal squamous cell carcinoma via induction of EMT[J]. Oncogene, 2016, 7(51): 84480. |
| [20] |
Xu Q, Deng F, Qin Y, et al. Long non-coding RNA regulation of epithelial-mesenchymal transition in cancer metastasis[J]. Cell Death Dis, 2016, 7(6): e2254. DOI:10.1038/cddis.2016.149 |
| [21] |
Chen FJ, Sun M, Li SQ, et al. Upregulation of the long non-coding RNA HOTAIR promotes esophageal squamous cell carcinoma metastasis and poor prognosis[J]. Mol Carcinogen, 2013, 52(11): 908-15. DOI:10.1002/mc.21944 |
| [22] |
Jing Z, Yongchao L, Wenhong Z, et al. Long non-coding RNA Linc00152 is involved in cell cycle arrest, apoptosis, epithelial to mesenchymal transition, cell migration and invasion in gastric cancer[J]. Cell Cycle, 2015, 14(19): 3112-23. DOI:10.1080/15384101.2015.1078034 |
| [23] |
Yang Y, Li H, Hou S, et al. The noncoding RNA expression profile and the effect of lncRNA AK126698 on cisplatin resistance in nonsmall-cell lung cancer cell[J]. Plos One, 2013, 8(5): e65309. DOI:10.1371/journal.pone.0065309 |
| [24] |
Li N, Shi K, Li W. TUSC7: A novel tumor suppressor long noncoding RNA in human cancers[J]. J Cell Physiol, 2018, 233(9): 6401-7. DOI:10.1002/jcp.26544 |
| [25] |
Wang Z, Wang Q, Wang J, et al. Overexpression of long non-coding RNA loc285194 suppresses cell proliferation and migration in hepatocellular carcinoma[J]. Int J Cancer, 2017, 10: 2521-9. |
| [26] |
Ding YC, Yu W, Ma C, et al. Expression of long non-coding RNA LOC285194 and its prognostic significance in human pancreatic ductal adenocarcinoma[J]. Int J Clin Exp Pathol, 2014, 7(11): 8065-8070. |
| [27] |
Cong M, Jing R, Li ZZ. Long noncoding RNA tumor suppressor candidate 7 functions as a tumor suppressor and inhibits proliferation in osteosarcoma[J]. Tumor Biol, 2016, 37: 9441-50. DOI:10.1007/s13277-015-4414-y |
| [28] |
Qi P, Xu MD, Shen XH, et al. Reciprocal repression between TUSC7 and miR-23b in gastric cancer[J]. Int J Cancer, 2018, 137(6): 1269-78. |
| [29] |
Chang ZW, Jia YX, Zhang WJ, et al. LncRNA-TUSC7/miR-224 affected chemotherapy resistance of esophageal squamous cell carcinoma by competitively regulating DESC1[J]. J Exp ClinCancers, 2018, 37(1): 56. |
| [30] |
Chao S, Yan G, Yang H, et al. Long Non-coding RNA TUSC7, a target of miR-23b, Plays tumor-suppressing roles in human gliomas[J]. Front Cell Neurosci, 2016, 10: 235. |
| [31] |
Wang Y, Liu Z, Yao B, et al. Long non-coding RNA TUSC7 acts a molecular sponge for miR-10a and suppresses EMT in hepatocellular carcinoma[J]. Tumor Biol, 2016, 37(8): 11429-41. DOI:10.1007/s13277-016-4892-6 |
 2020, Vol. 40
2020, Vol. 40

