2. 广东省医学图像处理重点实验室,广东 广州 510515;
3. 高端医学影像中心,中国科学院上海高等研究院,上海 201210
2. Guangdong Provincial Key Laboratory of Medical Image Processing, Southern Medical University, Guangzhou 510515, China;
3. Medical Imaging Center, Shanghai Advanced Research Institute, Shanghai 201210, China
阿尔茨海默病(AD)是最为常见的一种神经退行性疾病,生理表现为不可逆的神经元死亡和进展性的认知功能障碍[1]。轻度认知障碍(MCI)状态下的患者有较高风险转变为AD。因此,精确地诊断AD患者,尤其是将在一定时间段内可能转化为AD的MCI患者与认知状态稳定的MCI患者精确地区分开,对于病情预估和治疗方案的制定有重要的临床意义。随着计算机视觉领域的发展,将深度学习方法应用于脑部结构磁共振图像(sMRI)中实现AD相关疾病的诊断已经成为当前的研究热点。由于大脑中的许多区域不会因AD的影响而发生显著的结构改变,因此目前基于深度学习的诊断方法通常需要对大脑sMRI中受AD影响较为明显的区域进行定位。有研究提出了一种基于特征点的深度多示例学习框架来预测AD及其前驱症状[2];另有研究提出了一种分层全卷积网络模型,用来同时定位疾病相关区域和预测疾病类别[3];有文献将不同尺度的图像块送入卷积神经网络中,以预测AD相关的临床评价指标[4]。
目前取得较好效果的基于深度学习的AD诊断策略基本包括两步:从局部图像块提取特征;再将提取到的特征简单组合,进行进一步特征处理。但是,目前这些诊断策略的共同缺点是忽略不同图像块之间的拓扑关系。AD患者相比正常认知人群,其大脑sMRI中的图像块提取位置之间的空间位置关系必然是不同的。因此,这种拓扑结构可以提供大脑的结构信息,与图像的局部纹理信息结合,可以为AD的诊断提供更加可靠的依据。
为进一步提升AD疾病的自动诊断效果,本文提出了一种耦合的卷积-图卷积神经网络模型(CCGCN),利用大脑的局部纹理信息和全脑的拓扑信息来获得AD相关疾病的诊断结果。CCGCN模型首先从图像的局部区域提取纹理特征,再根据局部区域的空间拓扑结构进一步整合、处理特征。具体先使用组间比较,从大脑sMRI中获取疾病相关的关键区域;接着利用图像块层级特征提取网络从关键区域提取疾病相关的纹理特征;下一步,我们以图结构对各个关键区域间的空间位置关系进行建模,并以此为依据,构造全脑层级图卷积神经网络(b-GCN),对图像块层级特征提取网络提取到的疾病相关特征进行进一步融合,最终输出疾病标签的预测结果。CCGCN中的图像块层级特征提取网络和b-GCN分别进行预训练,然后进行组合,以端到端的训练方式进行微调,从而促进特征之间的局部-全局交流,确保模型参数不会陷入局部最优。
1 材料和方法 1.1 数据来源及预处理利用公开数据集The Alzhemer's Disease Neuroimaging Initiative(ADNI)[5],包括ADNI-1和ADNI-2数据集。根据ADNI数据库提供的信息[5],MMSE[23]评分在20~26分、同时CDR[24]评分为0.5或1的被试的疾病标签标记为AD组;MMSE评分在24~30分且CDR评分为0、无抑郁症状、无认知障碍、无焦虑症状的被试疾病标签标记为NC组;参考已有研究[2]的方法,根据MCI受试者是否会在首次扫描之后36月内转化为AD,进一步将ADNI-1和ADNI-2数据集中的MCI被试划分为稳定型MCI(即sMCI)和进行型MCI (pMCI)。
ADNI-1数据集中共有199例AD,167例pMCI,226例sMCI和229例NC被试;ADNI-2数据集中共有159例AD,38例pMCI,239例sMCI和200例NC被试,需注意的是,同时在ADNI-1和ADNI-2数据集中出现的被试已从ADNI-2数据集中移除。取以上被试的T1磁共振图像,作为实验数据。ADNI-1中的数据由1.5 T磁共振扫描仪获取,而ADNI-2的数据由3 T扫描仪获取。上述数据集中的被试分布见表 1。
| 表 1 ADNI数据集中的被试分布情况 Tab.1 Demographic information of the subjects included in the studied datasets |
为了更好地评估模型的性能,我们使用ADNI-1数据集进行训练,并使用ADNI-2数据集作为单独的测试集。特别在ADNI-1中,随机选择20%的数据来作为验证集,其余数据用作训练集。所有被试的T1 sMRI数据由一套标准的前处理流程进行处理,每个原始图像经过重采样规范化至1×1×1的体素和256×256×256的图像,然后进行去头骨、图像非均匀偏差校正[6],最后使用Colin27模板[7]作为参考,将每个图像对齐至相同方向。
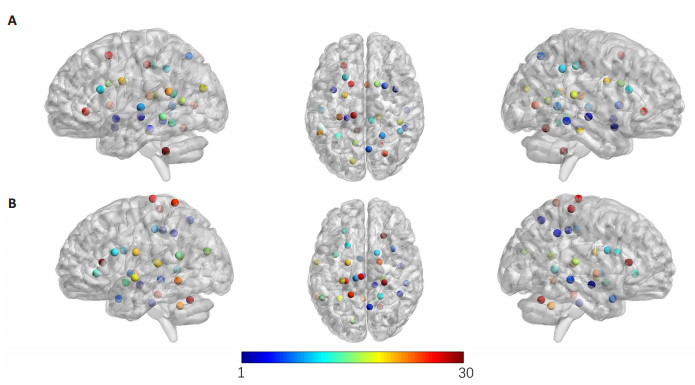
1.2 关键区域获取为定位大脑sMRI中对疾病最具区分度的区域,利用组间比较来获取AD组与NC组大脑结构最具统计学差异的区域。具体地,利用ANTs配准工具[8],将AD组和NC组经过预处理的大脑图像配准至模板空间,对每幅图像对应的变形场,计算其Jacobian行列式(JD)图像,JD图像中的每个体素值表示该被试在此位置的体积变化情况[9]。接下来,对AD组和NC组被试的JD图像进行体素层面的组间t检验,得到模板空间的P值图。将P值图中的局部极小值定义为AD组与NC组形态结构差异最大的区域。为避免区域之间的重叠,进一步设定空间欧式距离阈值来控制不同区域的距离,本实验设定为20 mm。由于较小的P值代表更显著的组间差异,我们根据P值的排序来选择前30个局部极值点,即疾病相关的特征点,作为关键区域的选取位置,图 1A中已标出30个特征点的位置。将同样的处理流程应用于pMCI组和sMCI组,得到组间差异最显著的30个特征点(图 1B)。值得注意的是,这些特征点的获取仅依赖于训练数据。在Colin27模板空间定义了这些特征点之后,利用逆配准将这些特征点变换回每个训练图像空间。对于新的测试图像,我们使用Zhang等[10]提出的方法实现快速的特征点检测。
|
图 1 30个最具区分度的大脑特征点区域在大脑中的空间位置分布 Fig.1 Distribution of 30 discriminative landmarks in different groups. A: 30 discriminative regions between AD and NC; B: 30 discriminative regions between pMCI and sMCI. |
基于获得的特征点,从每个MR图像中提取多个图像块作为特征表示。与已有文献的研究工作相同[4],以每个特征点为中心,提取24×24×24和48×48×48两个不同尺度的图像块,同时进一步对24×24×24的图像块上采样,使其尺寸达到48×48×48,然后将两个图像块拼接,作为后续模型的两个输入通道。为了减小特征点定位误差产生的影响,每次选取图像块时,对特征点在5× 5×5范围内执行随机抖动。以抖动后的特征点为中心获取图像块。
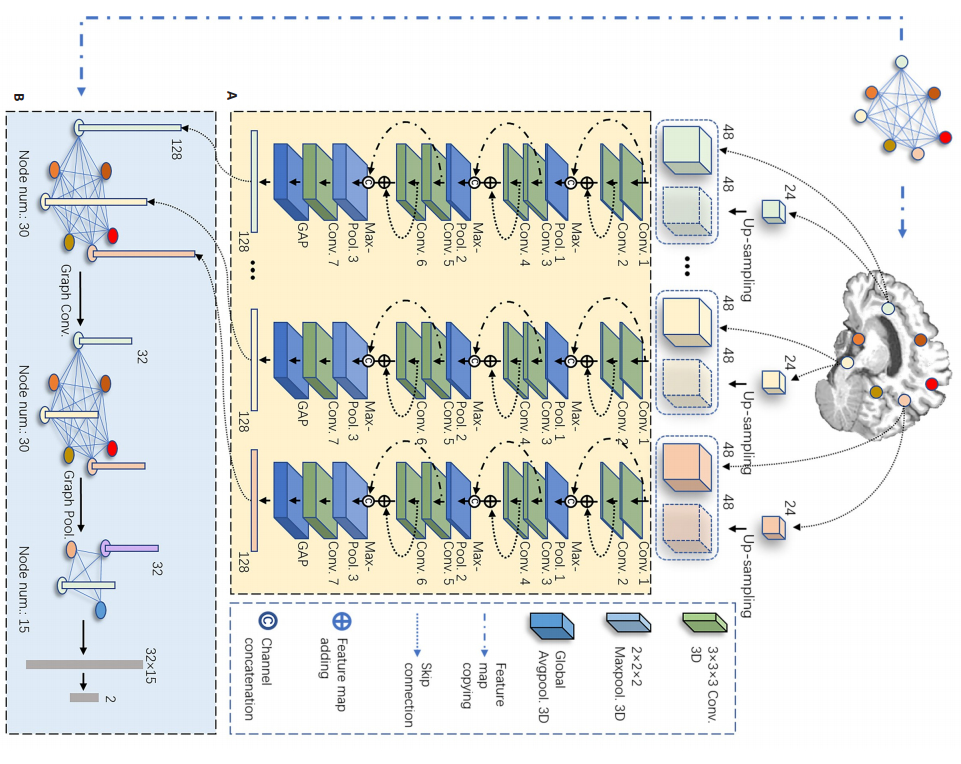
1.3 模型结构我们提出一种CCGCN模型(图 3),用于从不同的图像块中提取和处理疾病相关的特征。该模型主要有两个组成部分:图像块层级特征提取网络、b-GCN。
|
图 3 对于模型结构进行的消融实验结果 Fig.3 Results of the ablation experiments. A: Patch-level feature extraction network; B: Brain-level graph convolutional network. |
图像块层级特征提取网络:其作用是从对应的图像块中提取特征,其内部结构如图 2。网络的输入为两个同尺寸不同尺度的图像块(如前述),输出为标签相关特征向量。为生成标签相关特征,网络对输入图像块采用7个卷积层进行特征提取,其中每个卷积层后均加入批标准化操作和线性整流单元(ReLU)。网络的层间连接采用残差连接[11](以跳层连接和特征图相加实现)和密集连接[12](以特征图复制和按通道连接实现),以促进层间特征交流,避免优化过程中的梯度消失,增强网络的特征提取能力。最后一个卷积层的输出特征图经全局平均池化层[13]压缩为向量,再有全连接层对特征进行进一步的整合处理,最终输出与疾病标签相关的特征向量(本实验每个图像块上提取到的特征向量的维度定义为128维)。
|
图 2 耦合的卷积-图卷积神经网络模型结构 Fig.2 Structure of the coupled convolutional and graph convolutional network. A: Patch-level feature extraction network; B: Brain-level graph convolutional network. |
b-GCN:利用上文提到的特征点空间结构,可以构造图结构G(V, E),其中V为图结构的节点集合,E为图结构的节点间边的集合。将特征点定义为图的节点,节点间欧式距离值定义为图的边,每个特征点(记为k,特征点共有K = 30个)处的图像块提取到的μ维特征向量(μ = 128)定义为相应的节点处的特征向量λk。
由此,我们可以构造节点的特征矩阵H ∈ RK×μ:
| $ H = \left[ {\begin{array}{*{20}{c}} {{{({\lambda ^1})}^T}}\\ {{{({\lambda ^2})}^T}}\\ \vdots \\ {{{({\lambda ^K})}^T}} \end{array}} \right] $ | (1) |
同时,为了描述图结构中的边,即节点的邻接状态,我们定义di, j为第i、j个特征点间的欧式距离,则邻接矩阵A ∈ RK×K可表示为:
| $ A = \left[ {\begin{array}{*{20}{c}} 1 & {} & {\frac{1}{{{d_{1, 2}}}}} & \ldots & {\frac{1}{{{d_{1, K}}}}}\\ {\frac{1}{{{d_{2, 1}}}}} & 1 & {} & {} & {\frac{1}{{{d_{2, K}}}}}\\ {} & \vdots & {} & \ddots & \vdots \\ {\frac{1}{{{d_{K, 1}}}}} & {\frac{1}{{{d_{K, 2}}}}} & {} & \cdots & 1 \end{array}} \right] $ | (2) |
利用节点的特征矩阵和邻接矩阵,我们可以通过大脑中特征点的分布情况,对全脑的拓扑结构进行描述,并利用b-GCN对其进行处理(图 2)。参考Chen等[14]的工作,对于第l层图卷积层,经过图卷积操作输出的特征矩阵Hl+1可以表示为:
| $ {H^{l + 1}} = ReLU({A^l}{H^l}{W^l}) $ | (3) |
其中Wl ∈Rμ×μ'表示图卷积的系数矩阵,包含μ×μ'个可学习参数(本实验中μ' = 32)。ReLU(·)表示线性整流单元,为图卷积操作提供非线性操作。此外,为了进一步对拓扑空间进行自适应建模,参考Ying等[15]的工作,我们为b-GCN添加了图池化层。图池化操作由池化矩阵S∈RK×K'实现,其中K ' = 15。对于第l层池化层,池化后的邻接矩阵Apooll + 1和特征矩阵Hpooll + 1可表示为:
| $ A_{pool}^{l + 1} = {S^T}{A^l}S $ | (4) |
| $ H_{pool}^{l + 1} = {S^T}{H^l} $ | (5) |
经过b-GCN输出的特征矩阵拉伸为1维向量,经一层全连接层处理后,得到疾病标签的预测结果。
1.4 训练方法我们以一种部分-整体策略来训练CCGCN模型,利用从不同特征点位置提取的图像块,首先单独训练每个图像块层级特征提取网络。当图像块层级特征提取网络训练至收敛后,对于每个输入图像,可从图像上每个特征点对应的图像块上获取疾病标签相关的特征向量。接下来,利用这些特征向量,对全脑层级图卷积神经网络进行训练。以上的预训练过程的目的是降低接下来对CCGCN模型进行端到端训练的难度。在预训练结束后,利用预训练得到的参数来初始化CCGCN模型的相应参数,对整个模型执行端到端的训练,以促进各部分间特征交流,避免模型各个组成部分参数的局部最优。训练中,模型的优化采用自适应矩估计方法[16],使用Focal loss[17]作为损失函数,学习率设为10-4。模型的训练及测试均在安装单块NVIDIA GTX Titan Xp 12G显卡的计算机上进行。
1.5 实验为比较本研究提出的方法在AD及其前驱疾病的诊断任务上的有效性,我们在ADNI-2数据集上对比了CCGCN模型与4种现有方法(包括2种常用的机器学习方法和2种目前最为先进的方法)的诊断精度。4种方法分别为:基于感兴趣区域的方法(ROI)[18],基于体素测量的方法(VBM)[19],基于特征点的多示例学习(LDMIL)[2],基于先验信息的全卷积网络模型(wHFCN)[3]。实验在AD/NC诊断任务和pMCI/sMCI诊断任务上,分别对比了以上4种方法与CCGCN的诊断精度、敏感度、特异度以及受试者工作特性曲线下面积(AUC)。
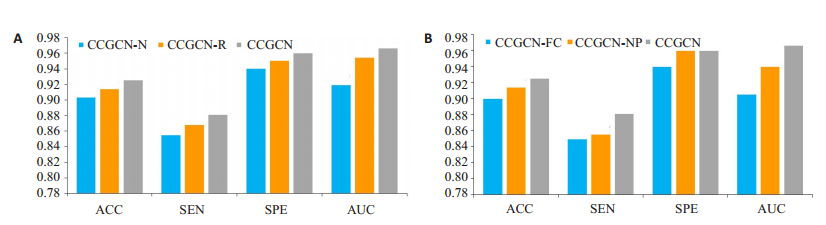
此外,为验证CCGCN模型各个组成部分的有效性,本研究设计了一系列消融实验,主要包括两部分:(1)针对图像块尺度卷积网络的消融实验:目前两种基于深度学习的方法中,一种提出的模型中不包含残差连接和密集连接[2],另一种提出的模型仅在1×1×1卷积操作前后增加了一次残差连接[3]。以上两种方法均未采取有效策略促进卷积层间特征交流。相比常规卷积网络,CCGCN模型的图像块尺度特征提取网络在3×3×3卷积层间添加了残差连接[11]和密集连接[12],以促进特征交流。我们对比了不含残差连接和密集连接的网络模型、包含残差连接的模型、包含残差连接和密集连接的模型(即CCGCN) 3个模型在疾病标签预测任务上的精度。(2)针对全脑尺度图卷积网络的消融实验:与目前直接利用全连接层对特征进行处理的方法不同,本文采用图卷积模型对全脑特征点之间的拓扑结构进行建模,自适应地学习该拓扑结构与疾病标签之间的关联。为验证此方法的有效性,我们对比了CCGCN模型结构和以四层全连接层[2]代替图卷积网络、不包含图池化的图卷积网络两种模型结构在前述4类任务上的效果。
2 结果 2.1 对比实验CCGCN模型与4种现有方法的诊断精度比较结果显示,CCGCN模型在AD/NC诊断任务上取得了最高的诊断精度,以及最大的AUC值(表 2)。
| 表 2 不同方法在AD/NC诊断任务与pMCI/sMCI诊断任务的测试结果 Tab.2 Results of AD/NC and pMCI/sMCI prediction tasks for data from 159 AD, 38 pMCI, 239 sMCI and 200 NC subjects |
针对图像块尺度特征提取网络的消融实验结果显示(图 3A),相比于常规卷积网络,添加残差连接可以实现模型整体性能的提升,在此基础上,增加密集连接可以进一步提升模型各分类性能指标。针对全脑尺度图卷积网络的消融实验结果显示(图 3B),以图卷积神经网络代替传统全连接层,可以提升模型的诊断性能,而增加图池化操作可以实现诊断性能的进一步提升。
3 讨论ADNI-2数据集上的对比实验结果提示,对于AD及其前驱疾病的诊断任务,基于深度学习的3种方法(LDMIL[2],wH-FCN[3]及CCGCN)显著优于基于传统机器学习的两种方法(ROI[18]和VBM[19]);在AD/NC诊断任务上,相比于已有方法,本文提出的CCGCN方法取得了最好的诊断效果,包括最高的ACC和最高的AD敏感度;在pMCI/sMCI诊断任务上,本文提出的CCGCN实现了目前最高的pMCI敏感度。CCGCN在pMCI/ sMCI诊断任务上实现了非常接近目前最先进方法的诊断精度,ACC与目前研究提出的最先进的方法[3]相差0.011),考虑到MCI被试数量有限,特别是pMCI被试,CCGCN在该任务上的优越性需要在更大规模数据集上进一步评估。由于训练集数据ADNI-1由1.5 T磁共振设备采集,而测试数据ADNI-2由3 T磁共振设备采集,因此CCGCN模型在测试数据上实现的良好的分类结果,证明了模型对不同成像条件下采集的数据具有很好的稳定性。
本研究由针对图像块尺度特征提取网络的消融实验结果可知,相比于常规卷积网络,添加残差连接和密集连接,均可以实现模型整体性能的提升。由针对全脑尺度图卷积网络的消融实验结果可知,利用图卷积神经网络模型对特征进行处理,可以在4类任务上取得比全连接层更好的效果,由此证明了利用全脑拓扑结构信息可以为疾病相关预测、年龄预测和性别分类提供依据。由针对全脑尺度图卷积网络的消融实验结果可知,在图卷积网络结构中增加图池化操作,可以使模型性能进一步提升,证明了图池化的图结构重构特性在CCGCN模型框架中的有效性。综上消融实验证明,在我们提出的CCGCN模型中,以图卷积神经网络对大脑拓扑结构进行建模,并以此为依据,对卷积神经网络提取到的特征进行进一步处理,可以实现诊断性能的进一步提升。
相比传统特征提取方法,作为一种自适应特征提取方法,深度学习可以分层次地从原始数据空间提取、融合特征,从而获得对于目标任务的更好的特征表示。大脑sMRI中疾病相关的特征,相对整幅图像而言非常稀疏,因此必须通过预定位,找到疾病影响最为显著的区域,进而使用神经网络(如CNN)在此区域进行特征提取。然而,上述处理方法,相当于将完整的大脑图像视为局部图像块的组合,而忽略了这些图像快的空间位置分布状态,即全脑的拓扑结构,与疾病标签之间的关联。为解决这一问题,更好地利用原始图像所提供的信息,从而获得更好的疾病预测结果,我们将卷积神经网络与图卷积神经网路耦合为一个整体,利用卷积神经网络对局部图像块特征进行抽取、处理,再利用图卷积神经网络,以大脑的空间拓扑结构为依据,对卷积神经网络抽取到的特征进行进一步处理。实验证明本文方法可以提高AD及其前驱症状的诊断效果,且有较好的稳定性,为临床实现AD及其前驱症状的计算机辅助诊断提出了一种新的方法。
为进一步提升疾病的诊断性能,为未来计算机辅助诊断在临床上的应用提供更好的方法,本文提出的方法仍有改进之处。本文方法中,图像块位置的确定过程独立于后续的特征提取和分类过程。考虑到图像块的定位任务与疾病诊断任务之间存在关联,以往的工作[3, 20]均在实验中设计方法,实现了病变区域定位任务和疾病诊断任务的联合优化。未来将参考已有工作,对模型进行调整,使图像块位置定位与疾病诊断任务可以利用其相关性实现共同优化,即将本文提出的耦合思想扩展至任务层面。本文提出的方法旨在对疾病标签进行预测,而未考虑其他因素(如年龄、性别、MMSE、CDR-SB等[21-24])与疾病标签之间的内在联系。作为一种可行方案,未来将在模型结构中嵌入多任务策略,自适应地学习其他因素与疾病标签之间的关系,为进一步提升疾病标签预测任务的精度和稳定性提供一种方法。由于阿尔茨海默病及其前驱症状与多种生理指标相关,如糖代谢、脑功能连接等[25-26],而T1 sMRI主要提供脑的结构信息,增加其他成像模态,如静息态功能磁共振成像、正电子发射计算机断层成像以及扩散张量成像等,可以为疾病诊断提供更多信息,从而进一步提升诊断精度和稳定性。
| [1] |
Jagust W. Vulnerable neural systems and the borderland of brain aging and neurodegeneration[J]. Neuron, 2013, 77(2): 219-34. DOI:10.1016/j.neuron.2013.01.002 |
| [2] |
Liu M, Zhang J, Adeli E, et al. Landmark-based deep multi-instance learning for brain disease diagnosis[J]. Med Image Analysis, 2018, 43(1): 157-68. |
| [3] |
Lian C, Liu M, Zhang J, et al. Hierarchical fully convolutional network for joint atrophy localization and alzheimer's disease diagnosis using structural MRI[J]. IEEE Transactions Pattern Analysis Machine Intellig, 2018, 39(2): 1-10. |
| [4] |
Liu M, Zhang J, Lian C, et al. Weakly supervised deep learning for brain disease prognosis using MRI and incomplete clinical scores[J]. IEEE Transactions Systems Man Cybernetics, 2019, 10(7): 1-12. |
| [5] |
Jack CR, Bernstein MA, Fox NC, et al. The Alzheimer's disease neuroimaging initiative (ADNI): MRI methods[J]. J Magnetic Resonance Imag, 2008, 27(4): 685-91. DOI:10.1002/jmri.21049 |
| [6] |
Sled JG, Zijdenbos AP, Evans AC. A nonparametric method for automatic correction of intensity nonuniformity in MRI data[J]. IEEE Transactions Med Imaging, 1998, 17(1): 87-97. |
| [7] |
Holmes CJ, Hoge RD, Collins L, et al. Enhancement of MR images using registration for signal averaging[J]. J Comp Assisted Tomography, 1998, 22(2): 324-33. |
| [8] |
Avants BB, Tustison NJ, Stauffer M, et al. The insight toolKit image registration framework[J]. Front Neuroinform, 2014, 8(2): 44-9. |
| [9] |
Hua X, Leow AD, Parikshak NN, et al. Tensor-based morphometry as a neuroimaging biomarker for Alzheimer's disease: an MRI study of 676 AD, MCI, and normal subjects[J]. NeuroImage, 2008, 43(3): 458-69. |
| [10] |
Zhang J, Liu M, Shen D. Detecting anatomical landmarks from limited medical imaging data using two-stage task-oriented deep neural networks[J]. IEEE Trans Imag Process, 2017, 26(10): 4753-64. DOI:10.1109/TIP.2017.2721106 |
| [11] |
He K, Zhang X, Ren S, et al. Deep residual learning for image recognition[J]. Conf Comp Vision Pattern Recogn, 2016, 5: 770-8. |
| [12] |
Huang G, Liu Z, Der Maaten LV, et al. Densely connected convolutional networks[J]. Conf Comp Vision Pattern Recogn, 2017, 13: 2261-9. |
| [13] |
Lin M, Chen Q, Yan S, et al. Network In Network[J]. arXiv: Neural Evolution Comp, 2013. |
| [14] |
Chen Z, Wei X, Wang P, et al. Multi-label image recognition with graph convolutional Networks[J]. Conf Comp Vision Pattern Recogn, 2019, 20: 5177-86. |
| [15] |
Ying Z, You J, Morris C, et al. Hierarchical graph representation learning with differentiable pooling[J]. Neural Inform Process Systems, 2018, 9: 4805-15. |
| [16] |
Kingma DP, Ba J. Adam: a method for stochastic optimization[J]. arXiv: Learning, 2014. |
| [17] |
Lin T, Goyal P, Girshick R, et al. Focal loss for dense object detection[J]. IEEE Trans Pattern Anal Mach Intell, 2018, 42(2): 318-27. |
| [18] |
Zhang D, Shen D. Multi-modal multi-task learning for joint prediction of multiple regression and classification variables in Alzheimers disease[J]. Neuroimage, 2012, 59(2): 895-907. DOI:10.1016/j.neuroimage.2011.09.069 |
| [19] |
Baron JC, Chételat G, Desgranges B, et al. In Vivo mapping of gray matter loss with voxel-based morphometry in mild alzheimer's disease[J]. 2001, 14(2): 298-309.
|
| [20] |
Li Q, Xing X, Sun Y, et al. Novel iterative attention focusing strategy for joint pathology localization and prediction of MCI progression[M]. Springer: Med Image Comp Assisted Intervent, 2019: 307-15.
|
| [21] |
Braak H, Thal DR, Ghebremedhin E, et al. Stages of the pathologic process in Alzheimer disease: age categories from 1 to 100 years[J]. J Neuropathol Exp Neurol, 2011, 70(11): 960-9. DOI:10.1097/NEN.0b013e318232a379 |
| [22] |
Mielke MM, Vemuri P, Rocca WA, et al. Clinical epidemiology of Alzheimer's disease: assessing sex and gender differences[J]. Clin Epidemiol, 2014, 6(1): 37-48. |
| [23] |
Chapman KR, Bingcanar H, Alosco ML, et al. Mini mental state examination and logical memory scores for entry into Alzheimer's disease trials[J]. Alzheimer's Res Therapy, 2016, 8(1): 9-19. DOI:10.1186/s13195-016-0176-z |
| [24] |
Obryant S, Lacritz LH, Hall J, et al. Validation of the new interpretive guidelines for the clinical dementia rating scale sum of boxes score in the national Alzheimer's coordinating center database[J]. JAMA Neurology, 2010, 67(6): 746-9. |
| [25] |
Edison P, Archer H, Hinz R, et al. Amyloid, hypometabolism, and cognition in Alzheimer disease: An 11C PIB and 18F FDG PET study[J]. Neurology, 2007, 68(7): 501-8. DOI:10.1212/01.wnl.0000244749.20056.d4 |
| [26] |
Wang L, Zang Y, He Y, et al. Changes in hippocampal connectivity in the early stages of Alzheimer's disease: evidence from resting state fMRI[J]. NeuroImage, 2006, 31(2): 496-504. DOI:10.1016/j.neuroimage.2005.12.033 |
 2020, Vol. 40
2020, Vol. 40

