2. 广东省医学图像处理重点实验室, 广东 广州 510515;
3. 上海联影智能医疗科技有限公司, 上海 200030
2. Guangdong Provincial Key Laboratory of Medical Image Processing, Southern Medical University, Guangzhou 510515, China;
3. Shanghai United Imaging Intelligence Co., Ltd., Shanghai 200030, China
头颈部肿瘤是全球最常见的恶性肿瘤之一[1]。图像引导的放射治疗是治疗头颈部癌症的有效手段, 目前已被广泛用于临床[2]。在头颈部放射治疗计划中, 技术人员首先获得靶区及其周围危及器官(OARs)的位置和边界, 来确定射线照射的剂量和区域。为了尽量避免过量照射对周围的正常组织造成放射性损伤, 提高治疗准确性, 准确勾画危及器官成为关键一步。
头颈部重要器官多且集中, 边界和体积差异较大, 解剖关系复杂, 这些因素增加了头颈部器官分割的挑战性。目前的临床应用中, 普遍通过交互系统手工勾画, 这种方法往往非常耗时, 且手工勾画的主观性较强, 依赖于医生的个人经验, 不同年资的医生之间, 甚至同一医生不同时间的勾画结果一致性不强, 勾画精度差异较大[3-7]。近年来, 研究人员提出了很多解决方法, 基于图谱的分割方法[8-14]将已有标注的模板图像与待分割图像进行非刚性配准, 根据配准得到的形变场, 将模板标注转换得到待分割图像的分割结果。这种方法对于分割形状分布较一致的器官效果较好, 然而由于肿瘤发展侵犯周围组织, 患者个体间器官差异以及与模板图像器官差异过大, 导致配准精度降低, 从而影响分割精确度。为了充分利用模板图像中的先验知识, 在单图谱分割基础上, 全局权重投票, 局部权重投票等方法采用多图谱配准策略, 将不同模板的信息融合后分割图像[15-18], 提高了分割的鲁棒性, 但也会导致器官分割出现解剖结构异变[19]。随着计算机硬件水平的提高, 一些研究将不同的分割算法进行组合来补偿单个算法的弱点[20-24]。如使用统计形状模型得到头颈部淋巴结在形状上的一致性特征, 对基于图谱配准得到的分割结果进行约束, 使得分割得到的器官形状尽量符合实际分布[25]。但在算法构建方面, 多个分割方法信息之间的融合模式以及初始化原则往往需要手动尝试, 容易陷入局部最优。
过去10年中, 深度学习已经成为机器学习中一个突出的研究领域。卷积神经网络(CNN)可通过卷积操作获取图像特征, 更有效地发掘图像中的信息, 因而在图像分析领域得到了广泛应用[26-29]。如Ibragimov等使用二维卷积神经网络, 对CT图像3个切面的图像分别进行训练, 应用到头颈部腮腺分割中[30]。相比于传统算法, 基于卷积神经网络的分割模型免去了人工选择特征的繁琐步骤, 能获得更好的分割性能和泛化能力。然而普遍采用的二维卷积神经网络, 对CT图像中切片之间的空间信息学习不足。基于三维卷积的深度学习网络对上述问题进行了优化, 但也大大增加了硬件消耗, 因此往往都是逐图像块进行处理, 导致模型计算存在冗余, 不易拓展。为了解决上述问题, 本文基于卷积神经网络对头颈部器官分割方法做了进一步的探究。基于目前性能稳定的通用分割网络V-Net, 本文加入SE模块用于提高模型特征学习能力, 同时使用分层次的级联分割方法, 用于改善算法性能。
1 方法概述本研究的主要任务是对CT图像中头颈部器官进行准确分割, 我们的方法在V-Net[31]基础上进行了进一步的优化, 引入了SE(Squeeze-and-Excitation)[32]结构和多尺度分割策略。该任务主要可分为以下步骤:数据预处理、模型训练、模型整合、以及数据测试, 整体结构如图 1所示。
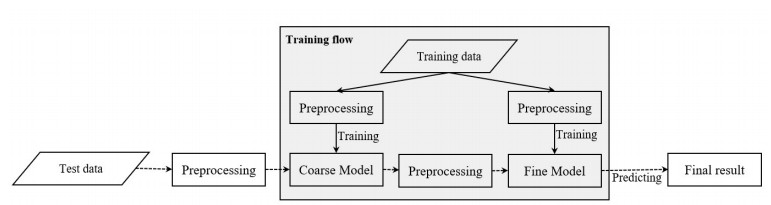
|
图 1 算法流程图 Fig.1 Algorithm flow chart. |
本文实验数据由上海联影智能医疗科技有限公司收集, 共包含50例头部至胸部区域的CT图像。实验数据中CT图像平面尺寸为512×512像素, 空间分辨率分布在0.97~1.2 mm之间, 切片数量分布在100~152层, 平均层厚为3 mm。数据集中22个头颈部器官均由高级医师手工标注, 作为金标准, 用于模型训练和性能评估。
1.2 数据预处理不同医院、设备以及采集参数会导致图像空间分辨率的不同, 使得单位体素中包含的空间信息量存在差异, 直接将不同空间分辨率的图像作为网络输入会使得模型训练产生震荡, 难以达到最优。为了解决这个问题, 我们将图像重采样为统一的分辨率, 确保相同图像尺寸下, 输入网络的图像包含的空间信息相似, 从而加快模型的收敛速度。具体的, 我们在粗分割阶段, 将图像重采样为较粗分辨率, 突出器官的结构、位置信息; 在细分割阶段, 将图像重采样为与原始图像接近的细分辨率, 尽可能保留图像纹理、灰度等细节信息。
为了增强头颈部器官之间的对比度, 我们对图像灰度值进行了标准化处理。基于待分割器官的灰度分布范围, 我们选择窗位为50 Hu, 窗宽为250 Hu, 将这一范围内CT图像灰值归一化至[-1, 1], 灰度值小于或大于这一范围的, 分别设为-1和1。
本文中使用三维卷积, 相对于二维卷积网络, 参数量显著增加, 考虑到GPU显存的限制, 在训练阶段, 我们从整个图像中随机采样出96×96×96大小的图像块作为网络输入, 以减少神经网络迭代过程中的计算量。
1.3 模型 1.3.1 基本结构基于深度学习的医学图像分割中, UNet[33]一经提出, 就表现出了优秀性能。U-Net是基于二维卷积的分割网络, 可对医学图像逐切片进行分割, 然后通过空间堆叠, 得到最终分割结果。后来在此基础上提出的V-Net, 采用三维卷积来引入更多空间信息。与临床治疗中, 资深医师观察感兴趣区域周围的多张切片再做判断的思路契合, 提升了算法性能[34]。
我们的网络架构基于V-Net, 由收缩通路和扩张通路组成, 呈对称结构, 所有的卷积核均为三维卷积。网络结构如图 2所示。相对于U-Net, V-Net将残差结构[35]引入卷积块(图 4A)。每个残差结构由两条路径组成, 第1条路径通过卷积层(卷积、批归一化、Relu组成的基本单元)对输入特征图进行一系列特征学习, 第2条路径为输入特征图的恒等映射。两条路径在最后相加作为输出, 保证了网络训练中梯度可以不间断的流动, 即使在非常深的网络中也可以更新参数。在残差模块(Residual Block)的基础上, 我们显式的建模特征图谱通道之间的相互依赖关系(图 4B), 在残差块卷积层结束后使用SE模块对每个通道的特征响应强度进行学习, 得到每个通道的重要程度, 自适应的增强有效特征的通道权重, 从而提高模块的表达能力。
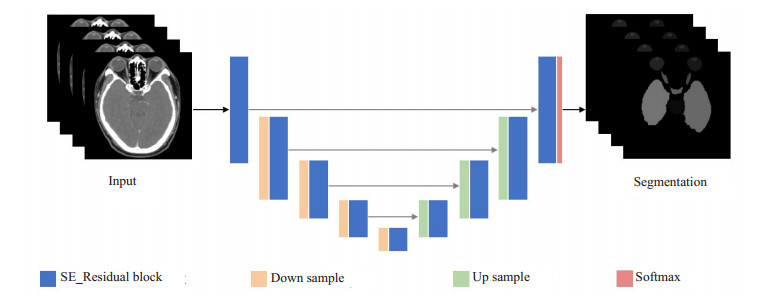
|
图 2 网络基本结构 Fig.2 Structure of the network. |
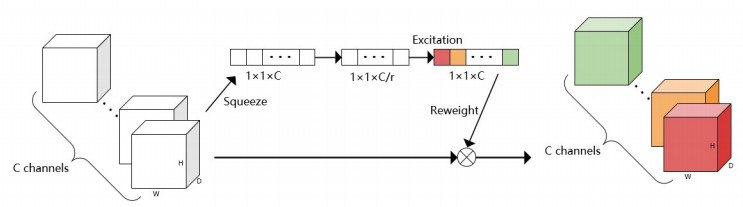
|
图 3 SE模块 Fig.3 The SE module. |
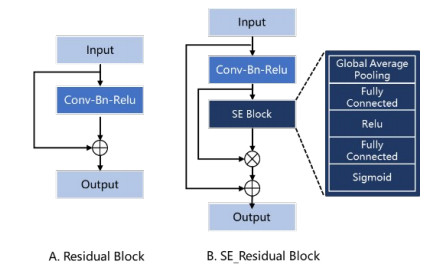
|
图 4 残差模块优化前后结构对比 Fig.4 Structure comparison before and after optimization of the Residual Block. A: Original Residual Block; B: Residual Block combined with SE Block. |
整个网络收缩通路包括5层SE_Residual Block, 卷积块之间由2×2×2大小的卷积层对特征图进行下采样。每进行1次下采样后, SE_Residual Block中增加一组卷积层进行特征提取。SE_Residual Block中最大卷积层个数为3组。扩张通路也包含5个SE_Residual Block, 通过2×2×2反卷积层上采样后连接。收缩通路与扩张通路之间加入跳跃连接, 补充卷积过程中丢失的细节信息, 从而提高分割精度。
1.3.2 SE模块在深度学习模型中,很多方法通过在网络的空间结构和深度上的改进来提升网络的性能。SE模块通过考虑特征通道之间的关系, 更为显式的建立特征通道之间的相互关系, 增加整个网络对特征的选择和捕捉能力, 从而加快网络的训练速度。在SE模块中, 首先通过全局平均池化, 将输入的特征图逐通道压缩至1×1大小的向量(图 3、图 4B)。加入全连接层和激活层, 引入非线性操作后, 再将各个向量归一化至(0, 1)作为每个特征通道对当前任务的重要程度, 最后将学得的权重与原始特征图相乘。在SE模块中包含了两个全连接操作, 中间加入Relu激活操作, 对向量维度分别进行了压缩和还原, 这里r代表通道压缩率, 以此增加模块的复杂性, 同时减少了模块参数量, 通过实验对比, 我们设r=32。
在Residual Block中加入SE模块, 作为SE_Residual Block, 对残差卷积得到的特征图在特征通道层面进行建模, 增加对分割任务更相关的特征权重来提升模型的特征表达能力(图 4B)。
1.3.3 多尺度策略现有的基于深度学习的分割算法都是在单一尺度上进行处理的。而很多医学影像数据(CT、MR)往往需要非常大的计算量, 考虑到目前的硬件资源, 直接将整个图像作为网络的输入是不现实的。目前常见的解决方法多是基于二维的, 将图像逐切片送入二维神经网络进行分割, 最后将分割结果进行堆叠, 得到整个图像的结果; 或者采用滑窗法从图像中采样出一些更小的图像块输入网络进行处理, 最后将各个小图像块的结果组合。这些方法往往不能获取较全面的上下文信息, 测试时间也随之增加。同时基于图像块的处理方法, 位于图像块边缘的分割结果组合后会不够平滑。也有一些方法直接将整个图像下采样到一个较粗的分辨率上进行分割, 然后上采样回原图像尺寸, 这种方法导致分割边缘出现锯齿状现象, 损失图像的细节。
针对上述问题, 本文提出了多尺度分割策略, 将分割任务分为定位和精分割两个子模型, 图像在不同尺度下重采样后分别输入子模型进行训练。在粗尺度定位阶段, 为了提高子模型对器官空间解剖位置的敏感性, 降低细节信息的干扰, 如灰度、纹理等, 输入图像块分辨率重采样为6 mm×6 mm×6 mm。在细尺度精分割阶段, 为了对已定位出的器官进行更加精细的分割, 尤其是对器官边界的准确区分, 输入图像块分辨率重采样为与原始图像接近的分辨率。由于训练阶段两个子模型是分开训练, 为了确保精分割模型仅仅对待分割器官的属性进行学习, 我们仅从待分割器官对应区域内随机采样得到输入图像块作为精分割模型输入。
在应用时, 两个单独训练的模型进行级联, 端到端得到输入图像的分割结果。测试图像首先通过粗尺度网络得到初始分割结果, 提取最大连通域去除分割离散点后, 得到分割结果的边界框, 再将边界框的边缘扩增10 mm, 确保待分割器官被完全包含在内(图 5)。根据边界框对应的位置, 将原始测试图像中截取到的相应区域经过预处理输入细尺度分割网络, 得到的精细分割结果重采样回原图分辨率后, 作为最终的分割结果。由于粗分割阶段仅在较低分辨率的图像上进行定位, 而细分割网络仅在由粗分割网络得到的感兴趣区域上进行, 因而我们的分割框架可以在有限的硬件资源下对整图进行快速测试, 同时不损失精度。
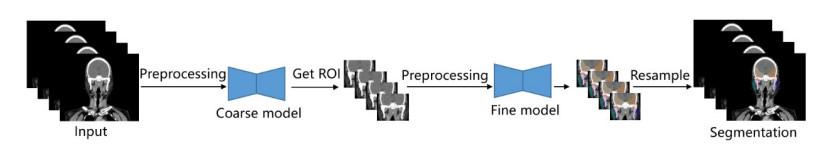
|
图 5 应用流程 Fig.5 Inference flow. |
医学图像分割的本质是对每一个体素进行分类, 在基于深度学习的分割方法中, 通常将交叉熵(Cross Entropy, CE)作为损失函数来优化网络。多类分割的交叉熵损失函数如下:
| $ Los{s_{CE}}{\rm{ = }} - \frac{1}{C}\sum\limits_{c = 1}^C {\sum {_i^M\begin{array}{*{20}{c}} {{g_c}\left( i \right)\log \left( {{p_c}\left( i \right)} \right) + }\\ {\left( {1 - {g_c}\left( i \right)} \right)\log \left( {1 - {p_c}\left( i \right)} \right)} \end{array}} } $ | (1) |
在上式中M表示全图的像素总数目, C表示类别数目, gc(i)∈{0, 1}表示金标准中体素i是否为c类的二值化结果, pc(i)∈[0, 1]为模型预测体素i为c类的概率。在医学器官分割中, 很多器官体积较小, 只占据了整个图像的很小部分, 类别不平衡现象非常普遍, 且训练样本中会存在大量易于分类的背景体素中。直接引入交叉熵作为损失函数, 会导致网络训练中过多的考虑背景体素的分类精度, 前景体素的训练损失会很容易被忽略, 易于分类的背景体素的损失将会主导网络的优化方向。
为了解决这个问题, 我们加入Dice作为损失函数。Dice系数(DSC)通过计算图像之间的重叠度来评估其相似性。它被广泛用于医学图像分割结果的评价。与交叉熵相比, DSC只计算前景体素, 而不考虑整个图像中有多少背景体素, 作为损失函数来优化分割网络可避免类别不平衡问题。Dice损失函数公式如下:
| $ Los{s_{Dice}}{\rm{ = }}\frac{1}{C}\sum\limits_{c = 1}^C {\left( {1 - \frac{{2\sum\limits_i^M {{h_c}\left( i \right){g_c}\left( i \right)} }}{{\sum\limits_i^M {{h_c}\left( i \right)} + \sum\limits_i^M {{g_c}\left( i \right)} }}} \right)} $ | (2) |
其中, M为输入图像的像素总数目, hc(i)∈{0, 1}表示在体素i处网络预测为类别c的二值化结果, gc(i) ∈{0, 1}定义与式(1)中相同。
在本研究中, 我们将交叉熵与Dice系数结合作为网络的损失函数, 对网络进行优化。其数学公式如下, 其中λ1=1, λ2=1:
| $ Loss = {\lambda _1}Los{s_{CE}} + {\lambda _2}Los{s_{Dice}} $ | (3) |
实验数据集中每个器官的类别标签均不同, 对称器官(如左右眼球)也被标注为不同的类别, 即每个类别都只有一个连通域, 因此, 我们对网络输出结果逐类别提取最大连通域, 从而消除可能存在的孤立分割。
1.6 实验细节使用多尺度策略在粗细两个尺度下分别训练卷积神经网络。具体的, 在粗尺度下, 图像分辨率重采样为6 mm×6 mm×6 mm, 然后在全图中随机采样出96×96× 96体素个数的图像块输入粗尺度分割网络进行训练; 在细尺度下, 将图像分辨率重采样为1 mm×1 mm×1 mm, 随后在标注区域内随机采样出96×96×96体素个数的图像块输入细尺度分割网络进行训练。模型运行在24 GB Nvidia TitanX GPU, 使用PyTorch深度学习框架。模型训练中, 学习率设为10-4, 批大小为12, 损失函数的优化方法为Adam算法[36]。
2 结果本文从实验数据中每次选取40例作为训练集, 10例作为测试集, 进行五折交叉验证实验, 并使用Dice系数来评估不同网络结构、单尺度网络和多尺度网络的分割性能。
2.1 网络架构比较为了比较本文提出的SE_Residual Block在头颈部分割任务上的有效性, 我们与使用Residual Block的VNet模型做了性能对比, 各个器官的实验结果如表 1所示。可以看到, 加入Residual Block的网络模型, 平均分割精度比V-Net网络提升了2.2%。在单尺度网络中, 基于SE_Residual Block的网络也能比V-Net网络获得更好的分割性能(表 2)。
| 表 1 不同网络结构下的各个器官分割结果的Dice系数比较 Tab.1 Comparison of Dice coefficients of individual organ segmentation results under different network structures |
| 表 2 不同尺度网络以及不同网络结构下的分割性能比较 Tab.2 Comparison of segmentation performance under different scale networks and different network structures |
本文分别使用单尺度、多尺度策略, 在两种网络结构上进行试验比较。在单尺度网络中, 输入图像首先重采样至体素大小为1 mm×1 mm×1 mm, 再从中随机采样出图像块输入网络进行训练(与多尺度网络输入尺寸相同)。在模型应用阶段, 由于显存限制, 本文使用滑窗法从测试图像中依次采样出200×200×200体素个数的图像块作为网络输入, 并在测试图像块各个方向扩充10 mm, 用于补充图像块周围的空间信息。具体性能比较如表 2所示, 实验结果表明:(1)同一尺度下, 加入SE_Residual Block的网络分割精度均优于V-Net网络; (2)基于不同的网络结构, 采用多尺度结构的网络分割精度均优于单尺度结构; (3)基于多尺度策略的分割算法可降低测试时间和显存消耗。
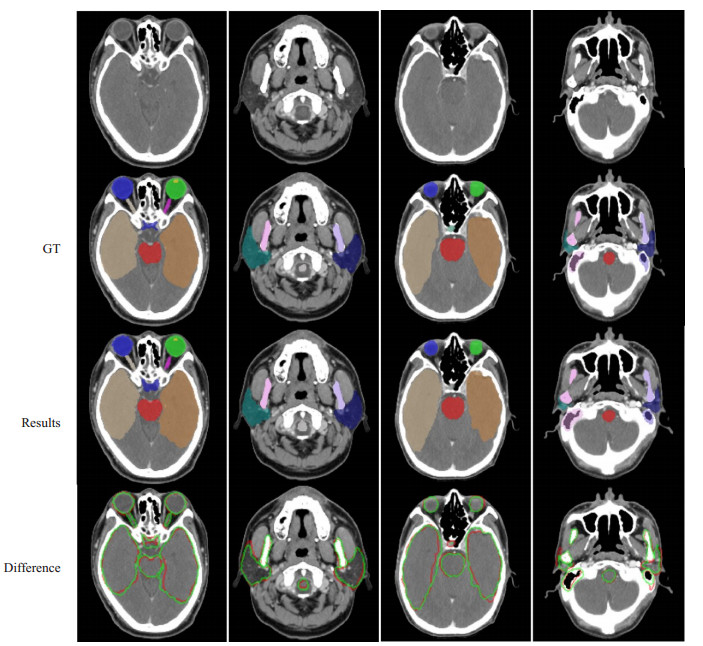
|
图 6 基于本文方法的实验结果 Fig.6 Segmentation results using the proposed method. The first row is the original image; the second is the ground-truth; the third is our results; the fourth line shows the difference between the segmentation result and the ground-truth, in which the white outline represents the ground-truth and the green outline represents the result. |
近些年绝大部分有关头颈部危及器官分割的研究都是基于MICCAI 2015头颈器官自动分割挑战赛提供的公开数据集进行验证以及性能评估, 但这个数据集仅仅包含双侧腮腺、双侧视神经和双侧下颌下腺在内的9个器官标注[37]。鼻咽癌靶区周围解剖结构复杂, 组织器官密集且形状体积差异较大, 实际的放射治疗中, 为了尽可能降低射线对患者的损害, 需要对靶区周围的数十个危及器官进行勾画。因此仅仅基于以上数据集中的9个器官标注进行研究评估, 是远远不够的。基于以上数据集有学者对待分割的多个器官建立活动轮廓模型, 夺得MICCAI 2015头颈器官分割挑战赛的冠军[24]; Ren等[29]人将分割不同器官的网络模型交错连接以便互相指导, 用于分割头颈部体积较小的组织。总体来说, 这个数据集器官标注较少, 不太适用于临床应用需求较高的算法研究。
也有一部分基于非公开数据集的头颈部危及器官分割研究。数据集的不一致导致不同算法之间的分割精度比较参考性不大。Tam等[23]使用多输出支持向量回归算法在收集的56例数据的13个器官上进行了研究评估。在医学图像分割领域, V-Net作为经典分割模型, 仍然具有非常鲁邦的性能, 本文与基于V-Net分割的方法进行比较。本文提出的方法对模型分割性能的提升是有效的。基于本文方法得到的分割结果对于不同形状、大小的器官都实现了成功分割, 尤其对于体积只有不到100体素个数的晶状体, 也实现了准确标注。同时也保留了视交叉、视神经等解剖结构特异性较大器官的结构特点。
器官分割的效率极大影响了放疗计划的实施, 决定了临床治疗效率。Ren等[29]使用卷积神经网络分割一例患者双侧视神经和视交叉的算法需要15 min。而本文粗细尺度下仅使用一个模型进行多器官分割训练, 在应用过程中, 通过多尺度策略逐步去除图像中的冗余信息, 降低算法计算量, 如表 2所示, 本文提出的方法可在3 s内完成22个危及器官的准确分割。
本文的研究中还存在一些提升空间和不足:(1)本文的网络输入都是将输入图像重采样为各个方向上体素大小一致的图像块, 这保证了训练中输入网络的空间信息量是相似的。然而医学图像层厚与切片内分辨率差异往往过大, 使用各向同性的图像块使得沿切片方向空间信息存在冗余, 也增大了模型的计算量。后续工作中将进一步研究调整网络卷积方式, 使用各向异性图像块作为网络输入, 减小网络的参数量; (2)本文在粗尺度分割阶段, 对图像进行了下采样, 这样降低了图像的大小, 但单独分割体积较小的器官时, 粗尺度阶段的预处理操作会导致定位失败。本文中头颈部器官分布集中, 体积较小的器官都位于头颈部器官内部, 因此, 粗分割结果得到的定位区域中也包含这些小尺寸器官, 对分割结果没有影响。这也启发我们在小器官的分割中, 可以将其周围的大器官作为先验信息用于定位, 从而增加模型分割的鲁棒性。
综上所述, 本文提出的方法能够提升现有方法的分割精度, 并且该方法速度更快, 可在3 s内从头颈部CT中分割出22个危及器官。有了更多的部位的训练数据, 本文方法可灵活拓展用于全身器官分割, 从而帮助医生提高临床实践的准确率和效率。
| [1] |
Torre LA, Bray F, Siegel RL, et al. Global cancer statistics, 2012[J]. CA Cancer J Clin, 2015, 65(2): 87-108. |
| [2] |
Dawson LA, Sharpe MB. Image-guided radiotherapy:rationale, benefits, and limitations[J]. Lancet Oncol, 2006, 7(10): 848-58. DOI:10.1016/S1470-2045(06)70904-4 |
| [3] |
Feng MU, Demiroz C, Vineberg KA, et al. Intra-observer variability of organs at risk for head and neck cancer:geometric and dosimetric consequences[J]. Int J Radiat Oncol Biol Phys, 2010, 78(3): S444-5. |
| [4] |
Hardcastle N, Tomé WA, Cannon DM. A multi-institution evaluation of deformable image registration algorithms for automatic organ delineation in adaptive head and neck radiotherapy[J]. Radiat Oncol, 2012, 7(1): 90. DOI:10.1186/1748-717X-7-90 |
| [5] |
Li XA, Tai A, Arthur DW, et al. Variability of target and normal structure delineation for breast cancer radiotherapy:an RTOG multi-institutional and multiobserver study[J]. Int J Radiat Oncol Biol Phys, 2009, 73(3): 944-51. DOI:10.1016/j.ijrobp.2008.10.034 |
| [6] |
Macchia ML, Fellin F, Amichetti M. Systematic evaluation of three different commercial software solutions for automatic segmentation for adaptive therapy in head-and-neck, prostate and pleural cancer[J]. Radiat Oncol, 2012, 7(1): 160. DOI:10.1186/1748-717X-7-160 |
| [7] |
Nelms BE, Tomé WA, Robinson G, et al. Variations in the contouring of organs at risk:Test case from a patient with oropharyngeal cancer[J]. Int J Radiat Oncol Biol Phys, 2012, 82(1): 368-78. DOI:10.1016/j.ijrobp.2010.10.019 |
| [8] |
Daisne J, Blumhofer A. Atlas-based automatic segmentation of head and neck organs at risk and nodal target volumes:a clinical validation[J]. Radiat Oncol, 2013, 8(1): 154. DOI:10.1186/1748-717X-8-154 |
| [9] |
Han X, Hoogeman MS, Levendag PC, et al. Atlas-Based Autosegmentation of Head and Neck CT Images[C]. International Conference on Medical Image Computing and Computer-assisted Intervention, 2008: 434-41.
|
| [10] |
Ramus L, Malandain G. Multi-atlas based segmentation: Application to the head and neck region for radiotherapy planning[C]. MICCAI Workshop Medical Image Analysis for the Clinic-A Grand Challenge, 2010: 281-8.
|
| [11] |
Zhang T, Chi Y, Meldolesi E, et al. Automatic delineation of on-line head-and-neck computed tomography images:toward on-line adaptive radiotherapy[J]. Int J Radiat Oncol Biol Phys, 2007, 68(2): 522-30. |
| [12] |
Faggiano E, Fiorino C, Scalco E, et al. An automatic contour propagation method to follow parotid gland deformation during head-and-neck cancer tomotherapy[J]. Phys Med Biol, 2011, 56(3): 775. |
| [13] |
Parraga A, Petterssson J, Susin A, et al. Non-rigid registration methods assessment of 3D CT images for head-neck radiotherapy[J]. Med Imaging 2007: Image Processing. Internat Society for Optics and Photoni, 2007, 6512: 65121H.
|
| [14] |
Parraga A, Susin A, Pettersson J, et al. Quality assessment of non-rigid registration methods for atlas-based segmentation in head-neck radiotherapy[C]. IEEE International Conference on Acoustics, Speech and Signal Processing-ICASSP'07, 2007, 1: 445-8.
|
| [15] |
Sabuncu MR, Yeo BTT, Van Leemput K, et al. A generative model for image segmentation based on label fusion[J]. IEEE Trans Med Imaging, 2010, 29(10): 1714-29. DOI:10.1109/TMI.2010.2050897 |
| [16] |
Artaechevarria X, Munoz-Barrutia A, Ortiz-De-Solorzano C. Combination strategies in multi-atlas image segmentation:application to brain MR data[J]. IEEE Trans Med Imaging, 2009, 28(8): 1266-77. DOI:10.1109/TMI.2009.2014372 |
| [17] |
Isgum I, Staring M, Rutten A, et al. Multi-atlas-based segmentation with local decision fusion-application to cardiac and aortic segmentation in CT scans[J]. IEEE Trans Med Imaging, 2009, 28(7): 1000-10. |
| [18] |
Fortunati V, Verhaart RF, van der Lijn F, et al. Tissue segmentation of head and neck CT images for treatment planning:a multiatlas approach combined with intensity modeling[J]. Med Phys, 2013, 40(7): 071905. DOI:10.1118/1.4810971 |
| [19] |
Sharp G, Fritscher KD, Pekar V, et al. Vision 20/20:Perspectives on automated image segmentation for radiotherapy[J]. Med Phys, 2014, 41(5): 050902. |
| [20] |
Fritscher KD, Peroni M, Zaffino P, et al. Automatic segmentation of head and neck CT images for radiotherapy treatment planning using multiple atlases, statistical appearance models, and geodesic active contours[J]. Med Phys, 2014, 41(5): 051910. DOI:10.1118/1.4871623 |
| [21] |
Lee MJ, Hong H, Chung JW. Automatic segmentation of vertebral arteries in CT angiography using combined circular and cylindrical model fitting[C]. Medical Imaging 2014: Image Processing. International Society for Optics and Photonics, 2014, 9034: 90344A.
|
| [22] |
Qazi AA, Kim JJ, Jaffray DA, et al. Probabilistic refinement of model-based segmentation: application to radiation therapy planning of the head and neck[C]. International Workshop on Medical Imaging and Virtual Reality, 2010: 403-10.
|
| [23] |
Tam CM, Yang X, Tian S, et al. Automated delineation of organs-atrisk in head and neck CT images using multi-output support vector regression[C]. International Society for Optics and Photonics, 2018, 10578: 1057824.
|
| [24] |
Mannion-Haworth R, Bowes M, Ashman A, et al. Fully automatic segmentation of head and neck organs using active appearance models[J]. Med Phys, 2015. |
| [25] |
Chen A, Deeley MA, Niermann KJ, et al. Combining registration and active shape models for the automatic segmentation of the lymph node regions in head and neck CT images[J]. Med Phys, 2010, 37(12): 6338-46. DOI:10.1118/1.3515459 |
| [26] |
Litjens G, Kooi T, Bejnordi BE, et al. A survey on deep learning in medical image analysis[J]. Med Image Analysis, 2017, 42(2): 60-88. |
| [27] |
Shen D, Wu G, Suk H. Deep learning in medical image analysis[J]. Annual Rev Biomed Engineering, 2017, 19(3): 221-48. |
| [28] |
Sahiner B, Pezeshk A, Hadjiiski LM, et al. Deep learning in medical imaging and radiation therapy[J]. Med Phys, 2019, 46(1): e1-e36. |
| [29] |
Ren X, Xiang L, Nie D, et al. Interleaved 3D-CNNs for joint segmentation of small-volume structures in head and neck CT images[J]. Med Phys, 2018, 45(5): 2063-75. DOI:10.1002/mp.12837 |
| [30] |
Ibragimov B, Xing L. Segmentation of organs-at-risks in head and neck CT images using convolutional neural networks[J]. Med Phys, 2017, 44(2): 547-57. DOI:10.1002/mp.12045 |
| [31] |
Milletari F, Navab N, Ahmadi SA. V-net: Fully convolutional neural networks for volumetric medical image segmentation[C]. International Conference on 3D Vision (3DV), 2016: 565-71.
|
| [32] |
Hu J, Shen L, Sun G. Squeeze-and-excitation networks[C]. Proceedings of the IEEE conference on computer vision and pattern recognition, 2018: 7132-41.
|
| [33] |
Ronneberger O, Fischer P, Brox T. U-net: Convolutional networks for biomedical image segmentation[C]. International Conference on Medical image computing and computer-assisted intervention, 2015: 234-41.
|
| [34] |
Li X, Chen H, Qi X, et al. H-DenseUNet:hybrid densely connected UNet for liver and tumor segmentation from CT volumes[J]. IEEE Trans Med Imaging, 2018, 37(12): 2663-74. DOI:10.1109/TMI.2018.2845918 |
| [35] |
He K, Zhang X, Ren S, et al. Deep residual learning for image recognition[C]. Proceedings of the IEEE conference on computer vision and pattern recognition, 2016: 770-8.
|
| [36] |
Kingma D, Ba J. Adam: A Method for Stochastic Optimization[C]. International Conference on Learning Representations, 2014.
|
| [37] |
Raudaschl PF, Zaffino P, Sharp GC, et al. Evaluation of segmentation methods on head and neck CT:auto-segmentation challenge 2015[J]. Med Physics, 2017, 44(5): 2020-36. DOI:10.1002/mp.12197 |
 2020, Vol. 40
2020, Vol. 40

