2. 慧影医疗科技 (北京) 有限公司, 北京 100000
2. Huiying Medical Technology Co., Ltd, Beijing 100000, China
软骨肉瘤和内生软骨瘤是较为常见的软骨源性骨肿瘤[1-4], 术前准确诊断对于二者治疗方式的选择和预后至关重要, 软骨肉瘤主要以手术切除为主, 而内生软骨瘤以随访或病灶刮除术为主[5-8]。影像学上鉴别软骨肉瘤与内生软骨瘤仍有一定困难, 尤其对于高分化软骨肉瘤与内生软骨瘤的鉴别仍面临较大挑战[3, 9-11]。影像组学通过提取医学图像中传统视觉难以观察到的信息, 定量图像分析来反映潜在的病理生理学的信息[12-16]。近年来影像组学已广泛应用于肿瘤的诊断、监测随访及生存预测等方面[17-21], 在骨肌系统的研究则相对少见[22-23], 在软骨源性骨肿瘤方面的研究更为少见。目前相关文献报道[24-26]为基于单一序列(T1WI)或单个层面的影像组学特征, 所获组学信息量少, 本研究在此基础上进一步深入研究, 基于MRI平扫T1WI和T2WI两个最基本序列上、肿瘤所有层面的全部信息以构建影像组学模型, 探讨其用于鉴别诊断软骨肉瘤与内生软骨瘤的价值, 并进行组学模型与放射科医师诊断效能对比。
1 资料和方法 1.1 研究对象回顾性分析2011年1月~2019年5月中山大学附属第五医院符合以下纳入标准及排除标准的患者资料, 包括患者性别、年龄、肿瘤定位以及病理组织学结果等信息。纳入标准:(1)经手术或穿刺活检病理证实为软骨肉瘤或内生软骨瘤的病例; (2)术前行MRI检查, 至少包含T1WI及T2WI-FS序列; (3)术前MRI检查与手术或活检穿刺检查间隔不超过3月。排除标准:(1)在MRI检查前接受了放疗或化疗; (2)患者同时患有软骨肉瘤或内生软骨瘤以外的其他肿瘤; (3)发生在椎骨的肿瘤; (4)肿瘤部位发生病理性骨折; (5)图像质量欠佳, 难以进行感兴趣区域(ROI)勾画及影像组学特征提取。
根据纳入标准和排除标准, 本研究最终纳入68例患者, 其中女性43例, 男性25例, 年龄7~88 (43.8±17.7)岁。包括27例软骨肉瘤及41例内生软骨瘤, 其中低级别(1级)软骨肉瘤17例, 高级别(2~3级)软骨肉瘤10例。以7:3的比例将病例随机分配到训练组与验证组中, 训练组用于建立诊断模型, 验证组用于验证模型鉴别诊断软骨肉瘤与内生软骨瘤的效能。
1.2 MRI检查方法采用德国SIEMENS 3.0T MR扫描仪进行术前检查, 所有检查至少包含T1WI和T2WI-FS序列。扫描序列及参数如下:(1) T1WI快速自旋回波序列:TR 640 ms, TE 9.9 ms, 反转角150°, 层厚5.0 mm, 间隔1 mm, 矩阵418×314, 回波链长度3。(2) T2WI脂肪抑制快速自旋回波序列:TR 2800 ms, TE 91 ms, 反转角150°, 层厚5.0 mm, 间隔1 mm, 矩阵256×256, 回波链长度17。本研究选取T1WI与T2WI-FS序列进行组学特征提取及分析。
1.3 图像特征提取与诊断模型构建 1.3.1 肿瘤ROI的勾画及分割将显示软骨肉瘤或内生软骨瘤所有层面的T1WI和T2WI-FS序列图像以DICOM格式导出, 上传至慧影放射组学云平台, 由两名放射科医师(分别具有影像诊断3年和5年经验)独立对图像进行分割, 优先选取轴位图像, 当轴位图像显示不理想时选取冠状位图像或矢状位图像, 沿肿瘤边缘逐层手动勾画肿瘤ROI, 提取整个肿瘤的图像信息(图 1)。
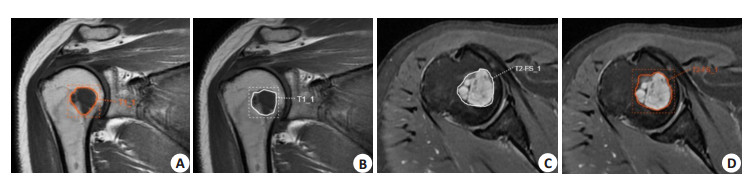
|
图 1 两名放射科医师在T1WI和T2WI-FS序列上沿肿瘤边缘独立逐层手动勾画ROI Fig.1 Regions of interesT (ROIs) manually depicted along the margin of tumor on T1WI and T2WI-FS sequences by two radiologists independently. The case is a 50-year-old woman with enchondroma in the right humerus. A, B: ROIs manually depicted along the margin of tumor on T1WI sequences by two different radiologists (with 3 and 5 years' experiences in radiology diagnosis) independently; C, D: ROIs manually depicted along the margin of tumor on T2WI-FS sequences by two different radiologists. |
将分割的肿瘤ROI在放射组学云平台(慧影医疗科技(北京)有限公司)上进行影像组学特征提取, 包括一阶统计量特征、灰度级共生矩阵(GLCM)、灰度级长度矩阵(GLRLM)、灰度级形状矩阵(GLSZM)、小波特征、平方值、平方根、对数特征、指数特征等。
在训练组中, 首先采用方差选择法对提取的组学特征进行初步筛选, 选择方差大于0.8的特征值进行下一步分析。然后采用单变量特征选择, 对分类变量选用卡方检验, 对连续型变量选用皮尔森相关系数, 进一步选出重要的组学特征, 保留P值小于0.05的特征进行下一步分析。最后通过最小绝对收缩与选择算子算法(LASSO)对组学特征进行降维, 筛选出最佳影像组学特征构建组学模型。
1.3.3 组学模型的建立与效能评估使用多因素逻辑回归(LR)分别构建基于T1WI和T2WI-FS序列的影像组学模型。影像组学模型的诊断效能使用敏感度、特异度、准确率(ACC)、尤登指数及受试者工作特征(ROC)曲线和曲线下面积(AUC)评估, 并分别对比基于两组序列图像的组学诊断模型在训练组与验证组中的诊断效能。
1.4 放射科医师采用常规MR图像诊断由第3名放射科医师(具有10年肌骨影像诊断经验)通过PACS系统对所有患者的MR图像进行诊断, 允许参考所有MR图像序列, 但对患者的临床信息、病理组织学结果均不知晓, 记录常规MRI影像学诊断结果, 在后续与影像组学模型的诊断结果作对比。
1.5 影像组学模型与放射科医师采用常规MR诊断的对比将所有病例应用到构建的组学模型中, 计算其特异度、敏感度、ACC、尤登指数、ROC曲线及AUC, 与放射科医师采用常规MR图像诊断进行对比。
1.6 统计学方法采用SPSS 20.0软件进行统计学分析。采用Kolmogorov Smirnov检验对定量资料进行正态性检验, 符合正态分布的计量资料用均数±标准差表示, 偏态分布数据用中位数(上、下四分位数)表示, 计数资料以频数表示。采用组内相关系数(ICC)评价2名医师提取肿瘤ROI组学特征的一致性。当ICC值 0.4, 表示一致性较差; 当ICC值为0.4~0.59, 表示一致性一般; 当ICC值为0.6~0.74, 表示一致性较好; 当ICC值>0.75, 表示一致性良好[27]。使用敏感度、特异度、ROC曲线及其95%置信区间(CI)评估影像组学模型及常规MR图像诊断的诊断效能。采用Medcalc 15.2软件比较ROC曲线的差异, AUC比较采用DeLong法[28-29]。所有检验均采用双侧检验, P < 0.05为差异有统计学意义。
2 结果本研究最终纳入68例病例, 共136组MR图像。其中训练组46例(软骨肉瘤18例, 内生软骨瘤28例), 验证组22例(软骨肉瘤9例, 内生软骨瘤13例)。患者的基本资料见表 1。
| 表 1 软骨肉瘤和内生软骨瘤患者的基本资料 Tab.1 Characteristic of patients with chondrosarcoma or enchondroma |
两名放射科诊断医师A与医师B的ICC值范围为0.779~0.923, 提示影像组学特征提取在组间有较高的一致性, 可重复性较好。
2.2 影像组学特征的筛选结果在训练组T1WI序列中, 从837个组学特征中使用方差选择法初步筛选出521个特征, 然后使用单变量特征选择法选出392个特征, 最终用Lasso算法降维筛选出10个影像组学特征(表 2), 包括一阶统计量特征1个, 纹理特征6个, 高阶统计量特征3个。
| 表 2 基于T1WI序列经LASSO算法降维后筛选出10个影像组学特征 Tab.2 Ten radiomics features selected from T1WI sequence based on LASSO algorithm |
在训练组T2WI-FS序列中, 从837个组学特征中使用方差选择法初步筛选出530个特征, 然后使用单变量特征选择法选出85个特征, 最终用Lasso算法降维筛选出11个影像组学特征(表 3), 包括一阶统计量特征2个, 纹理特征7个, 高阶统计量特征2个。
| 表 3 基于T2WI-FS序列经LASSO算法降维后筛选出11个影像组学特征 Tab.3 Eleven radiomics features selected from T2WI-FS sequence based on LASSO algorithm |
通过箱式点图对基于T1WI、T2WI-FS序列的组学特征进行分布描述(图 2、3)。
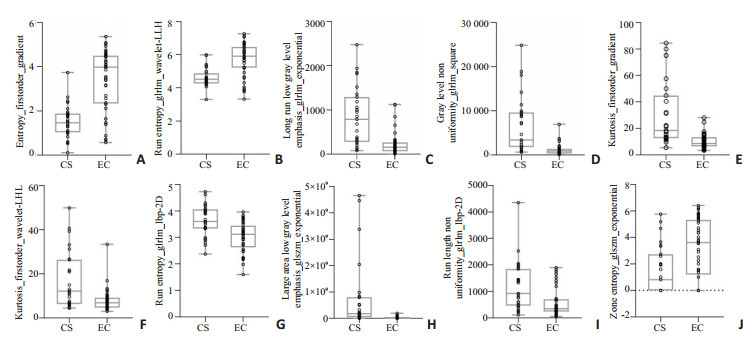
|
图 2 基于T1WI序列筛选的10个影像组学特征的箱形图及散点图 Fig.2 Box plots and scatter plots for 10 radiomics features selected from T1WI sequence. CS: Chondrosarcoma; EC: Enchondroma. Top and bottom line represents the maximum and minimum value. The middle line represents median value. Scatters represent values of radiomics features. |
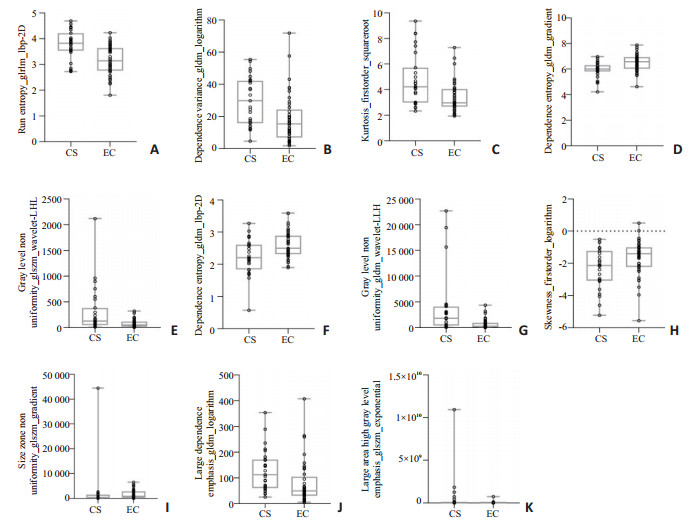
|
图 3 基于T2WI-FS序列筛选的11个影像组学特征箱形图及散点图 Fig.3 Box plots and scatter plots for 11 radiomics features selected from T2WI-FS sequence. CS: Chondrosarcoma; EC: Enchondroma. Top and bottom line represents the maximum and minimum value. The middle line represents median value. Scatters represent values of radiomics features. |
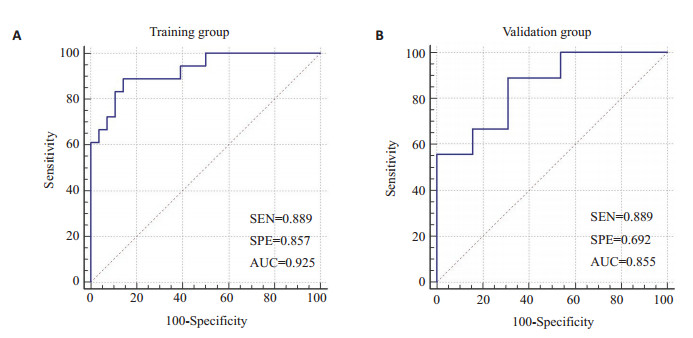
在训练组中, 基于T1WI序列筛选的10个组学特征及T2WI-FS序列筛选的11个组学特征, 分别使用多因素逻辑回归建立模型。在训练组中, 基于T1WI、T2WI-FS序列组学模型的敏感度分别为0.944和0.889, 特异度分别为0.964和0.857, ACC分别为0.957和0.891;在验证组中, 两者的敏感度分别为0.889和0.889, 特异度分别为0.846和0.692, ACC分别为0.864和0.773(表 4)。在T1WI、T2WI-FS序列中, 组学模型在训练组和验证组的ROC曲线(图 4、5)差异均无统计学意义(P>0.05)。
| 表 4 基于T1WI、T2WI-FS序列的逻辑回归组学模型在训练组和验证组中的诊断效能 Tab.4 The diagnostic accuracy of radiomics models using logistic regression analysis based on T1WI and T2WI-FS sequences in training group and validation group |
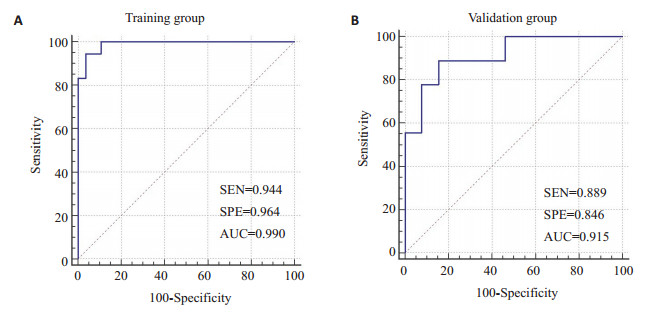
|
图 4 T1WI序列影像组学模型训练组和验证组的ROC曲线 Fig.4 Receiver operating characteristic (ROC) curve of the radiomics model based on T1WI for differentiation of chondrosarcoma from enchondroma in the training group (A) and validation (B) group. SEN: Sensitivity; SPE: Specificity; AUC: Area under the ROC curve. The AUCs of the training group (n=46) and the validation group (n=22) were 0.990 and 0.915, respectively. |
|
图 5 T2WI-FS序列影像组学模型训练组和验证组的ROC曲线 Fig.5 Receiver operating characteristic (ROC) curve of the radiomics model based on T2WI-FS for differentiation of chondrosarcoma from enchondroma in training group (A) and validation (B) group. The AUC of the training group (n=46) and the validation group (n=22) were 0.925 and 0.855, respectively. |
在T1WI序列中, 多因素逻辑回归分析构建的诊断模型=239.744-71.366×特征1-0.093×特征2+0.002×特征3-1.504×特征4+8.388×特征5-14.253×特征6+0.001×特征7+0.091×特征8-19.574×特征9-28.878×特征10 (表 2)。
在T2WI-FS序列中, 多因素逻辑回归分析构建的诊断模型=31.879-3.355×特征a+0.351×特征b-0.903×特征c-1.853×特征d+0.080×特征e-7.358×特征f-0.001×特征g-2.373×特征h-0.006×特征i-0.030×特征j+0.001×特征k (表 3)。
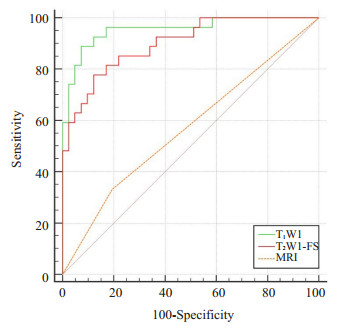
2.4 放射科医师采用常规MRI诊断与组学模型诊断效能的对比对于所有68例肿瘤, 放射科医师采用常规MR图像诊断的敏感度为0.333 (9/27), 特异度为0.805 (33/41), AUC为0.569 (95% CI:0.443~0.689)。将所有病例应用于T1WI、T2WI-FS序列的影像组学模型, 两者的敏感度分别为0.889、0.778, 特异度分别为0.927、0.878, T1WI组学模型与T2WI-FS组学模型之间的诊断效能差异无统计学意义(P=0.193), 而组学模型的诊断效能与放射科医师采用常规MRI诊断的差异有统计学意义(P < 0.01), 组学模型的诊断效能高于常规MRI诊断(表 5, 图 6)。
|
图 6 所有病例在T1WI、T2WI-FS序列的影像组学模型和采用常规MRI诊断的ROC曲线 Fig.6 Receiver operating characteristic (ROC) curve of radiomics model based on T1WI and T2WI- FS and conventional MRI for differentiation of chondrosarcoma from enchondroma (n=68). The AUC of the three groups were 0.955, 0.901 and 0.569, respectively. The diagnostic accuracy of the radiomics models was higher than that of conventional MRI (P < 0.01). |
| 表 5 所有病例应用于T1WI、T2WI-FS序列的影像组学模型并与常规MRI诊断对比 Tab.5 The comparison of diagnosis accuracy between radiomics models based on T1WI, T2WI-FS sequences and conventional MRI in all patients |
本研究选取了MRI平扫T1WI和T2WI-FS序列进行影像组学特征提取及筛选, 基于这两个序列的组学模型诊断效能差异均无统计学意义(P>0.05)。在所有病例中, 基于T1WI、T2WI-FS序列组学模型诊断效能均高于常规MRI诊断(P < 0.01)。
3.1 组学模型与放射科医师诊断效能的比较本研究表明, 基于T1WI、T2WI-FS序列的组学模型诊断效能均较高, 明显优于采用常规MRI诊断。与本研究结果不同, Fritz等[24]研究显示, 在鉴别诊断软骨肉瘤与内生软骨瘤, 根据常规MRI图像特征的敏感度为0.859, 特异度为0.808, AUC为0.870(95% CI:0.759~ 0.925), 根据纹理特征模型的敏感度为0.813, 特异度为0.904, AUC为0.898 (95% CI:0.828~0.946), 两者的诊断效能差异无统计学意义(P=0.458)。推测可能原因如下:(1)常规MR诊断软骨源性肿瘤主要是基于肿瘤的形态学、信号强度和强化特征等, 而没有考虑到纹理特征等肉眼不可见的组学特征; (2)曾有研究勾画每个肿瘤的最大层面提取纹理特征用以构建组学模型, 单个层面图像提供的影像组学信息有限; (3)此研究仅提取19个二维纹理特征, 包括4个一阶特征、4个二阶特征、11个高阶特征, 参与分析的特征过少不足以区分肿瘤的异质性, 另外在构建纹理特征模型时未对提取的特征进行筛选优化, 加入过多的无用特征可能令分类模型稳定性较差且带来负面影响。
与之相比, 本研究沿肿瘤边缘逐层勾画肿瘤ROI, 提取整个肿瘤的图像信息, 能够获取较完整的组学特征, 在T1WI和T2WI-FS序列上各提取了837个组学特征, 包含一阶统计量的直方图特征, 二阶的灰度共生矩阵、游程矩阵特征, 基于变换的高阶特征小波变换、几何参数等。在获取大量组学特征后, 笔者对所提取的特征进行筛选, 最后使用Lasso降维优化, 提取出对鉴别诊断最有价值的少数组学特征防止模型过度拟合, 从而提高模型的稳定性与可靠性, 这也是本研究结果较优的可能原因。此外, Fritz等[24]研究中, 医师阅片诊断并非采用完全盲法, 在进行阅片诊断前医师已知病灶为软骨性肿瘤, 因此常规MRI诊断准确性较高; 本研究中, 阅片者采用完全盲法, 更接近真实日常工作情景, 但不可避免受到研究者年资和经验的影响, 可能存在一定偏倚。
3.2 基于MRI平扫鉴别诊断软骨肉瘤与内生软骨瘤的序列选择软骨源性肿瘤在MR上表现有一定特点, 主要有T2WI呈明显高信号、增强扫描边缘强化和分隔强化、ADC值较高(弥散受限程度较轻)。不抑脂的T1WI及T2WI序列是最基本序列, 更能真实反映肿瘤组织的内部信息特征。然而临床上普遍存在过分倚重T2WI FS序列的误区, T2WI FS序列在脂肪抑制的同时也不可避免的掩盖了部分病变的信息。目前软骨肉瘤影像组学的相关研究仅使用T1WI一个序列提取组学特征[24-26], 本研究则采用MR平扫的两个最基本序列T1WI及T2WI-FS提取整个肿瘤的体积进行组学特征提取, 结果显示两个序列提取的组学特征均可用于鉴别软骨肉瘤与内生软骨瘤, 价值相当。笔者推测这与在T1WI序列上, 软骨肉瘤以中等信号为主而内生软骨瘤以低信号为主; 在T2WI-FS序列上, 除了两者信号强度存在差异, 软骨肉瘤较常出现软组织肿块以及邻近髓腔的信号发生改变[30], 这些因素均反应了肿瘤的异质性从而能为鉴别诊断提供组学特征信息。此外, 准确勾画肿瘤ROI直接影像提取的组学特征, 与周行知等[25]研究相比, 本研究进行了组间一致性分析, 两名放射科医师所提取的组学特征ICC值范围为0.779~0.923, 表明在T1WI和T2WI-FS序列上勾画肿瘤ROI可重复性较好, 所提取的影像组学特征有较高的可靠性。
3.3 本研究的局限性第一, 本研究为回顾性分析, 可能存在选择性偏倚; 第二, 样本未进行严格的图像标准化处理, 对研究结果可能造成一定的影响; 第三, 为单中心研究, 内生软骨瘤和软骨肉瘤的发病率相对较低, 故样本量偏小, 无法对低级别和高级别软骨肉瘤进行亚组分析, 使用组学模型鉴别诊断内生软骨瘤与低级别软骨肉瘤, 以及使用组学模型对软骨肉瘤进行病理分级的临床意义更大; 第四, 本研究基于MR平扫、且单个序列的组学模型用于鉴别软骨肉瘤与内生软骨瘤, 现阶段未进行两个序列的联合分析。
综上所述, 基于MR平扫T1WI和T2WI-FS序列构建的影像组学模型可用于鉴别软骨肉瘤与内生软骨瘤, 且明显优于放射科医师采用常规MRI序列的诊断效能。
| [1] |
Jundt G. Updates to the WHO classification of bone tumours[J]. Pathologe, 2018, 39(2): 107-16. |
| [2] |
Angelini A, Guerra G, Mavrogenis AF, et al. Clinical outcome of central conventional chondrosarcoma[J]. J Surg Oncol, 2012, 106(8): 929-37. DOI:10.1002/jso.23173 |
| [3] |
Crim J, Schmidt R, Layfield L, et al. Can imaging criteria distinguish enchondroma from grade 1 chondrosarcoma[J]. Eur J Radiol, 2015, 84(11): 2220-30. |
| [4] |
Doyle LA. Sarcoma classification:an update based on the 2013 World Health Organization classification of tumors of Soft tissue and bone[J]. Cancer, 2014, 120(12): 1763-74. DOI:10.1002/cncr.28657 |
| [5] |
Roberto AG, Inwards CY, Krishnan KU. Benign bone tumors-recent developments[J]. Semin Diagn Pathol, 2011, 28(1): 73-85. |
| [6] |
Schaser KD, Bail HJ, Haas NP, et al. Treatment concepts of benign bone tumors and tumor-like bone lesions[J]. Chirurg, 2002, 73(12): 1181-90. DOI:10.1007/s00104-002-0584-4 |
| [7] |
Fiorenza F, Abudu A, Grimer RJ, et al. Risk factors for survival and local control in chondrosarcoma of bone[J]. J Bone Joint Surg Br, 2002, 84: 93-9. DOI:10.1302/0301-620X.84B1.0840093 |
| [8] |
Lee FY, Mankin HJ, Fondren G, et al. Chondrosarcoma of bone:an assessment of outcome[J]. J Bone Joint Surg Am, 1999, 81(3): 326-38. DOI:10.2106/00004623-199903000-00004 |
| [9] |
谭一慜, 丁晓毅. 影像学检查在内生软骨瘤与Ⅰ级软骨肉瘤鉴别诊断中的价值[J]. 国际骨科学杂志, 2016, 37(3): 167-70. DOI:10.3969/j.issn.1673-7083.2016.03.008 |
| [10] |
Yoo HJ, Sung HH, Ja-Young C, et al. Differentiating high-grade from low-grade chondrosarcoma with Mr imaging[J]. Eur Radiol, 2009, 19(12): 3008-14. DOI:10.1007/s00330-009-1493-4 |
| [11] |
Douis H, Davies AM, James SL, et al. Can Mr imaging challenge the commonly accepted theory of the pathogenesis of solitary enchondroma of long bone[J]. Skeletal Radiol, 2012, 41(12): 1537-42. DOI:10.1007/s00256-012-1387-4 |
| [12] |
Gillies RJ, Kinahan PE, Hricak H. Radiomics:images are more than pictures, they are data[J]. Radiology, 2016, 278(2): 563-77. |
| [13] |
Larue RT, Defraene G, De Ruysscher D, et al. Quantitative radiomics studies for tissue characterization:a review of technology and methodological procedures[J]. Br J Radiol, 2017, 90(170): 20160665. |
| [14] |
Lambin P, Leijenaar RT, Deist TM, et al. Radiomics:the bridge between medical imaging and personalized medicine[J]. Nat Rev Clin Oncol, 2017, 14(12): 749-62. DOI:10.1038/nrclinonc.2017.141 |
| [15] |
O'connor JB. Cancer heterogeneity and imaging[J]. Semin Cell Dev Biol, 2017, 64: 48-57. DOI:10.1016/j.semcdb.2016.10.001 |
| [16] |
Lambin P, Rios-Velazquez E, Leijenaar R, et al. Radiomics:extracting more information from medical images using advanced feature analysis[J]. Eur J Cancer, 2012, 48(4): 441-6. DOI:10.1016/j.ejca.2011.11.036 |
| [17] |
Sun Y, Reynolds HM, Parameswaran B, et al. Multiparametric MRI and radiomics in prostate cancer:a review[J]. Australas Phys Eng Sci Med, 2019, 42(1): 3-25. DOI:10.1007/s13246-019-00730-z |
| [18] |
Li ZM, Lan Y, Xin W, et al. Diagnostic performance of mammographic texture analysis in the differential diagnosis of benign and malignant breast tumors[J]. Clin Breast Cancer, 2018, 18(4): e621-7. |
| [19] |
Wu W, Parmar C, Grossmann P, et al. Exploratory study to identify radiomics classifiers for lung cancer histology[J]. Front Oncol, 2016, 6: 71. |
| [20] |
Dinapoli N, Casa C, Barbaro B, et al. Radiomics for rectal cancer[J]. Transl Cancer Res, 2016, 5(4): 424-31. DOI:10.21037/tcr.2016.06.08 |
| [21] |
Islam A, Syed MR, Iftekharuddin KM. Multifractal texture estimation for detection and segmentation of brain tumors[J]. IEEE Trans Biomed Eng, 2013, 60(11): 3204-15. DOI:10.1109/TBME.2013.2271383 |
| [22] |
Ping Y, Ning M, Chao Z, et al. Comparison of radiomics machinelearning classifiers and feature selection for differentiation of sacral chordoma and sacral giant cell tumour based on 3D computed tomography features[J]. Eur Radiol, 2019, 29(4): 1841-7. |
| [23] |
Yan W, Lei X, Yang PF, et al. Survival prediction in high-grade osteosarcoma using radiomics of diagnostic computed tomography[J]. EBioMedicine, 2018, 34: 27-34. DOI:10.1016/j.ebiom.2018.07.006 |
| [24] |
Fritz B, Müller DA, Sutter R, et al. Magnetic resonance ImagingBased grading of cartilaginous bone tumors:added value of quantitative texture analysis[J]. Invest Radiol, 2018, 53(11): 663-72. DOI:10.1097/RLI.0000000000000486 |
| [25] |
周行知, 许镭, 林鹏, 等. 应用MR影像组学特征鉴别长骨内生软骨瘤与软骨肉瘤的研究分析[J]. 实用肿瘤杂志, 2019, 34(3): 219-26. |
| [26] |
Catharina SL, Christoph GL, Flosdorf K, et al. Diagnostic value of MRI-based 3D texture analysis for tissue characterisation and discrimination of low-grade chondrosarcoma from enchondroma:a pilot study[J]. Eur Radiol, 2018, 28(2): 468-77. |
| [27] |
Guidelines CD. Criteria, and rules of thumb for evaluating normed and standardized assessment instruments in psychology[J]. Psychol Assess, 1994, 6(4): 284-90. |
| [28] |
Er DL, Delong DM, Clarke-Pearson DL. Comparing the areas under two or more correlated receiver operating characteristic curves:a nonparametric approach[J]. Biometrics, 1988, 44(3): 837-45. DOI:10.2307/2531595 |
| [29] |
Goncalves L, Subtil A, Oliveira MR, et al. ROC Curve estimation:an overview[J]. Revstat-Statistical J, 2014, 12(1): 1-20. |
| [30] |
Bo-Bae C, Won-Hee J, Hee-Jung S, et al. Mr differentiation of lowgrade chondrosarcoma from enchondroma[J]. Clin Imaging, 2013, 37(3): 542-7. |
 2020, Vol. 40
2020, Vol. 40

