肾母细胞瘤是儿童泌尿系统中最常见的恶性肿瘤,据统计,肾母细胞瘤占所有儿科肿瘤的8%~10%,小于15岁的儿童及青少年中综合发病率约为0.1‰ [1-2]。随着手术、放化疗等个体化、规范化综合治疗逐渐应用于临床,肾母细胞瘤患儿的生存率有所提高,但仍有部分患儿如双侧肾母细胞瘤患儿、不能完全缓解、治疗后复发或转移的患儿,预后不佳[3]。因此,研究新的肿瘤预测标志物,探索参与肾母细胞瘤发生、发展和转移的机制是当前亟待解决的问题之一。
研究表明,微小RNA(miRNA)的异常表达能促进癌细胞增殖和转移,并能抑制癌细胞凋亡,从而促进肿瘤的发展[4]。其中,miR-155-5p被证实介导了多种肿瘤增殖和转移[5-6],但miR-155-5p在肾母细胞瘤中的的表达及作用尚未见文献报道。本研究通过检测miR-155-5p在肾母细胞瘤组织上的表达情况,同时在肾母细胞瘤G401细胞中上调和下调miR-155-5p的表达,观察对肿瘤细胞增殖、迁移及凋亡的影响。
1 资料和方法 1.1 研究对象选取2017年1月~2019年6月在重庆医科大学附属儿童医院泌尿外科行手术切除的肾母细胞瘤组织标本和瘤旁组织,共40例。其中男性21例,女性19例,年龄2月10 d~12岁(2.7±2.5)岁。患者在手术前均未接受过药物及放疗化疗处理。手术标本切下后,迅速将组织分离,无菌生理盐水冲洗2遍后,用4%多聚甲醛固定,另外取部分组织标本装于冻存管中置于液氮中速冻, 置入-80 ℃冰箱冷冻保存备用。本实验中标本的病理诊断由我院病理科高年资医生阅片诊断明确。本研究经重庆医科大学附属儿童医院伦理委员会批准,所有样本采集均获得患者本人及其家属的同意并均签署了知情同意书。
1.2 实验方法 1.2.1 RT-qPCR检测miR-155-5p在组织中的表达水平使用miRNA提取试剂盒(E.Z.N.A.TM miRNA Kit,OMEGA)分别提取肾母细胞瘤及瘤旁组织的miRNA,紫外分光光度计测其浓度,按逆转录试剂盒说明书(Mir-XTM miRNA First-Strand Synthesis Kit,TAKARA)操作进行反转录将RNA反转录为cDNA,RT-qPCR试剂盒(SYBR Premix Ex TaqTM Ⅱ,TAKARA)进行定量PCR扩增。引物(上海生工生物工程有限公司)序列如下:miR-155-5p上游引物为5'-TTAATGCTAATCGTG ATAGGGGT-3',下游引物和U6引物均为逆转录试剂盒提供。PCR仪(Bio-Rad),PCR扩增反应条件:95 ℃ 30 s,随后95 ℃ 5 s,60 ℃ 45 s,重复40个循环。以肿瘤组织为肿瘤组,瘤旁组织为正常对照组,肿瘤组织miR-155-5p的相对表达量用2-ΔΔCT法计算,每个样本按照相同条件检测3次,求均值。
1.2.2 肾母细胞瘤细胞株G401过表达及抑制miR-155-5p将肾母细胞瘤细胞株G401(中科院上海生物细胞研究所)接种于12孔细胞培养板中,置于37 ℃培养箱中培养24 h,当细胞密度达约70%~80%时进行转染操作,参照LipofectamineTM RNAiMAX转染试剂(Invitrogen)说明书将miR-155-5p mimics、inhibitor及各自相对应的阴性对照(上海吉玛制药技术有限公司)转染G401肾母细胞瘤细胞株,置于37 ℃培养箱中继续培养,分别将肿瘤细胞分为过表达组、过表达对照组、抑制组和抑制对照组。
1.2.3 RT-qPCR检测G401细胞miR-155-5p的表达情况取转染miR-155-5p mimics、inhibitor和阴性对照48 h的G401细胞,分别按照miRNA提取试剂盒说明书提取细胞miRNA,紫外分光光度计测其浓度。然后运用反转录试剂盒进行反转录,合成cDNA,运用定量SYBR green PCR试剂盒将cDNA在实时PCR仪上进行定量,U6为内参,反应模式为预变性95 ℃ 30 s;PCR反应变性95 ℃ 5 s,退火延伸60 ℃ 45 s,共40个循环。采用2-△△ct方法分析miR-155-5p在3种细胞中的相对表达水平。确定miR-155-5p转染成功后,行后续实验。
1.2.4 CCK-8检测过表达及抑制miR-155-5p对G401细胞增殖能力的影响将经过转染操作24 h的3组G401细胞以1×104/孔接种于96孔板中,每组设置5个复孔,置于37 ℃培养箱中培养0、1、2、3 d后,向每孔加入10 μL CCK-8溶液(Biosharp),注意避免产生气泡,置于37 ℃培养箱中孵育2 h后,用酶标仪测定吸光度A450 nm,实验重复3次。分析3组细胞的活力。
1.2.5 划痕实验检测过表达及抑制miR-155-5p对G401细胞迁移能力的影响将转染后24 h的各组G401细胞以1×106/孔接种于6孔板,待细胞汇合度达90%时,用10 μL的无菌枪头垂直方向划直线,PBS清洗3次去除脱落细胞后,加入2 mL无血清的新鲜McCoy's 5A培养基,分别在0、24、48 h于显微镜下拍照记录,用ImageJ软件测量迁移距离,计算各组细胞的划痕的相对距离[划痕相对距离(%)=24 h或48 h划痕宽度/0 h划痕宽度×100%]。实验重复3次。
1.2.6 流式细胞术实验检测过表达及抑制miR-155-5p对G401细胞细胞凋亡能力的影响收集转染48 h的各组G401细胞,经无EDTA的胰蛋白酶消化后,PBS洗涤两次,加入500 μL结合缓冲液重悬细胞,再加入膜联蛋白-V-异硫氰酸荧光素(Annexin V-FITC)和碘化丙啶各5 μL,避光、室温孵育15 min后,流式细胞仪检测各组细胞的凋亡率。每组实验均重复3次。
1.3 统计学方法数据分析采用SPSS 24.0统计软件,计量资料以均数±标准差表示,两组间均数的比较用独立样本t检验,P < 0.05为差异有统计学意义。
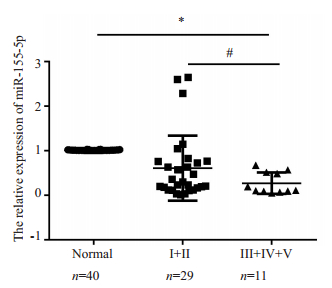
2 结果 2.1 miR-155-5p在肾母细胞瘤组织中低表达RT-qPCR结果显示,肾母细胞瘤组织中的miR-155-5p表达水平低于正常肾脏组织的表达水平,且患者临床TNM分期Ⅲ、Ⅳ、Ⅴ期(11例)的miR-155-5p表达量低于Ⅰ、Ⅱ期(29例)的患者,组间差异有统计学意义(P < 0.05,图 1)。
|
图 1 肾母细胞瘤组织和正常肾脏组织中miR-155-5p的表达 Fig.1 Expression of miR-454-3p in Wilms tumor tissues and normal kidney tissues. *P < 0.05 for comparison between tumor group and normal group; #P < 0.05 between stage Ⅲ/Ⅳ/Ⅴ and stage Ⅰ/Ⅱ Wilms tumor. |
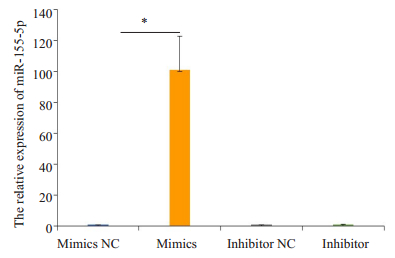
RT-qPCR结果显示,在miR-155-5p mimics转染组细胞的miR-155-5p表达水平高于阴性对照组(P < 0.05),miR-155-5p inhibitor转染组细胞的miR-155-5p表达水平无明显变化(图 2)。
|
图 2 在G401肾母细胞瘤细胞中成功过表达miR-155-5p Fig.2 Over-expression and knockdown of miR-155-5p in Wilms tumor G401 cells. *P < 0.05. |
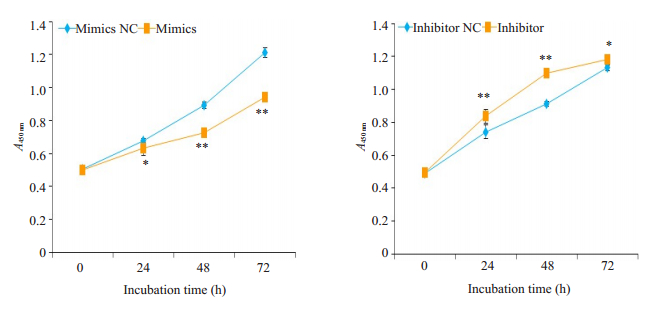
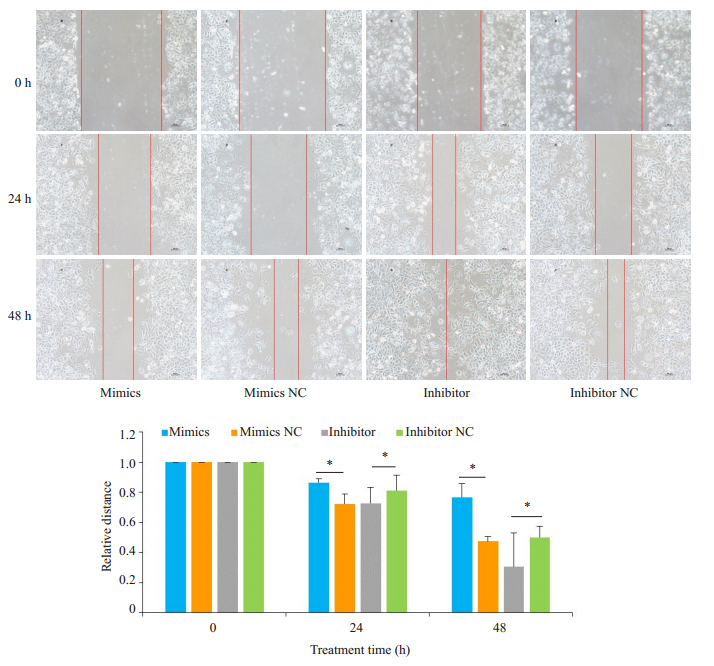
CCK-8检测结果显示,在G401细胞中过表达miR-155-5p后,细胞的生长受到了明显的抑制;反之,抑制miR-155-5p后,细胞的生长能力明显的增强,组间差异有统计学意义(P < 0.05,图 3)。划痕实验结果显示,miR-155-5p mimics转染组较阴性对照组细胞的迁移明显受抑制,而miR-155-5p inhibitor转染组细胞较阴性对照组的迁移能力明显增强,组间差异有统计学意义(P < 0.05,图 4)。
|
图 3 过表达miR-155-5p抑制G401肾母细胞瘤细胞增殖,下调miR-155-5p促进肿瘤细胞增殖 Fig.3 Overexpression of miR-155-5p inhibits the proliferation of Wilms tumor G401 cells, and knockdown of miR-155-5p promotes the proliferation of the tumor cells. *P < 0.05 vs miR-155-5p mimics NC group or inhibitor NC group. **P < 0.01. |
|
图 4 过表达miR-155-5p抑制G401肾母细胞瘤细胞迁移,下调miR-155-5p促进肿瘤细胞迁移 Fig.4 Overexpression of miR-155-5p inhibits the migration of Wilms tumor G401 cells, and knockdown of miR-155-5p promotes the migration of the tumor cells (*P < 0.05) |
流式细胞术结果显示,在G401细胞中过表达miR-155-5p后,细胞的凋亡能力得到了明显的促进;抑制miR-155-5p后,细胞的凋亡率明显降低,凋亡受到抑制,组间差异有统计学意义(P < 0.05,图 5)。
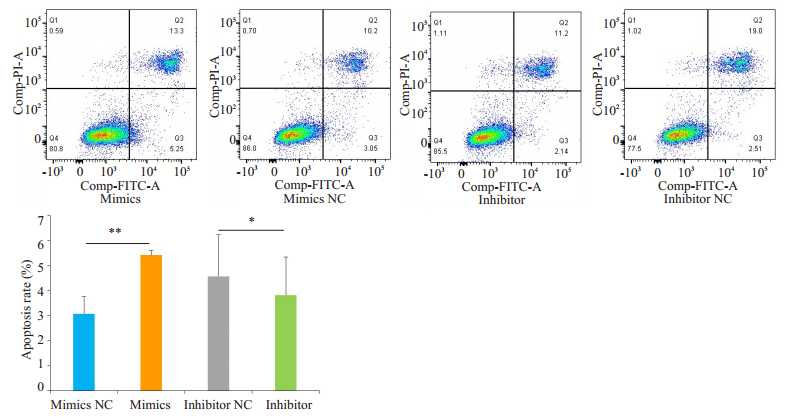
|
图 5 过表达miR-155-5p诱导G401肾母细胞瘤细胞凋亡,下调miR-155-5p抑制肿瘤细胞凋亡 Fig.5 Overexpression of miR-155-5p induces the apoptosis and knockdown of miR-155-5p suppresses the apoptosis of Wilms tumor G401 cells. *P < 0.05, **P < 0.01. |
肾母细胞瘤是小儿最常见的恶性实体肿瘤之一,其发病机制仍然不明确[7]。目前的研究认为患儿体内癌基因的异常激活、抑癌基因的缺乏或失活、相关基因的点突变、DNA复制、重排、转录或调控异常与肿瘤的发生都有关系[8-9]。近年来,运用基因芯片技术检测肾母细胞瘤组织及细胞系中miRNA表达情况,对比正常肾脏组织和细胞发现,在肾母细胞瘤中,众多miRNA含量变化明显、转录水平异常并且影响了疾病的发生、发展[10-11]。目前国内外关于miRNA在肾母细胞瘤中的具体机制研究还相对较少。国外主要研究肾母细胞瘤中调控miRNA的具体机制,如编码miRNA的DNA甲基化或其启动子周围染色质结构闭合使其表达沉默的相关机制,Myc基因激活直接与miRNA启动子相结合,进而抑制miRNA表达以及miRNA成熟过程的两个关键酶Drosha及Dicer1的异常导致miRNAs在生物转化途径中受阻等机制[12-15]。而国内主要研究具体miRNA在肾母细胞瘤中的表达及调控机制,如miR-572在肾母细胞瘤组织中表达上调,通过调控CDH1基因的表达,可促进肿瘤的转移以及上皮间质转化过程,miR-140-5p在肾母细胞瘤组织中表达下调,在肿瘤细胞中抑制TGFBR1和IGF1R的表达,从而抑制肿瘤的增殖,促进凋亡[16-18]。此外,miR-539和miR92a3p在肾母细胞瘤中可通过调控NOTCH1基因表达,进而抑制肿瘤细胞的增殖、转移及侵袭能力[19-20]。本研究应用RT-qPCR技术检测40例未经放化疗的肾母细胞瘤肿瘤组织,首次发现miR-155-5p在肿瘤组织中呈低表达,且与肿瘤的NWTS-5临床分期密切相关,分期越靠后其相对表达量越低,其可能与肾母细胞瘤的发生及发展过程密切相关。
miR-155-5p是一个典型的多功能miRNA,位于人类染色体21q21.3上,其作用机制还在不断的研究[21]。最初的研究主要发现miR-155-5p是一个致癌性的miRNA,其在多种肿瘤中表达水平显著增高,如非小细胞肺癌、肝癌、结直肠癌、口腔鳞状细胞癌和乳腺癌等[21-26]。miR-155-5p的作用特点复杂多样,在脊索瘤和肝癌中,miR-155-5p可通过直接靶向结合PTEN mRNA的3'-UTR,抑制PTEN的表达及抑癌作用,从而导致肿瘤细胞增殖能力增强而凋亡能力减弱,促进肿瘤的发生与转移[23, 27]。而在结直肠癌中,肿瘤细胞分泌的外泌体miR-155-5p可与BRG1编码序列结合,下调BRG1的表达,这一机制被认为是促进结直肠癌转移的关键因素;黑色素瘤细胞分泌的外泌体miR-155-5p表达也明显增强,可促进肿瘤相关成纤维细胞和内皮细胞增殖、迁移和血管生成的作用[6, 28-29]。本研究进一步在肾母细胞瘤细胞中过表达及抑制miR-155-5p,结果显示上调miR-155-5p可以抑制肾母细胞瘤细胞增殖、迁移,诱导肿瘤细胞凋亡,发挥了抑癌基因的作用;反之,下调miR-155-5p则可促进肾母细胞瘤G401细胞的增殖及迁移,同时抑制肿瘤细胞的凋亡,由此证实miR-155-5p在肾母细胞瘤中具有抑癌作用。最近研究表明,miR-155-5p在胃癌和骨肉瘤中的表达显著降低,过表达miR-155-5p也被证实具有抑癌作用,miR-155-5p作为抑癌基因的新功能受到了广泛关注[30-31]。
综上所述,在肾母细胞瘤中,miR-155-5p的表达显著下降,且与临床分期呈负相关性;同时,miR-155-5p可抑制肾母细胞瘤细胞的增殖与迁移过程,诱导肿瘤细胞发生凋亡。这为我们进一步探寻肾母细胞瘤的发病机制及其治疗靶点提供了新的思路和实验依据。但本研究中用于检测的组织样本尚不够多,未对这些病例进行随访,需更多的研究病例及随访来完善;且miR-155-5p调控肾母细胞瘤的具体机制以及miR-155-5p如何下调的机制还有待更加深入的研究。
| [1] |
Brok J, Lopez-Yurda M, Tinteren HV, et al. Relapse of wilms' tumour and detection methods: a retrospective analysis of the 2001 renal tumour study group-international society of paediatric oncology wilms' tumour protocol database[J]. Lancet Oncol, 2018, 19(8): 1072-81. DOI:10.1016/S1470-2045(18)30293-6 |
| [2] |
Deng CK, Dai R, Li XL, et al. Genetic variation frequencies in Wilms' tumor: a meta-analysis and systematic review[J]. Cancer Sci, 2016, 107(5): 690-9. DOI:10.1111/cas.12910 |
| [3] |
何大维. 进一步提升儿童肾母细胞瘤治疗水平[J]. 中华小儿外科杂志, 2017, 38(10): 721-4. DOI:10.3760/cma.j.issn.0253-3006.2017.10.001 |
| [4] |
Pichler M, Calin GA. MicroRNAs in cancer: from developmental genes in worms to their clinical application in patients[J]. Br J Cancer, 2015, 113(4): 569-73. DOI:10.1038/bjc.2015.253 |
| [5] |
Li N, Cui T, Guo WL, et al. MiR-155-5p accelerates the metastasis of cervical cancer cell via targeting TP53INP1[J]. Onco Targets Ther, 2019, 12(9): 3181-96. |
| [6] |
Al-Haidari A, Algaber A, Madhi R, et al. MiR-155-5p controls colon cancer cell migration via post-transcriptional regulation of human antigen R (HuR)[J]. Cancer Lett, 2018, 421(3): 145-51. |
| [7] |
Luo X, Deng CK, Liu F, et al. HnRNPL promotes Wilms tumor progression by regulating the p53 and Bcl2 pathways[J]. Onco Targets Ther, 2019, 12(11): 4269-79. |
| [8] |
Treger TD, Chowdhury T, Pritchard-Jones KA. The genetic changes of Wilms tumour[J]. Nat Rev Nephrol, 2019, 15(4): 240-51. DOI:10.1038/s41581-019-0112-0 |
| [9] |
Turnbull C, Perdeaux ER, Pernet D, et al. A genome-wide association study identifies susceptibility loci for Wilms tumor[J]. Nat Genet, 2012, 44(6): 681-4. DOI:10.1038/ng.2251 |
| [10] |
Chen KS, Stroup EK, Budhipramono A, et al. Mutations in microRNA processing genes in Wilms tumors derepress the IGF2 regulator PLAG1[J]. Genes Dev, 2018, 32(15/16): 996-1007. |
| [11] |
王家祥, 胡倩, 刘秋亮, 等. 肾母细胞瘤细胞株与正常胚肾细胞株微小RNA表达谱的差异分析[J]. 中华医学杂志, 2010, 90(26): 1845-8. DOI:10.3760/cma.j.issn.0376-2491.2010.26.014 |
| [12] |
Brueckner B, Stresemann C, Kuner R, et al. The human let-7a-3 locus contains an epigenetically regulated microRNA gene with oncogenic function[J]. Cancer Res, 2007, 67(4): 1419-23. DOI:10.1158/0008-5472.CAN-06-4074 |
| [13] |
Swierczynski S, Klieser E, Illig R, et al. Histone deacetylation meets miRNA:epigenetics and post-transcriptional regulation in cancer and chronic diseases[J]. Expert Opin Biol Ther, 2015, 15(5): 651-64. DOI:10.1517/14712598.2015.1025047 |
| [14] |
Chang TC, Yu D, Lee YS, et al. Widespread microRNA repression by Myc contributes to tumorigenesis[J]. Nat Genet, 2008, 40(1): 43-50. DOI:10.1038/ng.2007.30 |
| [15] |
Wu MK, Sabbaghian N, Xu B, et al. Biallelic DICER1 mutations occur in Wilms tumours[J]. J Pathol, 2013, 230(2): 154-64. DOI:10.1002/path.4196 |
| [16] |
Zhang C, Lv GQ, Cui LF, et al. MicroRNA-572 targets CDH1 to promote metastasis of Wilms' tumor[J]. Eur Rev Med Pharmacol Sci, 2019, 23(9): 3709-17. |
| [17] |
Liu Z, He F, Ouyang S, et al. miR-140-5p could suppress tumor proliferation and progression by targeting TGFBRI/SMAD2/3 and IGF-1R/AKT signaling pathways in wilms' tumor[J]. BMC Cancer, 2019, 19(1): 405-16. DOI:10.1186/s12885-019-5609-1 |
| [18] |
Wang HL, Lou CY, Ma N. miR-140-5p alleviates the aggressive progression of wilms' tumor through directly targeting TGFBR1 gene[J]. Cancer Manag Res, 2019, 11(9): 1641-51. |
| [19] |
Su HL, Wang XE, Song JJ, et al. MicroRNA-539 inhibits the progression of Wilms' tumor through downregulation of JAG1 and Notch1/3[J]. Cancer Biomark, 2019, 24(1): 125-33. DOI:10.3233/CBM-181972 |
| [20] |
Zhu S, Zhang L, Zhao Z, et al. MicroRNA92a3p inhibits the cell proliferation, migration and invasion of Wilms tumor by targeting NOTCH1[J]. Oncol Rep, 2018, 40(2): 571-8. |
| [21] |
Elton TS, Selemon H, Elton SM, et al. Regulation of the MIR155 host gene in physiological and pathological processes[J]. Gene, 2013, 532(1): 1-12. |
| [22] |
Xue X, Liu Y, Wang Y, et al. MiR-21 and MiR-155 promote non-small cell lung cancer progression by downregulating SOCS1, SOCS6, and PTEN[J]. Oncotarget, 2016, 7(51): 84508-19. |
| [23] |
Fu X, Wen HQ, Jing L, et al. MicroRNA-155-5p promotes hepatocellular carcinoma progression by suppressing PTEN through the PI3K/Akt pathway[J]. Cancer Sci, 2017, 108(4): 620-31. DOI:10.1111/cas.13177 |
| [24] |
Zhang GJ, Xiao HX, Tian HP, et al. Upregulation of microRNA-155 promotes the migration and invasion of colorectal cancer cells through the regulation of claudin-1 expression[J]. Int J Mol Med, 2013, 31(6): 1375-80. DOI:10.3892/ijmm.2013.1348 |
| [25] |
Rather MI, Nagashri MN, Swamy SS, et al. Oncogenic microRNA-155 down-regulates tumor suppressor CDC73 and promotes oral squamous cell carcinoma cell proliferation: implications for cancer therapeutics[J]. J Biol Chem, 2013, 288(1): 608-18. DOI:10.1074/jbc.M112.425736 |
| [26] |
Jiang S, Zhang HW, Lu MH, et al. MicroRNA-155 functions as an OncomiR in breast cancer by targeting the suppressor of cytokine signaling 1 gene[J]. Cancer Res, 2010, 70(8): 3119-27. DOI:10.1158/0008-5472.CAN-09-4250 |
| [27] |
Zhao KC, Li XE, Chen XX, et al. Inhibition of miR-140-3p or miR- 155-5p by antagomir treatment sensitize chordoma cells to chemotherapy drug treatment by increasing PTEN expression[J]. Eur J Pharmacol, 2019, 854(12): 298-306. |
| [28] |
Lan JQ, Sun L, Xu F, et al. M2 Macrophage-Derived exosomes promote cell migration and invasion in colon cancer[J]. Cancer Res, 2019, 79(1): 146-58. |
| [29] |
Zhou XC, Yan TL, Huang CM, et al. Melanoma cell-secreted exosomal miR-1555p induce proangiogenic Switch of cancerassociated fibroblasts via SOCS1/JAK2/STAT3 signaling pathway[J]. J Exp Clin Cancer Res, 2018, 37(1): 242-53. DOI:10.1186/s13046-018-0911-3 |
| [30] |
Wang M, Yang F, Qiu R, et al. The role of mmu-miR-155-5p-NF-B signaling in the education of bone marrow-derived mesenchymal stem cells by gastric cancer cells[J]. Cancer Med, 2018, 7(3): 856-68. DOI:10.1002/cam4.1355 |
| [31] |
Bhattacharya S, Chalk AM, Ng A, et al. Increased miR-155-5p and reduced miR-148a-3p contribute to the suppression of osteosarcoma cell death[J]. Oncogene, 2016, 35(40): 5282-94. DOI:10.1038/onc.2016.68 |
 2019, Vol. 39
2019, Vol. 39

