2. 重庆医科大学附属第二医院,重庆 400010;
3. 成都市第五人民医院,四川 成都 611130
2. Department of Hepatobiliary Surgery, Second Affiliated Hospital of Chongqing Medical University, Chongqing 400010, China;
3. Department of Gastroenterology, Fifth People's Hospital of Chengdu, Chengdu 611130, China
肝癌是第二常见的癌症相关死亡原因,近年来也成为我国癌症死亡的主要原因之一[1-2]。肝细胞肝癌患者在被确诊时往往已是中晚期,并常伴有肝内外的转移。肿瘤复发和转移仍然是肝癌治疗失败的主要原因[3-4]。因此,明确肝癌的分子机制,寻找一种有效的生物标志物来判断肝癌患者的高风险复发与转移是一个紧要的任务。
研究发现,DNA甲基化是在DNA甲基化转移酶(DNMTs)催化下,以S-腺苷甲硫氨酸为甲基供体,将活性甲基转移至DNA链中特定碱基[5]。在该化学修饰过程中,DNA甲基化可以改变染色质结构和转录因子的工作效率,从而调控目的基因的表达[6]。槟榔生物碱可募集DNMT3B来调节口腔癌中的miR-486-3p/DDR1轴而抬头看口腔癌的恶性发展[7]。TGF-β1/miR-200s/miR-221/DNMT3B调节环负责肿瘤相关成纤维细胞激活状态的维持,也是癌相关成纤维细胞促进乳腺癌恶性程度的必要机制[8]。同时,DNA甲基化可导致肿瘤抑制基因的失活,而在肝癌中观察到多个肿瘤抑制基因的甲基化而失去活性[6]。最新研究发现DNMT3B是哺乳动物基因重新甲基化所必需的关键酶,并且DNMT3B在多种恶性肿瘤中高表达,其可作为预防癌症复发的潜在治疗靶标[9-10]。然而,现有对肝癌中DNMT3B蛋白的研究并不清楚。因此,本研究检测DNMT3B在肝癌中的表达情况与临床意义,并检测沉默DNMT3B后Huh-7细胞增殖、迁移及侵袭能力的变化情况,深入探索DNMT3B在肝癌恶性发展过程中的具体分子机制,并探讨了DNMT3B作为肝癌的预后指标的可行性,为后续相关研究提供线索。
1 资料和方法 1.1 临床资料收集2008年5月~2013年5月在重庆医科大学附二院确诊的175例肝癌患者。所有的组织样本在肿瘤手术切除后立刻进行处理,用4%福尔马林液固定(pH=7),固定24 h后进行石蜡包埋,并制作成组织芯片。最终,肝癌的组织学上确诊由两名资深的重庆医科大学第二附属医院病理科病理学专家完成,其使用的方法是苏木精和伊红(HE)染色片检验法。所有研究对象都收集了完整的临床资料与随访信息。纳入标准:术后病理学检查证实为原发性肝脏细胞癌;随访时间超过5年;切缘无癌残留;临床资料完整。排除标准:缺少病理学诊断;病危或合并其他肿瘤;资料不全等影响疗效判断者。取肝癌组织及相应癌旁组织(距肿瘤边缘2 cm以上且经病理证实无癌细胞残余)于液氮中保存备用。
研究内容包括诊断时的年龄、性别、肿瘤大小、TNM分期、肝硬化、肝内转移、血管侵犯、乙型肝炎病毒感染和血清AFP水平(μmg/mL)。截止2018年7月,完成了对所有研究对象的随访与调查。本研究严格遵守赫尔辛基宣言和重庆医科大学附属第二医院伦理审查委员会的指导方针。
1.2 主要耗材与试剂人Huh-7细胞株购于中科院上海细胞所,胎牛血清、DNMEM培养液、青霉素/链霉素溶液(Hyclone),siRNA、转染试剂、EdU细胞增殖检测试剂盒(锐博生物),Western blot配胶试剂盒(康为生物),DNMT3B、LATS1、YAP1、Bcl-2、Bax、Ki-67、GAPDH及二抗(Abcam),Matrigel基质胶(BD),Transwell小室(Corning),CCK8试剂盒(凯基生物),总DNA提取试剂盒与总RNA提取试剂盒(天根生化科技),DNA甲基化试剂盒(ZYMO RESEARCH),荧光定量PCR试剂SYBR Premix EX Taq、逆转录试剂盒Prime Script RT Master Mix及甲基化特异性PCR试剂(TaKaRa)。
1.3 细胞培养与siRNA转染Huh-7细胞培养于含10%胎牛血清的DMEM培养液中,培养条件为37 ℃、5% CO2、100%饱和湿度。每隔1 d对细胞的生长情况进行观察,每2~3 d对细胞进行换液或传代。实验分为空白对照组(control)、阴性对照组(si-NC)和实验组(si-DNMT3B)。转染前取对数生长期的Huh-7细胞,以5×103细胞铺于96孔板,以4×104细胞铺于24孔板,2×105细胞铺于6孔板,采用Lipofectamine 3000试剂,将siRNA转染至Huh-7细胞。转染后48 h收集细胞,提取细胞总蛋白或总DNA等进行后续实验。
1.4 慢病毒转染实验分为空白对照组(control)、阴性对照组(NC)和实验组(LV-DNMT3B)。转染前取对数生长期的Huh-7细胞,以1×105细胞铺于6孔板,加入1 mL完全培养基,再加20 μL慢病毒。转染完成后收集细胞,提取细胞总蛋白或总DNA等进行后续实验。
1.5 qRT-PCR法检测DNMT3B基因的表达情况总RNA提取方法参照天根总RNA提取试剂盒说明书对各组Huh-7细胞进行RNA的提取。紫外分光光度计检测提取RNA到浓度及纯度。采用Takara公司的逆转录试剂盒将RNA反转录为cDNA。qRT-PCR以GAPDH为内参对照,采用2-ΔΔCt法分析定量PCR的结果。
1.6 甲基化特异性PCR检测LATS1甲基化水平总DNA提取方法参照天根总DNA提取试剂盒说明书对各组Huh-7细胞进行DNA的提取,用5 μg DNA进行亚硫酸氢盐处理,采用TaKaRa EpiTaq HS(for bisulfite-treated DNA)试剂盒进行PCR反应,具体方法参见其说明书。以凝胶电泳检测LATS1的甲基化水平。
1.7 Western blot检测DNMT3B以及相关蛋白的表达提取转染前后各组细胞的总蛋白。采用10%的SDS-PAGE分离胶分离蛋白质样品,随后进行转膜(300 mA,恒流湿转),用5%脱脂奶粉常温封闭1 h,加入DNMT3B、LATS1、YAP1、Bcl-1、Bax、Ki-67一抗4 ℃孵育过夜,TBST洗膜,二抗孵育(37 ℃,1 h),TBST洗膜等步骤。使用ECL化学发光试剂盒显影。所用一抗稀释比例为:DNMT3B(1:2000)、LATS1(1:3000)、YAP1(1 : 2000)、Bcl-1(1 : 3000)、Bax(1 : 2000)、Ki-67(1 : 3000)、GAPDH(1: 5000)。
1.8 CCK8检测细胞增殖能力根据转染后接种于96孔板的分组,待细胞贴壁后在0、24、48及72 h每孔加入10 μL CCK-8试剂,在37 ℃、5% CO2的细胞培养箱内孵育1 h,利用酶标仪检测A450 nm,取3个复孔的均值,绘制细胞生长曲线。
1.9 EdU细胞增殖检测细胞增殖能力根据转染后接种于96孔板的分组,待细胞贴壁后每孔加入100 μL EDU染料试剂,避光、室温、脱色摇床孵育30 min后,每孔再加入100 μL Hoechst 33342反应液,避光、室温、脱色摇床孵育30 min,在荧光显微镜下观察细胞EDU的摄取情况。
1.10 Transwell实验检测细胞迁移侵袭能力取转染后接种于6孔板的分组细胞,以无血清培养液悬浮为1×106/mL细胞浓度,接种于预铺好胶的Transwell小室的上室中,放入完全培养基条件的24孔板培养,37 ℃、5% CO2孵育8 h后甲醛固定并HE染色,在倒置显微镜下观察并计数细胞。
1.11 免疫组化为确定DNMT3B在肝癌中的表达情况,采用免疫组化染色检测样本组织芯片。二甲苯被用于脱蜡和水化,在梯度的酒精系列中脱水,在100 ℃ 10 mmol/L枸橼酸钠溶液(pH 6.0)中加热10 min进行抗原修复,在沸腾后冷却20 min,反复3次。在PBS(pH 7.2)冲洗后,在3%过氧化氢与山羊血清室温中分别封闭15 min以避免非特异性蛋白结合。此后,在切片上滴加DNMT3B单克隆抗体(1:300)并在4 ℃的温度下孵育过夜。接着,用生物素标记的二抗体孵育37 ℃ 30 min孵育,再与亲和素辣根酶在37 ℃孵育20 min。使用DAB对切片进行显色,显色后用流水冲洗15 min。切片使用1%苏木精复染。梯度乙醇脱水。最后,中性胶封闭。
1.12 免疫组织化学染色法的评分系统所有组织芯片的染色情况分析均由两位经验丰富的病理学专家进行独立评估,使用半定量评价方法得分,评分参数包括染色强度(范围0~3:0,无;1,弱;2,中;3, 强)和的阳性细胞百分比(范围0~4:0,0% < 5%;1,6% ~25%;2,26%~50%;3,51%~75%;4,76%~100%)。采用阳性细胞的比例与强度相加的计算方式来确定最终的染色分数。以总分≤4被定义为DNMT3B的低表达,而>4被定义为DNMT3B的高表达。
1.13 统计学分析使用SPSS 20.0软件对数据进行了分析处理,所有实验数据采用均数±标准差表示。实验组与对照组的比较采用独立样本t检验,多组间采用单因素方差分析。DNMT3B的表达与肝癌患者临床病理特征之间的关系采用卡方检验和Fisher精确检验。肿瘤无瘤生存率定义为在从尽可能切除肿瘤后到肿瘤复发之间的时间。肿瘤特异性生存率定义为从尽可能切除肿瘤后到患者因肿瘤原因死亡之间的时间。使用Kaplan-Meier与log-rank test检验来分析患者的肿瘤无瘤生存率与肿瘤特异性生存率。使用单因数与多因素Cox回归分析DNMT3B的表达对肝癌患者预后的影响。P < 0.05为差异有统计学意义。
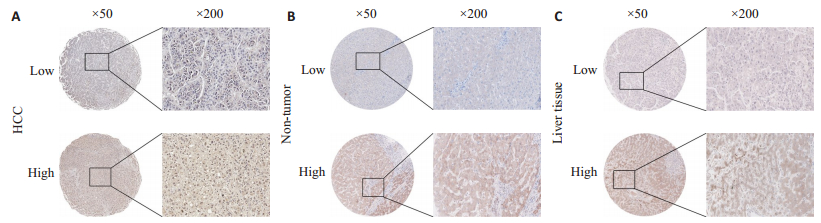
2 结果 2.1 患者的临床病理特征和DNMT3B的表达本研究共纳入175例患者,包括肝癌组织与配对的癌旁组织,男89例(50.8%),女86例(49.1%)。DNMT3B低表达组有男性患者27例,女性患者30例。DNMT3B高表达组有男性患者62例,女性患者56例。83例患者 < 45岁,92例患者≥45岁;DNMT3B低表达组 < 45岁的患者32例,≥45岁的患者25例,DNMT3B高表达组 < 45岁的患者51例,≥45岁的患者67例。所有研究对象的临床病理特征细节见表 1。DNMT3B主要表达在癌组织与癌旁组织的细胞核与部分胞浆中,结果表明,在肝癌中DNMT3B的表达率是67.4%(图 1A),配对癌旁的表达率是41.1%(图 1B),肝脏组织中的表达率是40%(图 1C)。
| 表 1 DNMT3B的表达情况与患者临床病理特征 Tab.1 DNMT3B expression and clinicopathological features of HCC patients |
|
图 1 DNMT3B在肝癌中的表达 Fig.1 Expression of DNMT3B in hepatocellular carcinoma (HCC) tissues. A: HCC; B: Non-tumor; C: Liver tissue. |
DNMT3B在肝癌中的表达在肿瘤大小(P=00.001)、血管侵犯(P=0.004)、肝内转移(P=0.018)的差异有统计学意义。然而,DNMT3B的表达在年龄、性别、AFP、肝硬化、HBV和TNM分期的差异无统计学意义(P>0.05,表 1)。
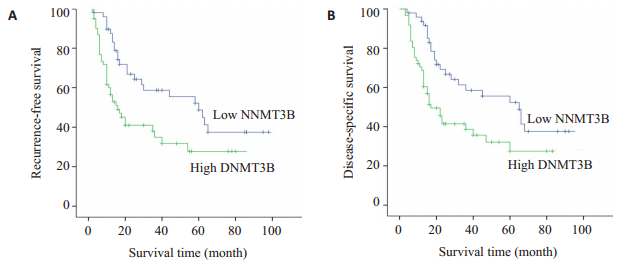
2.3 DNMT3B在肝癌中的表达与患者肿瘤无瘤生存率的关系DNMT3B在肝癌低表达组中的1年肿瘤无瘤生存率为91.1%,3年肿瘤无瘤生存率为68.5%。但DNMT3B在肝癌高表达组中的1年肿瘤无瘤生存率为77.8%,3年肿瘤无瘤生存率为48.7%。对数秩检验结果显示,肿瘤无瘤生存率在两组之间的差异有统计学意义(P=0.047,图 2A),肝癌DNMT3B低表达组的肿瘤无瘤生存率显著高于DNMT3B高表达组。
|
图 2 肝癌患者不同DNMT3B表达程度的肿瘤无瘤生存率与肿瘤特异性生存率 Fig.2 Tumor-free survival rate and tumor-specific survival rate of HCC patients with different levels of DNMT3B expression. A: Recurrence-free survival; B: Disease-specific survival. |
单因素、多因素分析显示,肿瘤大小、肝内转移、血管侵犯、DNMT3B的表达与肝癌中患者肿瘤无瘤生存率有显著相关性(P < 0.05,表 2)。
| 表 2 肝癌各种影响因素对患者无瘤生存率的影响 Tab.2 Influence of different factors on tumor-free survival of HCC patients |
DNMT3B在肝癌中低表达组的1年肿瘤特异性生存率为90.4%,3年肿瘤特异性生存率为64.3%。DNMT3B在肝癌中高表达组1年肿瘤特异性生存率分别为81.4%,3年肿瘤特异性生存率分别为45.5%。对数秩检验结果显示,两组总生存率差异有统计学意义(P=0.014,图 2B),肝癌DNMT3B低表达组的肿瘤特异性生存率高于DNMT3B高表达组。
单因素、多因素分析显示,肿瘤大小、肝内转移、血管侵犯、DNMT3B的表达与肝癌患者肿瘤特异性生存率有显著相关性(P < 0.05,表 3)。
| 表 3 肝癌各种影响因素对患者肿瘤特异性生存率的影响 Tab.3 Influence of different factors on tumor-specific survival rate of patients with HCC |
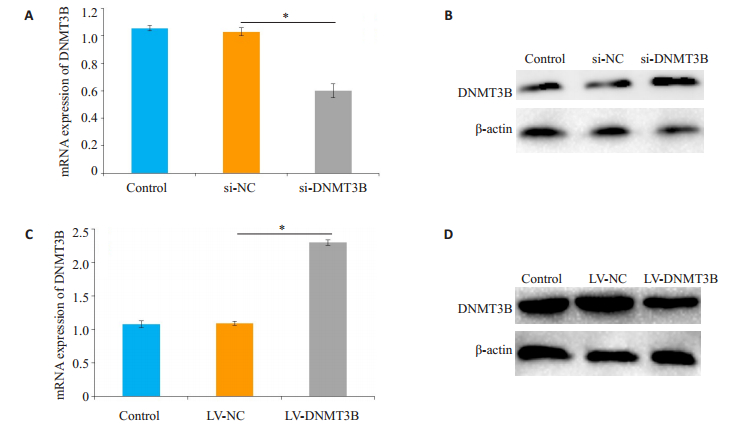
Huh-7细胞转染si-DNMT3B 48 h后,qRT-PCR检测显示,实验组细胞的DNMT3B mRNA水平显著下调,与空白对照组和阴性对照组比较差异有统计学意义(P=0.021,图 3A)。Western blot检测显示,实验组细胞的DNMT3B蛋白表达水平显著降低,与空白对照组和阴性对照组比较差异有统计学意义(P=0.037,图 3B)。Huh-7细胞转染慢病毒成功后,qRT-PCR检测显示,实验组细胞的DNMT3B mRNA水平显著上升,与空白对照组和阴性对照组比较差异有统计学意义(P=0.039,图 3C)。Western blot检测其蛋白质水平显示,实验组细胞的DNMT3B蛋白表达水平显著上升,与空白对照组和阴性对照组比较差异有统计学意义(P=0.011,图 3D)。
|
图 3 过表达与沉默DNMT3B后Huh-7细胞中DNMT3B的mRNA和蛋白水平 Fig.3 mRNA and protein expression of DNMT3B in Huh-7 cells transfected with si-DNMT3B and LV-DNMT3B. A: Expression of si-DNMT3B mRNA (*P < 0.05); B: Protein expression of si-DNMT3B; C: Expression of LVDNMT3B mRNA (*P < 0.05); D: Protein expression of LV-DNMT3B. |
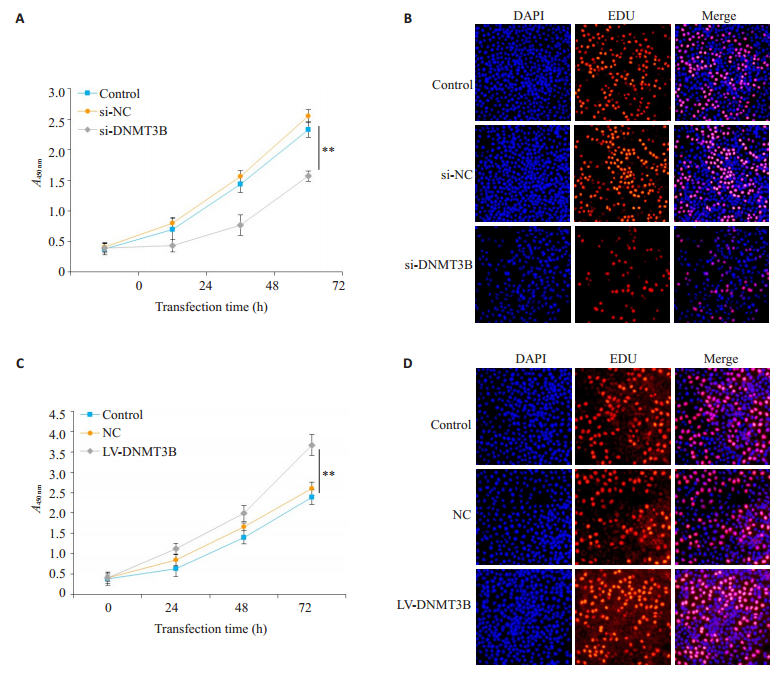
Huh-7细胞成功转染si-DNMT3B后,通过CCK-8实验检测细胞增殖情况,结果显示,与空白对照组和阴性对照组比较,随着转染时间的延长,实验组细胞的吸光度值下降速率逐渐增大,在转染后72 h降低最明显(P=0.018,图 4A)。通过细胞EDU摄取情况来检测细胞增殖情况,结果显示,实验组细胞的EDU摄取能力显著下降,与空白对照组和阴性对照组比较差异有统计学意义(P=0.033,图 4B)。Huh-7细胞转染慢病毒成功后,通过CCK-8实验检测细胞增殖情况,结果显示,与空白对照组和阴性对照组比较,随着转染时间的延长,实验组细胞的吸光度值逐渐升高,在转染后72 h升高的最为显著(P=0.011,图 4C)。通过细胞EDU摄取情况来检测细胞增殖情况,结果显示,实验组细胞的EDU摄取能力显著上升,与空白对照组和阴性对照组比较差异有统计学意义(P=0.021,图 4D)。
|
图 4 过表达与沉默DNMT3B后对Huh-7细胞增殖能力影响 Fig.4 Effect of transfection with si-DNMT3B and LV-DNMT3B on proliferation of Huh-7 cells. A, C: CCK8 assay of cell proliferation (**P < 0.05); B, D: Cell proliferation detected by EDU uptake. |
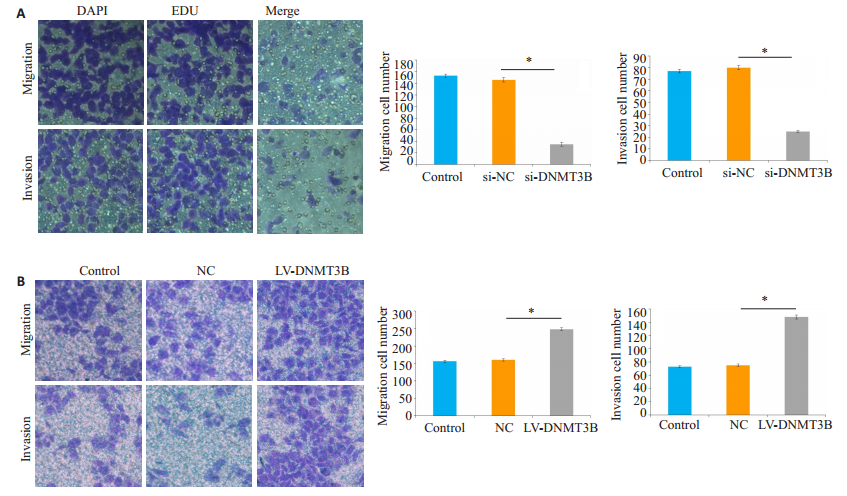
与空白对照组和阴性对照组比较,实验组细胞的侵袭迁移细胞数目减少,差异有统计学意义(P=0.016,图 5A)。而空白对照组和阴性对照组细胞侵袭迁移的数目差异没有统计学意义。Huh-7细胞转染慢病毒成功后,通过Transwell实验检测细胞侵袭迁移能力,结果显示,与空白对照组和阴性对照组比较,实验组细胞的侵袭迁移细胞数目显著增加,差异有统计学意义(P=0.014,图 5B)。而空白对照组和阴性对照组细胞侵袭迁移的数目差异没有统计学意义(P>0.05)。
|
图 5 过表达与沉默DNMT3B后对Huh-7细胞侵袭及迁移能力的影响 Fig.5 Effects of si-DNMT3B (A) and LV-DNMT3B (B) on invasion and migration of Huh-7 cells (*P < 0.05). |
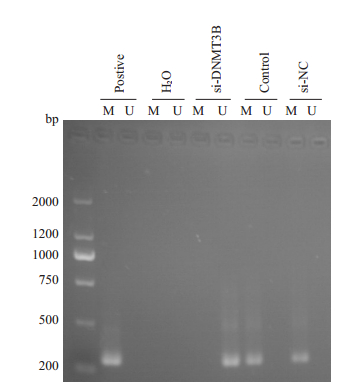
甲基化特异性PCR结果显示,与空白对照组和阴性对照组比较,实验组细胞的LATS1甲基化水平显著降低(P=0.001,图 6)。而空白对照组和阴性对照组细胞侵袭转移的数目差异没有统计学意义。
|
图 6 转染si-DNMT3B后对LATS1的甲基化水平影响 Fig.6 Effect of transfection with si-DNMT3B on methylation level of LATS1 in Huh-7 cells. |
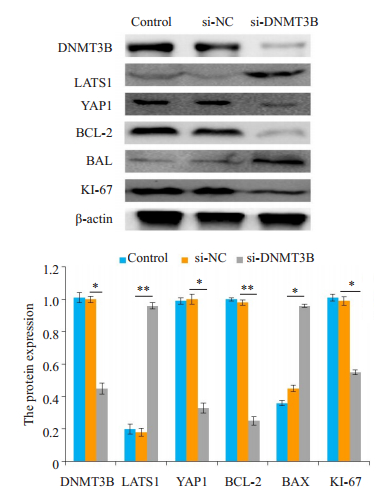
Western blot结果显示,与空白对照组和阴性对照组比较,实验组细胞的LATS1和BAX的表达水平显著升高(P < 0.05),同时,DNMT3B、YAP1、BCL-2、KI-67的表达水平显著降低(P < 0.05,图 7)。空白对照组和阴性对照组细胞侵袭转移的数目差异没有统计学意义。
|
图 7 沉默DNMT3B对Hippo信号通路相关基因表达的影响 Fig.7 Effect of silencing DNMT3B on the expression of Hippo signaling pathway-related genes in Huh-7 cells. *P < 0.05, **P < 0.01. |
研究表明肿瘤细胞中抑癌基因的甲基化失活是促进肿瘤转移的主要基础,当基因启动子区域出现甲基化改变,基因表达就会出错,其结果是基因表达沉默产生非正常蛋白质,干扰正常细胞的构成,使细胞的功能和身份识别遭到大规模破坏,细胞变异并导致癌症[11-12]。DNMT3B是一种从头甲基转移酶,可以完全甲基化靶基因的启动子区域,并且在许多恶性肿瘤中高表达,例如白血病[13],黑色素瘤[14]和膀胱癌[15]。最新研究发现DNMT3B对DNA的从头甲基化起到了主要作用,可使基因启动子区完全甲基化,其可作为防止抑癌基因甲基化失活的重要治疗靶点[16]。在肺癌中DNMT3B可介导MEG3甲基化而低表达,激活Akt/p70S6K/S6轴与HIF-1α,引起支气管上皮恶性转化,表明肿瘤细胞的恶化依赖抑癌基因的甲基化失活[17]。同样,在肝癌中DNMT3B的高表达可造成抑癌基因甲基化失活,直接影响肝癌患者术后肿瘤的复发与转移[18]。此外, 使用去甲基化药物抑制DNMT3B表达能降低了肿瘤抑制基因的甲基化程度,从而恢复这些基因的表达,显着降低肿瘤细胞的增殖和侵袭能力[19-20]。目前,作用于DNMT3B的去甲基化药物如地西他滨已经在患者中单独使用或与化疗联合使用,用于治疗抑癌基因的甲基化改变[21]。本研究结果与之前的研究一致,并进一步表明,DNMT3B的高表达能够增加患者的肿瘤体积,肝内转移和血管侵犯的情况。特别是,肝癌DNMT3B高表达组比DNMT3B低表达组表现出更多的恶性肿瘤的浸润性生长和较低生存率,这表明DNMT3B能够促进肿瘤细胞增殖、侵袭,并促进癌细胞的迁移。
那么,DNMT3B到底是如何促进肝癌细胞的增殖与侵袭的呢?研究表明,LATS1是Hippo信号通路的核心因子,并已明确LATS1基因启动子区存在大量可发生甲基化的CpG岛位点,在多种恶性肿瘤中LATS1发生DNA甲基化而失活,激活YAP1,调控多种促癌因子,推动肝癌的恶性发展[20-22]。LATS1基因在肝癌中存在高度甲基化并低表达的情况,LATS1的低表达导致YAP1活化,导致其活化进入细胞核,活化的YAP1激活MAGL并促进肿瘤的恶性发展[24]。作为Hippo信号通路的核心因子,LATS1在调节下游癌基因YAP的表达中起关键作用[25-27]。据报道LATS1的缺失导致各种类型的癌症的形成,包括神经胶质瘤[28]、胃癌[29]和转移性前列腺癌[30]。此外,LATS1活性受到肾细胞癌CpG岛甲基化的高度抑制,并导致46.7%(14/30)肾癌组织中LATS1低表达,表明LATS1高甲基化在肾癌中LATS1的下调中起重要作用[31]。由此推测,DNMT3B通过诱导LATS1的甲基化从而促进肝癌细胞的增殖和侵袭。
本研究通过免疫组化的方法检测了175例原发性肝癌患者的DNMT3B表达情况,我们分析了DNMT3B的表达与患者临床病理特征及预后的关系。本研究发现DNMT3B在肝癌中阳性表达率显著高于癌旁组织,DNMT3B在肝癌中的高表达与肿瘤大小、血管侵犯和肝内转移有密切的关系。通过分析在肝癌中DNMT3B高表达患者的生存率,本研究发现DNMT3B高表达组表现出较低的1年和3年肿瘤无瘤生存率与肿瘤特异性生存率。同时,高DNMT3B的表达与肝癌的不良预后有密切关系。为了进一步探讨DNMT3B在肝癌恶性发展中的作用,利用siRNA干扰DNMT3B的表达,检测Huh-7细胞中沉默DNMT3B后对细胞增殖与侵袭能力的影响。结果显示,沉默DNMT3B后Huh-7细胞的增殖与侵袭能力显著受到抑制。为进一步阐明沉默DNMT3B在Huh-7细胞中调控细胞增殖侵袭的分子机制,使用特异性PCR检测肝癌细胞LATS1的甲基化情况,结果发现沉默DNMT3B显著抑制LATS1的甲基化水平,恢复其正常表达,并抑制癌基因YAP1的表达,激活Hippo信号通路,发挥其抑癌作用进而抑制肝癌细胞的增殖与侵袭能力。
总之,本研究结果表明在肝癌中DNMT3B的表达量较高,DNMT3B的高表达与肿瘤大小、血管侵犯、肝内转移密切相关。生存分析结果显示DNMT3B高表达患者的肿瘤无瘤生存率与肿瘤特异性生存率更低。本研究表明DNMT3B的高表达能够增加肿瘤的恶性程度,同时降低患者的生存率。本研究在细胞水平验证了沉默DNMT3B对肝癌细胞增殖侵袭能力的影响以及其分子机制,为肝癌的治疗提供了理论基础。当然,本次临床研究仍有限,未来的研究中将更多的探讨DNMT3B影响肝癌发生发展的具体的机制以及针对DNMT3B基因特异性靶向治疗。
| [1] |
Siegel RL, Miller KD, Jemal A. Cancer statistics, 2017[J]. CA Cancer J Clin, 2017, 67(1): 7-30. DOI:10.3322/caac.21387 |
| [2] |
Greten TF, Wang XW, Korangy F. Current concepts of immune based treatments for patients with HCC: from basic science to novel treatment approaches[J]. Gut, 2015, 64(5): 842-8. DOI:10.1136/gutjnl-2014-307990 |
| [3] |
Chen W, Zheng R, Baade PD, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2): 115-32. DOI:10.3322/caac.21338 |
| [4] |
Pang RW. Biology of hepatocellular carcinoma[J]. Ann Surg Oncol, 2008, 15(8): 962-71. |
| [5] |
Wu H, Zhang Y. Reversing DNA methylation: mechanisms, genomics, and biological functions[J]. Cell, 2014, 156(1/2): 45-68. |
| [6] |
Subramaniam D, Thombre R, Dhar A, et al. DNA methyltransferases: a novel target for prevention and therapy[J]. Front Oncol, 2014, 4(1): 80-91. |
| [7] |
Chou ST, Peng HY, Mo KC, et al. MicroRNA-486-3p functions as a tumor suppressor in oral cancer by targeting DDR1[J]. J Exp Clin Cancer Res, 2019, 38(1): 281-92. DOI:10.1186/s13046-019-1283-z |
| [8] |
Tang X, Tu G, Yang G, et al. Autocrine TGF-β1/miR-200s/miR-221/ DNMT3B regulatory loop maintains CAF status to fuel breast cancer cell proliferation[J]. Cancer Lett, 2019, 452(1): 79-89. |
| [9] |
Okano M, Bell DW, Haber DA, et al. DNA methyltransferases Dnmt3a and Dnmt3b are essential for de novo methylation and mammalian development[J]. Cell, 1999, 99(3): 247-57. DOI:10.1016/S0092-8674(00)81656-6 |
| [10] |
Yang XJ, Han H, De Carvalho DD, et al. Gene body methylation can alter gene expression and is a therapeutic target in cancer[J]. Cancer Cell, 2014, 26(4): 577-90. DOI:10.1016/j.ccr.2014.07.028 |
| [11] |
Siegel RL, Miller KD, Jemal A. Cancer statistics, 2016[J]. CA Cancer J Clin, 2016, 66(1): 7-30. DOI:10.3322/caac.21332 |
| [12] |
Huang B, Jiang C, Zhang R. Epigenetics: the language of the cell[J]. Epigenomics, 2014, 6(1): 73-88. DOI:10.2217/epi.13.72 |
| [13] |
Niederwieser C, Kohlschmidt J, Volinia S, et al. Prognostic and biologic significance of DNMT3B expression in older patients with cytogenetically normal primary acute myeloid leukemia[J]. Leukemia, 2015, 29(3): 567-75. DOI:10.1038/leu.2014.267 |
| [14] |
Micevic G, Muthusamy V, Damsky W, et al. DNMT3b modulates melanoma growth by controlling levels of mTORC2 component RICTOR[J]. Cell Rep, 2016, 14(9): 2180-92. DOI:10.1016/j.celrep.2016.02.010 |
| [15] |
Qiu W, Lin J, Zhu Y, et al. Kaempferol modulates DNA methylation and downregulates DNMT3B in bladder cancer[J]. Cell Physiol Biochem, 2017, 41(4): 1325-35. DOI:10.1159/000464435 |
| [16] |
Chhabra R. miRNA and methylation: a multifaceted liaison[J]. Chembiochem, 2015, 16(2): 195-203. DOI:10.1002/cbic.201402449 |
| [17] |
Zhou C, Huang C, Wang J, et al. LncRNA MEG3 downregulation mediated by DNMT3b contributes to nickel malignant transformation of human bronchial epithelial cells via modulating PHLPP1 transcription and HIF-1α translation[J]. Oncogene, 2017, 36(27): 3878-89. DOI:10.1038/onc.2017.14 |
| [18] |
Saito Y, Kanai Y, Sakamoto M, et al. Expression of mRNA for DNA methyltransferases and methyl-CpG-binding proteins and DNA methylation status on CpG islands and pericentromeric satellite regions during human hepatocarcinogenesis[J]. Hepatology, 2001, 33(3): 561-8. DOI:10.1053/jhep.2001.22507 |
| [19] |
Wang N, Duan H, Zhang C, et al. The LINC01186 suppresses cell proliferation and invasion ability in papillary thyroid carcinoma[J]. Oncol Lett, 2018, 16(5): 5639-44. |
| [20] |
Zhou GX, Li XY, Zhang Q, et al. Effects of the hippo signaling pathway in human gastric cancer[J]. Asian Pac J Cancer Prev, 2013, 14(9): 5199-205. DOI:10.7314/APJCP.2013.14.9.5199 |
| [21] |
Viet CT, Dang D, Ye Y, et al. Demethylating drugs as novel analgesics for cancer pain[J]. Clin Cancer Res, 2014, 20(18): 4882-93. DOI:10.1158/1078-0432.CCR-14-0901 |
| [22] |
Moroishi T, Hayashi T, Pan WW, et al. The hippo pathway kinases LATS1/2 suppress cancer immunity[J]. Cell, 2016, 167(6): 1525-39. DOI:10.1016/j.cell.2016.11.005 |
| [23] |
He M, Zhou Z, Shah AA, et al. New insights into posttranslational modifications of hippo pathway in carcinogenesis and therapeutics[J]. Cell Div, 2016, 11(2): 4-15. |
| [24] |
Zhang J, Liu Z, Lian Z, et al. Monoacylglycerol lipase: a novel potential therapeutic target and prognostic indicator for hepatocellular carcinoma[J]. Sci Rep, 2016, 6(12): 35784-95. |
| [25] |
Wu H, Zhang W, Wu ZR, et al. miR-29c-3p regulates DNMT3B and LATS1 methylation to inhibit tumor progression in hepatocellular carcinoma[J]. Cell Death Dis, 2019, 10(2): 48-59. DOI:10.1038/s41419-018-1281-7 |
| [26] |
Li L, Dong L, Wang Y, et al. Lats1/2-Mediated alteration of hippo signaling pathway regulates the fate of bone marrow-derived mesenchymal stem cells[J]. Biomed Res Int, 2018, 26(8): 4387932-43. |
| [27] |
Nantie LB, Young RE, Paltzer WG, et al. Lats1/2 inactivation reveals hippo function in alveolar type Ⅰ cell differentiation during lung transition to air breathing[J]. Development, 2018, 145(21): 2506-17. |
| [28] |
Ji T, Liu D, Shao W, et al. Decreased expression of LATS1 is correlated with the progression and prognosis of glioma[J]. J Exp Clin Cancer Res, 2012, 31(3): 67-79. |
| [29] |
Xu Y, Yu X, Wei C, et al. Over-expression of oncigenic pesudogene DUXAP10 promotes cell proliferation and invasion by regulating LATS1 and β-catenin in gastric cancer[J]. J Exp Clin Cancer Res, 2018, 37(1): 13-24. DOI:10.1186/s13046-018-0684-8 |
| [30] |
Zhao B, Li L, Wang L, et al. Cell detachment activates the Hippo pathway via cytoskeleton reorganization to induce anoikis[J]. Genes Dev, 2012, 26(1): 54-68. DOI:10.1101/gad.173435.111 |
| [31] |
Chen KH, He J, Wang DL, et al. Methylation-associated inactivation of LATS1 and its effect on demethylation or overexpression on YAP and cell biological function in human renal cell carcinoma[J]. Int J Oncol, 2014, 45(6): 2511-21. DOI:10.3892/ijo.2014.2687 |
 2019, Vol. 39
2019, Vol. 39

