2. 陕西省中医药管理局中药药效机制与物质基础重点研究室,陕西 咸阳 712046;
3. 陕西省中药基础与新药研究重点实验室,陕西 咸阳 712046;
4. 中国兵器工业五二一医院,陕西 西安 710065;
5. 北京大学医学部药理学系,北京 100191
2. Key Laboratory of Pharmacodynamics and Material Basis of Chinese Medicine of Shaanxi Administration of Traditional Chinese Medicine, Xianyang 712046, China;
3. Shaanxi Key Laboratory of Traditional Medicine Foundation and New Drug Research, Xianyang 712046, China;
4. Department of Gastroenterology, 521 Hospital of Norinco Group, Shaanxi, Xi'an, 710065, China;
5. School of Basic Medicine, Peking University Health Science Center, Beijing, 100191, China
乳腺癌是女性最常见的癌症之一,约占全部癌症病例的23%,占癌症死亡病例的11.6% [1]。乳腺癌目前的主要治疗手段包括手术治疗,放射治疗,化学治疗,内分泌治疗,分子靶向治疗等。虽然以上治疗显著降低了乳腺癌死亡率,考虑到副作用和经济成本,乳腺癌患者迫切需要低毒性和低成本的新疗法。多项临床研究表明,定期服用阿司匹林可以提高乳腺癌的生存率[2-7]。此外,从阿司匹林治疗心血管疾病的大型随机试验中收集的数据显示,服用阿司匹林的受试者降低了癌症死亡率的风险[8]。近年来,多项研究表明阿司匹林在前列腺癌[9]、胰腺癌[10]和子宫内膜癌[11]等实体瘤中具有抗癌作用。MCF-7是一种常用的乳腺癌细胞株,被证明是一种适用于全球乳腺癌研究的模型细胞株,包括抗癌药物的研究[12]。我们前期实验表明阿司匹林可以抑制人乳腺癌MCF-7细胞的增殖,但阿司匹林抗人乳腺癌MCF-7细胞增殖的分子机制仍不清楚。
分子靶向治疗是指在肿瘤分子细胞生物学的基础上,利用肿瘤组织或细胞所具有的特异性结构分子作为靶点,使用某些能与这些靶分子特异结合的抗体、配体等达到直接治疗或导向治疗目的的一类疗法。在本研究中,我们引入生物信息学分析作为一种有效的方法,通过搜索公共数据库预测并分析阿司匹林抑制人乳腺癌细胞增殖的候选基因。
我们在DrugBank5.1.3中发现阿司匹林的直接作用蛋白靶点(DPTs),分析了阿司匹林DPTs的蛋白-蛋白相互作用(PPI)网络和信号通路,并筛选出6个基因(EDNRA,IKBKB,NFKB2,NFKBIA,PTGS2和TP53)作为阿司匹林在人乳腺癌中最重要的靶基因。同时,我们通过比较TCGA数据库中乳腺肿瘤样本与正常样本的转录组数据,发现乳腺肿瘤过表达的基因。我们找出了在人乳腺癌中阿司匹林最重要的靶基因和人乳腺癌样品中均出现的4个基因作为阿司匹林抗人乳腺癌增殖的潜在治疗靶点,并通过蛋白免疫印迹实验证实了我们的预测,并为分析阿司匹林甚至其他药物的作用靶点和作用机制提供新的方法。
1 材料和方法 1.1 识别阿司匹林直接作用靶点蛋白从Drugbank(https://www.drugbank.ca)中查找阿司匹林的分子信息及相应作用靶点,确定阿司匹林直接靶蛋白DPTs。
1.2 阿司匹林DPTs的蛋白-蛋白相互作用网络及其KEGG信号通路富集设定物种为人种,以直接关联不超过50个相互作用体,P < 0.05作为界限标准,采用STRING(https://string-db.org/cgi/input.pl)在线构建阿司匹林DPTs的PPI网络,并分析阿司匹林DPTs的KEGG信号通路。
1.3 探讨阿司匹林DPTs在人乳腺癌中的基因组学数据我们借助cBio Portal平台(cBio Cancer Genomics Portal)(https://cbioportal.org)和OncoPrint考察了乳腺肿瘤中6个阿司匹林DPTs(EDNRA,IKBKB,NFKB2,NFKBIA,PTGS2和TP53)的表达变化。其中EDNRA,IKBKB,NFKB2,NFKBIA,PTGS2和TP53及侵润性人乳腺癌作为限定条件。
1.4 鉴定阿司匹林对人乳腺癌潜在的治疗靶点基因通过癌症基因组图谱(The Cancer Genome Atlas,TCGA)(https://tcga-data.nci.nih.gov)数据库获取人乳腺癌癌症组织与正常组织的基因组数据,利用DECenter分析差异过表达基因(P < 0.05)(记为A组)。用STRING和Cytoscape预测6个阿司匹林DPTs相互关联的基因(记为B组)。将以上两组数据求交集,得到阿司匹林在人乳腺癌中潜在的治疗靶点基因。
1.5 阿司匹林治疗人乳腺癌潜在靶基因的基因本体论分析利用Gene Ontology进行基因本体论(GO;http://www.geneontology.org/)功能富集分析。
1.6 细胞培养MCF-7细胞由含10% FBS的DMEM培养基培养,经0.25%胰蛋白酶消化传代,置于37 ℃,5% CO2的细胞培养箱培养。实验组和对照组分别用阿司匹林和等量DMSO处理。
1.7 Western blot法检测MCF- 7中CDC25C,TPX2,CDC20,PLK1蛋白水平将人乳腺癌细胞MCF-7分为3组,分别经0、5、10 mmol/L阿司匹林处理24 h。倾去上清,PBS洗涤3次。用预冷的RIPA裂解液裂解30 min后,BCA试剂盒检测蛋白质浓度。每个样品中加入等体积的5× Loading buffer,混匀后,沸水浴煮10 min。每孔取40 μg样品上样,经12% SDS-PAGE分离,分离成功后将目的蛋白电转至PVDF膜,恒流0.2 A转膜2 h,室温条件下在5%脱脂奶粉溶液中封闭1 h,加1: 1000稀释的兔抗人GAPDH抗体、兔抗人CDC25C抗体、兔抗人TPX2抗体、兔抗人CDC20抗体、兔抗人PLK1抗体,4 ℃孵育过夜;TBST洗涤3次,加1 : 5000稀释的HRP标记的山羊抗兔IgG或HRP标记的山羊抗小鼠IgG,室温孵育1 h;TBST洗涤3次,以ECL发光法分析各蛋白的表达量。以GAPDH为内参。
2 结果 2.1 阿司匹林的直接作用靶点阿司匹林显弱酸性,可作为解热镇痛药、非甾体类消炎药、抗风湿药、心血管预防药、中枢神经系统药、溶栓药、抗血小板聚集药。阿司匹林直接作用的蛋白靶点共11个(PTGS1、PTGS2、AKR1C1、PRKAA1、EDNRA、IKBKB、TP53、HSPA5、RPS6KA3、NFKBIA和NFKB2)(表 1)。
| 表 1 使用DRUGBANK确认阿司匹林的直接作用靶点(DPTs) Tab.1 Identification of direct protein targets of aspirin by searching DRUGBANK |
采用STRING研究了11个阿司匹林直接作用靶点之间的蛋白-蛋白相互作用及相关的信号通路(图 1,表 2)。最显著前5条KEGG信号通路包括NF-κB信号通路,神经营养因子信号通路,胰岛素抵抗信号通路,肿瘤信号通路,小细胞肺癌信号通路。其中,肿瘤信号通路中包含6个阿司匹林直接作用靶点EDNRA,IKBKB,NFKB2,NFKBIA,PTGS2和TP53(表 2)。
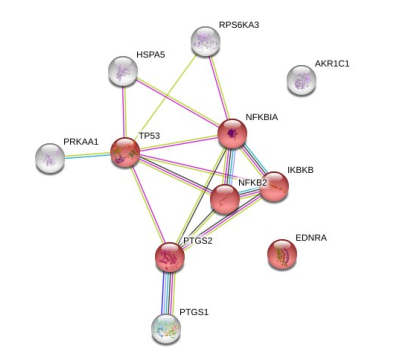
|
图 1 阿司匹林靶基因(PTGS1,PTGS2,AKR1C1,PRKAA1,EDNRA,IKBKB,TP53,HSPA5,RPS6KA3,NFKBIA,NFKB2)蛋白-蛋白相互作用(PPI)网络 Fig.1 Protein-protein interaction (PPI) network of the target genes of aspirin (PTGS1, PTGS2, AKR1C1, PRKAA1, EDNRA, IKBKB, TP53, HSPA5, RPS6KA3, NFKBIA, NFKB2). The 6 nodes in red color are related to cancer. |
| 表 2 阿司匹林直接作用靶点的top5 KEGG通路列表 Tab.2 Top 5 KEGG pathways associated with the DPTs of aspirin |
为了了解6个与肿瘤相关的阿司匹林DPTs(EDNRA,IKBKB,NFKB2,NFKBIA,PTGS2,TP53)的表达变化及功能,通过cBio portal研究这些基因在人乳腺癌患者中的遗传改变。我们检测了EDNRA,IKBKB,NFKB2,NFKBIA,PTGS2和TP53在人乳腺癌患者中的肿瘤基因组改变和临床表达特征。在3项人乳腺癌研究中(Broad研究[13]、TCGA研究[14]和France研究[15]),EDNRA,IKBKB,NFKB2,NFKBIA,PTGS2和TP536个基因的变化范围在0.6%到27%之间,TP53发生变化比例最大(图 2A)。我们通过3项研究总结了多种基因的变化,通过OncoPrint可视化结果表明,303例患者(27%)至少有一种EDNRA,IKBKB,NFKB2,NFKBIA,PTGS2和TP53发生改变(图 2B)。EDNRA和PTGS2的改变表现为基因扩增,纯和缺失和错义突变。IKBKB和NFKB2的改变表现为基因扩增,纯和缺失,错义突变和截短突变。NFKBIA的改变表现为基因扩增和错义突变。TP53的改变表现为基因扩增,纯和缺失,错义突变,非移码突变,种系突变和截短突变。
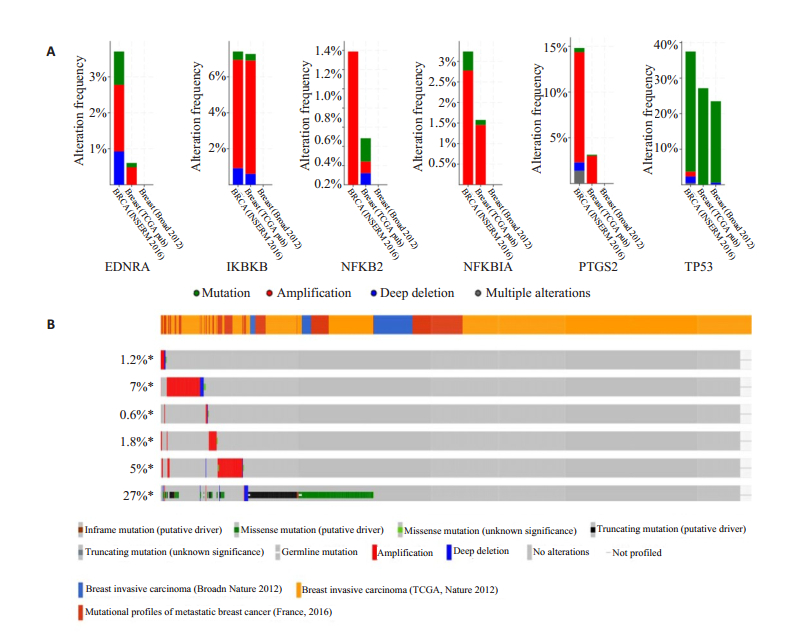
|
图 2 在cBio癌症基因组学网关于人乳腺癌研究中,分析6个阿司匹林DPTs(EDNRA,IKBKB,NFKB2,NFKBIA,PTGS2和TP53)的改变 Fig.2 Analysis of genetic alterations of 6 DPTs of aspirin (EDNRA, IKBKB, NFKB2, NFKBIA, PTGS2 and TP53) in human breast cancer studies embedded in cBio cancer genomics portal. A: Overview of the changes in EDNRA, IKBKB, NFKB2, NFKBIA, PTGS2 and TP53 in genomics datasets available in 3 different studies; B: Visual summary of mutations across a set of human breast cancer samples based on a query of the 6 genes in OncoPrint. Distinct genomic alterations including mutations, copy number alterations, exemplified by gene amplifications and homozygous deletions, are summarized and color coded (as % changes) in the affected genes in individual tumor samples. Each row represents a gene, and each column represents a tumor sample. |
基于STRING数据库的在线PPI分析,我们将与阿司匹林直接作用靶点相关的基因进行了可视化(图 3)。我们发现了很多与阿司匹林抗肿瘤作用相关的基因。这些基因可能为阿司匹林治疗人乳腺癌的研究提供新的思路。KEGG pathway分析表明,这些基因主要参与细胞周期,人类嗜T淋巴细胞病毒Ⅰ型感染,EB病毒感染,病毒性肿瘤,肿瘤等(表 3)。

|
图 3 与EDNRA,IKBKB,NFKB2,NFKBIA,PTGS2和TP53相关的基因 Fig.3 Visual display of the gene network connected to EDNRA, IKBKB, NFKB2, NFKBIA, PTGS2 and TP53. |
| 表 3 阿司匹林潜在作用靶点的top5 KEGG通路列表 Tab.3 TOP 5 KEGG pathways in the PPI of the DPTs of aspirin |
我们通过下载TCGA数据库中乳腺癌样本与乳腺正常组织的转录组数据,并进行差异基因分析,确定了乳腺癌中过表达的基因。1216例乳腺癌样本中,10 220个过表达差异基因。过表达前50位基因主要富集于卵母细胞减数分裂、细胞周期KEGG通路和孕酮介导的卵母细胞成熟(表 4)。
| 表 4 人乳腺癌top100过表达基因的KEGG通路列表 Tab.4 KEGG pathways in the top100 over-expressed genes in breast cancer vs normal tissue |
通过比较人乳腺癌中过表达基因(Top100)和与人乳腺癌中6个阿司匹林DPTs相互关联的基因(102个),我们发现了4个基因CDC25C、TPX2、CDC20和PLK1(图 4,表 5)是阿司匹林治疗人乳腺癌的潜在靶基因。
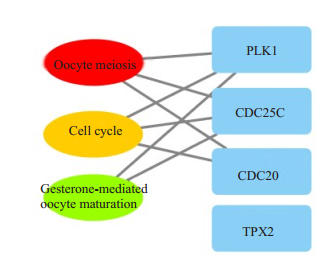
|
图 4 阿司匹林作用于乳腺癌的4个预测靶点的KEGG通路分析 Fig.4 KEGG pathway analysis of aspirin relatedfour predictive targets of breast cancer |
| 表 5 两组基因的交集基因 Tab.5 The intersection of genes between the two groups of genes |
对这4个基因的KEGG通路分析表明,CDC20,CDC25C和PLK1参与细胞周期和卵母细胞减数分裂通路,CDC25C和PLK1与孕酮介导的卵母细胞成熟信号相关(图 4)。这4个基因的功能注释表明PLK1与胞质分裂有关,CDC25C,TPX2和CDC20均与细胞周期有关(表 6)。
| 表 6 阿司匹林抗人乳腺癌增殖的潜在靶点的基因本体分析 Tab.6 Gene Ontology analysis of the potential targets of aspirin against human breast cancer proliferation |
为进一步验证生物信息学分析的阿司匹林抗乳腺癌细胞增殖的潜在靶点,我们评估了不同浓度阿司匹林处理24 h的MCF-7细胞中细胞周期和细胞分裂相关蛋白表达(图 5)。阿司匹林可以下调MCF-7中细胞周期及细胞分裂相关蛋白(CDC25C,TPX2,CDC20和PLK1)的表达,且呈剂量依赖性。
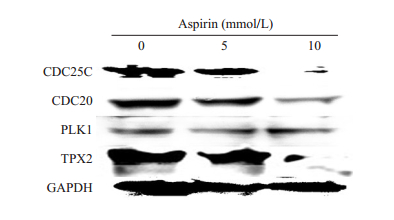
|
图 5 阿司匹林作用于MCF-7 24 h后,Western blot分析4个预测靶点(CDC25C, CDC20, PLK1和TPX2)的表达 Fig.5 Effects of aspirin on the expressions of the potential targets (CDC25C, CDC20, PLK1 and TPX2) of aspirin in MCF-7 cells. The cells were treated with aspirin at the indicated concentrations for 24 h. |
乳腺癌是全世界女性最常见的恶性肿瘤,发病率在逐年上升,且具有年轻化的趋势,其病死率居女性恶性肿瘤首位,为提高患者的生存率及生活质量,我们要寻求新的治疗方法以降低乳腺癌的复发率。大量的研究证明定期服用阿司匹林可以降低患癌症的风险[16-17]。亦有研究表明,使用阿司匹林可以降低乳腺癌的患癌率[18]。我们的前期研究结果显示:阿司匹林可抑制人乳腺癌细胞MCF-7的增殖能力。以往研究表明[19-20],细胞周期蛋白和细胞周期蛋白依赖性激酶(CDKs)的失调促进了肿瘤的生长。本研究中,我们借助系统生物信息学分析阐明阿司匹林及其靶蛋白在人乳腺癌MCF-7中的分子作用,通过分析TCGA数据库中乳腺癌与正常组织的过表达差异基因,进一步揭示阿司匹林抗人乳腺癌MCF-7增殖的潜在靶点(CDC25C,TPX2,CDC20,PLK1),主要参与细胞增殖及细胞分裂。
我们通过Western blot法分析验证了阿司匹林作用MCF-7后,细胞周期及细胞分裂相关蛋白(CDC25C,TPX2,CDC20,PLK1)的表达。这与前期研究结果是一致的[21-23],如Flavokawain A可以通过抑制CDC25C阻断G2期细胞增殖,诱导细胞凋亡;miR-142-3p靶向CDC25C,起到抑制肿瘤的作用;槲皮素-3-甲基醚通过CHK1-CDC25C-cyclin b1/CDK1途径引起G(2)/M阻滞,抑制人乳腺癌MCF-7及其拉帕替尼耐药细胞的增殖。TPX2基因与乳腺癌干细胞的发育密切相关,实验证明洛伐他汀通过作用TPX2发挥对乳腺肿瘤干细胞致敏作用[24]。沉默CDC20可以增加HER2阳性乳腺癌曲妥珠单抗获得性耐药细胞对曲妥珠单抗的敏感性[25]。PLK1是一种有丝分裂激酶,体内外实验证明PLK1抑制剂或敲除PLK1均能恢复HER2阳性乳腺癌对曲妥珠单抗的敏感性[26]。总而言之,4个基因(CDC25C,TPX2,CDC20,PLK1)与乳腺癌增殖有关。我们借助生物信息学和分子生物学确认它们是阿司匹林抑制HER2阳性乳腺癌增殖的潜在作用靶点。我们相信阿司匹林通过影响细胞周期起作用,阿司匹林可调节HER2阳性乳腺癌细胞中依赖于细胞周期蛋白丝氨酸/苏氨酸激酶PLK1的活性。我们的研究结果为阐明阿司匹林在抗乳腺癌增殖的分子机制提供了新的线索。
在药物发现过程中,确定药物靶标相互作用非常重要。尽管微阵列,蛋白质组学和其他高通量筛选分析方法已经用于寻找药物靶标,然而这些实验方法仍然非常昂贵、耗时,即使现在也难于普及。因此,各种计算模型被开发用来大规模预测药物靶标相互作用[27-28]。利用计算模型可以探索与阿司匹林作用相关的基因,便于进一步认识阿司匹林在疾病预防和治疗中的作用。然而,利用计算生物学预测药物靶点也存在一定的局限性,其主要依赖于数据库的完整性。越来越多的研究表明非编码RNA与基因表达和疾病进展息息相关,因此非编码RNA也是一类重要的药物靶点[27]。本研究中未发现阿司匹林可以作用的非编码RNA,是本研究的一个不足。我们拟将挖掘更多的数据库,探讨阿司匹林可能作用的非编码RNA靶点。
总之,系统生物信息学分析提供了一个便捷、灵活的方法,可以借助现有的药物信息通过PPI预测癌症发生可能的基因变化,帮助研究人员将基础研究转化为临床应用。这种分析方法可以减少实验室的冗余实验,节省资源。作为一种基于大数据源的新研究方法,系统生物信息学分析可进一步用于预测药物治疗疾病潜在的靶点基因。
| [1] |
Bray F, Ferlay J, Soerjomataram I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [2] |
Zhong SL, Zhang XH, Chen L, et al. Association between aspirin use and mortality in breast cancer patients: a meta-analysis of observational studies[J]. Breast Cancer Res Treat, 2015, 150(1): 199-207. DOI:10.1007/s10549-015-3300-z |
| [3] |
Huang XZ, Gao P, Sun JX, et al. Aspirin and nonsteroidal antiinflammatory drugs after but not before diagnosis are associated with improved breast cancer survival: a meta-analysis[J]. Cancer Causes Control, 2015, 26(4): 589-600. DOI:10.1007/s10552-015-0539-y |
| [4] |
Elwood PC, Morgan G, Pickering JE, et al. Aspirin in the treatment of cancer: reductions in metastatic spread and in mortality: a systematic review and Meta-Analyses of published studies[J]. PLoS One, 2016, 11(4): e0152402. DOI:10.1371/journal.pone.0152402 |
| [5] |
Blair CK, Sweeney C, Anderson KE, et al. NSAID use and survival after breast cancer diagnosis in post-menopausal women[J]. Breast Cancer Res Treat, 2007, 101(2): 191-7. DOI:10.1007/s10549-006-9277-x |
| [6] |
Fraser DM, Sullivan FM, Thompson AM, et al. Aspirin use and survival after the diagnosis of breast cancer: a population-based cohort study[J]. Br J Cancer, 2014, 111(3): 623-7. DOI:10.1038/bjc.2014.264 |
| [7] |
Holmes MD, Chen WY, Li L, et al. Aspirin intake and survival after breast cancer[J]. J Clin Oncol, 2010, 28(9): 1467-72. DOI:10.1200/JCO.2009.22.7918 |
| [8] |
Rothwell PM, Wilson M, Price JF, et al. Effect of daily aspirin on risk of cancer metastasis: a study of incident cancers during randomised controlled trials[J]. Lancet, 2012, 379(9826): 1591-601. DOI:10.1016/S0140-6736(12)60209-8 |
| [9] |
Assayag J, Pollak MN, Azoulay L. The use of aspirin and the risk of mortality in patients with prostate cancer[J]. J Urol, 2015, 193(4): 1220-5. DOI:10.1016/j.juro.2014.11.018 |
| [10] |
Fendrich V, Chen NM, Neef M, et al. The angiotensin-I-converting enzyme inhibitor enalapril and aspirin delay progression of pancreatic intraepithelial neoplasia and cancer formation in a genetically engineered mouse model of pancreatic cancer[J]. Gut, 2010, 59(5): 630-7. DOI:10.1136/gut.2009.188961 |
| [11] |
Matsuo K, Cahoon SS, Yoshihara K, et al. Association of LowDose aspirin and survival of women with endometrial cancer[J]. Obstet Gynecol, 2016, 128(1): 127-37. DOI:10.1097/AOG.0000000000001491 |
| [12] |
Comşa Ş, Cîmpean AM, Raica M. The story of MCF-7 breast cancer cell line: 40 years of experience in research[J]. Anticancer Res, 2015, 35(6): 3147-54. |
| [13] |
Banerji S, Cibulskis K, Rangel-Escareno C, et al. Sequence analysis of mutations and translocations across breast cancer subtypes[J]. Nature, 2012, 486(743): 405-9. |
| [14] |
Cancer Genome Atlas Network. Comprehensive molecular portraits of human breast tumours[J]. Nature, 2012, 490(7418): 61-70. DOI:10.1038/nature11412 |
| [15] |
Lefebvre C, Bachelot T, Filleron T, et al. Mutational profile of metastatic breast cancers: a retrospective analysis[J]. PLoS Med, 2016, 13(12): e1002201. DOI:10.1371/journal.pmed.1002201 |
| [16] |
Patrignani P, Patrono C. Aspirin and cancer[J]. J Am Coll Cardiol, 2016, 68(9): 967-76. DOI:10.1016/j.jacc.2016.05.083 |
| [17] |
Thorat MA, Cuzick J. Role of aspirin in cancer prevention[J]. Curr Oncol Rep, 2013, 15(6): 533-40. DOI:10.1007/s11912-013-0351-3 |
| [18] |
Clarke CA, Canchola AJ, Moy LM, et al. Regular and low-dose aspirin, other non-steroidal anti-inflammatory medications and prospective risk of HER2-defined breast cancer: the california teachers study[J]. Breast Cancer Res, 2017, 19(1): 52. DOI:10.1186/s13058-017-0840-7 |
| [19] |
Gnant M, Steger GG, Bartsch R. CDK4/6 inhibitors in luminal breast cancer[J]. Lancet Oncol, 2015, 16(1): 2-3. DOI:10.1016/S1470-2045(14)71188-X |
| [20] |
Santo L, Siu KT, Raje N. Targeting Cyclin-Dependent kinases and cell cycle progression in human cancers[J]. Semin Oncol, 2015, 42(6): 788-800. DOI:10.1053/j.seminoncol.2015.09.024 |
| [21] |
Jandial DD, Krill LS, Chen LX, et al. Induction of G2M arrest by flavokawain a, a Kava chalcone, increases the responsiveness of HER2-Overexpressing breast cancer cells to herceptin[J]. Molecules, 2017, 22(3): 462. DOI:10.3390/molecules22030462 |
| [22] |
Cao XC, Yu Y, Hou LK, et al. miR-142-3p inhibits cancer cell proliferation by targeting CDC 25C[J]. Cell Prolif, 2016, 49(1): 58-68. DOI:10.1111/cpr.12235 |
| [23] |
Li JX, Zhu F, Lubet RA, et al. Quercetin-3-methyl ether inhibits lapatinib-sensitive and -resistant breast cancer cell growth by inducing G2/M arrest and apoptosis[J]. Mol Carcinog, 2013, 52(2): 134-43. DOI:10.1002/mc.21839 |
| [24] |
Huang CY, Han Z, Wu DH. Effects of TPX2 gene on radiotherapy sensitization in breast cancer stem cells[J]. Oncol Lett, 2017, 14(2): 1531-5. DOI:10.3892/ol.2017.6277 |
| [25] |
Sabbaghi MA, Gil-Gomez G, Guardia CA, et al. Defective cyclin B1 induction in trastuzumab-emtansine (T-DM1) acquired resistance in HER2-positive breast cancer[J]. Clin Cancer Res, 2017, 23(22): 7006-19. DOI:10.1158/1078-0432.CCR-17-0696 |
| [26] |
Saatci Ö, Borgoni S, Akbulut Ö, et al. Targeting PLK1 overcomes TDM1 resistance via CDK1-dependent phosphorylation and inactivation of Bcl-2/xL in HER2-positive breast cancer[J]. Oncogene, 2018, 37(17): 2251-69. DOI:10.1038/s41388-017-0108-9 |
| [27] |
Chen X, Sun YZ, Zhang DH, et al. NRDTD: a database for clinically or experimentally supported non-coding RNAs and drug targets associations[J]. Database (Oxford), 2017(7): 1-6. |
| [28] |
Huang YA, You ZH, Chen X. A systematic prediction of DrugTarget interactions using molecular fingerprints and protein sequences[J]. Curr Protein Pept Sci, 2018, 19(5): 468-78. DOI:10.2174/1389203718666161122103057 |
 2019, Vol. 39
2019, Vol. 39

