临床上人工识读心电图(ECG)费时费力,诊断误差难以避免,为降低临床的诊断成本,如何借助计算机对ECG进行有效、准确的分析成为了生物信号领域的研究热点。但心律失常种类较多,不同病人发生心律失常时的ECG存在个体差异,自动检测和分类仍然是本领域的技术难点[1]。许多学者将ECG分析与深度学习结合起来,极大地推动了ECG分析算法的发展。有研究提出了一个基于人工神经网络的ECG异常检测方法[2],先提取ECG波形作为特征信息,然后进行模型的训练。但是,目前各种ECG波形提取方法的拟合能力较为一般[3],存在一定局限性。为了更好地反映样本的内在属性,有研究利用神经网络自动学习特征的特性,用卷积神经网络进行了ECG心拍正常和异常的检测[4],平均检测准确率达到了95.22%。然而该研究在训练集和测试集划分时采用的是“面向类” [5]分类思想,模型在测试见过的样本的时候获得很高的分数,但由于不同受试者之间存在个体差异性,所以导致其模型的泛化能力较差[6-7]。有研究基于“面向对象” [5]分类思想,首次将小波包熵和随机森林应用到ECG分类中,对心电信号进行小波分解,然后从分解系数中计算不同的熵作为代表特征,最后用随机森林建立心电分类模型[8]。虽然利用小波变换能提取多尺度特征,避免了对一些特征点的直接定位,但是缺点是难以使用这些小波变换得到的抽象特征[9]。
由于深度学习方法可以将特征学习和分类过程融合在一起,所以直接使用原始数据可以避免人工特征提取过程产生的偏差[9]。有文献使用了不同的深度学习方法[10-12],通过部分“专家介入”来对模型进行微调,大大减轻了个体差异带来的性能恶化,但对样本量少的类别的检测性能仍有提升空间。因此本文提出了一种利用卷积神经网络实现的深度学习方法,通过改进损失函数,有效地改善了样本不平衡对模型造成的消极影响。通过MIT-BIH心律失常数据库对比本文方法和其他文献方法,本文方法在考虑个体差异性的情况下,明显提高了分类性能,具有一定的先进性和创新性。
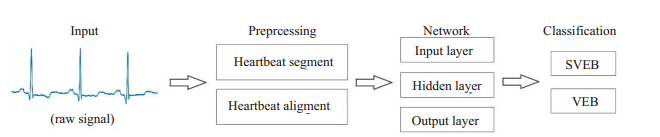
1 资料和方法系统的输入端输入原始心电信号,先经过预处理,把单个心拍从连续心电信号中分割出来,并将分割得到心拍进行对齐校正,然后进入网络进行分类器训练,最终输出分类决策结果(图 1)。
|
图 1 系统概述 Fig.1 Overview of the system. |
本文旨在创建一个鲁棒性强且高效的端到端心拍分类系统,因此使用原始的ECG信号作为数据输入,不作任何噪声滤除和基线修正等处理,可以最大程度上保留所有的心电信息,从而获得更好的表征进行分类[12]。
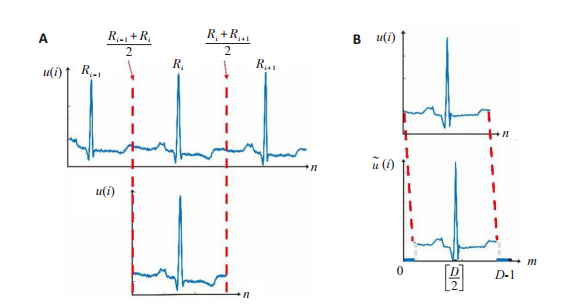
系统使用Xu等[12]提出的方法对连续心电信号进行心拍分割,先用Pan-Tompkins算法[13]得到R波位置,再根据其前后邻的R波位置分割出单个心拍(图 2A)。然后对每个心拍应用离差标准化[14],使所有的心拍都映射到相同的分布上,加快网络收敛速度,提高泛化能力。因上述截取到的每个心拍长度(采样点数目)各异,无法直接用于神经网络的训练,仍需作进一步校正,以确定一个具有普适性的心拍长度(D)。Xu等[12]将D取值为417个样本点,但是其忽略了导联脱落、强噪声等异常会使得心拍长度的统计出现偏差。本文统计了所有截取出来的心拍的长度,剔除了长度异常的心拍,并对剩下心拍的长度作单侧区间估计,取置信度为90%,得到置信区间为[0, 357]。本文取357作为本实验的最优心拍长度,即D=357,通过对其它不同D值时的心拍进行比较观察,当D=357时,每个心拍既能截取到足够的心动周期信息,也没有保留过多的冗余信息。心拍校正公式如下:
| $ {\tilde u_i}(m) = \left\{ {\begin{array}{*{20}{l}} {0, m < \left\lfloor {\frac{D}{2}} \right\rfloor - {n^*}\ \ or\ \ m > \left\lfloor {\frac{D}{2}} \right\rfloor + \left( {{L_i} - {n^*}} \right)}\\ {{u_i}\left( {m - \left\lfloor {\frac{D}{2}} \right\rfloor + {n^*}} \right), \ \ o\;{\rm{th}}\;erwise\ \ } \end{array}} \right. $ | (1) |
|
图 2 心拍分割(A)与心拍校正示例(B) Fig.2 Illustration of heartbeat segmentation (A) and heartbeat alignment (B). |
其中m=0, 1, …, D-1,
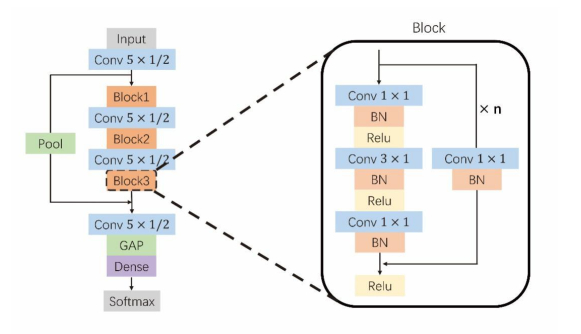
提出一个端到端的卷积神经网络心律失常分类模型架构(图 3)。网络输入为预处理后的一维ECG信号,长度为357,输出为每个心拍的预测分类的概率值。网络层中3种残差块(Block)的n值分别为1、2、3,每个Block之间的卷积层卷积核大小为5,步长为2。将第二层卷积层的输出与Block3的输出在通道上进行叠加,卷积之后做全局平均池化操作,最后软分类得到每个心拍的分类结果。
|
图 3 神经网络架构 Fig.3 Architecture of the proposed neural network. |
利用深度残差网络[15]的优点,在网络中间层使用跳跃式传递,一方面有利于梯度向下传播,另一方面,残差块中瓶颈[15]的存在可以使得网络参数极大减少。第二层卷积层与Block3之间的密集连接[16]可以使得网络在进行若干次卷积操作后依然可以直接利用到更原始的信息,同时有利于让梯度更好地向上传递。在主路径上,每个Block之间使用步长为2的卷积操作替代传统的池化层,同时起到特征提取和池化的作用,简化了网络结构。每一个卷积操作后都进行了批处理化[17],防止梯度弥散,使每一层输出的特征映射都映射到同一分布上,加快训练速度。在激活函数的选择上,使用卷积神经网络中最常用的非线性激活函数ReLu。然后用Network In Network [18]中提出的全局平均池化层替换传统卷积神经网络的全连接层。全局平均池化层对最后一层卷积的每一个特征映射求取均值,池化操作不需要进行参数更新,因此可以减少全连接层带来的参数数量,缩短网络训练时间,增强神经网络的表示能力,另一方面,又可以起到正则化的作用,防止过拟合[19]。最后将池化后的值经过Dense层连接成向量送进softmax得到各分类的概率值。
1.3 损失函数对于分类问题的损失函数,通常会先考虑交叉熵(Cross-Entropy),公式如下:
| $ C E=-\sum\limits_{i=1}^{c} y_{i}^{*} \log (\widehat{y_{i}}) $ | (2) |
其中yi为样本真实标签,
针对类别不平衡问题,有研究提出BatchWeighted Loss [20],公式如下:
| $ c{w_{i, clas{s_k}}} = 1 - \frac{{\sum\limits_{j = 1}^M {{1_{{y_{i, j = clas{s_{_k}}}}}}} }}{M} + \varepsilon $ | (3) |
| $ L=-\sum\limits_{j=1}^{M} c w_{i, y_{i j}} y_{i, j} \log \left(\hat{y}_{i, j}\right)+\lambda\|W\|_{2}^{2} $ | (4) |
其中yi, j表示第i个batch中第j个样本的标签,
| $ L = - \sum\limits_{i = 1}^m c {w_{i, \ \ clas{s_k}}}{\left( {1 - \widehat {{y_i}}} \right)^\gamma }{y_i}\log \left( {\widehat {{y_i}}} \right) + \lambda ||W||_2^2 $ | (5) |
γ是大于等于0的调制系数,用于调节类别学习的难易程度,当
使用目前国际上公认且热门的MIT-BIH心律失常数据库[21]验证本文提出的分类方法。该心律失常数据库包含了48条心电记录,取自47个不同的患者(记录编号201和202来自同一患者),每条30 min的记录都选自24 h心电记录中最具代表性的数据片段,采样率均为360 Hz。根据美国医疗仪器促进协会(AAMI)的国际标准(ANSI/AAMI EC57:2012)规范,MIT-BIH心律失常数据库中的15种心律失常类型分为了5类:正常或束支传导阻滞(N)、室上性异常(S)、心室异常(V)、融合类(F)和未知类(Q)(表 1)。
| 表 1 MIT-BIH心律失常数据库心拍类型 Tab.1 Heartbeat types in the MIT-BIH arrhythmia database |
依照AAMI规范,移除数据集中4条包含起搏心搏(P)的心电记录(102,104,107,217),并依照文献[22],以“面向对象”的思想,将数据集划分为训练集DS1和测试集DS2,每个集合各包含22条心电记录(表 2)。DS1和DS2的样本数量如表 3。
| 表 2 训练集DS1和测试集DS2中包含的ECG记录 Tab.2 ECG recordings included in the training dataset (DS1) and the test dataset (DS2) |
| 表 3 数据集详细 Tab.3 Dataset details |
根据ANSI/AAMI EC57规范,本研究专注于室上性异位心搏(SVEB)和室性异位心搏(VEB),即S类和Ⅴ类两类特定事件的表现,来衡量分类器的性能。表 3反映出数据集中存在严重的类别不平衡现象,S类心拍样本数量约占整个DS1的1.84%,Ⅴ类约占了DS1的7.42%。由于本文对损失函数作了改进,为验证新的损失函数在类别不平衡的情况有更好的学习能力,本文将实验分成两组,第一组实验采用不同的损失函数构成不同分类器,在相同的训练集DS1上训练,在测试集DS2上评估。第二组实验则是评估在引入不同程度“专家介入”情况下的表现。其中,第二组实验用于分类器训练的数据由两部分组成:全局(患者通用)和局部(特定于患者)数据,全局数据为DS1,局部数据包含DS2中每个患者最多前5 min的片段,但剩下的25 min片段对分类器来说仍是全新的[6]。局部数据包含了每个类别中相对少量的典型心拍,帮助模型学习一些未包括在全局数据集中的其它形式的心律失常[11]。该组实验通过分别引入前0、50、100和150个样本作为局部数据进行训练,引入的总样本数量少于测试集的1%。这符合AAMI允许最多使用每个病人记录前5 min的数据用于训练的规范[23]。
1.4.3 网络参数进行22折交叉验证[12],使用1cycle policy [24-25]设置学习率和动量,最小学习率为1e-2,最大学习率为1e-0,最小动量为0.85,最大动量为0.95。网络常规参数中,Batch大小为128,Epoch为150,权重衰减系数λ为0.008,调制系数γ为2。
1.5 评价指标使用灵敏度(SEN)、特异性(SPC)、受试者特性曲线(ROC)和AUC来反映模型性能。灵敏度和特异性公式如下:
| $ S E N=\frac{T P}{T P+F N} \times 100 \% $ | (6) |
| $ S P C=\frac{T N}{T N+F P} \times 100 \% $ | (7) |
TP表示真阳性样本个数,TN表示真阴性样本个数,FP表示假阳性样本个数,FN表示假阴性样本个数。SEN高意味着漏诊率低,SPC高意味着误诊率低。ROC曲线是以灵敏度为纵坐标,(1-特异性)为横坐标绘制的曲线。AUC是ROC曲线下面积,同时考虑分类器对于正例和负例的分类能力,在类别不平衡的情况下,依然能够对分类器的性能做出合理的评价。
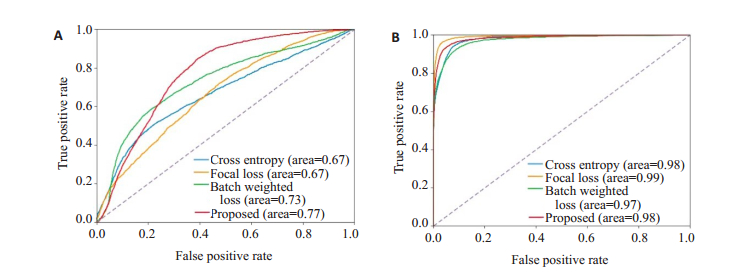
2 结果在不引入任何样本作为“专家介入”的条件下,模型在使用不同损失函数时的ROC曲线结果对比如图 4,本文提出的损失函数在S类事件的AUC为0.77,在Ⅴ类事件的AUC为0.98。
|
图 4 不同损失函数下的ROC曲线 Fig.4 ROC curves under different loss functions. A: ROC and AUC for SVEB; B: ROC and AUC for VEB. The AUC of the proposed loss function reached 0.77 and 0.98 for SVEB and VEB, respectively. |
本文提出的端到端心律失常分类系统与同样基于“面向对象”并且引入“专家知识”的文献在性能上作横向对比。其中,表 4引入了每个患者前5 min的片段作为局部数据的实验结果,本实验在S类事件上的灵敏度达到78.28%,在Ⅴ类事件上灵敏度达到98.88%。表 5和表 6是在特定类别上分别引入0、50、100和150个样本作为局部数据的实验结果,其中表 5是S类事件的数据结果比较,本文灵敏度和特异度分别为29.29%,65.81%,78.23%,82.25%和90.31%,96.18%,98.02%,97.04%;表 6是Ⅴ类事件的数据结果比较,本文灵敏度和特异度分别为78.02%,84.29%,91.99%,93.23%和99.49%,99.73%,97.63%,97.51%。
| 表 4 本研究和使用5 min数据介入的文献作性能比较 Tab.4 Performance comparisons between the reported systems and the proposed system with 5 min data intervention |
| 表 5 分别引入0、50、100、150个样本作为局部数据时在S类事件上的结果 Tab.5 Result of intervening 0, 50, 100, 150 samples as local data on SVEB |
| 表 6 分别引入0、50、100、150个样本作为局部数据时在Ⅴ类事件上的结果 Tab.6 Result of intervening 0, 50, 100, 150 samples as local data on VEB |
通过上述实验结果可知,本文提出的损失函数在S类和Ⅴ类事件上均获得了较高的AUC,证明新的损失函数对类别不平衡有积极作用。而在引入了5 min片段的实验中,本文在S类事件上的灵敏度相比文献[10-12]有了较大幅度提升;Ⅴ类事件上灵敏度同样高于现有的算法。此外,在引入0个样本,即不引入任何的专家交互的情况下,本文在两类事件上的表现均优于其它方法;在引入不同数量样本作为“专家介入”的情况下,本文提出的方法在Ⅴ类事件上的性能也优于其它方法。
神经网络的训练需要大量的人工标注的数据集,能够提供学习的样本越多,越能够反映出真实的数据分布情况,得到的模型的泛化误差就越小,但实际上,获取大规模有标签的数据需要巨大的成本。在无法获取足够多的训练样本的时候,往往考虑进行数据扩增。在二维图像处理上,一般可通过加噪、剪裁、旋转、尺度变化等方式对数据做扩增[28],但在对一维ECG信号做相同的处理反而会带来误差,甚至改变信号本身的含义。因此,在样本量不足且不平衡的情况下,本文通过改进损失函数,增加少样本类别的权重,增加易错分样本类别的权重,使网络专注于少数类和难识别类的学习,改善了类别不平衡带来的消极影响。
目前大部分文献[8, 12, 20]在S类事件上的灵敏度都普遍较低,且进行多分类任务时,S类样本容易被错分为N类。一方面是因为S类的样本量太少,难以反映出总体真实分布;另外,训练集DS1中的S类样本不够典型,与测试集DS2中的S类样本分布或者说真实的S类样本分布相差较大,不能起到一般代表性[29];并且,室上性异常心拍与正常类心拍的波形相似程度高[6],造成这两类样本在分布上重叠部分较多,容易错分。这些问题都给分类任务带来了一定的困难。本文通过改进训练方法、网络架构,结合残差网络和密集网络中的两种恒等映射,克服了神经网络随层数增加而导致退化的问题,使模型能够学习到更深层的特征,从而一定程度上改善了S类事件的性能。但从实验结果可看出,本文所用的方法仍有改进和提升的空间。考虑到心电信号的本质是一种时间序列,某一个时间点上的状态并不是独立的,而是和前面的输出也有关,CNN更多反映的是ECG的形态信息,而长短记忆网络[30]可以反映序列在时域上的关系,因此在处理时间序列问题上有明显的优势。本文在后续的工作中将结合CNN和长短记忆网络搭建网络并探究其组合对心律失常分类的影响。
综上所述,本文提出的基于心拍的端到端心律失常分类方法将人工智能与ECG信号分类识别相结合,较好地实现了分类任务,对S和Ⅴ类心律失常事件有良好的识别能力,达到了较高的灵敏度,为心律失常的自动分类提供了新的技术参考方案。
| [1] |
陈学琴.基于心电图分析的心律失常分类[D].北京: 北京交通大学, 2011. http://www.cnki.com.cn/Article/CJFDTotal-QHXB200903026.htm
|
| [2] |
Pandit D, Zhang L, Aslam N, et al. An efficient abnormal beat detection scheme from ECG signals using neural network and ensemble classifiers[C] // 8th Intern Conf Software Knowl Inform Managem Appl, 2014.
|
| [3] |
樊承柱.基于深度神经网络的心电图自动分类方法研究与实现[D].济南: 山东大学, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10422-1016165113.htm
|
| [4] |
Acharya UR, Fujita H, Oh SL, et al. Application of deep convolutional neural network for automated detection of myocardial infarction using ECG signals[J]. Inf Sci (Ny), 2017, 415(3): 190-8. |
| [5] |
de Chazal P, Reilly RB. A Patient-adapting heartbeat classifier using ECG morphology and heartbeat interval features[J]. IEEE Trans Biomed Eng, 2006, 53(12): 2535-43. DOI:10.1109/TBME.2006.883802 |
| [6] |
Huang H, Liu J, Zhu Q, et al. A new hierarchical method for interpatient heartbeat classification using random projections and RR intervals[J]. Biomed Eng Online, 2014, 13(1): 90-103. DOI:10.1186/1475-925X-13-90 |
| [7] |
Ye C, Bv K, Mt C. Heartbeat classification using morphological and dynamic features of ECG signals[J]. IEEE Trans Biomed Eng, 2012, 59(10): 2930-41. DOI:10.1109/TBME.2012.2213253 |
| [8] |
Li TY, Zhou M. ECG classification using wavelet packet entropy and random forests[J]. Entropy, 2016, 18(8): 285-98. DOI:10.3390/e18080285 |
| [9] |
金林鹏, 董军. 面向临床心电图分析的深层学习算法[J]. 中国科学:信息科学, 2015, 45(3): 398-416. |
| [10] |
Ince T, Kiranyaz S, Gabbouj M. A generic and robust system for automated patient-specific classification of ECG signals[J]. IEEE Trans Biomed Eng, 2009, 56(5): 1415-26. DOI:10.1109/TBME.2009.2013934 |
| [11] |
Serkan K, Turker I, Moncef G. Real-time patient-specific ECG classification by 1-D convolutional neural networks[J]. IEEE Trans Biomed Eng, 2015, 63(3): 225-37. |
| [12] |
Xu SS, Mak MW, Cheung CC. Towards End-to-End ECG classification with raw signal extraction and deep neural networks[J]. IEEE J Biomed Health Inform, 2018, 1(3): 287-99. |
| [13] |
Pan J, Tompkin WJ. A real-time QRS detection algorithm[J]. IEEE Transact Biomed Engin, 1985, 32(3): 230-6. |
| [14] |
Pollescha NL, Dale VH. Normalization in sustainability assessment: methods and implications[J]. Ecolog Econom, 2016, 130(3): 195-208. |
| [15] |
He K, Zhang X, Ren S, et al. Deep residual learning for image recognition[J]. Comput Vision Pattern Recogn, 2016, 29(8): 770-8. |
| [16] |
Huang G, Liu Z, Laurens VM, et al. Densely connected convolutional networks[C] // IEEE Conf CompVision Pattern Recogn, 2016: 2261-9.
|
| [17] |
Ioffe S, Szegedy C. Batch normalization: accelerating deep network training by reducing internal covariate shift[OL]. https://arxiv.org/abs/1502.03167, 2015.
|
| [18] |
Lin M, Chen Q, Yan S. Network in network[OL]. https://arxiv.org/abs/1312.4400, 2013.
|
| [19] |
周飞燕, 金林鹏, 董军. 卷积神经网络研究综述[J]. 计算机学报, 2017, 40(6): 1229-51. |
| [20] |
Sellami A, Hwang H. A robust deep convolutional neural network with batch-weighted loss for heartbeat classification[J]. Expert Syst Appl, 2019, 122(1): 75-84. |
| [21] |
Moody GB, Mark RG. The impact of the MIT-BIH arrhythmia database[J]. IEEE Eng Med and Biol, 2001, 20(3): 45-50. DOI:10.1109/51.932724 |
| [22] |
de Chazal P, O'dwyer M, Reilly RB. Automatic classification of heartbeats using ECG morphology and heartbeat interval features[J]. IEEE Trans Biomed Eng, 2004, 51(7): 1196-206. DOI:10.1109/TBME.2004.827359 |
| [23] |
Ecar A. Recommended practice for testing and reporting performance results of ventricular arrhythmia detection algorithms[J]. Biomed Instrum Technol, 1998, 6(1): 69-81. |
| [24] |
Smith L. Cyclical learning rates for training neural networks[J]. Comput Sci, 2017(11): 464-72. |
| [25] |
Smith LN. A disciplined approach to neural network hyper-parameters: part 1--learning rate, batch size, momentum, and weight decay[J]. Comput Sci, 2018, 45(4): 201-13. |
| [26] |
Al Rahhal MM, Bazi Y, Alhichri H, et al. Deep learning approach for active classification of electrocardiogram signals[J]. Inf Sci (Ny), 2016, 345(7): 340-54. |
| [27] |
Sayantan G, Kien P, Kadambari K. Classification of ECG beats using deep belief network and active learning[J]. Med Biol Eng Comput, 2018, 56(10): 1887-98. DOI:10.1007/s11517-018-1815-2 |
| [28] |
Perez L, Wang J. The effectiveness of data augmentation in image classification using deep learning[OL]. https://arxiv.org/abs/1712.04621, 2017.
|
| [29] |
Kiranyaz S, Ince T, Hamila R, et al. Convolutional neural networks for patient-specific ECG classification[C] // Conf Proc IEEE Eng Med Biol Soc, 2015: 2608-11.
|
| [30] |
Graves A. Long short-term memory[M]. Berlin: Springer, 2012.
|
 2019, Vol. 39
2019, Vol. 39

