胃癌是世界上常见的恶性肿瘤之一,严重威胁到人类健康。据统计,2008年全球新增胃癌病例约占全球癌症新增病例数的8%。我国胃癌的发病率及病死率均位居恶性肿瘤前列,仅次于肺癌和肝癌[1-2]。据估计,2015年中国共发现胃癌新案例679 100例,死亡人数高达49.8万[3]。虽然胃癌在手术,化疗和放射治疗方面取得了进展,但胃癌导致的病死率仍位居前列[4]。目前胃癌发生的机制尚未阐明,缺乏早期诊断标志物。
miRNA(miR)通过靶向特定的癌基因或抑癌基因,在肿瘤的发生、侵袭、转移中起着重要作用,例如miRNA-137在转录因子HMGA1的调控下,通过靶基因FMNL2抑制结直肠癌的侵袭和转移[5]。miRNA- 192/215通过调控下游多个靶基因促进胃癌细胞的增殖和侵袭[6-8]。同一个miRNA在不同类型肿瘤中, 将会涉及多个靶基因,参与多条信号通路,因此会分别起着癌基因或抑癌基因的作用。例如miRNA-223在前列腺癌和鼻咽癌中,分别靶向作用原癌基因ITGA3/ITGB1 [9]及MAFB [10],抑制细胞的侵袭和迁移行为;然而在胃癌和肺癌中,miRNA-223确起着癌基因的作用,促进肿瘤的侵袭和转移。本课题组通过利用芯片筛选差异表达miRNA,选择miRNA-340在胃癌中的发生机制进行研究。本课题首先利用miRNA芯片筛选胃癌中差异表达的miRNA,通过比较正常细胞株与胃癌细胞株中的表达差异表达,进行筛选,结果显示,在多个差异表达的miRNA中,miRNA-340在胃癌细胞株中表达明显降低,并且miRNA-340被发现与乳腺癌、骨肉瘤、胰腺癌和子宫内膜癌等肿瘤发生发展密切相关,由此选择miRNA-340作为研究对象,我们利用RT-PCR,MTT及EdU细胞增殖实验,裸鼠成瘤实验等检测miRNA-340在胃癌中的表达及增殖情况。随后,通过在线软件Targetscan、David数据库及Starbase分别对miRNA-340的相互作用靶点进行生物信息分析。本课题意在探讨miRNA-340在胃癌组织中的表达情况及对肿瘤细胞的增殖能力影响;揭示胃癌中miRNA-340可能的作用机制及为胃癌的机制研究提供新的研究靶点。
1 材料和方法 1.1 主要试剂1640培养液和新生胎牛血清(Hyclone),RNA提取试剂盒(Invitrogen),TaqMan反转录试剂盒、实时荧光定量-PCR试剂盒,PCR引物(锐博)。
1.2 细胞培养、转染及胃癌组织收集胃正常细胞株HFE-145、胃癌细胞株BGC-823、由本课题组保存。细胞均用含10%胎牛血清的1640培养液,置于37 ℃、CO2体积分数为5%的培养箱中培养,取生长状态良好的细胞用于实验。分别将细胞接种在96孔板及6 cm培养皿中,融合度约为50%,接种后24 h,分别在HFE-145和BGC-823细胞中转染miRNA-340 mimics和inhibitors(浓度为60 nmol/L),8 h后换液。
胃癌组织标本21对,所有标本均来源于广州医科大学第一附属医院,并经病理诊断证实;癌旁组织,均为癌及癌旁配对组织。收集患者的年龄、性别、肿瘤大小、病理分级等临床资料。
1.3 细胞总RNA的提取及质量检测取生长状态良好的细胞株按105/mL接种于25 cm2细胞培养瓶,离心后弃上清液,加TRIzol试剂(Invitrogen),48 h后利用TRIzol抽取总RNA,实验步骤按照说明书进行。使用1%琼脂糖凝胶电泳检测总RNA的完整性,测定A260/280比值来检测RNA纯度,RNA纯度检测合格,RNA产物置-80 ℃保存,备用。
1.3 miRNA芯片检测和数据分析取约100 μg的总RNA, 使用小分子RNA的分离试剂盒(Ambion's miRNA Isolation Kit)进行小分子RNA的分离,使用小分子RNA的Cy3荧光标记试剂盒(Agilent's miRNA Complete Labeling and Hyb Kit)将小分子RNA进行3-pCp荧光标记(Agilent, Santa Clara, CA, USA)。将标记好的小分子RNA与m i R NA芯片进行杂交反应,芯片使用美国Agilent公司的Human miRNA Microarray(V2)对样品进行杂交。Human miRNA Microarray(V2)芯片即为Human mi RNA芯片,含有723条人类miRNA。每张miRNA芯片中包含有1500左右个特异性探针、对照探针以及无探针的空白对照。杂交后的芯片应用Agilent Microarray Scanner(Agilent, version A.7.0.1)进行扫描分析。相对表达水平≥对照组的2倍或 < 0.5倍的miRNA为差异表达的miRNA。
1.4 miRNA-340的相互作用circRNA及下游靶基因的生物信息分析通过targetscan在线预测miRNA-340的下游靶基因;通过DAVID数据库对miRNA-340下游靶基因进行聚类分析,预测相关靶基因可能参与的信号通路。通过在线软件starbase(http://starbase.sysu.edu.cn/agoClipRNA.php?source=circRNA)分析miRNA-340的相互作用的circRNAs。
1.5 QRT-PCR总RNA提取及cDNA合成;取约300 mg,-80 ℃保存的组织,利用TRIzol(Invitrogen, Carlsbad, California, USA)提取组织总RNA。所得RNA经琼脂糖凝胶电泳确定无降解后,取3 μg总RNA,利用miRNA-340特异性的茎-环逆转录试剂盒合成cDNA。通过PCR扩增看家基因以检测cDNA的质量。将合格的cDNA分装,-20 ℃保存,备用。
QPCR:反应体系为15 μL,一切操作均参考试剂说明书,以U6为对照,不加模板为阴性对照,每个样品为3个复孔。通过2-△△Ct方法分析QPCR获得相对基因表达差异。每个样本的CT值求得平均后,减去各自的内参的平均Ct值,得到△Ct值。需要比较的两个样本之间的△Ct值相减得到△△Ct值。然后求2-△△Ct获得相对基因表达差异的倍数。
1.6 MTT细胞增殖实验将状态良好的细胞消化后精确计数,以103/孔接种于96孔板中,在培养的第1~5天分别取出,加入5 mg/mL的四氮唑蓝盐(MTT),孵育箱培养4 h,每孔再分别加入二甲基亚砜(DMSO),室温孵育10 min,轻微振荡15 min,在酶标仪上读取的吸光度A570 nm。各组设3个重复孔,取平均值,绘制细胞体外生长曲线。
1.7 EdU细胞增殖实验EdU增殖实验根据Ribbio Inc.公司Edu试剂说明书进行操作。简言之,在6 cm培养皿上接种细胞BGC-823,转染48 h后,细胞用50 μm EdU孵育5 h,在内室温下用4%多聚甲醛固定30 min,随后PBS冲洗2次。再用0.5% Triton X-100培养10 min,最后染色溶液中染色30 min,所有图像共聚焦显微镜拍摄。
1.8 体内成瘤实验BGC-823细胞用胰酶消化制成单细胞悬液,无血清的培养基洗3遍后,细胞计数。取4周龄雌性BALB/c裸鼠5只,分别将miRNA-340inhibitor组和inhibitor NC组细胞分别注射到裸鼠左侧背部皮下及右侧背部皮下,注射细胞数为2×106,每隔3~5 d用游标卡尺测量瘤体直径,连续观察20 d。
1.9 统计学处理应用SPSS 16.0统计学分析软件,对所得数据进行统计学处理,样本均数的比较用配对t检验。P < 0.05表示差异有统计学意义。
2 结果 2.1 miRNA芯片检测结果通过miRNA芯片筛选,BGC823、HFE145细胞中差异表达的miRNA,结果发现多个miRNA表达上调或下调。其中miRNA-340在胃癌细胞株BGC-823细胞中表达水平明显下调(表 1),随后对miR-340的表达进行检测。
| 表 1 miRNA芯片筛选GC细胞中差异表达的miRNA Tab.1 Differentially expressed miRNAs in GC identified by miRNAmicroarray data |
miRNA-340在胃癌组织中的表达明显低于正常胃粘膜组织(P < 0.05,图 1)。21对胃癌组织中,与癌旁正常组织相比,miRNA-340在17例组织中低表达,4例组织中表达增高,因此,miRNA-340在胃癌发生发展中可能是潜在的抑癌基因。
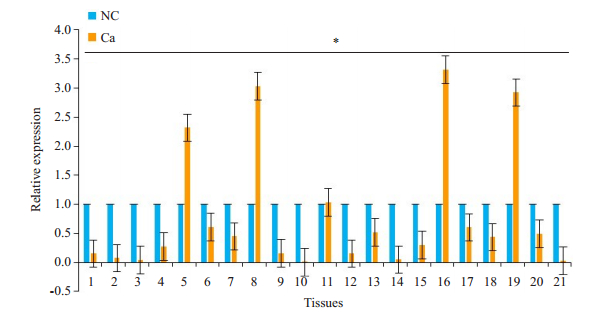
|
图 1 miRNA-340在胃癌组织中的表达情况 Fig.1 Expression of miRNA-340 in gastric cancer tissues (Ca) is significantly lower than that in adjacent normal tissues (NC). *P < 0.05. |
抑制miRNA-340,与对照组相比,BGC-823细胞增殖能力明显增强(图 2B),差异具有显著统计学意义(P < 0.0001);反之,转染miRNA-340mimics,HFE145细胞明显抑制细胞的增殖能力(图 2A)。抑制miRNA-340,与对照组相比,BGC-823细胞增殖能力明显增强(图 3B),差异具有显著统计学意义(P < 0.0001);反之,miRNA-340mimics能明显抑制细胞的增殖能力(图 3A)。
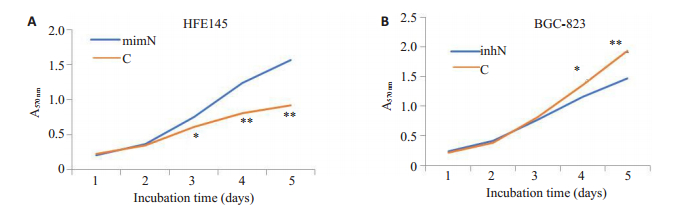
|
图 2 MTT实验检测miRNA-340对细胞增殖能力的影响 Fig.2 MTT assay for assessing the effect of miRNA-340 expression on cell proliferation. A: miR-340 mimics suppresses proliferation of HFE145 cells; B: miRNA-340 inhibitor promotes proliferation of BGC-823 cells. **P < 0.0001 vs NC mimics; **P < 0.0001 vs NC inhibitor. mimNC: NC mimics; 340 mim: miRNA-340 mimics; InhNC: NC inhibitors; 340inh: miRNA-340 inhibitor; NC: Negative control. |
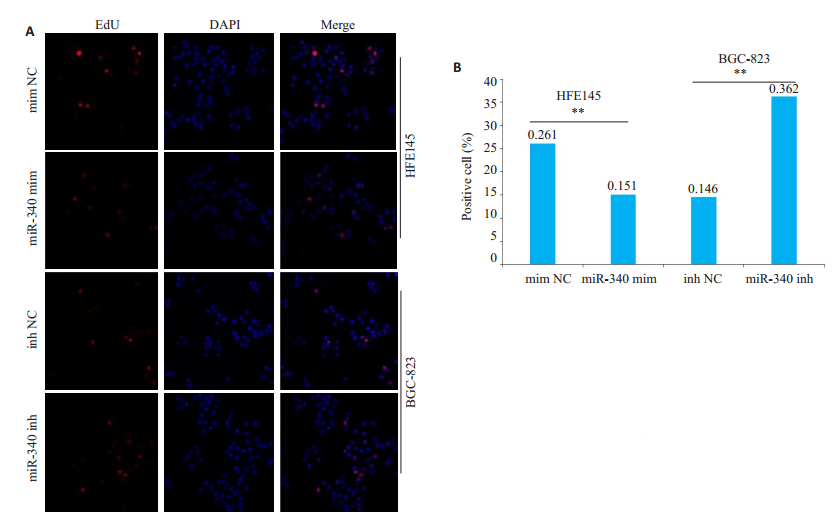
|
图 3 EdU实验检测miRNA-340对细胞增殖能力的影响 Fig.3 EdU assay for assessing the effect of miRNA-340 on cell proliferation. A: miR-340 mimics suppresses proliferation of HFE145 cells; B: miRNA-340 inhibitor promotes proliferation of BGC-823 cells. **P < 0.0001 vs NC mimics; **P < 0.0001 vs NC inhibitors. mimNC: NC mimics; 340 mim: miRNA-340 mimics; InhNC: NC inhibitors; 340inh: miRNA-340 inhibitor; NC: Negative control. |
抑制miRNA-340,与对照组相比,BGC-823细胞增殖能力明显增强,差异具有显著统计学意义(P < 0.0001)(图 4)。
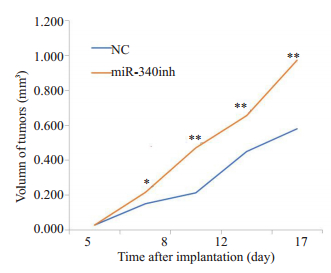
|
图 4 体内成瘤实验检测细胞增殖能力的影响 Fig.4 Tumor formation assay for assessing the effect of miR-340 expression on cell proliferation in nude mice. miR-340 inhibitor obviously increases xenograft volume in nude mice. *P < 0.001, **P < 0.0001. 340inh:miRNA- 340inhibitor; NC: Negative control. |
由于miRNA通过下游靶基因参与肿瘤的增殖、侵袭和转移。因此,本研究通过targetscan在线预测miRNA-340的下游靶基因;通过DAVID数据库对miRNA-340下游靶基因进行聚类分析,预测相关靶基因可能参与的信号通路。结果显示,miRNA-340下游预测的靶基因中,共预测出1391个靶基因,其中有3个及以上的保守结合部位的靶基因有21个(表 2),其中多个靶基因均参与肿瘤的发生及侵袭相关的基因。例如,与细胞粘附相关的原癌基因CDON,激动蛋白相关的MYO9A等基因(表 3)。在1391个靶基因中,选取其中有2个及以上保守结合位点的186个靶基因进行KEGG通路分析,结果显示,186个靶基因参与吗miRNA在肿瘤的发生机制,Wnt通路的激活及多种肿瘤相关的通路(表 4)。
| 表 2 通过Targetscan在线预测miRNA-340下游的靶基因 Tab.2 Downstream target genes of miRNA-340 predicted by Targetscan |
| 表 3 通过starbase在线预测miRNA-340相互作用的circRNAs Tab.3 circRNAs predicted to interact with miRNA-340 by starBase |
| 表 4 通过DAVID数据库预测miRNA-340下游的靶基因参与的信号通路 Tab.4 Signaling pathways involving the predicted target genes of miRNA-340 predicted using the DAVID database |
为了了解miRNA-340相互作用的circRNA,我们通过在线软件starbase预测了miRNA-340的相互作用的circRNAs。结果显示有多个circRNA与miRNA-340相互作用,根据clip ExpNum的高低,我们选取前10个circRNA作为与miRNA-340作用最强的sponge circRNA(表 3)。
3 讨论miRNA-340在肿瘤中的研究较多,主要集中在乳腺癌[11]、骨肉瘤[12]、胰腺癌[13]、子宫内膜癌[14]、肺癌[15]、肝细胞癌[16]、宫颈癌[17]等肿瘤中对靶基因的调控研究。在这些肿瘤中发现,miRNA-340均具有抑癌基因的作用,通过不同的靶基因抑制肿瘤的生长、侵袭和转移。乳腺癌研究发现,miRNA-340分别通过负向调控原癌基因c-Met、MYO10抑制乳腺癌细胞的增殖、侵袭和转移[18-19]。Huang等[20]研究结果表明,miRNA-340/MDM2/ p53轴可有效的抑制前列腺癌细胞的生长。因此,前列腺癌中miRNA-340有望成为miRNA靶向治疗的新靶点。神经系统肿瘤中,miRNA-340靶向作用SOX2、CDK6,cyclin-D1及cyclin-D2等多个靶基因,抑制肿瘤的的增殖和侵袭[21-23]。此外,在非小细胞肺癌[24]、结直肠癌[25]等,均发现miRNA-340为具有抑癌作用,抑制肿瘤的生长和侵袭。本课题首先利用miRNA芯片筛选胃癌中差异表达的miRNA,通过比较正常细胞株与胃癌细胞株中的表达差异表达,进行筛选。结果显示,在多个差异表达的miRNA中,miRNA-340在胃癌细胞株中表达明显降低。因此将对miRNA-340在胃癌中的表达机制进行进一步的研究。首先利用RT-PCR检测胃癌组织中miRNA-340的表达情况,结果显示,miRNA-340在胃癌中低表达,结果提示miRNA-340在人胃癌中可能扮演着抑癌基因的角色。由此我们推测miRNA-340可能调控胃细胞的生物学功能,通过体外MTT与EdU细胞增殖实验中我们发现,下调miRNA-340的表达能促进胃癌细胞的生长,上调miRNA-340的表达则能抑制正常胃黏膜细胞的生长。两个细胞增殖实验结果均表明miRNA-340对胃癌细胞的增殖能力起到抑制作用;最后通过构建裸鼠体内成瘤模型,在体内实验中进一步验证了我们的猜想,即miRNA-340在胃癌发生中抑制肿瘤细胞的增殖。本研究揭示了胃癌中miRNA-340的表达及增殖情况,为胃癌的发生机制奠定了一定的基础。
环状RNA(circRNAs)是一类来自外显子转录本的内源性RNA,多数由非共性反向剪接而成,广泛且多样地存在于哺乳动物的细胞中,区别于传统线性RNA的新型形式,在转录后水平对基因表达起到重要的调节作用[26-27]。Circular最早于20世纪70年代在RNA病毒中被发现[28]。Hsu和Coca-Prados [29]在1979年,利用电子显微镜第1次观察到RNA以环状的形式存在于真核细胞的细胞质中,富集在包浆中具有牢固的环状结构。随后,在酵母的线粒体中也发现了circRNA。随着深度RNA测序及规模化生物细细技术的发展,研究者才真正发现在生物体内大量存在环化的RNA分子。circRNA由于形成闭合的环状,具有良好的保守性及组织特异性,不易被核酸降解,在生物体内非常的稳定存在胞质中。
CircRNA对miRNA的sponge功能是存在的,主要通过吸附作用,抑制miRNA的功能,从而抑制miRNA下游靶基因的作用。目前研究表明,miRNA-340在多种肿瘤组织中呈低表达,且具有一定的抑癌功能,例如:抑制细胞增殖、迁移等。结合本研究的结果,我们推测其在胃癌中发挥抑癌基因的功能。本研究通过预测软件分析发现,miRNA-340相互作用的circRNA,据结果推测出多个circRNA与miRNA-340相互吸附,发挥sponge的功能,抑制miRNA的功能,可能调控miRNA下游靶基因的作用,共同调节肿瘤的发生发展及转移。所以我们检测到胃癌组织中miRNA-340呈低表达,可能跟其相互作用的circRNA吸附作用有关,从而导致其表达降低。circRNA高表达,推测是原癌基因,发挥促癌作用,这将抑制相互作用的抑癌miRNA来发挥作用。
预测的circRNA-CCND1的母基因,能参与编码周期蛋白cyclin D1,因此circRNA-CCND1对细胞周期的调控具有一定的价值[30]。circRNA在人体细胞中广泛表达,表达水平甚至超过了线性异构体的10倍之多,且由于circRNA拥有miRNA应答元件,能与miRNA相互作用,因此,circRNA如“spong”一般吸附miRNA,作为竞争性内源性RNA调整miRNA的表达,抑制其功能[31]。这些特性使得circRNA作为新型临床诊断标记物的应用上具有明显优势。本课题通过生物信息分析,筛选了部分与miRNA-340相互作用的circRNA,无疑将为胃癌的早期诊断、药物治疗及预后判断等方面提供新的思路与靶点,为胃癌的机制研究与临床应用架起了一座桥梁。
本研究也存在一些不足之处。本实验中,对细胞的功能检测只检测了增殖能力,miR-340对胃癌细胞的侵袭、迁移、周期等生物学功能尚未阐述。因此,我们将在下一步补充相应的生物功能实验来进一步完善miRNA-340对细胞生物学功能的影响。circRNA与miR-340间的相互作用也需仍需进一步的阐述与验证。
通过我们本次的研究,证明miRNA-340在人胃癌的发生发展中扮演着抑癌基因的角色,可能与多个circRNA相互作用共同参与胃癌的发生和发展。本研究能为将来miRNA-340作为胃癌早期诊断因子、预后评估因子、靶向治疗等相关临床应用提供理论基础。
| [1] |
Jemal A, Bray F, Center MM, et al. Global cancer statistics[J]. CA Cancer J Clin, 2011, 61(2): 69-90. DOI:10.3322/caac.v61:2 |
| [2] |
Chen WQ, Zheng RS, Zeng HM, et al. The updated incidences and mortalities of major cancers in China, 2011[J]. Chin J Cancer, 2015, 34(3): 502-7. |
| [3] |
Chen W, Zheng R, Baade PD, et al. Cancer statistics in China, 2015[J]. CACancer J Clin, 2016, 66(2): 115-32. DOI:10.3322/caac.21338 |
| [4] |
Bray F, Ferlay J, Soerjomataram I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. DOI:10.3322/caac.v68.6 |
| [5] |
Liang L, Li X, Zhang X, et al. MicroRNA-137, an HMGA1 target, suppresses colorectal cancer cell invasion and metastasis in mice by directly targeting FMNL2[J]. Gastroenterology, 2013, 144(3): 624-635. DOI:10.1053/j.gastro.2012.11.033 |
| [6] |
Jin Z, Selaru FM, Cheng Y, et al. MicroRNA-192 and -215 are upregulated in human gastric cancer in vivo and suppress ALCAM expression in vitro[J]. Oncogene, 2011, 30(13): 1577-85. DOI:10.1038/onc.2010.534 |
| [7] |
Zhang XJ, Peng Y, Huang Y, et al. Inhibition of the miR-192/215-Rab11-FIP2 axis suppresses human gastric cancer progression[J]. Cell Death Dis, 2018, 9(7): 778. DOI:10.1038/s41419-018-0785-5 |
| [8] |
Zhang XJ, Peng Y, Huang Y, et al. SMG-1 inhibition by miR-192/-215 causes epithelial-mesenchymal transition in gastric carcinogenesis via activation of Wnt signaling[J]. Cancer Med, 2018, 7(1): 146-56. DOI:10.1002/cam4.2018.7.issue-1 |
| [9] |
Kurozumi A, Goto Y, Matsushita R, et al. Tumor-suppressive microRNA-223 inhibits cancer cell migration and invasion by targeting ITGA3/ITGB1 signaling in prostate cancer[J]. Cancer Sci, 2016, 107(1): 84-94. DOI:10.1111/cas.12842 |
| [10] |
Yang WY, Lan X, Li DM, et al. MiR-223 targeting MAFB suppresses proliferation and migration of nasopharyngeal carcinoma cells[J]. BMC Cancer, 2015, 15: 461. DOI:10.1186/s12885-015-1464-x |
| [11] |
Maskey N, Li DF, Xu H, et al. MicroRNA-340 inhibits invasion and metastasis by downregulating ROCK1 in breast cancer cells[J]. Oncol Lett, 2017, 14(2): 2261-7. DOI:10.3892/ol.2017.6439 |
| [12] |
Su P, Mu S, Wang Z. Long noncoding RNA SNHG16 promotes osteosarcoma cells migration and invasion via sponging miRNA-340[J]. DNACell Biol, 2019, 38(2): 170-5. |
| [13] |
Yang DY, Li YT, Zhao DQ. Curcumin induces apoptotic cell death in human pancreatic cancer cells via the miR-340/XIAP signaling pathway[J]. Oncol Lett, 2017, 14(2): 1811-6. DOI:10.3892/ol.2017.6321 |
| [14] |
Xie W, Qin W, Kang YL, et al. MicroRNA-340 inhibits tumor cell proliferation and induces apoptosis in endometrial carcinoma cell line RL95-2[J]. Med Sci Monit, 2016, 22(5): 1540-6. |
| [15] |
Fernandez S, Risolino M, Verde P. A novel miRNA-mediated STOP sign in lung cancer: miR-340 inhibits the proliferation of lung cancer cells through p27(KIP1[J]. Mol Cell Oncol, 2015, 2(2): e977147. DOI:10.4161/23723556.2014.977147 |
| [16] |
Xie L, Chen Z, Liu H, et al. Effects of miR-340 on hepatocellular carcinoma by targeting the DcR3 gene[J]. Dig Liver Dis, 2018, 50(3): 291-6. DOI:10.1016/j.dld.2017.10.024 |
| [17] |
Xiao HM, Yu LJ, Li FD, et al. MiR-340 suppresses the metastasis by targeting EphA3 in cervical cancer[J]. Cell Biol Int, 2018, 42(9): 1115-23. DOI:10.1002/cbin.v42.9 |
| [18] |
Wu ZS, Wu Q, Wang CQ, et al. miR-340 inhibition of breast cancer cell migration and invasion through targeting of oncoprotein c-Met[J]. Cancer, 2011, 117(13): 2842-52. DOI:10.1002/cncr.v117.13 |
| [19] |
Chen CP, Sun ZL, Lu X, et al. miR-340 suppresses cell migration and invasion by targeting MYO10 in breast cancer[J]. Oncol Rep, 2016, 35(2): 709-16. DOI:10.3892/or.2015.4411 |
| [20] |
Huang K, Tang Y, He L, et al. MicroRNA-340 inhibits prostate cancer cell proliferation and metastasis by targeting the MDM2-p53 pathway[J]. Oncol Rep, 2016, 35(2): 887-95. DOI:10.3892/or.2015.4458 |
| [21] |
Presneau N, Eskandarpour M, Shemais T, et al. MicroRNA profiling of peripheral nerve sheath tumours identifies miR-29c as a tumour suppressor gene involved in tumour progression[J]. Br J Cancer, 2013, 108(4): 964-72. |
| [22] |
Li X, Gong X, Chen J, et al. miR-340 inhibits glioblastoma cell proliferation by suppressing CDK6, cyclin-D1 and cyclin-D2[J]. Biochem Biophys Res Commun, 2015, 460(3): 670-7. DOI:10.1016/j.bbrc.2015.03.088 |
| [23] |
Das S, Bryan K, Buckley PG, et al. Modulation of neuroblastoma disease pathogenesis by an extensive network of epigenetically regulated microRNAs[J]. Oncogene, 2013, 32(24): 2927-36. DOI:10.1038/onc.2012.311 |
| [24] |
Fernandez S, Risolino M, Mandia N, et al. miR-340 inhibits tumor cell proliferation and induces apoptosis by targeting multiple negative regulators of p27 in non-small cell lung cancer[J]. Oncogene, 2015, 34(25): 3240-50. DOI:10.1038/onc.2014.267 |
| [25] |
Takeyama H, Yamamoto H, Yamashita S, et al. Decreased miR-340 expression in bone marrow is associated with liver metastasis of colorectal cancer[J]. Mol Cancer Ther, 2014, 13(4): 976-85. DOI:10.1158/1535-7163.MCT-13-0571 |
| [26] |
Memczak S, Jens M, Elefsinioti A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency[J]. Nature, 2013, 495(7441): 333-8. DOI:10.1038/nature11928 |
| [27] |
Capel B, Swain A, Nicolis S, et al. Circular transcripts of the testisdetermining gene Sry in adult mouse testis[J]. Cell, 1993, 73(5): 1019-30. DOI:10.1016/0092-8674(93)90279-Y |
| [28] |
Pearce MB, Jayaraman A, Pappas C, et al. Pathogenesis and transmission of swine origin A(H3N2)v influenza viruses in ferrets[J]. Proc NatlAcad Sci USA, 2012, 109(10): 3944-9. DOI:10.1073/pnas.1119945109 |
| [29] |
Hsu MT, Coca-Prados M. Electron microscopic evidence for the circular form of RNA in the cytoplasm of eukaryotic cells[J]. Nature, 1979, 280(5720): 339-40. DOI:10.1038/280339a0 |
| [30] |
Xue J, Liu Y, Luo F, et al. Circl 00284, via miR-217 regulation of EZH2, is involved in the arsenite-accelerated cell cycle of human keratinocytes in carcinogenesis[J]. Biochim Biophys Acta Mol Basis Dis, 2017, 1863(3): 753-63. DOI:10.1016/j.bbadis.2016.12.018 |
| [31] |
Khan I, Kerwin J, Owen K, et al. Correction: Registered report: A coding-independent function of gene and pseudogene mRNAs regulates tumour biology[J]. Elife, 2015, 4: e13015. DOI:10.7554/eLife.13015 |
 2019, Vol. 39
2019, Vol. 39

