细胞周期蛋白依赖性激酶抑制因子2A(CDKN2A)和细胞周期蛋白依赖性激酶抑制因子2B(CDKN2B)的基因产物是一种肿瘤抑制因子,参与调控细胞周期,延缓细胞增殖,同时,在干细胞/祖细胞的细胞衰老过程中发挥着既定的作用,促进机体衰老[1-2]。研究表明,CDKN2A、CDKN2B基因的调控位点发生改变,会产生与衰老表型有关的疾病,如动脉粥样硬化疾病、2型糖尿病(T2D)、青光眼和一些恶性肿瘤[3]。在哺乳动物基因组中,DNA甲基化是一种表观遗传机制,在DNA甲基转移酶的作用下,将一个甲基基团转移到胞嘧啶的C5位置,形成5-甲基胞嘧啶[4]。基因组DNA甲基化水平的高低,与衰老疾病有着密切联系,如癌症、心血管疾病、T2D等[5]。衰老以一种复杂的形式来改变基因组DNA甲基化,一般来说,随着年龄的增长,基因组DNA甲基化水平呈降低趋势[1]。
目前CDKN2A和CDKN2B的相关研究虽然探讨了相关基因与衰老的关系,但是都未明确甲基化水平与年龄等临床特征之间的关系,而本研究通过对两基因甲基化水平进行测定并分析,从而获得两基因之间甲基化水平的关系及与衰老之间的关系,有利于对细胞、组织、器官等衰老研究提供思路和方向。
1 资料和方法 1.1 研究对象从浙江省宁波市第一医院收集体检者530名,包括男性284名,女性246名。纳入标准:(1)年龄≥30岁;(2)无肿瘤者、无严重肝肾功能不全者、无血液疾病者、无传染病者等。排除标准:(1)年龄≤29岁;(2)阿尔茨海默病患者;(3)冠心病患者等。本研究经宁波大学医学院伦理委员会讨论通过及患者知情同意。
1.2 血液采集及一般临床资料体检者空腹8 h后,清晨抽取肘静脉血5 mL,置于EDTA管中,防止血液凝固,并保存于-80 ℃。采集一般临床资料包括年龄、性别、既往史、个人史等。LDL、TC、HDL、TG、ApoA、ApoB、ApoE等含量采用Beckman AU5800全自动生化分析仪测定。
1.3 DNA提取及浓度测定全基因组DNA严格按照E.Z.N.A.TM Blood试剂盒(Omega Bio-Tek)说明书进行提取,同时使用Nanodrop 2000分光光度计(Thermal Scientific)对DNA浓度进行测定。将提取好的DNA保存于-20 ℃,等待用于后步实验。
1.4 基因组DNA亚硫酸氢盐修饰使用EZ DNA Methylation-Gold Kit(Zymo Research)试剂盒对上步提取好的DNA进行亚硫酸氢盐修饰。最终得到的效果是将基因组DNA中未甲基化的胞嘧啶转化为尿嘧啶,但已甲基化的胞嘧啶及其他碱基保持不变。
1.5 CDKN2A、CDKN2B基因甲基化水平的测定将经亚硫酸氢盐修饰过的DNA进行qMSP扩增,反应在Light Cycler 480(Roche)中进行。反应体系共20 μL,包括10 μL SYBRGreen I Master(Roche),1 μL经亚硫酸氢盐修饰过的DNA,8 μL ddH2O及上下游引物各0.5 μL。反应条件如下:95 ℃预变性,持续时间10 min;接下来45次循环,包括95 ℃下变性20 s,58 ℃下退火20 s,72 ℃下延伸30 s;熔融曲线分析,95 ℃下15 s,60 ℃下1 min;最后,在40 ℃下进行时长10 min的延伸。
1.6 统计学分析所有统计在SPSS 18.0版中进行。所有的图在GraphPad Prism 6软件中绘制。符合正态检验的数据用均数±标准差表示,不符合正态检验的数据用中位数(四分位数)表示,分类资料以例数(百分数)表示。使用Spearman相关性检验来评估CDKN2A、CDKN2B基因甲基化与生化指标之间的相关性。P < 0.05认为差异具有统计学意义。
2 结果 2.1 一般临床基本资料本实验共收集530名样本,其中男性284名,女性246名。吸烟者145名,高血压患者264名,T2D患者68名。LDL、TC、ApoA、ApoB、ApoE、Lp(a)、CRP、ALT、AST、ALP等含量按照中位数(四分位数)呈现(表 1)。
| 表 1 参与者临床基线表 Tab.1 Baseline clinical data of the participants |
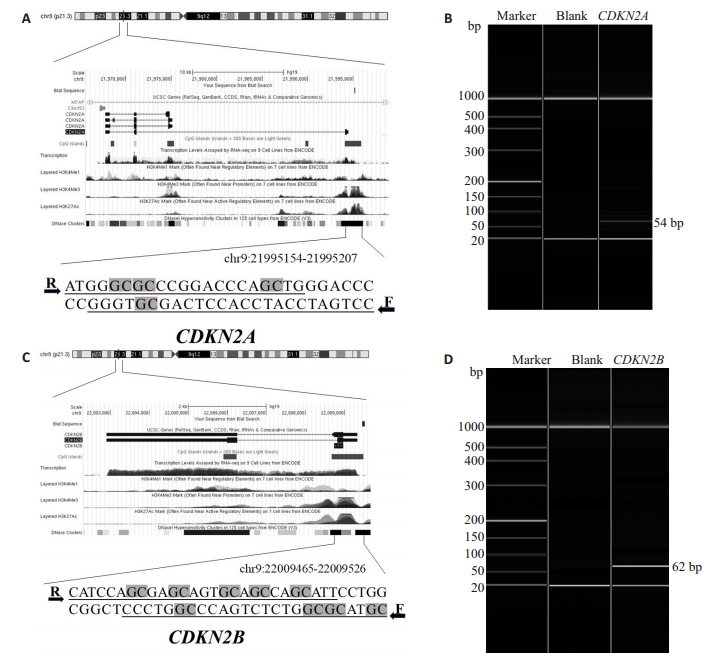
由于甲基化往往发生在CpG二核苷酸的位置,我们选择了启动子区域CpG岛位置的位点,CDKN2A(chr9:21995154-21995207)和CDKN2B(chr9:22009465- 22009526)进行研究(图 1A、C)。毛细管电泳证实CDKN2A和CDKN2B基因的qMSP扩增产物长度分别为54 bp和62 bp(图 1B、D)。
|
图 1 CDKN2A、CDKN2B基因信息图及代表性电泳结果 Fig.1 CDKN2A and CDKN2B gene information maps and representative electrophoresis results. A, C: Location and functional annotation of the target sequence from University of California Santa Cruz Genome Browser based on Human 2009 (GRCh37/hg19). The target sequences are located in the CpG island region of CDKN2A and CDKN2B, and the primers are underlined and CpG sites are in grey. F and R are forward and reverse primers, respectively. B, D: Electrophoresis of a representative qMSP product. |
qMSP检测发现,在总体中,CDKN2A基因甲基化水平与CDKN2B基因甲基化水平呈正相关(r=0.622,P=4.E-58);分层分析发现,男性组和女性组中CDKN2A基因甲基化水平与CDKN2B基因甲基化水平呈正相关(男性:r=0.571,P=6.E-26;女性:r=0.679,P=2.E-34)。根据吸烟史、糖尿病和高血压进行调整后发现,女性组中CDKN2A基因甲基化水平与CDKN2B基因甲基化水平呈正相关(r=0.252,P=1.E-04)。
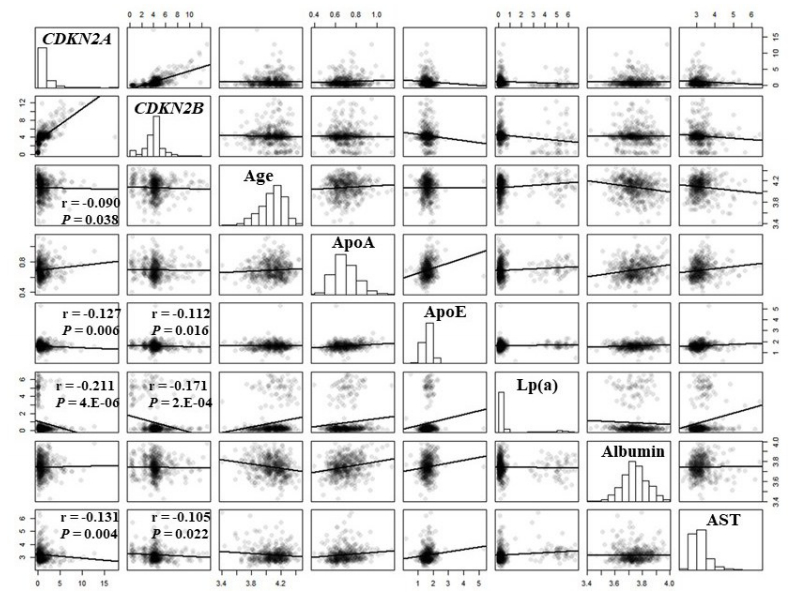
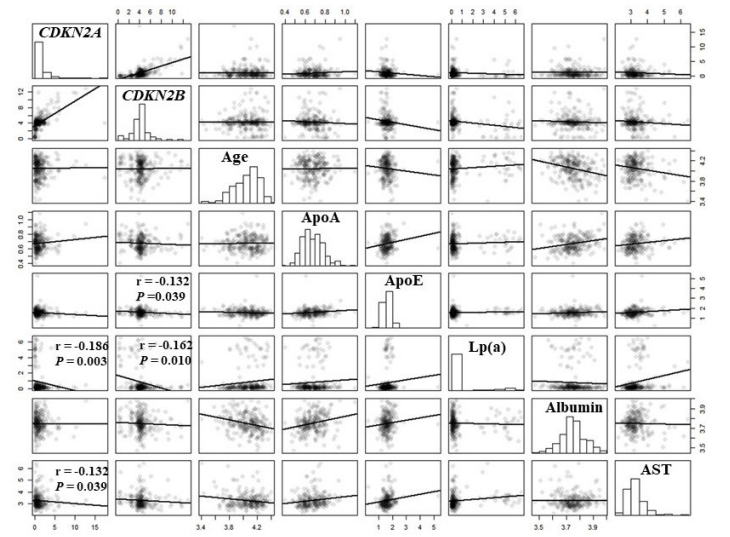
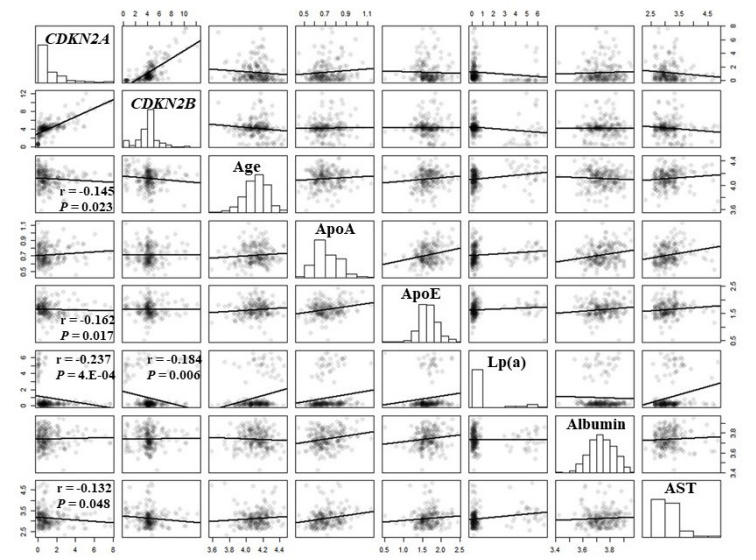
2.4 基因DNA甲基化与临床表型之间的相关性在总体样本、女性组中,CDKN2A基因甲基化水平与年龄呈负相关,而在男性中无统计学意义(总体:图 2;男性:图 3;女性:图 4)。在总体组、男性组、女性组中,CDKN2B基因甲基化水平与年龄也呈负相关,但无统计学意义(总体:图 2;男性:图 3;女性:图 4)。女性组中的CDKN2A基因甲基化水平与LDL呈正相关(P=0.013,表 2)。在男性组中,发现CDKN2B基因甲基化水平与HDL呈负相关(P=0.013,表 2)。在总体中,发现CDKN2A、CDKN2B基因甲基化水平与TG呈负相关(CDKN2A:P=0.049;CDKN2B:P=0.034,表 2)。在总体和女性组中,CDKN2A基因甲基化水平与ApoE呈负相关(总体:图 2;女性:图 4)。在总体和男性组中,CDKN2B基因甲基化水平均与ApoE呈负相关(总体:图 2;男性:图 3)。总样本及性别分层后发现CDKN2A基因甲基化水平与AST含量呈负相关(总体:图 2;男性:图 3;女性:图 4)。在总体中,CDKN2B基因甲基化水平与AST含量呈负相关(图 2)。总样本及性别分层后均发现CDKN2A、CDKN2B基因甲基化水平与Lp(a)含量呈负相关(图 2~4)。
|
图 2 总体样本组中各阳性指标之间的相关性分析图 Fig.2 Correlation analysis diagram of the positive indicators in the overall sample. |
|
图 3 男性组中各阳性指标之间的相关性分析图 Fig.3 Correlation analysis diagram of positive indicators in the male sample. |
|
图 4 女性组中各阳性指标之间的相关性分析图 Fig.4 Correlation analysis diagram of positive indicators in the female sample. |
| 表 2 基因甲基化与表型之间的相关性 Tab.2 Correlation between gene methylation and phenotype |
本研究发现在总体、男性组、女性组中,CDKN2A基因甲基化水平与CDKN2B基因甲基化水平呈正相关。在总体样本、女性组中,CDKN2A基因甲基化水平与年龄呈负相关。女性组中的CDKN2A基因甲基化水平与LDL呈正相关。在男性组中,发现CDKN2B基因甲基化水平与HDL呈负相关。在总体中,发现CDKN2A、CDKN2B基因甲基化水平与TG呈负相关。
荟萃分析结果表明,CDKN2A高甲基化与结直肠癌的淋巴血管浸润、淋巴结转移和近端肿瘤位置之间存在显著相关性,同时,CDKN2A高甲基化可能是结直肠癌患者预后不良的预测因素[6]。Hu等[7]分别对72例儿童急性淋巴细胞白血病患儿和12例免疫血小板减少症患儿进行骨髓穿刺和甲基化特异性聚合酶链反应分析,并进行改良端粒重复扩增方案分析,发现CDKN2B基因高甲基化与端粒酶活性上调显著相关,CDKN2B基因高甲基化、端粒酶活性的增加与患儿童急性淋巴细胞白血病的风险上升相关。Arya等[8]研究发现,在甲状旁腺腺瘤中,CDKN2A、CDKN2B基因呈高甲基化,而在正常的甲状旁腺组织中未发现基因的异常甲基化,同时还表明,CDKN2A、CDKN2B的高甲基化会导致其基因表达下降,从而促进散发性甲状旁腺肿瘤的发生。以上相关研究的样本均来源于组织,并表明CDKN2A和CDKN2B基因高甲基化与肿瘤的发生、发展过程相关,而我们的研究样本却来源于外周血,且发现基因甲基化水平随着年龄的增长而呈下调的趋势,这可能是甲基化水平的组织特异性造成的[9]。以上结果证实CDKN2A/ CDKN2B基因的调控机制复杂,未来尚需进一步深入研究其表达调控机制。总而言之,我们的研究表明,CDKN2A基因甲基化水平与CDKN2B基因甲基化水平呈正相关,且在总体样本、女性组中,CDKN2A基因甲基化水平与年龄呈负相关。
此外,CDKN2A、CDKN2B甲基化水平与性别关系密切。Alves等通过对93例星形细胞肿瘤提取得到的DNA采用甲基化特异性PCR和免疫组织化学方法进行CDKN2A启动子甲基化分析和p16INK4a表达分析,发现启动子甲基化使CDKN2A失活是星形细胞瘤中常见的事件,且与患者的年龄和性别有关[10]。与此同时,基因启动子甲基化模式随着年龄和激素水平的变化而变化[11-12]。在本研究中,仅在女性中观察到CDKN2A基因甲基化水平与年龄呈负相关的现象,进一步验证了该基因甲基化水平存在性别差异的理论。
HDL、LDL、TG等脂质代谢相关蛋白参与衰老机制[13-15]。有研究表明,HDL不但具有清除细胞脂质的能力,还可能通过直接干预衰老信号或生存因子KLOTHO调节衰老过程[16]。另外,Postmus等[17]发现LDL遗传风险特征与家族长寿有关。HDL、LDL、TG均是衰老相关疾病如冠心病(CHD)发生发展的重要参与者[18]。此外,Lettre等[19]通过全基因组关联研究发现,CDKN2A、CDKN2B基因与CHD的发生与发展有关。lnc-RNA ANRIL位于CDKN2A、CDKN2B临近位置,在血管平滑肌细胞中敲除ANRIL,将导致CDKN2A、CDKN2B表达显著变化,细胞生长减少[20]。临床研究亦发现,ANRIL可能通过调控CDKN2A、CDKN2B的表达影响CHD的发生发展[21]。本研究中,我们发现CDKN2A、CDKN2B基因甲基化水平和HDL、LDL、TG指标显著相关,这给CHD的发生机制、诊断和治疗提供新的研究思路。
本研究采用甲基化特异性实时定量聚合酶链技术(qMSP)测定CDKN2A、CDKN2B基因DNA甲基化水平。qMSP结合了MSP和实时PCR技术,具有高灵敏度,适用于任何CpG岛,使用DNA样本量少,动态范围大等优点[22-23]。同时,qMSP比COBRA分析、亚硫酸氢盐测序省时省力,也不需要昂贵的焦磷酸测序[24-26]。此外,我们选用较大的样本量,在统计学上更具有代表性,误差会相对缩小。本文的不足之处在于没有进行相关机制实验,但是并不影响我们的研究结果。
综上所述,CDKN2A、CDKN2B基因甲基化水平与正常衰老密切相关,而CDKN2A、CDKN2B基因甲基化水平的异常增高可能会增加人们患癌症、T2D、心血管疾病的风险,从而影响人们的寿命和生活水平。
| [1] |
Keyes MK, Jang H, Mason JB, et al. Older age and dietary folate are determinants of genomic and p16-specific DNA methylation in mouse colon[J]. J Nutr, 2007, 137(7): 1713-7. DOI:10.1093/jn/137.7.1713 |
| [2] |
Sato S, Kawamata Y, Takahashi A, et al. Ablation of the p16(INK4a) tumour suppressor reverses ageing phenotypes of klotho mice[J]. Nat Commun, 2015, 6: 7035. DOI:10.1038/ncomms8035 |
| [3] |
Jeck WR, Siebold AP, Sharpless NE. Review:a meta-analysis of GWAS and age-associated diseases[J]. Aging Cell, 2012, 11(5): 727-31. DOI:10.1111/j.1474-9726.2012.00871.x |
| [4] |
Moore LD, Le T, Fan G. DNA methylation and its basic function[J]. Neuropsychopharmacology, 2013, 38(1): 23-38. DOI:10.1038/npp.2012.112 |
| [5] |
Hamidi T, Singh A K, Chen T. Genetic alterations of DNA methylation machinery in human diseases[J]. Epigenomics, 2015, 7(2): 247-65. DOI:10.2217/epi.14.80 |
| [6] |
Xing X, Cai W, Shi H, et al. The prognostic value of CDKN2A hypermethylation in colorectal cancer:a meta-analysis[J]. Br J Cancer, 2013, 108(12): 2542-8. DOI:10.1038/bjc.2013.251 |
| [7] |
Hu Q, Chen X, Liu S, et al. Methylation of CDKN2B CpG islands is associated with upregulated telomerase activity in children with acute lymphoblastic leukemia[J]. Oncol Lett, 2017, 13(4): 2115-2120. DOI:10.3892/ol.2017.5710 |
| [8] |
Arya AK, Bhadada SK, Singh P, et al. Promoter hypermethylation inactivates CDKN2A, CDKN2B and RASSF1A genes in sporadic parathyroid adenomas[J]. Sci Rep, 2017, 7(1): 3123. |
| [9] |
Wan J, Oliver VF, Wang G, et al. Characterization of tissue-specific differential DNA methylation suggests distinct modes of positive and negative gene expression regulation[J]. BMC Genomics, 2015, 16: 49. DOI:10.1186/s12864-015-1271-4 |
| [10] |
Alves MK, Faria MH, Neves Filho EH, et al. CDKN2A promoter hypermethylation in astrocytomas is associated with age and sex[J]. Int J Surg, 2013, 11(7): 549-53. DOI:10.1016/j.ijsu.2013.05.030 |
| [11] |
Brunet A, Berger SL. Epigenetics of aging and aging-related disease[J]. J Gerontol A Biol Sci Med Sci, 2014, 69(Suppl 1): S17-20. DOI:10.1093/gerona/glu042 |
| [12] |
Kouzmenko A, Ohtake F, Fujiki R, et al. Hormonal gene regulation through DNA methylation and demethylation[J]. Epigenomics, 2010, 2(6): 765-74. DOI:10.2217/epi.10.58 |
| [13] |
Morgan AE, Mooney KM, Wilkinson SJ, et al. Cholesterol metabolism:A review of how ageing disrupts the biological mechanisms responsible for its regulation[J]. Ageing Res Revi, 2016, 27: 108-124. DOI:10.1016/j.arr.2016.03.008 |
| [14] |
Pantsulaia I, Iobadze M, Kikodze N, et al. Lipid profile and cytokines interactions during successful aging[J]. Georgian Med News, 2015(243): 46-51. |
| [15] |
Goh VH, Hart WG. The association of metabolic syndrome and aging with cognition in Asian men[J]. Aging Male, 2014, 17(4): 216-22. DOI:10.3109/13685538.2014.968772 |
| [16] |
Walter M. Interrelationships among HDL metabolism, aging, and atherosclerosis[J]. Arterioscler Thromb Vasc Biol, 2009, 29(9): 1244-50. DOI:10.1161/ATVBAHA.108.181438 |
| [17] |
Postmus I, Deelen J, Sedaghat S, et al. LDL cholesterol still a problem in old age? A Mendelian randomization study[J]. Int J Epidemiol, 2015, 44(2): 604-12. |
| [18] |
吴燕丹. TC/HDL-C、LDL-C/HDL-C、TG/HDL-C与冠心病不同程度相关性[J]. 临床误诊误治, 2014, 27(6): 64-67. DOI:10.3969/j.issn.1002-3429.2014.06.027 |
| [19] |
Lettre G, Palmer C D, Young T, et al. Genome-wide association study of coronary heart disease and its risk factors in 8, 090 African Americans:the NHLBI CARe Project[J]. PLoS Genet, 2011, 7(2): e1001300. DOI:10.1371/journal.pgen.1001300 |
| [20] |
Congrains A, Kamide K, Oguro R, et al. Genetic variants at the 9p21 locus contribute to atherosclerosis through modulation of ANRIL and CDKN2A/B[J]. Atherosclerosis, 2012, 220(2): 449-55. |
| [21] |
王娜, 张学军. 冠心病患者外周血中lncrna-anril的表达水平及其临床意义[J]. 天津医科大学学报, 2019, 25(1): 54-8. |
| [22] |
Herman J G, Graff J R, Myohanen S, et al. Methylation-specific PCR:a novel PCR assay for methylation status of CpG islands[J]. Proc Natl Acad Sci USA, 1996, 93(18): 9821-6. DOI:10.1073/pnas.93.18.9821 |
| [23] |
Lo Y M, Wong I H, Zhang J, et al. Quantitative analysis of aberrant p16 methylation using real-time quantitative methylation-specific polymerase chain reaction[J]. Cancer Res, 1999, 59(16): 3899-903. |
| [24] |
Colella S, Shen L, Baggerly K A, et al. Sensitive and quantitative universal Pyrosequencing methylation analysis of CpG sites[J]. Biotechniques, 2003, 35(1): 146-50. DOI:10.2144/03351md01 |
| [25] |
Fraga M F, Esteller M. DNA methylation:a profile of methods and applications[J]. Biotechniques, 2002, 33(3): 632, 634, 636-49. |
| [26] |
Mikeska T, Bock C, El-Maarri O, et al. Optimization of quantitative MGMT promoter methylation analysis using pyrosequencing and combined bisulfite restriction analysis[J]. J Mol Diagn, 2007, 9(3): 368-81. |
 2019, Vol. 39
2019, Vol. 39

