寨卡病毒(ZIKV)是一种黄病毒科病毒,于1947年在乌干达首次被发现并从Zika森林的猴子体内分离,因此命名为Zika virus [1-2]。近年来,科学家们已证实ZIKV感染与胎儿脑部发育异常、神经系统疾病等并发症[3-4]密切相关。由于其导致的严重疾病和快速的传播速度,世界卫生组织于2016年2月将ZIKV疫情升级为全球关注的健康紧急事件[5],ZIKV迄今已扩散至全球80多个国家。遗憾的是,目前还没有治疗ZIKV感染的疫苗或特效药上市[6],因此,急需开发抗ZIKV药物。为了广泛筛选抗ZIKV药物,我们需要建立一种高效、简便、特异性高、准确可靠的方法。
目前抗寨卡病毒药物的体外筛选方法主要有以下几种:(1)实时荧光定量PCR法(RT-PCR),采用RT-PCR试剂盒检测病毒基因组或蛋白的表达量[7-8];(2)MTT法,由于ZIKV感染可能会导致细胞死亡,加入MTT,有活力的细胞才会产生紫色甲臜,用DMSO溶解后检测A570 nm,是一种基于细胞水平的药物活性筛选方法[9-11];(3)表面等离子共振法(SPR),实时跟踪在天然状态下化合物分子与病毒蛋白的相互作用,是一种基于靶点(分子水平)的高通量药物筛选方法[11];(4)ELISA方法,用专用的酶标板包被捕获抗体过夜,再加入病毒上清或细胞裂解液(抗原),然后加入二抗,显色,检测A450 nm [12-13]。上述方法中,RT-PCR法与SPR法的检测价格昂贵且SPR对仪器要求高;MTT法的准确性不高(低滴度的ZIKV感染引起的细胞病变不一定会导致细胞死亡);传统的ELISA方法步骤繁琐,耗时长。本研究拟开发一种基于细胞的ELISA方法直接检测细胞内的ZIKV蛋白的相对表达量。
ZIKV基因组RNA入胞后在内质网经翻译生成多聚蛋白,与此同时,病毒和宿主蛋白酶(NS2B-NS3蛋白酶和信号肽酶)将多聚蛋白裂解成3种结构蛋白:衣壳蛋白、包膜蛋白以及前体膜(prM),和7种非结构(NS)蛋白(NS1、NS2A、NS2B、NS3、NS4A、NS4B和NS5)[14-16]。其中,E蛋白是病毒感染和复制的必需蛋白质,介导病毒与细胞相关受体的结合,促进病毒与细胞膜的融合,也是黄病毒感染的主要抗原标志物,可用于开发新型的治疗和诊断工具[17-18]。
在精液淀粉样纤维对ZIKV性传播影响的研究中使用了基于细胞的ELISA方法[19],但是该方法中应用的一抗是黄病毒属E蛋白的广谱抗体,由于ZIKV与其他黄病毒在基因结构上高度相似[20-21],可能有交叉反应,导致方法特异性不高。另外,细胞培养板孔中的细胞空隙没有被封闭可能导致背景过高,影响结果准确性。
本研究通过优化基于细胞的ELISA方法,将培养好的细胞固定在96孔板上,增加封闭步骤降低背景值,加入ZIKV抗体与之特异性结合,从而省去了用抗体包被过夜的环节,大大缩短了检测时间,最终建立一种简便快捷、特异性高且准确可靠的药物筛选方法。
1 材料和方法 1.1 实验材料 1.1.1 细胞株和毒株Vero-143细胞:非洲绿猴肾细胞,由广东省疾病预防控制中心病原微生物检验所提供,用含10%胎牛血清、1%双抗的高糖DMEM细胞培养基培养。
ZIKV毒株:Z16006(GenBank:955589.1)血液分离株(浙江首例),亚洲谱系,由广东省疾病预防控制中心病原微生物检验所提供,经本科室通过Vero-143细胞扩增,于-80 ℃冰箱保存。
1.1.2 主要试剂DMEM高糖培养基、双抗、胎牛血清(FBS)、0.25%胰蛋白酶/EDTA(Gibco);Zika E蛋白多抗(GTX133314,GeneTex);辣根过氧化物酶HRP标记的二抗(CST);二甲亚砜DMSO(Zymed);甲基纤维素(Sigma);冰醋酸(天津市大茂化学试剂厂);多聚甲醛(PFA)(北京雷根生物技术有限公司);吐温20(广州化学试剂厂)。用于验证方法的木脂素类化合物双细辛酮(代号C1)由石菖蒲植物中分离得到。
1.1.3 实验仪器二级生物安全柜(ESCO);恒温CO2细胞培养箱(Thermo);高速离心机(Eppendorf);酶标仪(Tecan);-80 ℃冰箱(青岛海尔股份有限公司);倒置光学显微镜(Zeiss)。
1.2 实验方法 1.2.1 ZIKV病毒感染及加药Vero-143细胞以1×105/mL、每孔100 μL接种于96孔细胞培养平板,培养过夜;次日,弃去旧培养基,细胞用PBS洗涤2次;寨卡病毒稀释至100 TCID50,每孔细胞加入50 μL,感染1 h;吸出病毒接种液,每孔加入200 μL含药维持培养基(含2% FBS、1%双抗的DMEM),每个浓度设置3个复孔,同时设置阴性对照组、仅加病毒的阳性对照组,继续培养96 h。
1.2.2 ELISA法检测ZIKV E蛋白细胞培养物弃上清,用PBS洗涤2次;加入4%多聚甲醛,室温下固定20 min;加入冰冻的无水甲醇,于4 ℃通透细胞5 min;PBS洗涤1次;加入寨卡病毒E蛋白一抗,37 ℃孵育1 h;用洗涤液(含0.3%吐温20)洗涤4次;加入HRP-标记的二抗,37 ℃孵育1 h;洗涤5次,加入TMB底物,室温下避光显色5 min;加入1 mmol/L的硫酸终止反应,用酶标仪检测450 nm处的吸光度值(A450 nm)。
1.2.3 RT-PCR实验检验ELISA方法的准确性Vero-143细胞以2×105/mL、1 mL/孔接种于12孔板,培养过夜;弃去旧培养基,按上述设置空白对照组、病毒对照组、化合物C1处理组,培养72 h后,提取各组细胞总RNA,参照Takara逆转录试剂盒说明书,将mRNA逆转录为cDNA;随后按Promega RT-PCR试剂盒说明书进行RTPCR检测,E引物序列:上游引物GGTGGGACTTGG GTTGATGT,下游引物ATGTCACCAGGCTCCCT TTG;内参基因GADPH引物序列:上游引物TTGCAT CGCCAGCGCATC,下游引物TCGCCCCACTTGAT TTTGGA。根据各组样品的CT值来计算2-ΔΔCт,算出各组样品相对于病毒对照组的RNA水平。
1.2.4 检测ELISA方法的交叉反应性Vero-143细胞以1×105/mL、每孔100 μL接种于96孔细胞培养平板,培养过夜;次日,弃去旧培养基,细胞用PBS洗涤2次;登革病毒、寨卡病毒分别稀释至100 TCID50,每孔细胞加入50 μL,感染1 h;吸出病毒接种液,每孔加入200 μL维持培养基,各设置3个复孔,同时设置阴性对照组,继续培养96 h。使用倒置荧光显微镜拍摄细胞病变情况,接着用已建立的ELISA方法检测登革病毒、寨卡病毒组及阴性对照组的寨卡病毒E蛋白,见1.2.2。
1.3 数据处理实验结果采用Graphpad Prism 5软件进行作图及数据的统计分析,两组间比较采用Student's t检验,多组比较则用One-way ANOVA进行分析。P<0.05为差异有统计学意义。
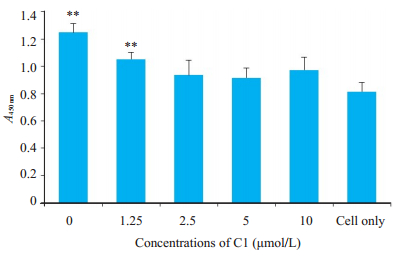
2 结果 2.1 初步建立的E蛋白ELISA实验方法导致高背景按照1.2.1的方法培养Vero-143细胞并加入寨卡病毒感染细胞,将化合物C1稀释成不同浓度加入培养物中,使其终浓度依次为0、1.25、2.5、5、10 μmol/L,未加病毒和药物的细胞为背景对照。然后按照1.2.2的ELISA步骤检测细胞中E蛋白的表达量(图 1),C1具有抗寨卡病毒活性,但背景对照值太高,导致所有结果都偏高,可能影响结果的准确性,也容易产生假阳性。
|
图 1 化合物C1抑制寨卡病毒复制的E蛋白ELISA检测结果 Fig.1 Detection of inhibitory effect of compound C1 on zika virus replication using E protein-based ELISA. **P < 0.01 vs cells only group. |
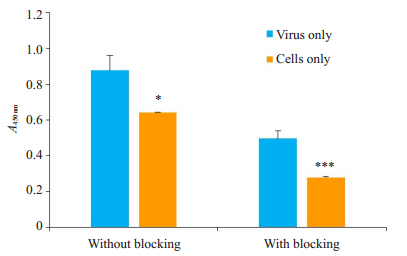
ELISA实验背景高的可能原因有抗体浓度、封闭液及洗涤液的成分和浓度等,我们初步建立的寨卡病毒E蛋白ELISA方法中,未用到封闭步骤,由于细胞固定于孔板后,孔表面尚有未被占据的空隙,用封闭的方法可以排除后续步骤中干扰物质的再吸附,因此,我们增加了2%的脱脂牛奶封闭的步骤。具体操作如下:吸去甲醇,用PBS洗涤后,加入2%的脱脂牛奶,150 μL/孔,在37 ℃封闭1 h。然后洗涤液洗涤3次。接下来按照1.2.2的方法加入一抗(1:1000稀释),继续完成后续步骤,二抗为1:2000稀释。背景对照组的A450 nm值降低0.22,病毒对照组的值也较封闭前降低0.23(图 2)。
|
图 2 2%的脱脂牛奶封闭能降低背景 Fig.2 The background was reduced by blocking with 2% skimmed milk. *P < 0.05, ***P < 0.001 vs without blocking group. |
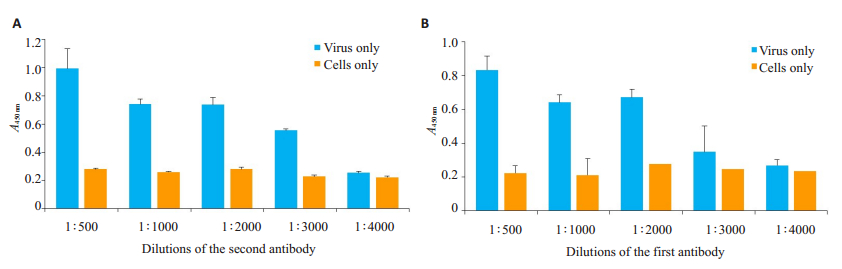
接下来,在增加封闭步骤的基础上,我们对一抗、二抗的浓度进行了摸索。首先固定一抗稀释倍数为1:1000,对二抗进行1:500、1:1000、1:2000、1:3000、1:4000稀释,结果如图 3A所示,二抗1:500、1:1000、1:2000稀释,病毒组的A450 nm值较为适宜(0.60~1.00),背景对照值也维持在0.20左右,为了节约抗体成本,二抗稀释倍数定为1:2000。然后也对一抗进行1:500、1:1000、1:2000、1:3000、1:4000稀释,结果如图 3B所示,一抗1:500、1:1000、1:2000稀释时,病毒组的A450 nm值较为适宜(0.60~1.00),背景对照值相差不大,为了节约成本,一抗稀释倍数也确定为1:2000。
|
图 3 抗体稀释倍数的确定 Fig.3 Determination of the dilution ratio of antibodies in ELISA. A: Determination of the dilution ratio of the 2nd antibody; B: Determination of the dilution ratio of the 1st antibody. |
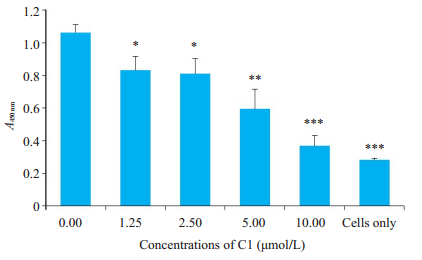
在前面的研究中,我们对基于细胞的ELISA方法进行了优化:孵育一抗前增加封闭步骤、E蛋白多抗作2000倍稀释、二抗作2000倍稀释,可大大降低背景,提高方法的准确性。接着我们应用优化后的ELISA方法检验化合物C1的抗寨卡病毒效果。结果发现,化合物C1呈浓度依赖性地抑制寨卡病毒E蛋白的合成(如图 4),优化后的E蛋白ELISA方法能满足检测寨卡病毒的需求,所有检测值都在合理范围内。
|
图 4 用ELISA方法检测化合物C1的抗病毒效果 Fig.4 Antiviral effect of C1 detected by ELISA. *P < 0.05, **P < 0.01, ***P < 0.001 vs virus only group. |
如前所述,RT-PCR方法是检测寨卡病毒的常用方法,我们最后采用RT-PCR法进一步检验本研究中建立的ELISA法的准确性。寨卡病毒感染细胞后,用浓度梯度的化合物C1持续处理72 h后,提取细胞总RNA,RT-PCR检测细胞内E RNA的合成水平。实验结果如图 5所示,化合物C1浓度依赖抑制E RNA的合成,与ELISA方法的结果基本一致,说明优化后的ELISA方法检测寨卡病毒E蛋白表达水平的结果是准确可靠的,可以作为RT-PCR法的补充或替代方法。
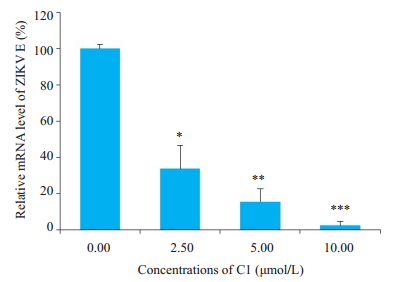
|
图 5 RT-PCR法检验ELISA方法的准确性 Fig.5 Validation of the accuracy of ELISA by RT-PCR. *P < 0.05, **P < 0.01, ***P < 0.001 vs virus only group. |
寨卡病毒与登革病毒的E蛋白在结构上相似度非常高,在应用ELISA法检测时可能会发生交叉反应。为了考察该ELISA方法的特异性,我们对比了寨卡病毒和登革病毒。如图 6A所示,加入100TCID50的登革病毒和寨卡病毒分别感染细胞96 h,ZIKV组和DENV组的细胞都发生了明显的细胞病变效应(CPE);相应的ELISA结果如图 6B,ZIKV组的A450 nm达到1.29,而DENV组的A450 nm与阴性对照组(背景)的A450 nm值没有显著性差异,约为0.34。以上结果提示,我们建立的ELISA方法对登革病毒没有交叉反应,对寨卡病毒具有高度特异性。
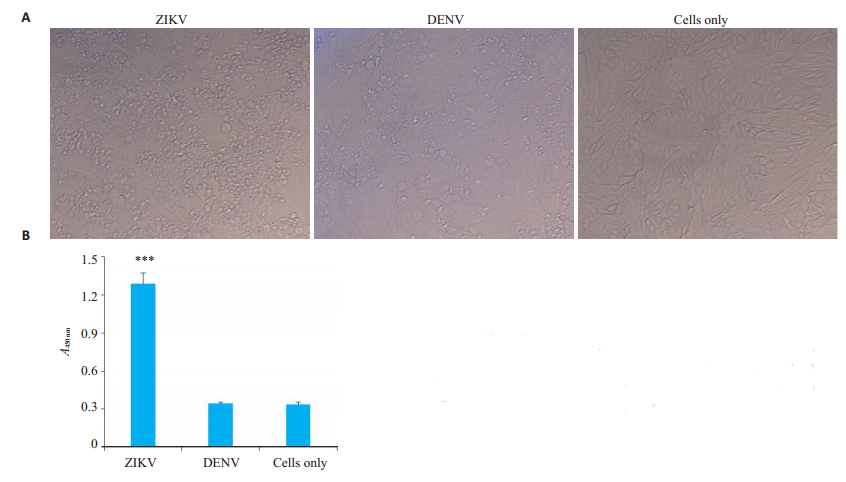
|
图 6 ELISA方法对登革病毒的交叉反应性 Fig.6 Cross reaction of ELISA with dengue virus (Original magnification: × 20). A: Morphology of Vero cells infected with 100TCID50 ZIKV and DENV; B: Cross reaction of ELISA with dengue virus. ***P < 0.001 vs cells only group. |
寨卡病毒并非新兴的病毒,由于寨卡病毒感染与胎儿小头症和睾丸损伤等[11]密切关联,完全超越此前对虫媒黄病毒的固有认识,因此迫切需要用新眼光来研究这个老病毒。目前对于寨卡病毒的检测、诊断方面的技术一般依赖于RT-PCR方法[22-23]。
如前所述,目前抗寨卡病毒药物筛选的方法有RTPCR、MTT、SPR、传统的包被ELISA、基于细胞的细胞病变或抑制噬斑形成等方法[24]。预包被的ELISA方法也是检测寨卡病毒的主要方法之一,检测血清中的寨卡病毒抗体或抗原[11, 25-26],但目前还没有非包被ELISA方法用于抗寨卡病毒药物筛选的报道。本研究中,我们建立的非包被ELISA方法不仅可用于寨卡病毒的检测,还可以用于抗寨卡病毒药物的体外筛选。非包被ELISA方法,即直接在细胞培养板上操作,省去了传统ELISA法中的抗体包被步骤,既节省时间又节省成本。尤其是当上清液中的病毒滴度不够高时,这种检测胞内病毒的方法便是首选,因为细胞是病毒的主要储库,胞内的病毒量远远高于上清液。
由于培养孔中细胞空隙的存在,部分一抗会与空隙非特异性结合,导致背景过高,容易出现假阳性。为了降低背景,我们在孵育一抗前加入2%脱脂牛奶封闭1 h,结果发现,增加封闭步骤可以在不降低检测准确性的同时显著降低背景。除此之外,一抗、二抗的浓度也是影响背景值的重要因素。接下来,根据抗体说明书推荐的稀释范围,分别对一抗、二抗作1:500、1:1000、1:2000、1:3000、1:4000稀释,考察仅加病毒组、细胞对照组(背景)的A值大小。最终确定较理想的实验条件:增加封闭步骤、一抗1:2000稀释、二抗1:2000稀释。
Vicenti等成功应用qRT-PCR方法检测细胞内E蛋白RNA的表达水平来进行抗寨卡病毒药物筛选[27]。接下来,为了评估优化后的ELISA方法,我们同时将ELISA方法、RT-PCR方法应用于抗寨卡病毒药物筛选,对药物抗病毒效果进行了一致性研究。结果显示,ELISA方法检测到化合物C1能浓度依赖性地抑制寨卡病毒E蛋白的合成,且RT-PCR的结果与ELISA方法的结果基本一致,化合物C1同样浓度依赖性地降低E蛋白RNA的相对表达水平,这充分证实了本研究中优化后的ELISA方法的可靠性。
我们还通过考察该ELISA方法对登革病毒的敏感性,研究该方法对其它黄病毒的交叉反应,在两种病毒对同种细胞产生相当的病变效应的情况下,寨卡病毒组的检测值达到1.29,而登革病毒组的检测值与阴性对照组没有显著性差异,说明该方法与登革病毒不存在交叉反应。另外,我们还分别用小鼠来源的4G2抗血清和NS1单抗建立了相应的非包被ELISA方法(数据未列出),也得到了稳定的结果,这间接说明该ELISA方法的可靠性。因此,本研究建立的ELISA方法可以用于抗寨卡病毒药物筛选或者作为其它筛选方法的补充。
| [1] |
Dick GW, Kitchen SF, Haddow AJ. Zika virus.I.Isolations and serological specificity[J]. Trans R Soc Trop Med Hyg, 1952, 46(5): 509-20. DOI:10.1016/0035-9203(52)90042-4 |
| [2] |
Macnamara FN. Zika virus: a report on three cases of human infection during an epidemic of jaundice in Nigeria[J]. Trans R Soc Trop Med Hyg, 1954, 48(2): 139-45. DOI:10.1016/0035-9203(54)90006-1 |
| [3] |
Mlakar J, Korva M, Tul N, et al. Zika virus associated with microcephaly[J]. N Engl J Med, 2016, 374(10): 951-8. DOI:10.1056/NEJMoa1600651 |
| [4] |
Rasmussen SA, Jamieson DJ, Honein MA. Zika virus and birth defects - reviewing the evidence for causality[J]. N Engl J Med, 2016, 374(20): 1981-7. DOI:10.1056/NEJMsr1604338 |
| [5] |
WHO. The History of Zika Virus. Available online: http://www.who.int/emer-gencies/zika-virus/history/en/ (accessed on 17 July 2018).
|
| [6] |
Saiz JC, Martin-Acebes MA. The race to find antivirals for Zika virus[J]. Antimicrob Agents Chemother, 2017, 61(6): e00411-17. |
| [7] |
Karim MR, Fout GS, Johnson CH, et al. Propidium monoazide reverse transcriptase PCR and RT-qPCR for detecting infectious enterovirus and norovirus[J]. J Virol Methods, 2015, 219(7): 51-61. |
| [8] |
Balm MN, Lee CK, Lee HK, et al. A diagnostic polymerase chain reaction assay for Zika virus[J]. J Med Virol, 2012, 84(9): 1501-5. DOI:10.1002/jmv.v84.9 |
| [9] |
Stepanenko AA, Dmitrenko VV. Pitfalls of the MTT assay: Direct and off-target effects of inhibitors can result in over/ underestimation of cell viability[J]. Gene, 2015, 574(2): 193-203. |
| [10] |
Muller JA, Harms M, Schubert A, et al. Development of a highthroughput colorimetric Zika virus infection assay[J]. Med Microbiol Immunol, 2017, 206(2): 175-85. DOI:10.1007/s00430-017-0493-2 |
| [11] |
Adcock RS, Chu YK, Golden JE, et al. Evaluation of anti-Zika virus activities of broad-spectrum antivirals and NIH clinical collection compounds using a cell-based, high-throughput screen assay[J]. Antiviral Res, 2017, 138(2): 47-56. |
| [12] |
Shan C, Ortiz DA, Yang YJ, et al. Evaluation of a novel reporter virus neutralization test for serological diagnosis of Zika and dengue virus infection[J]. J Clin Microbiol, 2017, 55(10): 3028-36. DOI:10.1128/JCM.00975-17 |
| [13] |
Kuno G, Chang G. Full-length sequencing and genomic characterization of Bagaza, Kedougou, and Zika viruses[J]. Arch Virol, 2007, 152(4): 687-96. DOI:10.1007/s00705-006-0903-z |
| [14] |
Chambers TJ, Hahn CS, Galler R, et al. Flavivirus genome organization, expression, and replication[J]. Annu Rev Microbiol, 1990, 44: 649-88. DOI:10.1146/annurev.mi.44.100190.003245 |
| [15] |
Shi Y, Gao GF. Structural biology of the Zika virus[J]. Trends Biochem Sci, 2017, 42(6): 443-56. DOI:10.1016/j.tibs.2017.02.009 |
| [16] |
Dai L, Wang Q, Song H, et al. Zika virus envelope protein and antibody complexes[J]. Subcell Biochem, 2018, 88: 147-68. DOI:10.1007/978-981-10-8456-0 |
| [17] |
Saiz JC, Jimenez de Oya N, Blazquez AB, et al. Host-Directed antivirals: a realistic alternative to fight Zika virus[J]. Viruses, 2018, 10(9): e453. DOI:10.3390/v10090453 |
| [18] |
Mueller JA, Harms M, Krueger F, et al. Semen inhibits Zika virus infection of cells and tissues from the anogenital region[J]. Nat Commun, 2018, 9(1): 2207. |
| [19] |
Gunawardana SA, Shaw RH. Cross-reactive dengue virus-derived monoclonal antibodies to Zika virus envelope protein: Panacea or Pandora's box?[J]. BMC Infect Dis, 2018, 18(1): 641. DOI:10.1186/s12879-018-3572-0 |
| [20] |
Barrows NJ, Campos RK, Powell ST, et al. A screen of FDAApproved drugs for inhibitors of Zika virus infection[J]. Cell Host Microbe, 2016, 20(2): 259-70. DOI:10.1016/j.chom.2016.07.004 |
| [21] |
He AL, Brasil P, Siqueira AM, et al. The emerging Zika virus threat: a guide for dermatologists[J]. Am J Clin Dermatol, 2017, 18(2): 231-6. |
| [22] |
Singh RK, Dhama K, Karthik K, et al. Advances in diagnosis, surveillance, and monitoring of Zika virus: an update[J]. Front Microbiol, 2018, 8(1): 2677. |
| [23] |
Steinhagen K, Probst C, Radzimski C, et al. Serodiagnosis of Zika virus (ZIKV) infections by a novel NS1-based ELISA devoid of cross-reactivity with dengue virus antibodies: a multicohort study of assay performance, 2015 to 2016[J]. Euro Surveill, 2016, 21(50): 30426. DOI:10.2807/1560-7917.ES.2016.21.50.30426 |
| [24] |
Lee KH, Zeng HQ. Aptamer-Based ELISA assay for highly specific and sensitive detection of Zika NS1 protein[J]. Anal Chem, 2017, 89(23): 12743-8. DOI:10.1021/acs.analchem.7b02862 |
| [25] |
De Ory F, Paz Sanchez-Seco M, Vazquez AA, et al. Comparative evaluation of indirect immunofluorescence and NS-1-Based ELISA to determine Zika Virus-Specific IgM[J]. Viruses, 2018, 10(7): e379. DOI:10.3390/v10070379 |
| [26] |
Kikuti M, Tauro LB, Moreira PS, et al. Diagnostic performance of commercial IgM and IgG enzyme- linked immunoassays (ELISAs) for diagnosis of Zika virus infection[J]. Virol J, 2018, 15(1): 108. DOI:10.1186/s12985-018-1015-6 |
| [27] |
Vicenti I, Boccuto A, Giannini A, et al. Comparative analysis of different cell systems for Zika virus(ZIKV)propagation and evaluation of anti-ZIKV compounds in vitro[J]. Virus Res, 2018, 244(1): 64-70. |
 2019, Vol. 39
2019, Vol. 39

