表观遗传参与生命活动多个方面,调节机体发育和细胞分化,与多种疾病的发生发展密切相关[1-3]。组蛋白翻译后修饰是重要的表观遗传特征[4]。染色质免疫共沉淀(ChIP)是一种研究组蛋白修饰的重要技术,主要包括交联的ChIP(X-ChIP)与非交联的ChIP(N-ChIP)。N-ChIP没有甲醛交联试剂破坏抗原表位,其抗原与抗体的亲和力比交联后的细胞亲和力更高,特异性更强,因此有更好的信噪比[5]。由于组蛋白和DNA结合稳定,可以采用非交联ChIP技术进行检测[6]。
N-ChIP主要过程包括对活性细胞进行裂解,酶切释放出核小体,特异性抗体与核小体组蛋白进行免疫共沉淀得到目的染色质,蛋白经过消化后得到目的DNA [7-8]。ChIP产物可以进行定量PCR检测或者构建二代测序文库进行全基因组检测,得到组蛋白修饰信息[9-11]。非交联ChIP-seq技术原理简单,但是其实验过程存在一些技术难点[7]。针对这些技术难点,本研究致力于对建库方法的优化,解决N-ChIP-seq建库中的一些问题。传统的ChIP建库采用末端修复、接头连接、文库富集的方法。但是传统建库过程步骤繁琐,且建库后测序数据覆盖度存在一定问题[12]。本研究使用一种新的建库方法,即Tn5转座酶片段化法进行N-ChIP的建库,并将两种建库方法进行实验操作流程、库检结果、测序质量的分析比较。
1 材料和方法 1.1 细胞株鼻咽癌细胞株HONE1,包括EBV阳性和阴性两种亚型,是由姚开泰、Ronald Glaser等人建立的低分化鼻咽癌细胞系[13],由本研究所储存。
1.2 主要试剂和仪器1640培养基、胎牛血清以及青霉素/链霉素双抗、非必需氨基酸购自Gibco。END Prep mix2、DNA纯化磁珠、2 × Phanta Master Mix,TruePrep Index kit v2 for illumina、TruePrep DNALibrary Prep kit v2 for illumina均购自南京诺唯赞生物科技有限公司;DNA纯化试剂盒、胶回收试剂盒均购自Qiagen;微球菌核酸酶(Micrococcal nuclease,MNase)购自Worthington;Quick Ligse Kit(NEB);Protein A/G磁珠(Pierce),蛋白酶K(Invitrogen)。其余粉末均购自Sigma-aldrich。旋转仪(Kylin Bell)、离心机(Eppendorf)、PCR仪(Applied Biosystems)、荧光定量PCR仪(Applied Biosystems)、Qubit(Invitrogen)。
1.3 主要试剂的配方细胞裂解液的配方为:80 mmol/L NaCl,10 mmol/L Tris-HCl pH 7.5,10 mmol/L丁酸钠,6 mmol/L MgCl2,1 mmol/L CaCl2,250 mmol/L蔗糖;MNase酶切缓冲液的配方为:10 mmol/L NaCl,10 mmol/L Tris-HCl pH7.5,10 mmol/L丁酸钠,3 mmol/L MgCl2,1 mmol/L CaCl2,250 mmol/L蔗糖;1×IP稀释缓冲液配方为16.7 mmol/L Tris pH 8.0,1.2 mmol/L EDTA,167 mmol/L NaCl,1.1% TritonX-100,0.01% SDS,10 mmol/L丁酸钠;TSE缓冲液配方为:20 mmol/L Tris-HCl pH8.1,2 mmol/L EDTA,1% Triton X-100,0.1% SDS,300 mmol/L NaCl,10 mmol/L丁酸钠;LiCl缓冲液配方为:50 mmol/L Tris PH8.0,0.25 mol/L LiCl,0.5% NP40,0.5%脱氧胆酸钠,10 mmol/L丁酸钠。
1.4 细胞培养及处理将贴壁细胞HONE1培养在RPMI 1640完全培养基中(10%胎牛血清以及1%的非必需氨基酸和1%的青霉素/链霉素双抗),在5% CO2压力条件下,37 ℃的恒温培养箱中培养。待细胞生长密度为约80%时,用细胞刮轻轻将细胞刮下来,用PBS洗1遍,在1500 r/min的转速在4 ℃离心5 min。收集细胞后,用1 mL细胞裂解液在冰上裂解10 min,在2500 r/min的转速在4 ℃离心5 min,弃上清,沉淀为细胞核。
1.5 ChIP样品的准备将细胞核用酶切缓冲液重悬,用微球菌核酸酶(MNase)在37 ℃孵育5 min。加入0.2 mol/L的EDTA/ EGTA混合液终止酶解。产物在12 000 r/min转速下,4 ℃离心5 min,得到释放后的核小体。将酶切后的染色质进行电泳验证,选择合适酶切浓度。在样品中加入IP稀释缓冲液,在最后加入2 μL组蛋白修饰抗体(如H3K4me1、H3K4me3抗体等),样品放于旋转仪上,在4 ℃冰箱里旋转过夜。
1.6 目的染色质的富集与纯化样品置于冰上,加入Protein A/G磁珠,放于旋转仪上,在4 ℃冰箱里旋转4 h。将样品放于磁力架上,放置5 min后,将未结合的上清液移入新的离心管中,放于4 ℃保存。用1 mL冷的1×稀释缓冲液、1×TSE缓冲液、LiCl缓冲液、1×Tris-HCl各洗2遍,弃上清。最后用33 μL的ChIP洗脱缓冲液重悬磁珠,加入1 μL 10 mg/mL蛋白酶K,55 ℃条件下消化1 h。取上清,用33 μL的ChIP洗脱缓冲液洗两遍磁珠并合并上清。严格按照说明书,用DNA纯化试剂盒、Qubit定量检测试剂盒将DNA进行纯化、定量。最终得到30 μL洗脱缓冲液洗脱的DNA样品。
1.7 ChIP文库的富集 1.7.1 传统建库首先进行DNA的末端修复。反应体系为:DNA(2.86 ng/μL)30 μL,END Prep mix 215 μL,ddH2O加至65μL。反应条件为:20℃15min,65℃15min,4 ℃保存。用1.8×DNA纯化磁珠(117 μL)纯化样品,严格按照说明书,用10 μL纯水洗脱,取上清。然后将DNA末端加接头。反应体系为:DNA 10 μL,2×Quick Ligse缓冲液12.5 μL,10 μmol/L Adaptor 1 μL,Quick Ligase 1.5 μL。在常温静置反应15 min;用1×DNA纯化磁珠(25 μL)纯化,最后用23 μL ddH2O洗脱,取上清。进行PCR文库扩增。配制37 μL PCR混合液:2×Phanta Master Mix34.3 μL,10 μmol/L Multiplexing引物1.37 μL,10 μmol/L Multiplexing index1.37 μL。PCR反应体系为:在DNA23 μL,PCR混合液27 μL。PCR反应条件:95 ℃预变性3 min;95 ℃ 15 s,65 ℃ 15 s,72 ℃ 30 s,5个循环;4 ℃保存。将PCR产物置于冰上,用定量PCR仪进行监测反应体系为:PCR产物5 μL,PCR混合液10 μL,20×evagreen 1 μL,ddH2O加至20 μL。反应条件与PCR一致,确定还需要增加的循环数x个。完成PCR反应:95 ℃ 15 s,65 ℃ 15 s,72 ℃ 30 s,x个循环;72 ℃最终延伸5 min;4 ℃保存。用1×DNA纯化磁珠(50 μL)进行纯化,纯化方法按照磁珠说明书进行。用10 μL ddH2O进行洗脱,加入0.5×DNA纯化磁珠(5 μL)进行片段选择,取上清,1×DNA纯化磁珠(15 μL)再次进行纯化,最终用20 μL ddH2O洗脱。最终用Qubit进行文库定量。
1.7.2 Tn5转座酶建库一般得到的DNA浓度较低,用TruePrep DNA Library Prep kit v2 for illumines进行1ng起始DNA量的文库富集。Tn5片段化反应体系:5×TTBL4 μL,DNAxμL,TTE Mix V 15 μL,ddH2O加至20 μL。反应条件:55 ℃ 10 min;10 ℃保存。立即向反应产物中加入5×TS,室温静置5 min终止片段化过程。然后进行文库富集。PCR反应体系:片段化DNA25 μL,5 × TAB 10 μL,N5XX 5 μL,N7XX 5 μL,TAE 1 μL,ddH2O加至50 μL。PCR仪中反应条件:72 ℃ 3 min;98 ℃预变性30 s;98 ℃ 15 s,60 ℃ 30 s,72 ℃ 3 min,5个循环;4 ℃保存,将PCR产物置于冰上,进行PCR监测确定循环数后继续进行PCR反应。DNA文库纯化、片段选择、定量与与传统建库方法一致。
1.8 文库质检与测序使用Bioanalyzer检测文库DNA片段的完整性及插入片段大小,用定量PCR对文库DNA拷贝数进行精确定量。文库检测合格后,按照有效浓度及目标下机数据量的需求将不同文库混合使用Illumina高通量测序平台(HiSeq)进行双端测序。
1.9 NGS生物信息学分析质量控制软件fastp(version 0.19.6)可以去除原始测序数据(Raw Data)中测序质量低的测序读长(Reads),切掉Reads中的测序接头序列。
用比对软件bowtie2将质量控制之后的数据(Clean Data)比对回参考基因组,去除PCR重复(软件:sambamba),提取比对质量大于30的测序读长(软件:samtools);将比对到基因组上的测序读长数目标准化为RPKM(Reads Per Kilobase per Million mapped reads)值(软件:deeptools),并得到bigwig文件用于信号峰图可视化(可视化工具:IGV)。
比对后分析包括样本间Reads基因组范围覆盖度的Pearson相关性计算(deeptools工具);Reads在基因组上显著富集区域的鉴定(Peak Calling)(HOMER工具);信噪比评估参数FRiP(Fragment Ratio in Peaks)值的计算(软件bedtools,samtools);以及不同建库方法得到的富集峰区域之间重叠关系的评估(R包ChIPseeker和ChIPpeakAnno)。
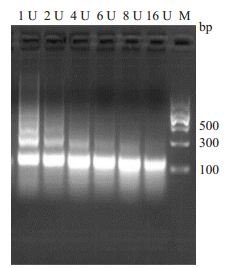
2 结果 2.1 MNase酶切验证与浓度选择由于不同类型细胞对MNase的敏感性各异,因此ChIP之前需要选择MNase的最适浓度。我们使用1、2、4、6、8、16 U浓度梯度的MNase切割DNA,电泳检测DNA片段大小。当DNA片段集中在1~2个核小体长度的位置时的酶浓度为适宜。从图 1可以看出,对于HONE1细胞系,8 U的MNase酶切割的DNA片段长度集中在1~2个核小体。
|
图 1 MNase酶切测试 Fig.1 MNase digestion test. |
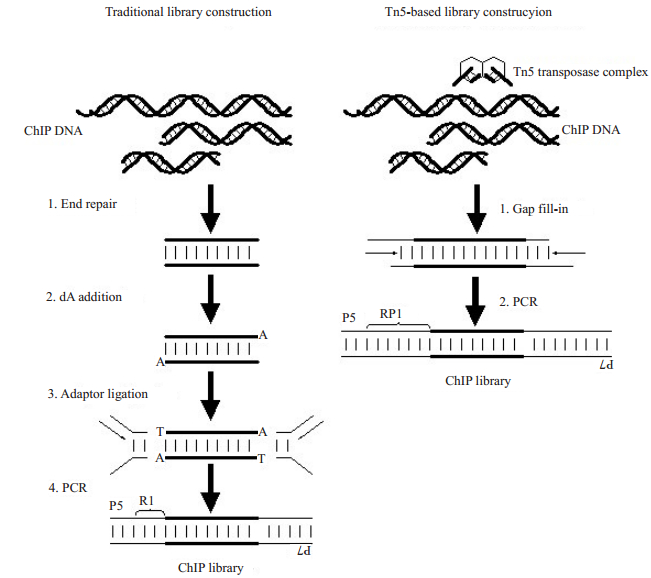
传统ChIP-seq文库的构建,包括末端修复、末端加入接头、文库的富集以及文库片段的选择,而Tn5转座酶建库将目的DNA片段化后直接进行文库的富集(图 2)。在建库过程中,Tn5转座酶建库使用1 ng DNA起始量,需要11~15个PCR循环数;一般传统建库的起始量要求50~200 ng,PCR循环数为15~20个。
|
图 2 传统建库(左图)与Tn5片段化建库(右图)策略流程 Fig.2 Diagram illustrating the conventional (left) and Tn5 fragmentation-based (right) strategies for DNA library construction. |
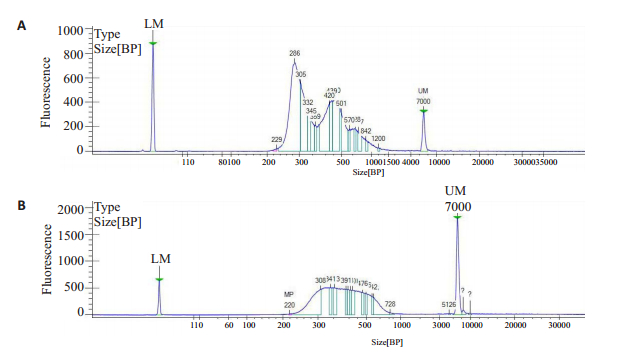
图 3A为传统建库后库检结果,文库大小集中在285 bp或450 bp,这代表了插入片段为一个或者两个核小体DNA片段大小。图 3B中,Tn5片段化建库后的文库主峰在250~500 bp范围内,得到的片段更加均一。
|
图 3 传统建库与Tn5片段化建库后H3K4me1ChIP文库质检 Fig.3 Quality assessment of H3K4me1 ChIP libraries constructed by conventional (A) and Tn5 fragmentation-based (B) methods. |
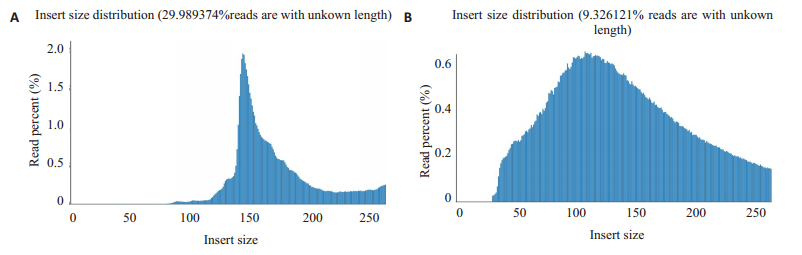
组蛋白修饰的非交联ChIP理论上能得到的DNA片段长度为一个组蛋白上大小(146 bp)。在质量控制过程中,对传统建库和Tn5转座酶建库的H3K4me1 ChIP文库插入片段大小进行分布统计。传统建库文库插入片段长度集中在一个组蛋白大小的位置,含有约30%的片段拖尾(>300 bp);Tn5转座酶建库后文库片段大小主要分布在100~150 bp之间,仅9%的片段拖尾(>300 bp)。两种建库方法得到的DNA片段大小均合理,传统建库得到的插入片段更加集中(图 4)。
|
图 4 Fastp插入文库大小分布统计比较 Fig.4 Statistics of fastp library insertion length. A: Conventional library; B: Tn5 transposase library. |
H3K4me1 ChIP实验的目的是为了富集H3K4me1结合的DNA片段,将高通量测序之后得到的Reads比对回参考基因组后,用IGV可视化软件查看信号峰图文件,Reads富集区域就是H3K4me1区域,在图中反映为信号峰图。图 5显示两种建库方式获得的信号富集峰图基本一致。
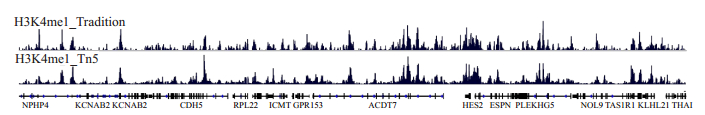
|
图 5 H3K4me1 ChIP-seq Reads覆盖峰图 Fig.5 H3K4me1 ChIP-seq Reads coverage peak map. Peaks shown in the map represent enriched region. |
两种建库方法得到的测序数据用HOMER软件鉴定Reads在基因组上显著富集区域(Peak Calling)。Tn5转座酶建库方法获得99 073个富集峰,传统建库方法获得85 060个富集峰。统计两种建库方法获得的富集峰的重叠关系,有63 897个Peak是两种建库方法共有的,占Tn5建库富集峰总数的64.45%,占传统建库富集峰总数的75.12%(图 6)。
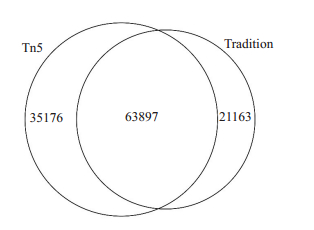
|
图 6 传统建库与Tn转座酶建库后文库富集区域Venn图 Fig.6 Venn diagram of library enrichment region with conventional and Tn5 fragmentation-based DNA library construction. |
为评估Tn5转座酶建库的稳定性,用Tn5转座酶方法对H3K4me3 ChIP三次生物学重复文库的建立,并计算3次重复试验得到的Reads在全基因组覆盖度的Pearson相关性系数。结果显示3次生物学重复的pearson相关系数接近1(图 7);同时IGV可视化局部信号分布峰图显示,Tn5转座酶建库3次生物学重复获得的Reads富集信号峰高度一致(图 8)。MACS2鉴定3次试验Reads覆盖富集区域,计算FRiP值。FRiP值越大,背景越低。Tn5转座酶建库后H3K4me3 ChIP信噪比分别为58.69%、84.19%、73.93%。
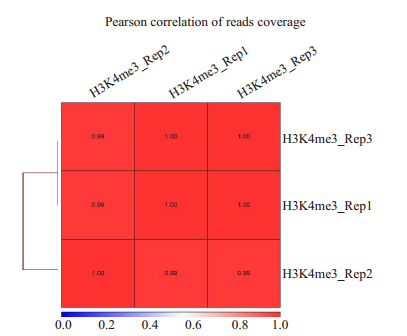
|
图 7 Tn转座酶建库后H3K4me3 ChIP pearson相关分析 Fig.7 Correlation analysis of H3K4me3 ChIP pearson after Tn5 transposase library construction. |
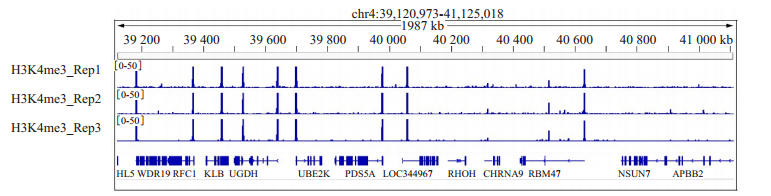
|
图 8 Tn5转座酶建库后H3K4me3 ChIP-seq信号峰图相似性分析 Fig.8 Similarity analysis of H3K4me3 ChIP-seq signal peak map after Tn5 transposase library construction. |
染色质免疫沉淀及其高通量DNA测序(ChIP-seq)是一种有价值且广泛使用的分子生物学技术,用于检测DNA与蛋白质相互作用[14]。许多组蛋白修饰在基因表达调控发挥重要重要[15-16],非交联ChIP技术可更好地检测基因组内组蛋白修饰的变化[17-18]。目前,下一代染色质高通量测序的广泛应用,为表观遗传学的研究提供了更好的平台[19-20]。ChIP-seq数据质量评估对其应用与表观遗传学研究非常重要[21-22]。目前普遍认证的测序程序具有非常高的质量和可靠性,ChIP-seq数据的质量问题一般出现在实验过程,如抗体特异性和灵敏度,染色质片段化,测序文库的制备过程中应用的PCR扩增轮数以及测序深度等方面直接影响数据的质量[23-25]。染色质免疫共沉淀后产物一般产量非常少[26],为得到足够的与组蛋白相互作用的DNA的信息量,建库过程在整个实验中非常关键。本研究将传统的建库方法与Tn5转座酶建库方法进行比较,总结Native ChIP-seq建库方法的更优方案。
建库过程中,初始DNA的量、PCR循环数、纯化过程都会影响最终的DNA文库[27]。由于ChIP文库构建是一个特异性富集与组蛋白相互作用的DNA的过程,得到的ChIP产物为1~2个核小体(146 bp/292 bp),加上引物序列120 bp,片段长度符合二代测序要求。ChIP建库中,DNA起始量一般少于50 ng,运用传统建库方法需要更多ChIP产物DNA,而Tn5转座酶建库可以用于1 ng起始DNA的建库。传统建库PCR扩增循环数一般为12~20个,Tn5转座酶建库PCR循环数为11~15个,提示Tn5转座酶建库比传统建库方法因为PCR扩增产生的偏向更小。此外,Tn5转座酶建库将DNA片段化后直接进行富集,操作更加简便,而传统建库中步骤繁琐,在实验操作过程中难免引起DNA的丢失,导致实验失败可能性更高。
二代测序分析主要包括3部分:质量控制,比对,比对后分析[28]。从测序质量上看,Tn5转座酶建库和传统建库有很高的一致性,而且得到富集峰更多。为进一步验证,我们使用另一种组蛋在修饰抗体H3K4me3,用Tn5转座酶建库方法进行N-ChIP-seq三次生物学重复。信噪比(FRiP)是检验数据质量的重要参数。FRiP值越大,富集区域越多,背景越低[29]。数据评估分析显示,生物学重复性良好,FRiP值达到50%~80%,提示Tn5转座酶建库后能得到更多富集峰和更低的背景,这进一步说明Tn5转座酶建库在ChIP-seq技术中的优势。此外,非交联ChIP与交联的ChIP类似,研究报道ChIPmentation用Tn5转座酶建库[30-31],提示Tn5转座酶建库在ChIP技术的建库中具有优势。与N-ChIP不同,ChIPmentation方法适用于交联的ChIP,其Tn5反应在磁珠上进行,不适用于N-ChIP [32]。本研究Tn5转座酶建库运用于N-ChIP,无需交联,可将DNA直接用Tn5转座酶片段化、富集DNA文库,富集效率更高。
综上所述,本研究中,与传统建库方法相比较,Tn5转座酶建库可以避免繁琐的流程,提高效率,减少实验失败可能。Tn5转座酶进行随机切割,并同时在末端加入接头,更加节约时间,效率更高,摆脱繁琐的建库流程,Tn5转座酶建库可以进行微量DNA的建库,克服了传统建库初始量过低以及后续的DNA丢失、背景高的不足,其技术难度低于传统建库,但测序质量优于传统建库。结果表明Tn5转座酶建库方法适用于ChIP-seq,在简化操作同时可以获得高度可重复结果和好的信噪比。本研究通过将两种二代测序建库方法进行比较,有助于N-ChIP-seq技术的应用及推广,也为不同技术在建库方法的选择上提供参考。
| [1] |
Kanwal R, Gupta S. Epigenetics and cancer[J]. J Appl Phy, 2010, 109(2): 598-605. |
| [2] |
Dupont C, Armant DR, Brenner CA. Epigenetics: definition, mechanisms and clinical perspective[J]. Semin Reprod Med, 2009, 27(5): 351-7. DOI:10.1055/s-0029-1237423 |
| [3] |
Novak K. Epigenetics changes in cancer cells[J]. MedGenMed, 2004, 6(4): 17. |
| [4] |
Kagohara LT, Stein-O'brien GL, Kelley D, et al. Epigenetic regulation of gene expression in cancer: techniques, resources and analysis[J]. Brief Funct Genomics, 2018, 17(1): 49-63. |
| [5] |
Jordan-Pla A, Visa N. Considerations on experimental design and data analysis of chromatin immol/lunoprecipitation experiments[M]. Methods Mol Biol, 2018: 9-28.
|
| [6] |
Hao BT, Naik AK, Watanabe A, et al. An anti-silencer- and SATB1- dependent chromatin hub regulates Rag1 and Rag2 gene expression during thymocyte development[J]. J Exp Med, 2015, 212(5): 809-24. DOI:10.1084/jem.20142207 |
| [7] |
Milne TA, Zhao K, Hess JL. Chromatin immol/Lunoprecipitation (ChIP)for analysis of histone modifications and chromatin-associated proteins[M]. Methods Mol Biol, 2009: 409-23.
|
| [8] |
Schmidt D, Wilson MD, Spyrou CA, et al. ChIP-seq: using highthroughput sequencing to discover protein-DNA interactions[J]. Methods, 2009, 48(3): 240-8. DOI:10.1016/j.ymeth.2009.03.001 |
| [9] |
田李, 张颖, 赵云峰. 新一代测序技术的发展和应用[J]. 生物技术通报, 2015, 31(11): 1-8. |
| [10] |
Lightbody G, Haberland V, Browne F, et al. Review of applications of high-throughput sequencing in personalized medicine: barriers and facilitators of future progress in research and clinical application[J]. Brief Bioinform, 2018. |
| [11] |
Heather JM, Chain B. The sequence of sequencers: The history of sequencing DNA[J]. Genomics, 2015, 107(1): 1-8. |
| [12] |
Gu H, Smith ZD, Bock C, et al. Preparation of reduced representation bisulfite sequencing libraries for genome-scale DNA methylation profiling[J]. Nat Protoc, 2010, 6(4): 468-81. |
| [13] |
Glaser R, Zhang HY, Yao KT, et al. Two epithelial tumor cell lines (HNE-1 and HONE-1)latently infected with Epstein-Barr virus that were derived from nasopharyngeal carcinomas[J]. Proc Natl Acad Sci USA, 1989, 86(23): 9524-8. DOI:10.1073/pnas.86.23.9524 |
| [14] |
Medina-Gali R, Bello-Perez M, Martinez-Lopez A, et al. Chromatin immunoprecipitation and high throughput sequencing of SVCVinfected zebrafish reveals novel epigenetic histone methylation patterns involved in antiviral Immune response[J]. Fish Shellfish Immunol, 2018, 82(4): 514-21. |
| [15] |
Karlic R, Chung HR, Lasserre J, et al. Histone modification levels are predictive for gene expression[J]. Proc Natl Acad Sci USA, 2010, 107(7): 2926-31. DOI:10.1073/pnas.0909344107 |
| [16] |
Seitan VC, Hao BT, Tachibana-Konwalski KA, et al. A role for cohesin in T-cell-receptor rearrangement and thymocyte differentiation[J]. Nature, 2011, 476(7361): 467-U126. DOI:10.1038/nature10312 |
| [17] |
Northrup DL, Zhao K. Application of ChIP-Seq and related techniques to the study of immune function[J]. Immunity, 2011, 34(6): 830-42. DOI:10.1016/j.immuni.2011.06.002 |
| [18] |
Gilfillan GD, Hughes T, Sheng Y, et al. Limitations and possibilities of low cell number ChIP-seq[J]. BMC Genomics, 2012, 13: 645. DOI:10.1186/1471-2164-13-645 |
| [19] |
杜玲, 刘刚, 陆健, 等. 高通量测序技术的发展及其在生命科学中的应用[J]. 中国畜牧兽医, 2014, 41(12): 109-16. |
| [20] |
Chaitankar V, Karakulah G, Ratnapriya RA, et al. Next Generation sequencing technology and genomewide data analysis: perspectives for retinal research[J]. Prog Retin Eye Res, 2016, 55(6): 1-31. |
| [21] |
Nakato R, Shirahige K. Recent advances in ChIP-seq analysis: from quality management to whole-genome annotation[J]. Brief Bioinform, 2017, 18(2): 279-90. |
| [22] |
Bailey T, Krajewski P, Ladunga I, et al. Practical guidelines for the comprehensive analysis of ChIP-seq data[J]. PLoS Comput Biol, 2013, 9(11): 1003326. DOI:10.1371/journal.pcbi.1003326 |
| [23] |
Landt SG, Marinov GK, Kundaje AA, et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia[J]. Genome Res, 2012, 22(9, SI): 1813-31. DOI:10.1101/gr.136184.111 |
| [24] |
Mendoza-Parra M. Gronemeyer H, assessing quality standards for ChIP-seq and related massive parallel sequencing-generated datasets: when rating goes beyond avoiding the crisis[J]. Genom Data, 2014, 10(2): 268-73. |
| [25] |
Mendoza-Parra M, Saravaki V, Cholley PE, et al. Antibody performance in ChIP-sequencing assays: from quality scores of public data sets to quantitative certification[J]. F1000Res, 2016, 12(5): 54. |
| [26] |
Mukhopadhyay A, Deplancke B, Walhout AJ, et al. Chromatin immol/Lunoprecipitation (ChIP) coupled to detection by quantitative real-time PCR to study transcription factor binding to DNA in caenorhabditis elegans[J]. Nat Protoc, 2008, 3(4): 698-709. DOI:10.1038/nprot.2008.38 |
| [27] |
Hashimoto M, He Y, Yeung ES. On-line integration of PCR and cycle sequencing in capillaries: from human genomic DNA directly to called bases[J]. Nucleic Acids Res, 2003, 31(8): e41. DOI:10.1093/nar/gng041 |
| [28] |
Chen S, Huang T, Zhou Y, et al. AfterQC: automatic filtering, trimmol/Ling, error removing and quality control for fastq data[J]. BMC Bioinformatics, 2017, 18(Suppl 3): 80. |
| [29] |
Simonsen AT, Hansen MC, Kjeldsen E, et al. Systematic evaluation of signal-to-noise ratio in variant detection from single cell genome multiple displacement amplification and exome sequencing[J]. BMC Genomics, 2018, 19(1): 681. DOI:10.1186/s12864-018-5063-5 |
| [30] |
Sundaram AY, Hughes T, Biondi S, et al. A comparative study of ChIP-seq sequencing library preparation methods[J]. BMC Genomics, 2016, 17(1): 816. DOI:10.1186/s12864-016-3135-y |
| [31] |
Schmidl C, Rendeiro AF, Sheffield NC, et al. ChIPmentation: fast, robust, low-input ChIP-seq for histones and transcription factors[J]. Nat Methods, 2015, 12(10): 963-5. DOI:10.1038/nmeth.3542 |
| [32] |
Gustafsson C, De Paepe A, Schmidl CA. High-throughput ChIPmentation: freely scalable, single day ChIPseq data generation from very low cell-numbers[J]. BMC Genomics, 2019, 20(1): 59. DOI:10.1186/s12864-018-5299-0 |
 2019, Vol. 39
2019, Vol. 39

