2. 国家卫健委抗体技术重点实验室,南京 211166;
3. 南京医科大学附属上海一院临床医学院消化科,上海 201600;
4. 兴化市人民医院消化内科,江苏 兴化 225700
2. Key Laboratory of Antibody Technology of National Health Commission, Nanjing 211166, China;
3. Department of Gastroenterology, Shanghai General Hospital Affiliated to Nanjing Medical University, Shanghai 201600, China;
4. Department of Gastroenterology, Xinghua People's Hospital, Xinghua 225700, China
胃癌是消化道的高发恶性肿瘤,中国又是胃癌高发国家,严重威胁着人们的健康[1]。尽管手术方法已逐渐改进,新辅助化疗已广泛应用,但中晚期胃癌的死亡率仍居高不下[2]。胃癌患者的高死亡率主要是由于肿瘤的复发和转移[3],而这个过程与基因异常表达、细胞增殖、侵袭迁移有很大关系[4]。因此,研究基因对细胞增殖、侵袭迁移的影响,可为胃癌的治疗提供新的思路。
蛋白酶体a7亚基(PSMA7)是近年来新发现的基因,定位于与多种癌症基因扩增相关的染色体区[5-6],同时又是蛋白酶体的一个亚基,参与组成泛素蛋白酶体系统[7],与肿瘤的发生、发展及转移密切相关[6, 8]。研究发现PSMA7在多种肿瘤组织如肝癌[9-10]、结直肠癌[11]、宫颈癌[7]、胰腺癌[12]等中高表达。在原发性肝癌中,PSMA7高表达还与肝硬化的程度、淋巴结转移和病理分期有关[13]。在结直肠癌中,PSMA7高表达不仅跟肝转移相关,还是判断患者生存预后的一个独立指标[14]。但PSMA7在胃癌中的表达及作用尚未见报道。本研究通过体内和体外实验,旨在探索PSMA7对胃癌生物学行为的影响,以期为胃癌的诊治提供新的思路。
1 资料和方法 1.1 研究对象选取2016年12月~2017年12月在兴化市人民医院外科行胃癌根治术的胃癌患者60例,取其手术切除的癌组织及配对癌旁组织。其中男性50例,女性10例,患者年龄47~83岁,平均年龄68.1岁。肿瘤组织取自胃癌癌巢,癌旁组织取自距肿瘤边缘5 cm外经病理检查确诊的正常组织。患者在手术前均未接受过放化疗及免疫治疗等处理,不伴有其它部位肿瘤。手术标本切下后,一部分组织放冻存管中置于液氮中速冻并保存;另一部分组织进行石蜡包埋,切片用于病理诊断和免疫组织化学分析。本研究经兴化市人民医院伦理委员会批准,所有样本采集均获得患者本人及其家属的同意并均签署了知情同意书。
1.2 实验方法 1.2.1 免疫组化法分析PSMA7在组织中的表达水平取上述各组织标本进行免疫组化染色(SP法),采用已知的免疫组织化学染色阳性标本作为阳性对照,以磷酸盐缓冲液(PBS)代替一抗作为阴性对照。PSMA7阳性反应主要定位于细胞质中,少部分位于细胞核中,染色为淡黄色至棕褐色深浅不等。按染色强度和阳性细胞所占百分比进行评分。(1)染色强度:无染色=0分,淡黄色=1分,棕黄色=2分,棕褐色=3分;(2)阳性细胞:无=0分,1%~25%=1分,26%~50%=2分,5l%~75%=3分,76%~100%=4分。染色强度和阳性细胞所占百分比评分之和≥3分为阳性表达。每张切片均由3位高年资病理医生独立评分,取平均值。
1.2.2 Western blotting检测人胃癌细胞株MKN28、SGC7901、MKN45及正常胃粘膜上皮细胞GES-1(南京医科大学抗体实验室保存)中PSMA7蛋白的表达情况收集上述4株处于对数生长期的细胞,裂解细胞(RIPA裂解液+PMSF)后,提取总蛋白,BCA测定蛋白浓度,调整蛋白浓度相同后煮样,配胶、上样,电泳、转膜、牛奶封闭,加入小鼠抗人PSMA7单克隆抗体(稀释比1:1000,LSBio)和作为内参的小鼠抗人的GAPDH抗体(稀释比1:2000,CST)过夜,次日洗膜后加入HRP标记的兔抗小鼠IgG抗体(稀释比1:4000,Santa Cruz),洗膜后滴加ECL显色液,覆盖PVDF膜,曝光拍照并记录结果,采用Image-Pro Plus 6.0软件检测灰度值。
1.2.3 PSMA7干扰序列设计PSMA7 shRNA及对照质粒均购自PPL公司。shRNA序列:shRNA-1:5'-CC CATTCTTCGCTCTGTGTTT-3';shRNA-2:5'-CTCCT CCATTTCCGTCTACTA-3';shRNA-3:5'-CCTCTTCC AAGTGGAGTA-3';sh- NC:5'-TTCTCCGAACGTGT CACGT-3'。
1.2.4 质粒转染将胃癌细胞接种于6孔板,培养箱中过夜,待细胞完全贴壁转染;取3 μg质粒加入100 μL OPTI-MEM培养基稀释,另取100 μL OPTI-MEM培养基稀释6 μL Lipo2000转染试剂(Invitrogen),将二者混匀,室温静置20 min;往上述200 μL转染复合物中加入800 μL无抗生素培养基,混匀后加入6孔板内,细胞培养箱中继续培养6 h,更换培养基;48 h后收集细胞,行Western blot,检测转染shRNA后PSMA7蛋白的变化,转染效率最好者用于后续实验。
1.2.5 胃癌细胞株SGC7901慢病毒干扰PSMA7慢病毒(PPL),病毒载体为pPLK/GFP+Puro-PSMA7 shRNA-3,将胃癌细胞株接种于96孔细胞培养板中,置于5% CO2、37 ℃培养箱中培养,当细胞密度达约70%~80%时进行转染操作,将病毒原液取出,冰浴融化,用完全培养基按1:50比例稀释,同时加入转染增强剂Polybrene(吉玛),混匀后加入;感染8 h后,更换为新鲜的DMEM培养基,继续培养72 h;细胞消化传代,次日贴壁后更换含0.5 μg/mL嘌呤霉素的细胞培养基,(提前做嘌呤霉素药筛实验,确定最佳浓度)筛选2周,荧光显微镜下观察转染效率,待细胞绿色荧光率达100%,即获得稳定表达的细胞株,经qRT-PCR验证转染效率,后改为含0.25 μg/mL嘌呤霉素的细胞培养基扩增培养。
1.2.6 qRT--PCR检测SGC7901细胞中PSMA7的表达情况将转染shPSMA7-3和Control的SGC7901细胞,分别加入1 mL的Trizol裂解液(Invitrogen)提取细胞总mRNA,紫外分光光度计测其浓度。然后运用反转录试剂盒(TaKaRa)进行反转录,合成cDNA,运用定量SYBR green PCR试剂盒(TaKaRa)将cDNA在实时PCR仪上进行定量,GAPDH为内参,反应条件为预变性95 ℃ 5 min;变性95 ℃ 10 s;退火60 ℃ 30 s;延伸95 ℃ 10 s,共40个循环。采用2-△△CT方法分析PSMA7在2种细胞中的相对表达水平。确定PSMA7 shRNA-3转染成功,进行后续实验。
1.2.7 CCK-8检测干扰PSMA7对SGC7901细胞增殖能力的影响将经过转染的两组待测细胞(1×105/mL)接种于96孔板中,置于含5% CO2、37 ℃培养箱中培养24、48、72 h后,分别向每孔加入10 μL CCK-8溶液(同仁),锡箔纸包好避光,置于37 ℃培养箱中孵育2 h后,用酶标仪测定450 nm处的吸光度,重复3次。
1.2.8 克隆形成实验检测干扰PSMA7对SGC7901细胞增殖能力的影响取对数生长期的两组待测细胞(103/mL),用胰蛋白酶消化后用完全培养基重悬并吹打成单细胞悬液,将重悬的单细胞悬液接种于6孔板中,密度为300/孔,置于含5% CO2、37 ℃细胞培养箱中培养,当6孔板中出现肉眼可见的克隆时终止培养,弃去细胞培养上清液,用PBS小心浸洗2遍后,加入4%多聚甲醛固定15 min,移除多聚甲醛,用PBS小心浸洗2遍后加适量结晶紫染色液(谷歌生物)染色20 min,然后用流水缓慢洗去染色液,空气中干燥后,以显微镜下大于50个细胞或肉眼可见的典型集落为标准,计算克隆形成数目。
1.2.9 Transwell实验检测干扰PSMA7对SGC7901细胞迁移、侵袭能力的影响实验前12 h将两组待测细胞的培养基更换为无血清培养基培养,加入200 μL无血清培养基稀释的各组细胞悬液(2×105/mL)于Transwell小室(未铺或铺Matrigel基质胶)上室表面,小室下层孔板中加入含10%血清的完全培养液800 μL,分别置于含5% CO2、37 ℃细胞培养箱内培养48 h,棉签轻轻擦拭上层小室内的液体,PBS轻柔洗2次,甲醇固定20 min后行结晶紫染色20 min,移去结晶紫,PBS柔洗2次晾干后,倒置显微镜下随机选取膜上5个不同视野,计算穿过小室的细胞个数,取平均值计数并统计分析。
1.2.10 动物实验分析PSMA7对裸鼠皮下成瘤的影响取两组对数生长期细胞,胰蛋白酶消化后,用无血清培养基洗涤重悬制成单细胞悬液(5×107/mL)。取5周龄左右的BALB/c裸鼠,分别于裸鼠左右两侧腋窝处皮下注射细胞悬液各200 μL(左侧为对照组,右侧为实验组),每隔3 d观查记录裸鼠皮下成瘤体积,记录裸鼠皮下移植瘤最大长径a和短径b,并运用如下公式:Volume(mm3)=a×b2/2计算移植瘤体积,处死小鼠后完整剥离皮下移植瘤并测量其大小和称质量。
1.3 统计学方法数据分析采用SPSS 20.0统计软件,计量资料以均数±标准差表示,两组间的比较用独立样本t检验,以P < 0.05为差异有统计学意义。
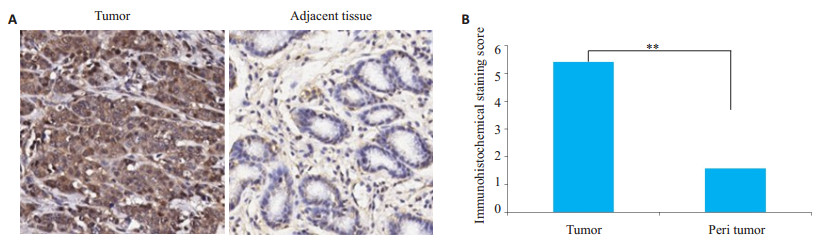
2 结果 2.1 PSMA7在组织中的免疫组化结果在胃癌组织中,PSMA7呈高表达,且主要表达在细胞质;在癌旁组织,PSMA7则呈低表达(图 1A)。胃癌组织中PSMA7表达水平明显高于癌旁组织,差异有统计学意义(P < 0.01,图 1B)。
|
图 1 胃癌组织和配对癌旁组织中PSMA7的表达 Fig.1 Expression of PSMA7 in gastric cancer tissues and paired adjacent tissues detected with immunohistochemical staining (A)and quantitative analysis of the results (B) (Original magnification: ×100). **P < 0.01. |
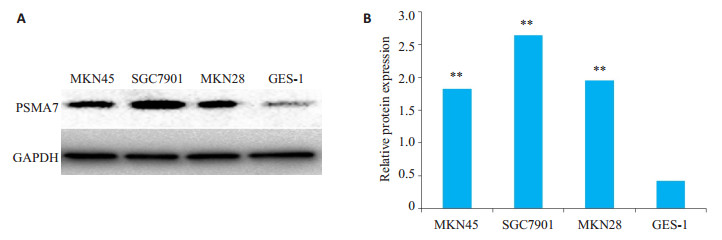
胃癌细胞中PSMA7的表达水平明显高于GES-1(图 2A),以SGC7901表达最高,差异有统计学意义,用于后续实验(P < 0.01,图 1B)。
|
图 2 胃癌细胞中PSMA7蛋白的表达 Fig.2 Expression of PSMA7 protein in different gastric cancer cells. A: Expression of PSMA7 protein detected with Western blotting; B: Quantitative analysis of the results. **P < 0.01 vs GES-1 cells. |
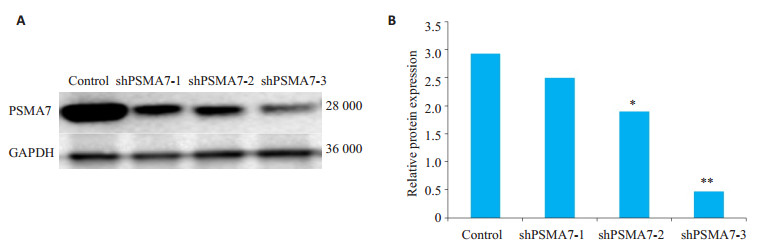
应用Western blot法检测转染shPSMA7-1、shPSMA7-2、shPSMA7-3及对照组细胞中PSMA7的表达。结果显示(图 3A),转染shRNA后,PSMA7的蛋白表达水平降低,shPSMA7-3降低最明显(图 3B),差异有统计学意义(P < 0.01,P < 0.05),选用其用于后续实验。
|
图 3 转染shRNA对胃癌细胞SGC7901中PSMA7蛋白的表达影响 Fig.3 Expression of PSMA7 protein in SGC7901 cells transfected with shRNA detected by Western blotting (A) and quantitative analysis of the results (B). **P < 0.01, *P < 0.05 vs Control. |
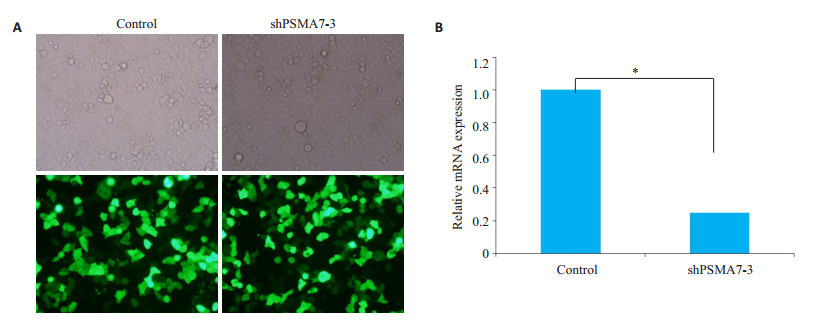
荧光显微镜观察结果显示,SGC7901细胞转染率约100%(图 4A)。qRT-PCR结果显示在转染shPSMA7-3组,细胞的PSMA7 mRNA表达水平显著低于对照组(P < 0.05,图 4B),稳转细胞株成功建立。
|
图 4 PSMA7shRNA载体在胃癌细胞株中转染效率的检测 Fig.4 Detection of transfection efficiency of shRNA vector in SGC7901 cell lines. A: Lentivirus- infected SGC7901 cells under visible light and green fluorescence; B: Expression of PSMA7 mRNA in lentivirus-infected SGC7901 cells detected with qRT-PCR(Original magnification: ×400). *P < 0.05. |
转染shPSMA7-3后,CCK8检测结果显示,细胞的增殖能力明显弱于对照组,差异有统计学意义(P < 0.05,图 5A)。同时细胞克隆形成实验结果显示,细胞克隆形成数目明显少于对照组,差异有统计学意义(P < 0.01,图 5B)。
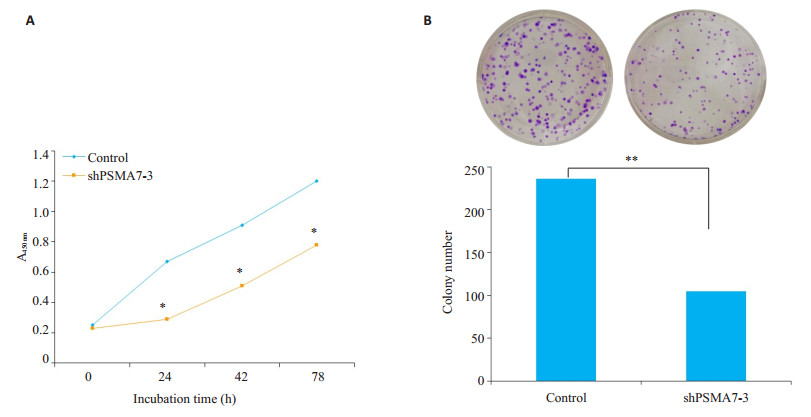
|
图 5 干扰PSMA7对胃癌细胞株SGC7901增殖能力的影响 Fig.5 Effects of PSMA7 interference on proliferation in SGC7901 cell. A: PSMA7 knockdown suppresses proliferation of SGC7901 cells; B: PSMA7 knockdown suppresses clone formation of SGC7901 cells. *P < 0.05 vs Control, **P < 0.01. |
转染shPSMA7-3后,采用Transwell小室实验观察下调PSMA7表达后对胃癌细胞株SGC7901细胞迁移和侵袭能力的影响,发现干扰组迁移(6A)和侵袭能力(6B)明显弱于对照组,差异有统计学意义(P < 0.01,图 4)。
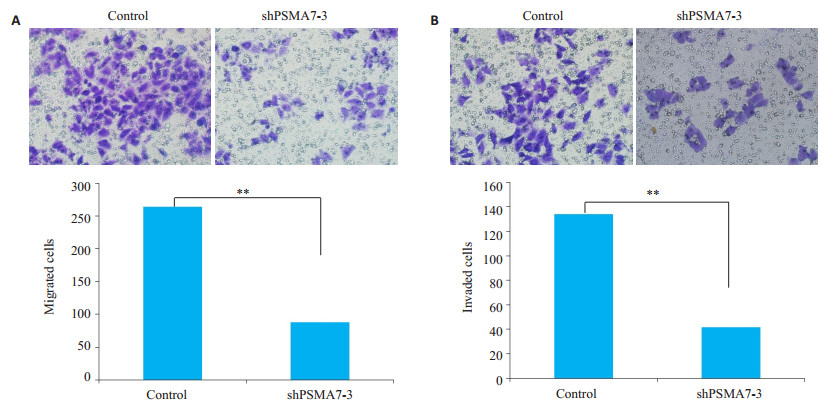
|
图 6 干扰PSMA7对胃癌细胞株SGC7901迁移和侵袭能力的影响 Fig.6 Effects of PSMA7 interference on the migration and invasion of SGC7901 cells. A: PSMA7 knockdown suppresses migration of SGC7901 cells; B: PSMA7 knockdown suppresses invasion of SGC7901 cells (original magnification: ×200). **P < 0.01. |
筛选好稳转shPSMA7-3及对照组的细胞株后,制备成肿瘤单细胞悬液注射至裸鼠皮下,研究干扰PSMA7对裸鼠皮下成瘤能力的影响。结果提示:在稳定干扰PSMA7组,瘤体组织的生长显著慢于对照组,接着对两组的瘤体体积和质量分别测量观察,结果表明干扰PSMA7后,瘤体组织的体积变小(图 7A),质量变轻(图 7B),差异有统计学意义(P < 0.05,图 5)。
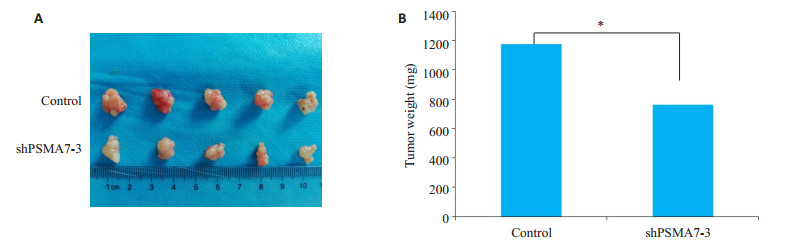
|
图 7 干扰PSMA7对裸鼠皮下瘤的影响 Fig.7 Effects of PSMA7 knockdown on growth of xenograft derived from SGC7901 cells in nude mice. A: PSMA7 knockdown reduced xenograft volume; B: PSMA7 knockdown lowered xenograft weight. *P < 0.05. |
近年来,由于内镜检查的广泛普及及早癌诊疗技术的迅速开展,胃癌作为全球常见的恶性肿瘤之一,早期胃癌患者的预后得到一定改善[15],然而中晚期胃癌患者仍存在较高的死亡率[16]。胃癌患者的高死亡率主要是由于肿瘤的复发和转移[17],而这个过程是一个多基因改变、多步骤参与的病理生理过程[18]。其中,在胃癌的发生发展过程中,胃癌相关基因的异常表达是影响胃癌恶性进程的重要因素[19]。以往很多学者的研究已经发现了许多与胃癌分子诊断或预后相关的异常表达基因[20-22],但真正应用临床并取得疗效的胃癌靶向药物却屈指可数[23],又由于现有的胃癌靶向药物对患者预后和总生存率的提高有限[24]。因此,寻找新的胃癌分子诊断和治疗靶点,探讨胃癌进展的相关机制,对改善胃癌预后和提高患者生存率具有重要意义[25]。
PSMA7基因定位于20q13.33染色体,该区域跟许多肿瘤的扩增有关,基因编码蛋白由248个氨基酸组成[8];另外PSMA7是还20 s蛋白酶体复合物的一个α亚基,参与组成了泛素蛋白酶体系统,在蛋白酶体的活性调控中发挥重要作用[26]。基于PSMA7在染色体定位及其在蛋白酶体中的结构,发现其可与调节细胞周期、转录、细胞凋亡等的调节蛋白[27],以及细胞趋化、血管形成和细胞粘连等过程的相关蛋白相互作用[26],在肿瘤的发生、进展、侵袭、转移等过程中起到重要作用[28-30]。
Scotto等[31]在子宫颈癌中检测到PSMA7的表达水平升高,且其表达水平与多种原癌基因表达水平相关。在原发性肝癌中的研究发现,PSMA7在细胞质和细胞核中均有表达,且在细胞核中癌组织表达高于癌旁组织,但在细胞质中癌组织表达和癌旁组织无差异性[13]。在肺癌中的研究发现,PSMA7在癌和癌旁组织的细胞质和细胞核内均有表达,但在肺腺癌细胞株中PSMA7主要表达在细胞质[28]。以往的研究都支持PSMA7在癌组织或癌细胞中高表达这一理论,但PSMA7表达定位于细胞质还是细胞核存在着差异,这可能与PSMA7在不同组织或者细胞中的胞质和胞核中的作用不同有关。本研究发现PSMA7主要表达在细胞质,且在胃癌组织中呈高表达,而在配对癌旁组织中呈低表达,癌组织和癌旁组织表达的差异性表明PSMA7可能参与胃癌恶性转化的进程。
随后在细胞实验中,本研究运用基因转染技术下调PSMA7表达,结果显示在下调PSMA7表达后,胃癌细胞SGC7901的增殖和侵袭迁移能力明显减弱;Qin等也发现干扰PSMA7表达可使人髓性白血病细胞株K562细胞S期比例降低,从而抑制K562细胞增殖[32]。也有研究发现干扰PSMA7后,可以抑制结肠癌RKO细胞的侵袭和迁移能力,但不影响细胞增殖[9]。本研究结果则发现,干扰PSMA后,对细胞增殖、侵袭和迁移都有影响。最后在动物实验中,本研究通过皮下注射PSMA7的干扰稳定株,观察裸鼠皮下成瘤能力。结果显示下调PSMA7表达后,裸鼠皮下成瘤的生长速度减慢、瘤体体积缩小、质量减轻。体内实验证明,干扰PSMA7可以抑制裸鼠皮下成瘤能力。Hu et al[9]等的研究也发现,干扰PSMA7可能通过影响肌动蛋白骨架的组装,抑制裸鼠体内RKO细胞的成瘤能力。本研究先后通过人体标本、细胞实验证明PSMA7在胃癌中高表达,通过细胞实验和动物实验证明,干扰PSMA7可以抑制胃癌细胞的增殖、侵袭和迁移及体内成瘤能力,而增殖、侵袭迁移能力又是肿瘤发生发展的关键因素,这些结果使我们有理由相信,PSMA7在胃癌的发生发展过程中起到一定作用。
基于以往的研究和本研究的结果,我们预测PSMA7可能作为胃癌的一个新的标志基因促进胃癌细胞增殖、侵袭和转移进而参与其恶性转化。本文尚有一些不足,如胃癌患者病例数尚不够多,也未能阐明PSMA7如何促进胃癌增殖、侵袭转移等的恶性生物学行为,这也是我们下步研究的目标。但我们的研究结果已经初步显示了PSMA7在胃癌进展中发挥重要作用,为胃癌的诊断和预后评估提供了一个新的潜在分子标志物和理论依据。
| [1] |
陈万青, 郑荣寿, 张思维, 等. 2012年中国恶性肿瘤发病和死亡分析[J].
中国肿瘤, 2016, 40(1): 1-8.
DOI: 10.3969/j.issn.1674-4136.2016.01.001. |
| [2] |
杜奕奇, 蔡全才, 方军, 等. 中国早期胃癌筛查流程专家共识意见(草案) (2017年, 上海[J].
胃肠病学, 2018, 23(2): 92-7.
DOI: 10.3969/j.issn.1008-7125.2018.02.007. |
| [3] |
Okugawa Y, Mohri Y, Tanaka K, et al. Metastasis-associated protein is a predictive biomarker for metastasis and recurrence in gastric cancer[J].
Oncol Rep, 2016, 36(4): 1893-900.
DOI: 10.3892/or.2016.5054. |
| [4] |
Sitarz R, Skierucha M, Mielko JA, et al. Gastric cancer: epidemiology, prevention, classification, and treatment[J].
Cancer Manag Res, 2018, 10: 239-48.
DOI: 10.2147/CMAR. |
| [5] |
Jia YX, Song T, Wei CW, et al. Negative regulation of MAVSMediated innate immune response by PSMA7[J].
J Immunol, 2009, 183(7): 4241-8.
DOI: 10.4049/jimmunol.0901646. |
| [6] |
Hu XT, Chen W, Wang D, et al. The proteasome subunit PSMA7 located on the 20q13 amplicon is overexpressed and associated with liver metastasis in colorectal cancer[J].
Oncol Rep, 2008, 19(2): 441-6.
|
| [7] |
Ren CC, Yang L, Liu L, et al. Effects of shRNA-mediated silencing of PSMA7 on cell proliferation and vascular endothelial growth factor expression via the ubiquitin-proteasome pathway in cervical cancer[J].
J Cell Physiol, 2019, 234(5): 5851-62.
|
| [8] |
Du H, Huang X, Wang S, et al. PSMA7, a potential biomarker of diseases[J].
Protein Pept Lett, 2009, 16(5): 486-9.
DOI: 10.2174/092986609788167824. |
| [9] |
Hu XT, Chen W, Zhang FB, et al. Depletion of the proteasome subunit PSMA7 inhibits colorectal cancer cell tumorigenicity and migration[J].
Oncol Rep, 2009, 22(5): 1247-52.
|
| [10] |
Shi YY, Wang HC, Yin YH, et al. Identification and analysis of tumour-associated antigens in hepatocellular carcinoma[J].
Br J Cancer, 2005, 92(5): 929-34.
DOI: 10.1038/sj.bjc.6602460. |
| [11] |
Honma K, Takemasa I, Matoba R, et al. Screening of potential molecular targets for colorectal cancer therapy[J].
Int J Gen Med, 2009, 2: 243-57.
|
| [12] |
Ohnami S, Matsumoto N, Nakano M, et al. Identification of genes showing differential expression in antisense K-ras-transduced pancreatic cancer cells with suppressed tumorigenicity[J].
Cancer Res, 1999, 59(21): 5565-71.
|
| [13] |
黄湘, 邹浩元, 杨宇辉, 等. 人蛋白酶体α7亚基在原发性肝细胞肝癌中的表达及临床意义[J].
细胞与分子免疫学杂志, 2012, 28(6): 640-2.
|
| [14] |
胡晓彤, 陈薇, 王达, 等. 大肠癌中蛋白酶体亚基PSMA7的高表达与肝转移的关系[J].
中华肿瘤杂志, 2008, 30(7): 515-8.
DOI: 10.3321/j.issn:0253-3766.2008.07.009. |
| [15] |
Kim JH, Kim SS, Lee JH, et al. Early detection is important to reduce the economic burden of gastric cancer[J].
J Gastric Cancer, 2018, 18(1): 82-9.
DOI: 10.5230/jgc.2018.18.e7. |
| [16] |
左婷婷, 郑荣寿, 曾红梅, 等. 中国胃癌流行病学现状[J].
中国肿瘤临床, 2017, 44(1): 52-8.
DOI: 10.3969/j.issn.1000-8179.2017.01.881. |
| [17] |
Kim HJ, Oh SC. Novel systemic therapies for advanced gastric cancer[J].
J Gastric Cancer, 2018, 18(1): 1-19.
DOI: 10.5230/jgc.2018.18.e3. |
| [18] |
Ichikawa H, Nagahashi M, Shimada YA, et al. Actionable genebased classification toward precision medicine in gastric cancer[J].
Genome Med, 2017, 9(1): 93-104.
DOI: 10.1186/s13073-017-0484-3. |
| [19] |
Tsukamoto Y, Uchida T, Karnan S, et al. Genome-wide analysis of DNA copy number alterations and gene expression in gastric cancer[J].
J Pathol, 2010, 216(4): 471-82.
|
| [20] |
Deng X, Xiao Q, Liu F, et al. A gene expression-based risk model reveals prognosis of gastric cancer[J].
Peer J, 2018, 6(4): e4204.
|
| [21] |
Zheng HC, Li J, Shen DF, et al. BTG1 expression correlates with pathogenesis, aggressive behaviors and prognosis of gastric cancer: a potential target for gene therapy[J].
Oncotarget, 2015, 6(23): 19685-705.
|
| [22] |
Caiazza C, Mallardo M. The roles of miR-25 and its targeted genes in human cancer[J].
Microrna, 2018, 5(2): 113-9.
|
| [23] |
Yuan DD, Zhu ZX, Zhang X, et al. Targeted therapy for gastric cancer: Current status and future directions (Review) J][J].
Oncol Rep, 2016, 35(3): 1245-54.
DOI: 10.3892/or.2015.4528. |
| [24] |
Feng F, Liu JQ, Wang F, et al. Prognostic value of differentiation status in gastric cancer[J].
BMC Cancer, 2018, 18(1): 865-71.
DOI: 10.1186/s12885-018-4780-0. |
| [25] |
周娜, 刘栋, 张传涛, 等. 新一代测序技术在鉴定晚期胃癌治疗新靶点中的应用[J].
肿瘤研究与临床, 2018, 30(2): 89-92.
DOI: 10.3760/cma.j.issn.1006-9801.2018.02.004. |
| [26] |
Zheng XW, Chen F, Zhang Q, et al. Salivary exosomal PSMA7: a promising biomarker of inflammatory bowel disease[J].
Protein Cell, 2017, 8(9): 686-95.
DOI: 10.1007/s13238-017-0413-7. |
| [27] |
黄湘, 钟裕恒, 谭家余, 等. PSMA7对A549细胞中RB途径的影响[J].
中国免疫学杂志, 2017, 33(4): 516-9.
|
| [28] |
Tan JY, Huang X, Luo YL. PSMA7 inhibits the tumorigenicity of A549 human lung adenocarcinoma cells[J].
Mol Cell Biochem, 2012, 366(1/2): 131-7.
|
| [29] |
Li DP, Dong QC, Tao QP, et al. c-Abl regulates proteasome abundance by controlling the Ubiquitin-Proteasomal degradation of PSMA7 subunit[J].
Cell Rep, 2015, 10(4): 484-96.
DOI: 10.1016/j.celrep.2014.12.044. |
| [30] |
Li N, Zhang Z, Zhang W, et al. Calcineurin B subunit interacts with proteasome subunit alpha type 7 and represses hypoxia-inducible factor-1 alpha activity via the proteasome pathway[J].
Biochem Biophys Res Commun, 2011, 405(3): 468-72.
DOI: 10.1016/j.bbrc.2011.01.055. |
| [31] |
Scotto L, Narayan G, Nandula SV, et al. Integrative genomics analysis of chromosome 5p gain in cervical cancer reveals target over-expressed genes, including Drosha[J].
Mol Cancer, 2008, 7(1): 58-67.
DOI: 10.1186/1476-4598-7-58. |
| [32] |
秦涛, 范翠青, 朱宁, 等. 小RNA干扰蛋白酶体亚基α7抑制K562细胞增殖[J].
中国医学科学院学报, 2013, 35(6): 601-6.
DOI: 10.3881/j.issn.1000-503X.2013.06.003. |
 2019, Vol. 39
2019, Vol. 39

