成簇规律间隔短回文重复序列及相关蛋白9 (CRISPR/Cas9)是一种新型基因编辑技术[1]。可实现目的基因的敲除、插入、定点替换等[2], 为基因治疗开启了新方向。已有文献报道CRISPR/Cas9技术用于地中海贫血[3]及杜氏肌营养不良(DMD/BMD)[4]等遗传疾病的实验研究。
葡萄糖-6-磷酸脱氢酶(G6PD)是磷酸戊糖途径(PPP)的关键酶[5], 可保护细胞免受氧化损伤。G6PD缺乏症是由于X染色体上G6PD基因突变导致红细胞破坏并溶血的一种最常见的血液系统遗传病[6], 临床表现主要为新生儿黄疸、急性溶血性贫血和慢性非球形红细胞溶血性贫血[7]。对于G6PD缺乏症患者, 目前尚无根治疗法, 主要以预防及在疾病发作时给予对症治疗, 严重者治疗效果不佳。因此, 在基因水平上修复其突变基因将有望根治这一疾病, 改善治疗现状, 具有较好的应用前景。
目前, 虽有CRISPR/Cas9技术纠正多种遗传疾病的研究报道[3-4], 但尚未用于G6PD缺乏症的研究。HEK239T是实验室常见的模式细胞, 对于基因修饰、编辑等研究具有重要作用[8]。本实验选取G6PD基因常见突变位点c.392G>T (p.131G>V), 利用CRISPR/Cas9技术构建敲除G6PD基因c.392G>T(p.131G>V)突变位点, 为后续研究G6PD缺乏症的基因修复构建稳定的HEK293T细胞模型。
1 材料和方法 1.1 材料和试剂HEK293T细胞系、PX458质粒(实验室保存); 结晶紫染液、4%甲醛(实验室配置); 胎牛血清(ExCell Bio); DMEM培养基(Gibco); 氨苄西林抗生素(索莱宝); PBS磷酸盐结晶粉末(鼎国生物); 青霉素/链霉素100倍浓缩液、0.25% EDTA胰蛋白酶(Genview); Neofect转染试剂(零客创智生物技术); 酵母提取物、胰蛋白胨(Oxoid); 无内毒素质粒提取试剂盒(美基生物); 琼脂糖粉末(Gene); DH5α感受肽细胞、血液/组织/细胞基因组DNA提取试剂盒、琼脂糖凝胶回收试剂盒(天根生物); BbsⅠ核酸内切酶、EnGen™突变检测试剂盒、T4快速连接试剂盒、Q5 Hotstar高保真多聚核酸酶、T7E1核酸内切酶(NEB); Premix Taq(TaKaRa); 蛋白Marker (Thermo); ECL发光液(Millipore); 兔源G6PD抗体(CST); 兔源GAPDH抗体(武汉三鹰); 辣根酶标记山羊抗兔IgG抗体(北京中杉金桥); CCK8试剂盒(Genecopoeia); 6-磷酸葡萄糖脱氢酶测定试剂盒(中山生物工程); 维生素K3 (MCK); sgRNA合成、引物合成、测序服务均由北京六合华大基因有限公司完成。
1.2 实验方法 1.2.1 sgRNA和目的片段引物设计根据CRISPR/Cas9的工作原理, 利用在线设计工具(http//crispr.mit.edu/)针对G6PD基因5号外显子c.392G>T (p.131G>V)突变位点设计向导RNA(sgRNA), 选择四对评分较高的sgRNA, 在正义链模板的5'端添加CACC, 反义链模板的5'端添加AAAC。同时依据目的基因的序列, 利用NCBI引物设计工具设计CRISPR/Cas9敲除鉴定引物G6PD-5F: 5'-TGTCTGTGCTGCCTGCTTT-3', G6PD-5R: 5'-TCC ACGAAACACCGCCTTT-3'。
1.2.2 sgRNA-PX458质粒的构建用T4多聚核苷酸激酶使合成的单链sgRNA退火形成二聚体, 同时利用BbsⅠ核酸内切酶酶切PX458质粒并切胶回收, 再用T4连接酶将退火后的sgRNA与胶回收的PX458质粒连接过夜, 然后将质粒转化到DH5α感受态细胞中, 涂于氨苄青霉素抗性的LB平板上, 37℃培养12~14 h, 挑取单个菌落摇菌, 菌液提取质粒并测序鉴定。
1.2.3 HEK293T细胞培养及质粒转染将HEK293T细胞用10%FBS的DMEM培养基常规培养, 转染前1 d将HEK293T细胞铺在10 cm平板中, 使转染时细胞密度生长至60%~80%为宜。按照Neofect转染试剂说明书加入10 μL质粒进行转染, 6 h后换液。24 h后在荧光显微镜下观察GFP绿色荧光, 确保质粒成功转染到细胞中。
1.2.4 流式细胞仪分选GFP+的HEK293T细胞并筛选单克隆细胞转染HEK293T细胞48 h后, PBS缓冲液重悬细胞, 用流式细胞分选仪分选GFP阳性细胞, 将GFP阳性细胞采用有限稀释法接种至96孔板中, 待细胞形成单克隆集落后, 再接种于24孔板内培养, 长满后再接种至6孔板培养。
1.2.5 突变效率的验证及敲除G6PD基因c.392G>T (p.131G>V)突变位点HEK293T细胞株的鉴定根据c.392G>T (p.131G>V)突变位点设计的引物5F和5R, 目的片段长度599 bp, 突变位点前后序列长度分别为336 bp和262 bp, 前后相差74 bp。提取分选后细胞的DNA, 用Q5 HotStar高保真多聚核酸酶PCR扩增目的片段并切胶回收, 回收产物进行T7E1酶切, 验证突变效率。筛选的单克隆细胞在6孔板内长满后, 提取基因组DNA, 经PCR扩增后将产物交给北京六合华大基因有限公司测序, 测序结果与目的序列进行比对。
1.2.6 敲除G6PD基因c.392G>T (p.131G>V)突变位点后细胞G6PD mRNA、G6PD蛋白的表达和细胞功能的变化将敲除G6PD基因c.392G>T (p.131G>V)突变位点后的细胞扩增培养, 提取总RNA, 检测RNA浓度并按照逆转录试剂盒说明书逆转录为cDNA, 跑定量PCR检测其G6PD mRNA的表达情况。取部分细胞沉淀提取蛋白质, 进行SDS-PVDF电泳, 室温封闭1 h后, 用稀释好的兔源G6PD抗体孵育过夜, TBST洗膜, 辣根酶标记山羊抗兔IgG抗体室温孵育1 h, 洗膜后, 用ECL发光液检测结果, 重复3次, 利用Image J软件计算目的条带和内参条带光密度值。取部分细胞沉淀, 用G6PD酶活性测定试剂盒(比值法)测定细胞G6PD酶活性的改变。将对数生长期的细胞以1.5×104/mL的密度, 传至96孔板, 每孔200 μL, 每组设计4个复孔, 37℃培养24、48、72、96、120、144 h, 每孔加入10 μLCCK8试剂, 振荡混匀后培养2~3 h, 读取450 nm处吸光度(A)来测定细胞的增殖功能。将两组对数生长期的细胞铺板到六孔板, 每孔细胞数5×105, 37℃培养6 h后每孔加入维生素K3, 使其浓度依次为0、5、10、15、20、40 µmol, 培养12 h后用4%甲醛固定30 min, 结晶紫染色, 根据染色深浅及面积判断细胞存活多少。
1.2.7 统计学处理采用GraphPad Prism 5.0软件进行统计学分析, 两组间采用独立样本的T test比较, 以P < 0.05表示差异具有统计学意义。
2 结果 2.1 sgRNA靶位点的选择和设计为了提高CRISPR/Cas9的敲除效率, 选择4对评分较高的针对G6PD基因5号外显子c. 392G>T (p.131G> V)突变位点的sgRNA (图 1), 在5'端添加相应碱基(表 1), 并设计敲除鉴定引物序列, 送北京六合华大基因有限公司合成。
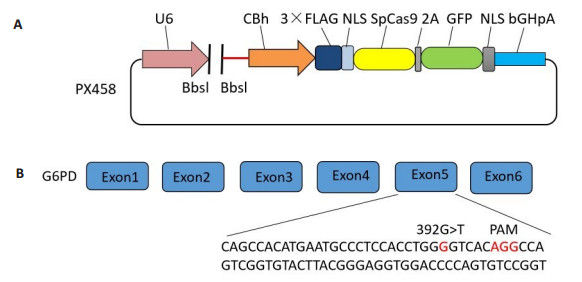
|
图 1 靶向G6PD基因c.392G>T (p.131G>V)突变位点的sgRNA设计 Fig.1 Design of sgRNA targeting G6PD c.392G>T(p.131G>V) mutation site. A: Structure of PX458 plasmid; B: Recognition sequence of sgRNAtargeting Exon 5. |
| 表 1 sgRNA寡核苷酸序列 Tab.1 Sequences of sgRNA oligonucleotides |
用BbsⅠ酶切PX458质粒并胶回收, 将sgRNA退火成双链, 再用T4连接酶将sgRNA与酶切后胶回收的质粒连接。连接产物经转化、摇菌、提质粒后, 送北京六合华大基因有限公司测序, 结果显示4对sgRNA序列均正确插入PX458质粒(图 2), 提示载体构建成功。
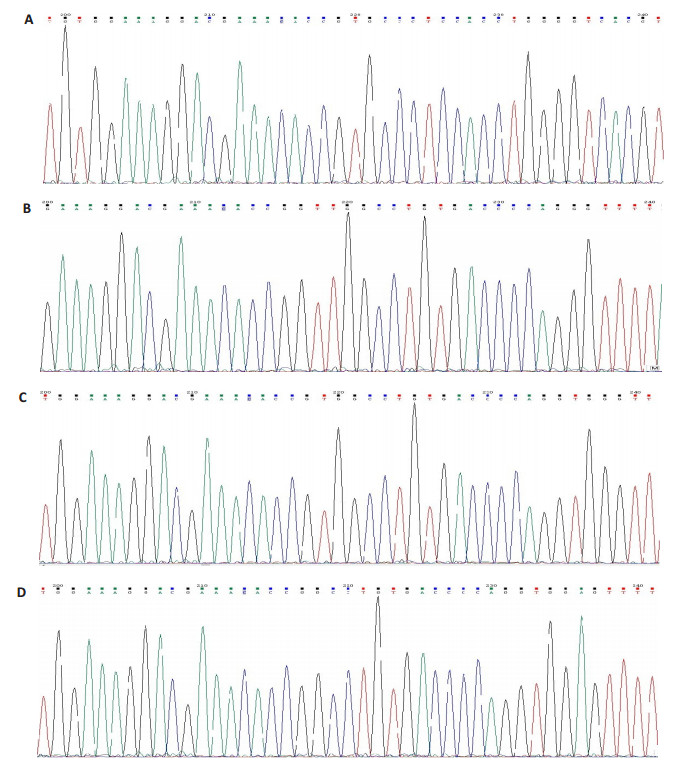
|
图 2 PX458-sgRNA测序图 Fig.2 Sequence maps of sgRNA1-PX458 (A), sgRNA2-PX458 (B), sgRNA3-PX458 (C) and sgRNA4-PX458 (D). |
质粒转染后24 d在荧光显微镜下观察HEK293T细胞及其荧光强度, 4对sgRNA-PX458质粒转染后的细胞均可见绿色荧光, 表明质粒转染成功(图 3), 且荧光强度高, 表明质粒转染效率较高。
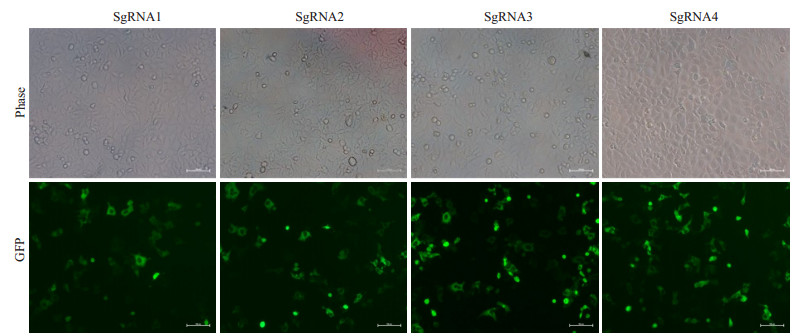
|
图 3 转染sgRNA-PX458质粒24 h后HEK293T细胞GFP表达 Fig.3 GFP expression in HEK293T cells at 24 h after transfection with sgRNA-PX458 plasmid (Original magnification: 10×10). |
PCR扩增分选后细胞的目的片段, 并用2%琼脂糖凝胶电泳(图 4A), T7E1酶切产物用2%琼脂糖凝胶电泳条带(图 4B), 可见sgRNA1、2、3均出现被T7E1内切酶剪切的两条带, 说明转染后细胞DNA被编辑, 使用Image J软件分析条带灰度值之比和公式(% Modification=100×[1-(1-fraction cleaved)1/2])计算突变率(图 4C), 4对sgRNAs对HEK293T细胞DNA编辑效率分别为6.79%、12.41%、12.54%、2.19%。
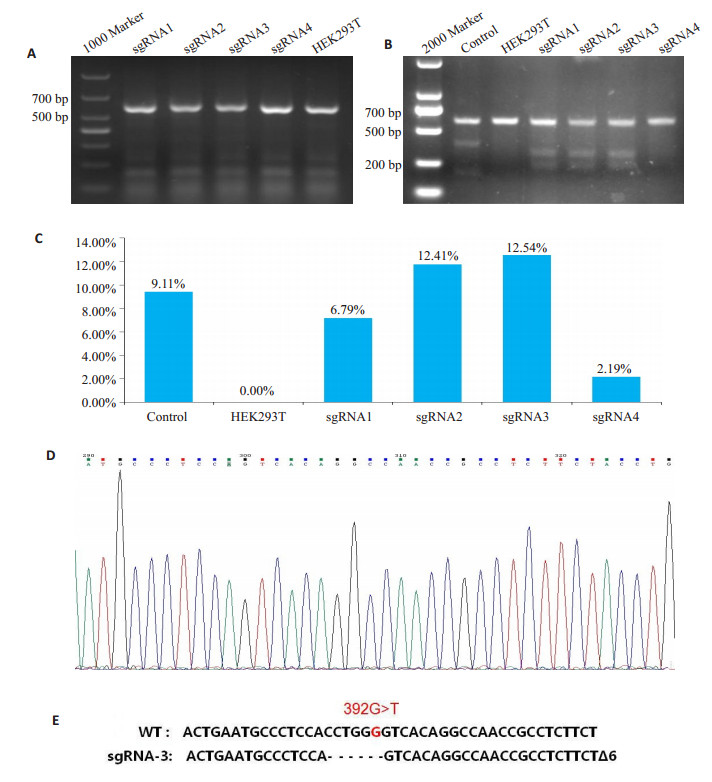
|
图 4 T7E1酶切和DNA测序结果 Fig.4 Results of T7E1 digestion and DNA sequencing for verifying stable G6PD gene knockout in HEK293T cells. A: PCR amplification of the target fragment of HEK293T cells; B: T7E1 cleavage of mismatched fragment, showing positive results for sgRNA 1, 2, and 3; C: Image J software analysis of the gray value of the band to calculate the mutation rate; D: Monoclonal cell sequencing showing successful knockouts; E: Comparison of c.392G>T(p.131G>V) sequence in sgRNA-3 with the original sequence. |
流式分选的GFP阳性细胞, 采用有限稀释法筛选单克隆HEK293T细胞株, 待细胞在6孔板内扩增后, 取部分细胞提取基因组DNA, 经PCR扩增后送北京六合华大基因有限公司测序, 测序结果与原序列进行比对(图 4D), 有一株细胞发生了6个碱基的缺失突变, 缺失碱基包括G6PD基因c.392G>T (p.131G>V)突变位点, G6PD基因c.392G>T(p.131G>V)突变位点敲除的HEK293T细胞模型构建成功。
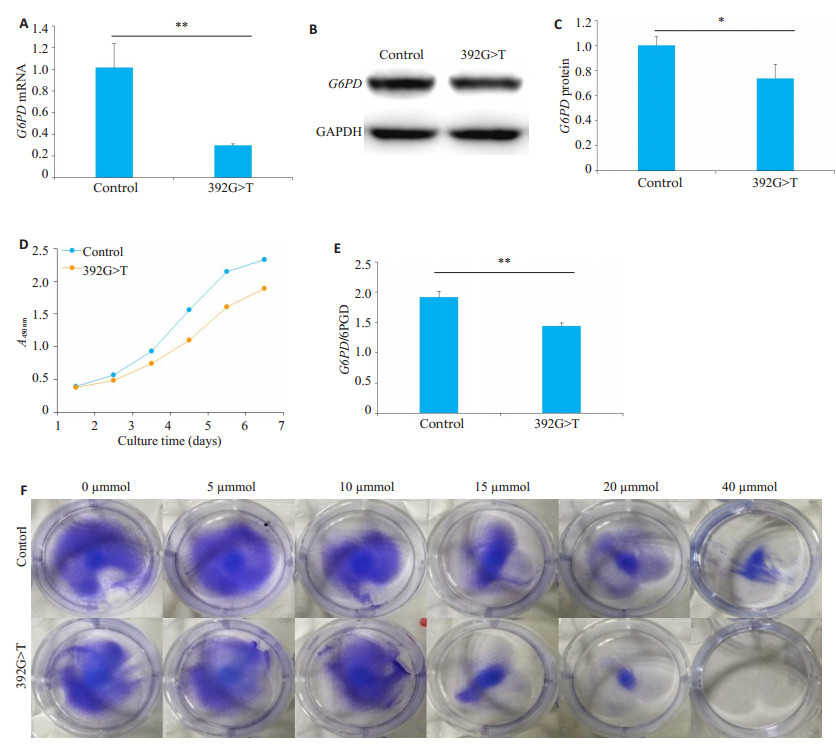
2.5 敲除G6PD基因c.392G>T (p.131G>V)突变位点后HEK293T细胞G6PD基因表达情况与细胞功能的改变将敲除G6PD基因c.392G>T(p.131G>V)突变位点后的HEK293T细胞扩增培养, 提取RNA, 逆转录定量PCR验证其G6PD mRNA的表达水平, 经T检验, 敲除G6PD基因c.392G>T(p.131G>V)突变位点后G6PD mRNA表达下降(P < 0.005, 图 5A)。Western blot结果显敲除G6PD基因c.392G>T(p.131G>V)突变位点后HEK293T细胞G6PD蛋白表达水平降低(图 5B、C, P < 0.05)。利用CCK8法检测稳定敲除G6PD基因c.392G>T(p.131G>V)突变位点后细胞增殖能力, 结果示敲除G6PD基因c.392G>T(p.131G>V)突变位点后HEK293T细胞增殖能力下降(图 5D); 取部分HEK293T细胞沉淀测定G6PD酶活性, G6PD基因c.392G>T (p.131G>V)突变位点敲除后细胞G6PD酶活性降低10%~20% (P < 0.005, 图 5E)。使用G6PD缺乏症患者禁用的药物维生素K3来观察敲除G6PD基因c.392G>T (p.131G>V)突变位点后HEK293T细胞对药物使用的影响, 用药后实验组细胞死亡明显多于对照组, 且随着药物浓度升高差异越大(图 5F)。
|
图 5 G6PD c.392G>T (p.131G>V)突变位点敲除后对HEK293T细胞G6PD表达和功能的影响 Fig.5 Expression of G6PD and cell function in HEK293T cells knockout G6PD c.392G>T (p.131G>V). A: The expression of G6PD mRNA in HEK293T cells with G6PD c.392G>T(p.131G>V) knockout; B: Expression of G6PD protein detected by Western blotting in HEK293T cells with G6PD c.392G>T(p.131G>V) knockout. GAPDH was used as the loading control. C: Statistical analysis of the protein levels; D: Effect of G6PD c.392G>T(p.131G>V) knockout on the proliferation of HEK293T cells; E: Activity of G6PD enzyme in HEK293T cells with G6PD c.392G>T(p.131G>V) knockout; F: Vitamin K3 induced apoptosis in HEK293T cells with G6PD c.392G>T(p.131G>V) knockout. *P < 0.05, **P < 0.01. |
葡萄糖-6-磷酸脱氢酶(G6PD)缺乏症是最常见的人类酶缺陷, 全球超过4亿人受累[9], 我国G6PD缺乏症患病率约为0.2-5.48%, 呈南高北低趋势[10]。目前报道的G6PD基因突变型有217种[11], 多为单个碱基置换导致的错义突变, 在中国已发现30多种突变类型, 最主要的突变位点为c.392G>T (p.131G>V)、c.95A>G (p.32H>R)、c.1376G>T(p.459R>L)和c.1388G>A(p.463R>H)[12]。研究表明, G6PD缺乏症与疟疾分布有显著的地理相似性[13-14], 而临床最常用的抗疟药物——伯氨喹, 服用后常引起G6PD缺乏症患者严重溶血[15]。此外, 磺胺类、镇痛药、砜、亚甲蓝、维生素K3、萘和拉布立酶等[16-18], 均可引起G6PD缺乏症患者发生急性溶血。G6PD缺乏还可导致机体易受病毒感染, 如冠状病毒229E[19]和肠道病毒71型[20]等, 且多种感染性疾病如甲型肝炎病毒A[21]和巨细胞病毒[22]均被认为是G6PD缺乏症患者急性溶血的触发因素。G6PD缺乏症是新生儿发生病理性黄疸的主要原因, 且高胆红素血症可诱发胆红素脑病, 留下严重后遗症甚至死亡[23], 给家庭造成极大的精神压力和经济负担。目前针对G6PD缺乏症患者的治疗以预防和对症治疗为主, 在基因水平上修复这些突变位点将改善这一现状。而且CRISPR/ Cas9具有非常强的基因编辑能力, 已经有该技术应用于多种遗传性疾病的研究报道, 如四氢生物蝶呤(BH4)缺乏症[24]、杜氏肌营养不良[4]、α1-抗胰蛋白酶缺乏症[25]、X-连锁慢性肉芽肿病[26]及单基因血液系统遗传病范尼可贫血(FA)[27]、血友病B[28]、β-地中海贫血[3]和镰状细胞性贫血[29]等。由此可见, 从基因水平上修复G6PD缺乏症患者的突变位点, 进而治疗这一疾病, 具有较好的可行性和临床应用前景。
本实验利用CRISPR/Cas9系统成功构建敲除G6PD基因c.392G>T(p.131G>V)突变位点的HEK293T细胞系, DNA测序结果表明该细胞株中G6PD基因5号外显子产生了6 bp的缺失突变, 说明sgRNA3-PX458质粒转染HEK293T细胞后, sgRNA3靶向G6PD基因5号外显子, 随后Cas9核酸酶在靶位点产生DNA双链断裂[2], 细胞DNA在修复过程中, 产生了6 bp的缺失突变[30], 导致细胞G6PDmRNA的下降, 引起G6PD蛋白表达降低, 从而导致细胞G6PD酶活性的降低。本研究还初步探讨了敲除G6PD基因c.392G>T (p.131G>V)突变位点后, 对HEK293T细胞的增殖能力及细胞抗氧化能力的影响。敲除G6PD基因c.392G>T (p.131G>V)突变位点后, HEK293T细胞增殖能力明显下降。维生素K3在细胞内有电子转移的作用, 具有氧化功能, 是G6PD缺乏症患者禁用的药物, 在细胞培养液中加入维生素K3, 敲除G6PD基因c.392G>T (p.131G>V)突变位点后的HEK293T细胞死亡明显多于对照组, 且随着维生素K3浓度的增加差异越大。Lichun Tang等[31]运用sgRNA和同源供体复合的Cas9蛋白注入单细胞人胚胎中, 证明有效的同源重组可介导G6PD基因中点突变的校正。已报道的G6PD基因突变中约83.9%为单基因突变, 其中约一半为WHO-I型[11], 导致严重的慢性溶血性贫血, 临床治疗效果不佳。根据sgRNA引导cas9蛋白在PAM区上游的3~8碱基处切割目的DNA[32]的工作原理, 利用CRISPR/ Cas9技术可修复包括c.392G>T (p.131G>V)、c.1376G> T(p.459R>L)、c.1388G>A(p.463R>H)和c.95A>G (p.32H>R)等我国最常见几种突变的大部分突变位点。本实验成功构建了敲除G6PD基因c.392G>T (p.131G>V)突变位点的HEK293T稳定细胞株, 为其基因修复奠定了基础。
CRISPR/Cas9系统虽然操作简便, 但转染效率和脱靶效应影响其基因编辑效率。针对不同序列设计的sgRNA特异性高低不一, 使得CRISPR/Cas9系统在不同基因中体现出的编辑效率大相径庭。虽然T7E1酶切后计算出的突变效率较高, 但在筛选单克隆细胞时发现部分敲除位点不包含c.392G>T (p.131G>V)突变位点。因此, 本实验还需进一步优化实验条件以提高CRISPR/Cas9系统的靶向编辑效率, 如更换外源CRISPR/Cas9系统转染方式、采用两对sgRNA同时引导Cas9蛋白对靶位点前后序列同时切割等[33], 为后期在患者来源的细胞中进行基因修复筛选出最佳条件。
| [1] |
Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J].
Science, 2013, 339(6121): 819-23.
DOI: 10.1126/science.1231143. |
| [2] |
Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering[J].
Cell, 2014, 157(6): 1262-78.
DOI: 10.1016/j.cell.2014.05.010. |
| [3] |
Xie F, Ye L, Chang JC, et al. Seamless gene correction of β-thalassemia mutations in patient-specific iPSCs using CRISPR/Cas9 and piggyback[J].
Genome Res, 2014, 24(9): 1526-33.
DOI: 10.1101/gr.173427.114. |
| [4] |
Long C, Mcanally JR, Shelton JM, et al. Prevention of muscular dystrophy in mice by CRISPR/Cas9-mediated editing of germline DNA[J].
Science, 2014, 345(621): 1184-8.
|
| [5] |
Beutler E, Duparc S, G6PD Deficiency Working Grp. Glucose-6-phosphate dehydrogenase deficiency and antimalarial drug development[J].
Am J Trop Med Hyg, 2007, 77(4): 779-89.
DOI: 10.4269/ajtmh.2007.77.779. |
| [6] |
Cappellini MD, Fiorelli G. Glucose-6-phosphate dehydrogenase deficiency[J].
Lancet, 2008, 371(9606): 64-74.
DOI: 10.1016/S0140-6736(08)60073-2. |
| [7] |
Luzzatto L, Nannelli C, Notaro R. Glucose-6-Phosphate dehydrogenase deficiency[J].
Adv Hum Genet, 2016, 30(2): 373-93.
|
| [8] |
Mao YY, Yan RH, Li A, et al. Lentiviral vectors mediate long-term and high efficiency transgene expression in HEK 293T cells[J].
Int J Med Sci, 2015, 12(5): 407-15.
DOI: 10.7150/ijms.11270. |
| [9] |
Nkhoma ET, Poole C, Vannappagari V, et al. The global prevalence of glucose-6-phosphate dehydrogenase deficiency:A systematic review and meta-analysis[J].
Blood Cells Mol Dis, 2009, 42(3): 267-78.
DOI: 10.1016/j.bcmd.2008.12.005. |
| [10] |
Laouini N, Bibi A, Ammar H, et al. Glucose-6-phosphate dehydrogenase deficiency in Tunisia:molecular data and phenotypegenotype association[J].
Mol Biol Rep, 2013, 40(2): 851-6.
DOI: 10.1007/s11033-012-2124-8. |
| [11] |
Gómez-Manzo S, Marcial-Quino J, Vanoye-Carlo A, et al. Glucose-6-Phosphate dehydrogenase:update and analysis of new mutations around the world[J].
Int J Mol Sci, 2016, 17(12): E2069.
DOI: 10.3390/ijms17122069. |
| [12] |
张格, 于洁, 李蕙, 等. 31例G6PD缺乏症患儿基因突变与临床表现分析[J].
中国小儿血液与肿瘤杂志, 2015, 20(6): 299-304.
DOI: 10.3969/j.issn.1673-5323.2015.06.006. |
| [13] |
Monteiro WM, Franca GP, Melo GC, et al. Clinical complications of G6PD deficiency in Latin American and Caribbean populations:systematic review and implications for malaria elimination programmes[J].
Malar J, 2014, 13(1): 1-13.
|
| [14] |
Sirugo G. Reassessing an old claim:Natural selection of hemizygotes and heterozygotes for G6PD deficiency in Africa by resistance to severe malaria[J].
Am J Hematol, 2013, 88(5): 436.
DOI: 10.1002/ajh.v88.5. |
| [15] |
Monteiro WM, Val FF, Siqueira AM, et al. G6PD deficiency in Latin America:systematic review on prevalence and variants[J].
Mem Inst Oswaldo Cruz, 2014, 109(5): 553-68.
DOI: 10.1590/0074-0276140123. |
| [16] |
Pamba A, Richardson ND, Carter N, et al. Clinical spectrum and severity of hemolytic anemia in glucose 6-phosphate dehydrogenase-deficient children receiving dapsone[J].
Blood, 2012, 120(20): 4123-33.
DOI: 10.1182/blood-2012-03-416032. |
| [17] |
Luzzatto L, Seneca E. G6PD deficiency:a classic example of pharmacogenetics with on-going clinical implications[J].
Br J Haematol, 2014, 164(4): 469-80.
DOI: 10.1111/bjh.12665. |
| [18] |
Sherwood GB, Paschal RD, Adamski J. Rasburicase-induced methemoglobinemia:case report, literature review, and proposed treatment algorithm[J].
Clin Case Rep, 2016, 4(4): 315-9.
DOI: 10.1002/ccr3.495. |
| [19] |
Wu YH, Tseng CP, Cheng ML, et al. Glucose-6-phosphate dehydrogenase deficiency enhances human coronavirus 229E infection[J].
J Infect Dis, 2008, 197(6): 812-6.
DOI: 10.1086/587999. |
| [20] |
欧俊斌, 张翠梅, 付四毛, 等. G6PD缺陷与肠道病毒71型手足口病的关系[J].
中国当代儿科杂志, 2013, 15(9): 751-5.
|
| [21] |
Bhutra A, Giri PP, Ganguly N. Acute hepatic failure with hemolytic anemia due to Hepatitis A infection with coexistent glucose-6-phosphate dehydrogenase deficiency[J].
Pediatr Infect Dis, 2015, 7(2): 50-2.
DOI: 10.1016/j.pid.2015.04.002. |
| [22] |
Farah R, Sbeit W, Nassar F, et al. Cytomegalovirus colitis and haemolytic anaemia in a glucose-6-phosphate dehydrogenase-deficient immunocompetent patient[J].
Eur J Gastroenterol Hepatol, 2003, 15(9): 1029-31.
DOI: 10.1097/00042737-200309000-00014. |
| [23] |
Kaplan M, Hammerman C, Bhutani VK. The preterm infant:a HighRisk situation for neonatal hyperbilirubinemia due to glucose-6-Phosphate dehydrogenase deficiency[J].
Clin Perinatol, 2016, 43(2): 325-40.
DOI: 10.1016/j.clp.2016.01.008. |
| [24] |
Ishikawa T, Imamura K, Kondo T, et al. Genetic and pharmacological correction of aberrant dopamine synthesis using patient iPSCs with BH4 metabolism disorders[J].
Hum Mol Genet, 2016, 25(23): 5188-97.
|
| [25] |
Smith C, Abalde-Atristain L, He C, et al. Efficient and allele-specific genome editing of disease loci in human iPSCs[J].
Mol Ther, 2015, 23(3): 570-7.
DOI: 10.1038/mt.2014.226. |
| [26] |
Sürün D, Schwäble J, Tomasovic A, et al. High efficiency gene correction in hematopoietic cells by donor-template-free CRISPR/Cas9 genome editing[J].
Mol Ther NucleicAcids, 2018, 10: 1-8.
|
| [27] |
Osborn MJ, Lonetree CL, Webber BR, et al. CRISPR/Cas9 targeted gene editing and cellular engineering in fanconi anemia[J].
Stem Cells Dev, 2016, 25(20): 1591-603.
DOI: 10.1089/scd.2016.0149. |
| [28] |
Huai C, Jia CQ, Sun RL, et al. CRISPR/Cas9-mediated somatic and germline gene correction to restore hemostasis in hemophilia B mice[J].
Hum Genet, 2017, 136(7): 875-83.
DOI: 10.1007/s00439-017-1801-z. |
| [29] |
Huang XS, Wang Y, Yan W, et al. Production of gene-corrected adult beta globin protein in human erythrocytes differentiated from patient iPSCs after genome editing of the sickle point mutation[J].
Stem Cells, 2015, 33(5): 1470-9.
DOI: 10.1002/stem.1969. |
| [30] |
Mali P, Esvelt KM, Church GM. Cas9 as a versatile tool for engineering biology[J].
Nat Methods, 2013, 10(10): 957-63.
DOI: 10.1038/nmeth.2649. |
| [31] |
Tang L, Zeng Y, Du H, et al. CRISPR/Cas9-mediated gene editing in human zygotes using Cas9 protein[J].
Mol Genet Genomics, 2017, 292(3): 525-33.
DOI: 10.1007/s00438-017-1299-z. |
| [32] |
Mali P, Yang LH, Esvelt KM, et al. RNA-Guided human genome engineering via Cas9[J].
Science, 2013, 339(6121): 823-6.
DOI: 10.1126/science.1232033. |
| [33] |
Liang XQ, Potter J, Kumar S, et al. Enhanced CRISPR/Cas9-mediated precise genome editing by improved design and delivery of gRNA, Cas9 nuclease, and donor DNA[J].
J Biotechnol, 2017, 241: 136-46.
DOI: 10.1016/j.jbiotec.2016.11.011. |
 2019, Vol. 39
2019, Vol. 39

