乳腺癌是全球女性中最常见的恶性肿瘤之一。中国女性乳腺癌发病率在过去20年中呈现持续上升趋势, 并以全球乳腺癌发病速度的2倍增长[1]。预计到2030年, 全球乳腺癌的发病人数和死亡人数将分别达到264万和170万[2]。尽管化疗在乳腺癌的综合治疗中发挥着关键作用, 但仍有部分乳腺癌患者因耐药而导致化疗失败。研究表明, 多数肿瘤的发生和耐药与DNA修复基因的修复能力密切相关, 而核苷酸还原酶M1亚基(RRM1)是DNA损伤修复系统中的关键物质之一[3]。研究发现, RRM1的遗传多样性可影响接受紫杉醇化疗的乳腺癌患者的临床疗效[4], 其在蛋白表达水平也和肿瘤的发生发展相关并影响肿瘤对化疗的敏感性[5-7]。研究表明乳腺癌组织中RRM1的mRNA水平显著高于正常乳腺组织, 其表达水平越高预后越差, 表明其与乳腺癌的发生、发展紧密相联[8]。目前对该基因的研究主要集中在非小细胞肺癌、结肠癌等肿瘤类型中, 而关于乳腺癌方面的研究暂无报道。本研究拟通过沉默RRM1基因的表达, 探究RRM1对乳腺癌药物耐药的影响, 为改善乳腺癌化疗效果提供一定的参考。
1 材料和方法 1.1 材料和试剂人乳腺癌MCF-7细胞购自南京科佰生物科技有限公司; MCF-7/R(本实验室诱导)、MCF-7/R/siRNA (本实验室构建); RRM1特异性siRNA干扰序列(siRRM1)和siRNA阴性对照序列(si-NC)由广州市锐博生物科技有限公司设计和合成, 序列信息见表(表 1); EdU增殖检测试剂盒购自广州市锐博生物科技有限公司; RPMI 1640和胎牛血清购于Gibco; 四甲基偶氮唑蓝(MTT)和紫杉醇购自Sigma; 脂质体Lipofectamine 2000购自Invitrogen; SYBR Green、cDNA逆转录试剂盒购自TAKARA; BCA定量试剂盒购自Thermo; RRM1、p53、p21、cyclin B、Bcl-2、p-AKT、AKT和GAPDH抗体购自CST; 细胞周期和细胞凋亡检测试剂盒购自上海碧云天生物技术有限公司。4~6周BALB/c雌性免疫缺陷型裸鼠购自南方医科大学动物中心。
| 表 1 siRNA干扰序列 Tab.1 Sequence of the small interfering RNAs (siRNAs) oligonucleotide |
人乳腺癌细胞MCF-7于添加有10%胎牛血清的DMEM培养基中, 37℃、5% CO2细胞培养箱中长期传代培养。通过高浓度短时间紫杉醇处理获得紫杉醇耐药株(MCF-7/R), 具体方法如下:将MCF-7细胞暴露于含4 µmol/L紫杉醇、10%胎牛血清的DMEM培养基中, 37℃、5% CO2细胞培养箱中培养20 d, 筛选出的细胞在无紫杉醇培养液中扩增7 d, 用于后续实验[9]。筛选能稳定抑制RRM1基因表达的细胞株MCF-7/R/siRNA, 具体方法如下:将靶向RRM1的特异性siRNA序列定向克隆入逆转录病毒载体RNAi-Ready pSIREN-RetroQ, 构建的载体通过测序、酶切鉴定后, 脂质体介导下转染到包装病毒细胞株中, 嘌呤霉素筛选并挑选细胞克隆, 收获病毒上清, 将病毒上清转染到MCF-7/R细胞中, 经过嘌呤霉素筛选得到稳定抑制RRM1表达的MCF-7/R/siRNA细胞株, 并分别通过Western blot和qRT-PCR方法鉴定抑制RRM1表达的效果[10], 构建成功后用于后续实验。按照Lipofectamine 2000 Reagent说明书进行siRNA的转染, 转染组转染si-RRM1, 对照组转染等剂量的si-NC。空白组只加转染试剂。转染12 h后即可换液进行给药处理, 继续培养48 h后检测各个指标。
1.2.2 MTT法检测细胞耐药性细胞处于对数生长期时, 以2500/孔的细胞量接种96孔板, 并于37℃、5% CO2细胞培养箱中过夜培养使其贴壁。加入不同浓度的紫杉醇, 同时设只加细胞的对照孔和不加细胞的调零孔于细胞培养箱中药物处理72 h。每孔加20 μL MTT溶液, 细胞培养箱中继续孵育4 h, 去除孔内细胞液, 并加入100 μL DMSO, 振荡10 min使结晶溶解后, 用多功能微孔板检测仪检测各孔吸光度值A490 nmol/L。细胞生长抑制率=[1(-加药孔A490 nmol/L-调零孔的A490 nmol/L/对照孔A490 nmol/L-调零孔的A490 nmol/L)]×100%。采用GraphPad Prism 5.0软件计算紫杉醇对细胞的半数抑制浓度(IC50), 用以确定耐药倍数(RI), RI=耐药细胞的IC50/敏感细胞的IC50。上述实验重复3次取平均值作为最终实验结果。
1.2.3 Western blot检测蛋白表达细胞处于对数生长期时, 以1.5×105/孔的细胞量接种6孔板, 并过夜培养使其贴壁。给予药物处理48 h后, 裂解细胞, 4℃, 12 000 g, 离心30 min以后, 吸取上清转移到离心管中。采用BCA(bicinchoninic acid)法对蛋白浓度定量, 步骤同BCA蛋白定量检测试剂盒。蛋白样品和上样缓冲液充分混合后, 沸水浴5 min。通过15%SDS-PAGE电泳90 min后转移至PVDF膜, 封闭液(5% milk/TBST)封闭1 h, 加一抗(1: 1000稀释), 4℃摇床孵育过夜, TBST洗膜4次, 10 min/次, 加二抗(1: 4000稀释)室温摇床缓慢孵育1 h, TBST洗膜4次, 加入ECL发光液显色, 通过多功能成像分析仪曝光拍照, 使用Image J软件进行蛋白条带灰度分析, GAPDH作为内参。
1.2.4 qRT-PCR检测RRM1 mRNA的表达将不同转染组的细胞接种于6孔培养板内, 培养48 h后, 收集细胞样品, Trizol法提取总RNA, 参照试剂盒说明逆转录为cDNA。引物序列来自美国国立生物技术信息中心NCBI数据库。反应条件为: 95℃预变性30 s, 95℃变性5 s, 60℃退火、延伸34 s, 反应40个循环。采用相对定量qPCR法, 以GAPDH做内参基因, 检测组织内RRM1目的基因mRNA的相对表达量, 通过下式计算:相对表达量=2-△△Ct, 其中△△Ct=(Ct处理组目的基因-Ct处理组内参基因) (-Ct对照组目的基因-Ct对照组内参基因), 其中以对照组目的基因的相对表达量为1, 并与处理组进行比较。扩增基因的特异性引物序列为: GAPDH: 5'- GCACCGTCAAGGCTGAGAAC-3'(forward), 5'-TG G TGAAGACGCCAGTGGA-3(' reverse); RRM1: 5'-G AGCAGGGCCCATACGAAAC-3'(forward), 5'-CCCA GGATCTGAGCAGTGGAA-3'(reverse)。
1.2.5 EdU细胞增殖实验取对数生长期的细胞, 以2× 103/孔的密度接种于96孔板, 过夜贴壁后, 给予siRNA转染24 h后换液并给予药物处理48 h后, EdU标记细胞并进行细胞固定以及Apollo染色。最后, 每孔加入100 μL的1×Hoechst 33342反应液进行DNA染色, 避光、室温、脱色摇床孵育30 min后, 弃去染色反应液; 每孔每次加入100 μL的PBS清洗3次; 染色完成后立即置于荧光倒置显微镜下观察并拍照。
1.2.6 流式细胞术检测细胞凋亡和周期收集各组细胞悬液于离心管, 合并用胰酶消化的细胞, 4℃, 1000 r/min, 离心5 min。弃上清, 加入1 mL预冷的PBS重悬细胞, 转移至1.5 mL的无菌EP管, 4℃, 1000 r/min, 离心5 min, 重复2遍, 缓慢吸取上清并丢弃(可以保留大约50 μL的培养液, 以免吸走细胞); 加入500 μL AnnexinV结合液重悬细胞后, 加入5 μL AnnexinV-FITC冰上避光孵育5 min, 10 μL PI轻轻混匀冰上避光孵育10 min后使用流式细胞仪检测细胞凋亡; 收集各组细胞悬液于离心管, 合并用胰酶消化的细胞, 4℃, 1000 r/min, 离心5 min。弃上清, 加入1 mL预冷的PBS重悬细胞, 转移至1.5 mL的无菌EP管, 4℃, 1000 r/min, 离心5 min, 重复2遍, 缓慢吸取上清并丢弃; 加入1 mL预冷的70%乙醇, -20℃条件下固定过夜; 固定完成后, 4℃, 1000 r/min, 离心5 min沉淀细胞, 加入1 mL预冷的PBS重悬细胞, 4℃, 1000 r/min, 离心5 min, 重复2遍, 加入PI染液500 μL充分混匀后于37℃水浴避光孵育30 min后使用流式细胞仪检测细胞周期。
1.2.7 裸鼠乳腺癌移植瘤模型的建立及体内抗肿瘤活性实验采用常规培养细胞移植法, 实验分为4组: MCF- 7/R组、MCF-7/R/siRNA组、MCF-7/R + PTX治疗组、MCF-7/R/siRNA+PTX治疗组, 每组5只鼠。取对数生长期的MCF-7/R、MCF-7/R/siRNA细胞, 0.05%胰蛋白酶消化后制成细胞悬液, 台盼蓝染色证实细胞活力95%以上, 充分吹打后取200 μL (9×106细胞)背部皮下注射, 并继续在SPF级环境下饲养。观察肿瘤生长情况, 肿瘤体积达到150 mm3后开始腹腔注射紫杉醇(15 mg/kg) 200 μL, 每3 d注射1次, 共治疗3周。治疗期间, 电子天平记录每只裸鼠体质量, 绘制裸鼠生长曲线; 游标卡尺测量皮下肿瘤长径(a)和短径(b), 并计算体积:体积(mm3)=a×b2/2。3周后颈椎脱臼法处死裸鼠, 切取肿瘤并拍照记录。
1.3 生存分析使用Kaplan-Meier Plotter(http://kmplot.com/analysis/)分析RRM1基因的表达对乳腺癌患者生存率的影响, 采用Kaplan-Meier法分析并绘制生存曲线, 通过log-rank检验法比较生存曲线的差异, 以P < 0.05表示差异有统计学意义。
1.4 统计学方法实验数据用SPSS17.0软件进行数据分析, 计量资料采用均数±标准差表示, 采用GraphPad Prism 5.0作图, 两样本间比较采用t检验, 组间比较使用单因素方差分析, P < 0.05为差异具有统计学意义。
2 结果 2.1 生存分析分析差异基因RRM1的表达水平与乳腺癌患者无复发生存率之间的关系。RRM1基因的表达改变与乳腺肿瘤患者的无复发生存率显著负相关, 即RRM1高表达患者的生存率均显著低于低表达患者(P < 0.05, 图 1)。
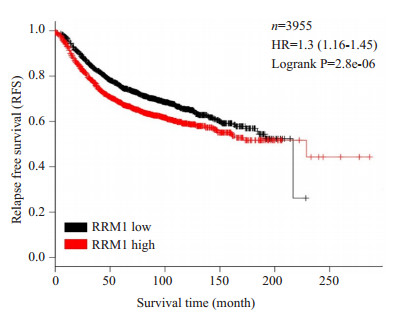
|
图 1 RRM1的表达与乳腺癌患者生存预后的关系(Kaplan Meier plotter生存分析曲线) Fig.1 Relationship between RRM1 expression and relapsefree survival time of breast cancer patients based on the data from publicly available databases. |
MCF-7/R细胞RRM1蛋白表达量显著高于MCF-7细胞(P < 0.01, 图 2)。MCF-7/R细胞中, 转染si-RRM1-04组细胞的RRM1蛋白表达量和mRNA的表达量显著降低(P < 0.001), 且沉默效果优于其它si-RRM1序列, 所以下述实验选择si-RRM1-04作为干扰序列, 而转染si-NC组与对照组相比没有统计学差异(图 3)。
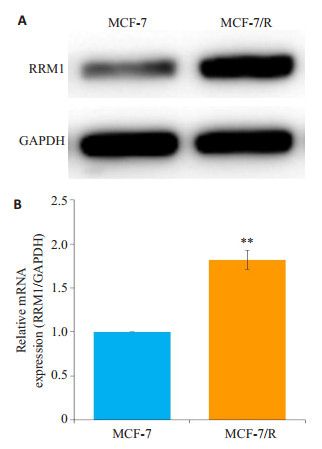
|
图 2 乳腺癌细胞MCF-7和MCF-7/R中RRM1蛋白及mRNA的表达 Fig.2 Expression of RRM1 protein and mRNA in human breast cancer MCF-7 and MCF-7/R cells. A: Western blotting for detecting RRM1 protein in different cells; B: Quantitative mRNA expression of RRM1 relative to GAPDH. **P < 0.01 vs MCF-7 group. |
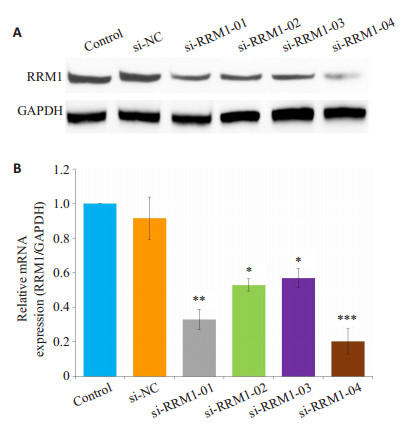
|
图 3 乳腺癌耐药细胞MCF-7/R转染不同siRNA序列后RRM1蛋白及mRNA的表达 Fig.3 Protein and mRNA expressions of RRM1 in MCF-7/R cells after transfection. A: Western blotting for RRM1 protein in each group; B: Quantitative mRNA expression of RRM1 relative to GAPDH. ***P < 0.001; **P < 0.01; *P < 0.05 vs Control group. |
使用MTT方法检测的结果如图 4显示, 与MCF-7细胞相比, MCF-7/R细胞对紫杉醇具有明显的耐药性, 其IC50和RI分别为395.62±87.31 nmol/Lol/L (P < 0.05)和12.33, 与MCF-7/R (si-NC)组相比无显著差异。转染MCF-7/R (si-RRM1-04)组细胞对紫杉醇的IC50和IR分别为142.95±32.44 nmol/Lol/L和4.46, 明显低于MCF- 7/R (si-NC)组(P < 0.05, 表 2)。
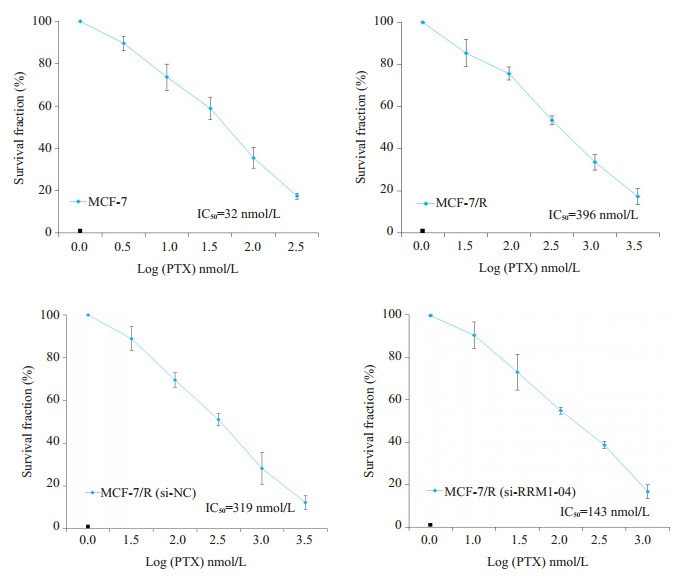
|
图 4 紫杉醇对乳腺癌细胞或乳腺癌耐药细胞细胞增殖的影响 Fig.4 Effect of paclitaxel on proliferation of MCF-7 or MCF-7/R cells after siRNA transfection. The cells were incubated with paclitaxel for 72 h. |
| 表 2 沉默RRM1基因逆转MCF-7/R细胞耐药性 Tab.2 RRM1 gene silencing reverses drug resistance of MCF-7/R cells in different groups (n=3, Mean±SD) |
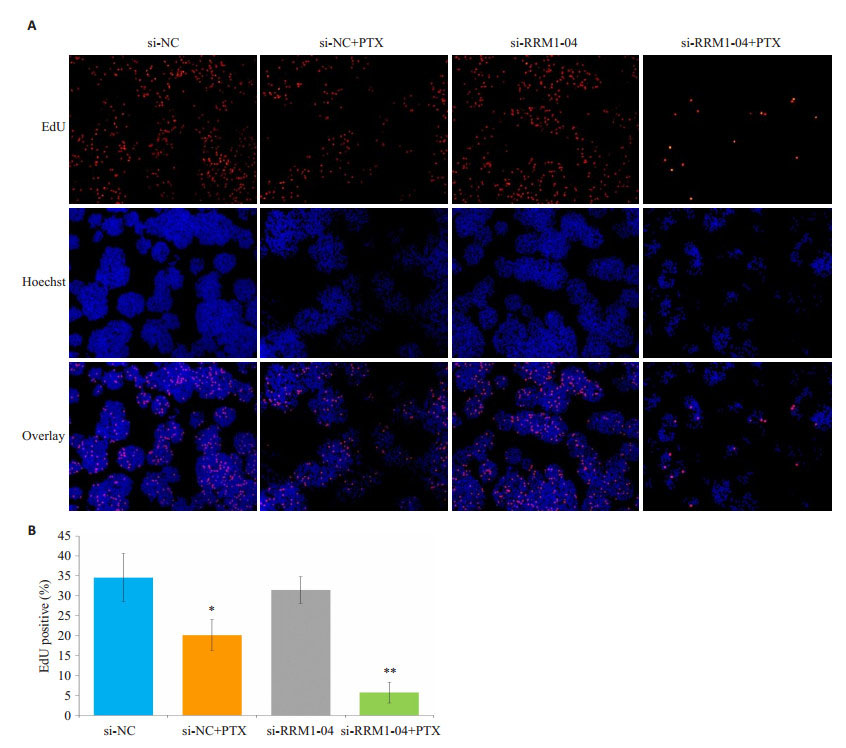
通过EdU实验检测沉默RRM1基因后对MCF-7/R细胞增殖能力的影响。与对照组相比, 转染si-RRM1- 04后给予紫杉醇处理组的细胞, 其增殖能力显著下降(P < 0.01), 而转染si-NC后给予紫杉醇处理组的细胞增殖能力的变化无统计学差异(图 5A)。使用Image J对不同染色的细胞进行计数, 其中蓝色代表所有细胞核, 红色代表处于增殖状态的细胞核, EdU positive(%)= (红色细胞数/蓝色细胞数) ×100%, 使用该指标对不同组细胞增殖能力进行比较(图 5B)。
|
图 5 乳腺癌耐药细胞MCF-7/R转染不同siRNA序列后对细胞增殖的影响 Fig.5 Effect of siRNA transfection on the proliferation of MCF-7/R cells. A: EDU staining detection for cell proliferation; B: Statistical analysis of cell proliferation detected by EDU staining. **P < 0.01; *P < 0.05 vs si-NC group. |
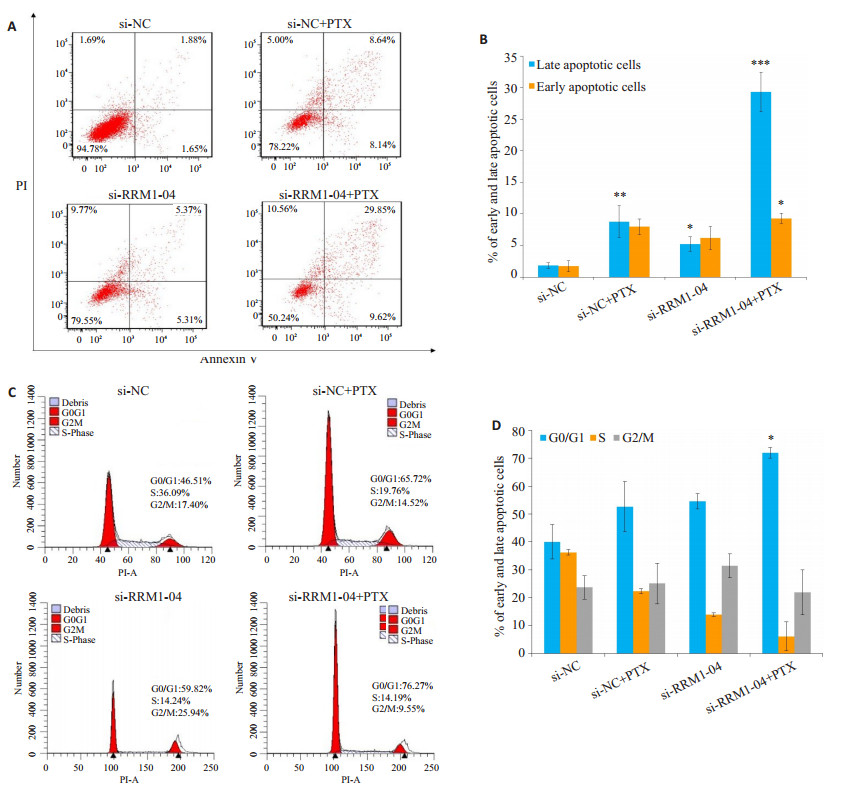
流式细胞仪检测结果表明, RRM1基因沉默以后可促进MCF-7/R细胞的凋亡, 对比差异具有统计学意义(P < 0.05), 再使用35 nmol/L紫杉醇处理48 h, MCF-7/R细胞的晚期凋亡率由(1.95±0.42)%达到(23.17±3.95)%, 显著高于si-NC+PTX组, 差异具有统计学意义(P < 0.001)。从对细胞的总凋亡率影响结果来分析, si-NC+ PTX组的早期、晚期凋亡率和坏死率共为21.78%, siRRM1-04+PTX组的早期、晚期凋亡率和坏死率共为49.76%。从该结果来看, si-NC+PTX组和si-RRM1-04+ PTX组统计学差异显著(P < 0.01)。细胞周期检测显示G0/G1期细胞比例由(45.13 ± 8.55)%达到(77.64 ± 11.928)%, 差异具有统计学意义(P < 0.05), 而G2/M期细胞无显著性变化(图 6)。使用协方差分析以排除siRNA的转染对细胞周期和凋亡的影响, 从而更为准确的评估RRM1基因沉默以后对MCF-7/R细胞紫杉醇抗性的影响, 结果显示si-RRM1+PTX组细胞凋亡率仍显著高于si-NC+PTX组(P < 0.05)。
|
图 6 乳腺癌细胞MCF-7/R转染不同siRNA序列后对细胞凋亡和周期的影响 Fig.6 Effect of RRM1 silencing on apoptosis and cell cycle distribution of MCF-7/R cells in different groups. A: PI-Annexin V flow cytometry for cell apoptosis; B: statistical analysis of cell apoptosis detected by PI-Annexin V flow cytometry; C: Flow cytometric analysis of the cell cycles; D: Quantitative cell cycle analysis.***P < 0.001;**P < 0.01;*P < 0.05 vs si-NC group. |
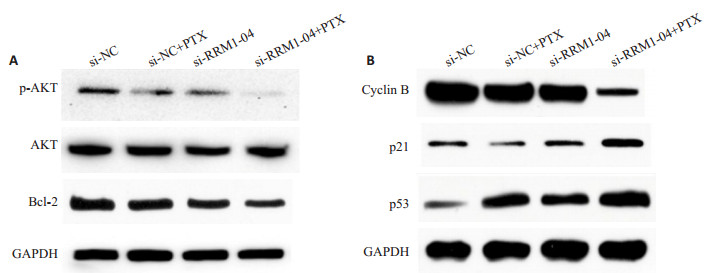
Western blot检测了各组蛋白提取物中一些凋亡标志物的表达水平, 结果显示RRM1沉默后再给予紫杉醇处理降低了p-AKT的表达, 对总蛋白AKT无明显影响, 抑制凋亡蛋白Bcl-2表达降低(图 7A)。细胞周期相关蛋白cyclin B表达水平下调, 而p21和p53的蛋白表达水平则出现上调(图 7B)。
|
图 7 乳腺癌细胞MCF-7/R转染不同siRNA序列后对细胞凋亡和周期相关蛋白表达的影响 Fig.7 Expression levels of apoptosis-and cell cycle-related proteins in MCF-7/R cells after transfection with different siRNA sequences. A: Western blotting for p-AKT, AKT, and Bcl-2 proteins in MCF-7/R cells; B: Western blotting for cyclin B, p21 and p53 proteins. |
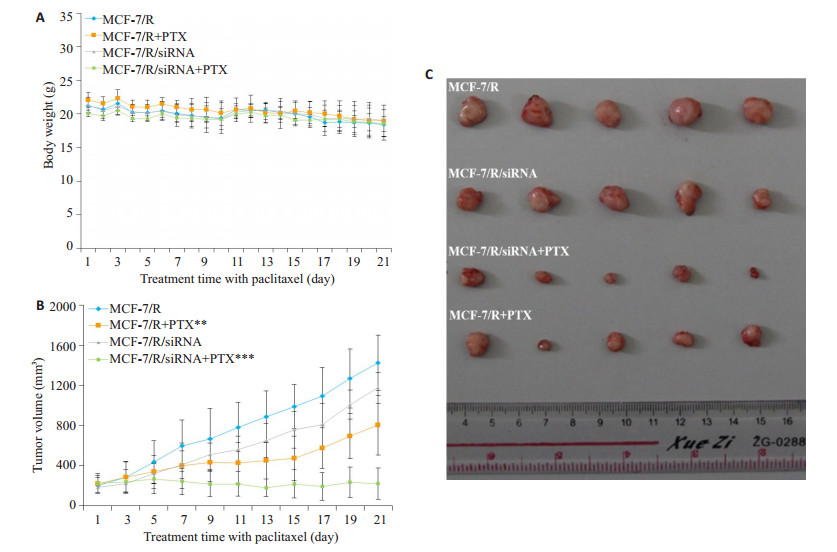
当肿瘤异种移植瘤体积达到150 mm3后, 用紫杉醇腹腔注射(15 mg/kg)给予治疗, MCF-7/R组或MCF-7/ R/siRNA组作为对照。结果显示在给药期间各组裸鼠没有明显的体质量减轻(图 78A), 表明治疗并未产生明显的毒性作用。接种MCF-7/R+PTX治疗组肿瘤体积明显小于MCF-7/R组(P < 0.01), 而接种MCF-7/R/siRNA+ PTX治疗组各时间点肿瘤体积相比MCF-7/R组显著减小(P < 0.001, 图 8B、C)。
|
图 8 沉默RRM1基因紫杉醇的体内抗肿瘤活性 Fig.8 In vivo anticancer efficacy of paclitaxel in nude mice bearing xenografts derived from MCF-7/R or MCF-7/R cells with or without siRNA transfection. A: Body weight of nude mice in different groups; B: Tumor volume in the nude mice in different groups; C: Tumor size in the nude mice in different groups. ***P < 0.001; **P < 0.01 vs MCF-7/R group. |
乳腺癌是世界范围内最常见的危害女性健康的癌症, 近些年来发病率显著提高。紫杉醇作为乳腺癌临床化疗的一线常用药, 是乳腺癌化疗中活性最强、效果较好的化疗药物之一[11]。然而, 在紫杉醇化疗过程中, 其肿瘤耐药问题成为影响疾病治疗效果的关键因素。核苷酸还原酶M1亚基(RRM1)作为DNA合成通路中的限速酶, 主要作用是为DNA修复提供原料[12], 在多种肿瘤中存在过度表达或异常激活[13], 在此同时, RRM1还介导肿瘤细胞的化疗耐药[14-15], 在非小细胞肺癌、胰腺癌、结肠癌、乳腺癌、白血病等多种细胞系中的研究中[16-24]也证实了RRM1表达水平与细胞对吉西他滨的敏感性呈现显著负相关。然而, RRM1是否参与乳腺癌化疗过程中紫杉醇耐药及RRM1在乳腺癌化疗耐药中的具体作用机制尚未见报道。我们通过对乳腺癌患者的生存曲线分析发现RRM1的表达与乳腺癌患者的无复发生存率呈负相关, 与上述研究报道的结果一致。接下来, 我们发现了人乳腺癌紫杉醇耐药细胞MCF-7/R中RRM1的表达水平显著高于紫杉醇敏感细胞MCF-7 (P < 0.01)。
我们设计并合成了一系列靶向RRM1的siRNA序列, 转染MCF-7/R细胞后根据siRNA序列对RRM1在基因和蛋白水平的干扰作用, 筛选出特异而高效的靶向沉默RRM1的siRNA序列, 通过MTT和EdU实验检测RRM1特异性沉默以后对乳腺癌细胞增殖的影响。结果显示, 转染特异性si-RRM1的耐药细胞其增殖受到抑制, 而转染阴性si-NC组细胞生长状况与空白对照组无明显差异。同时, 与空白对照组相比(395.62±87.31)nmol/L, 紫杉醇对转染特异性si-RRM1的耐药细胞IC50显著降低(142.95±32.44)nmol/L, EdU染色阳性细胞占比显著下降(P < 0.01), 而对转染阴性si-NC组细胞的IC50和EdU染色阳性细胞占比变化并不明显, 说明RRM1特异性沉默以后紫杉醇可显著抑制乳腺癌耐药细胞增殖。在裸鼠异种移植肿瘤模型实验中, 接种MCF-7/R/ siRNA细胞并给予紫杉醇治疗组的裸鼠肿瘤体积相比对照组显著减小(P < 0.001), 也说明了沉默RRM1后紫杉醇体内抗肿瘤活性增强(P < 0.001)。上述研究结果表明沉默RRM1可提高MCF-7/R细胞的化疗敏感性, 在一定程度上逆转乳腺癌紫杉醇的化疗耐药。
肿瘤化疗耐药的分子机制错综复杂, 既涉及药物转运蛋白、药物代谢相关蛋白表达的增加, 也与药物作用靶蛋白结构发生改变或表达减少以及抗细胞凋亡等相关[25]。肿瘤细胞是通过重新编码其生存信号通路而躲避细胞毒性治疗[26]。由于凋亡通路的抑制为肿瘤细胞在高压环境下的生存提供了有利条件, 研究普遍认为致癌基因为肿瘤细胞提供了内在耐药的可能[27]。由于大多数化学药物治疗是通过凋亡而引发细胞死亡, 同样诱导凋亡也可用于预防肿瘤的发生发展以及化疗耐药的产生[28]。RRM1不仅可以调节核苷酸还原酶的活性、决定底物的特异性[29-30], 另外, RRMl还可以激发G2监测点功能[31], 增加DNA损伤后的修复和凋亡[32-33]。据此, 我们推测, 靶向沉默RRM1后增加紫杉醇诱导乳腺肿瘤细胞凋亡进而影响其发生发展以及化疗耐药的产生。通过流式检测靶向沉默RRM1后细胞周期及凋亡, 结果显示, 与转染阴性si-NC组细胞凋亡情况相比, 紫杉醇对转染特异性si-RRM1-04的耐药细胞组晚期凋亡比例显著升高(P < 0.001)。为了进一步评估细胞的凋亡, 我们检测了抗细胞凋亡相关蛋白Bcl-2、p53及p-AKT表达的变化。结果显示, 与si-NC组相比, 靶向沉默RRM1后给予紫杉醇处理可明显降低p-AKT (P < 0.001)、Bcl-2 (P < 0.05)蛋白的表达水平, 对AKT总蛋白无明显影响, 而作为抑癌基因的p53, 其蛋白表达水平显著上升, 具有显著统计学差异(P < 0.001)。因此si-RRM1提高MCF- 7/R细胞化疗敏感性, 逆转乳腺癌紫杉醇化疗耐药与诱导细胞凋亡增加有关。
综上所述, 我们的研究结果首次证实沉默RRM1可提高MCF-7/R细胞化疗的敏感性, 逆转乳腺癌紫杉醇化疗耐药, 并与诱导细胞凋亡增加有关。为研究RRM1在乳腺癌中的作用机制提供了理论基础, 但本文就其具体作用机制阐述不足, 我们后续将作深入研究。
| [1] |
Wang F, Yu Z. Current status of breast cancer prevention in China[J].
Chronic Dis Transl Med, 2015, 1(1): 2-8.
DOI: 10.1016/j.cdtm.2015.02.003. |
| [2] |
Akarolo-anthony SN, Ogundiran TO, Adebamowo CA. Emerging breast cancer epidemic:evidence from Africa[J].
Breast Cancer Res, 2010, 12(4): S8.
|
| [3] |
Glück S. Nab-Paclitaxel for the treatment of aggressive metastatic breast cancer[J].
Clin Breast Cancer, 2014, 14(4): 221-27.
DOI: 10.1016/j.clbc.2014.02.001. |
| [4] |
党存曙, 张帆, 杨明, 等. 不同分期原发性肝癌患者肿瘤组织MVD与Survivin表达的相关性研究[J].
临床和实验医学杂志, 2017, 16(16): 1584-8.
DOI: 10.3969/j.issn.1671-4695.2017.16.007. |
| [5] |
Yan X, Chen X, Liang H, et al. miR-143and miR-145syn-ergistically regulate ERBB3 to suppress cell proliferation and invasion in breast cancer[J].
Mol Cancer, 2014, 24(13): 220.
|
| [6] |
袁慎俊, 胡卫, 陈涛. RNA干扰在肿瘤治疗中的研究进展[J].
中国全科医学, 2016, 19(13): 1367-70, 1374.
|
| [7] |
杨列, 张建平. RRM1和ERCC1在乳腺癌组织中的表达及临床意义[J].
现代中西医结合杂志, 2016(33): 3689-90.
DOI: 10.3969/j.issn.1008-8849.2016.33.014. |
| [8] |
Lee SY, Im SA, Park YH, et al. Genetic polymorphisms of SLC28A3, SLC29A1 and RRM1 predict clinical outcome in patients with metastatic breast cancer receiving gemcitabine plus paclitaxel chemotherapy[J].
Eur J Cancer, 2014, 50(4): 698-705.
DOI: 10.1016/j.ejca.2013.11.028. |
| [9] |
Matsumoto A, Hayashida T, Takahashi M, et al. Antitumor effect of lapatinib and cytotoxic agents by suppression of E2F1 in HER2-positive breast cancer[J].
Mol Med Rep, 2018, 18(1): 958-64.
|
| [10] |
Shinoda S, Kaino S, Amano S, et al. Deferasirox, an oral Iron chelator, with gemcitabine synergistically inhibits pancreatic cancer cell growth in vitro and in vivo[J].
Oncotarget, 2018, 9(47): 28434-44.
|
| [11] |
Ferguson S, Zheng SY, Xiu J, et al. Pan-cancer profiles of brain metastases:prioritization of therapeutic targets[J].
Neuro Oncol, 2017, 19(6): 40-1.
|
| [12] |
陈芹, 周彩存, 张颉. ERCC1、RRM1和BRCA1在非小细胞肺癌中的表达及预后意义[J].
肿瘤, 2007, 27(9): 719-22.
|
| [13] |
Turkson J, Jove R, et al. STAT proteins:novel molecular targets for cancer drug discovery[J].
Oncogene, 2000, 19(56): 6613-26.
DOI: 10.1038/sj.onc.1204086. |
| [14] |
王天晓, 王颖莹, 张中庆. STAT3小分子干扰RNA增强多柔比星的抗肿瘤活性[J].
药学学报, 2013, 48(1): 52-8.
DOI: 10.3969/j.issn.1008-7303.2013.01.07. |
| [15] |
Han Z, Feng J, Hong Z, et al. Silencing of the STAT3 signaling pathway reverses the inherent and induced chemoresistance of human ovarian cancer cells[J].
Biochem Biophys Res Commun, 2013, 435(2): 188-94.
DOI: 10.1016/j.bbrc.2013.04.087. |
| [16] |
Davidson JD, Ma L, Flagella M, et al. An increase in the expression of ribonucleotide reductase large subunn 1 is associated with gemcitabine resistance in non-small cell lung cancer cell lines[J].
Cancer Res, 2004, 64(11): 3761-6.
DOI: 10.1158/0008-5472.CAN-03-3363. |
| [17] |
Bepler G, Kusmartseva I, Sharma S, et al. RRM1 modulated in vitro and in vivo efficacy of gemcitabine and platinum in non-small-cell lung cancer[J].
J Clin Oncol, 2006, 24(29): 4731-7.
DOI: 10.1200/JCO.2006.06.1101. |
| [18] |
Nakahira S, Nakamori S, Tsujie M, et al. Involvement of dbonucleotide reductase M1 subunit overexpression ln gemcitabine resistance of human pancreatic cancer[J].
Int J Cancer, 2007, 120(6): 1355-63.
DOI: 10.1002/(ISSN)1097-0215. |
| [19] |
Nakano Y, Tanno S, Koizumi K, et al. Gemcitabine chemoresistance and molecular markers associated with gemcitabine transport and metabolism in human pancreatic cancer cells[J].
Br J Cancer, 2007, 96(3): 457-63.
DOI: 10.1038/sj.bjc.6603559. |
| [20] |
Smid K, Bergman AM, Eijk PP, et al. Micro-array analysis of resistance for gemcitabine results in increased expression of ribonucleotide reductase subunits[J].
Nucleosides Nucleotides Nucleic Acids, 2006, 25(9-11): 1001-7.
DOI: 10.1080/15257770600890269. |
| [21] |
Bergman AM, Eijk PP, Ruiz van Haperen VW, et al. In vivo induction of resistance to gemcitabine results in increased expression of ribonucleotide reductase subunit M1 as the major determinant[J].
Cancer Res, 2005, 65(20): 9510-16.
DOI: 10.1158/0008-5472.CAN-05-0989. |
| [22] |
van de Wiel MA, Costa JL, Smid K, et al. Expression microarray analysis and oligo array comparative genomic hybridization of acquired gemcitabine resistance in mouse colon reveals selection for chromosomaI aberrations[J].
Cancer Res, 2005, 65(22): 10208-13.
DOI: 10.1158/0008-5472.CAN-05-0760. |
| [23] |
Goan YG, Zhou B, Hu E, et al. Overexpression of ribonucleotide reductase as a mechanism of resistance to 2, 2-difluorodeoxycytidine in the human KB cancer cell line[J].
Cancer Res, 1999, 59(17): 4204-7.
|
| [24] |
Dumontet C, Fabianowska-Majewsda K, Mantincic D, et al. Common resistance mechanisms to deoxynucleoside analogues in vadants of the human erythroleukaemic line K562[J].
Br J Haematol, 1999, 106(1): 78-85.
DOI: 10.1046/j.1365-2141.1999.01509.x. |
| [25] |
Lecca P. Methods of biological network inference for reverse engineering cancer chemoresistance mechanisms[J].
Drug Discov Today, 2014, 19(2): 151-63.
|
| [26] |
Rycenga HB, Long DT. The evolving role of DNA inter-strand crosslinks in chemotherapy[J].
Curr Opin Pharmacol, 2018, 41: 20-6.
DOI: 10.1016/j.coph.2018.04.004. |
| [27] |
Hirotsu Y, Nakagomi H, Amemiya K, et al. Intrinsic HER2 V777L mutation mediates resistance to trastuzumab in a breast cancer patient[J].
Med Oncol, 2017, 34(1): 3.
|
| [28] |
Johnstone RW, Ruefli AA, Lowe SW. Apoptosis:a link between cancer genetics and chemotherapy[J].
Cell, 2002, 108(2): 153-64.
DOI: 10.1016/S0092-8674(02)00625-6. |
| [29] |
Aye Y, Stubbe J. Clofarabine 5'-di and-triphosphates inhibit human ribonucleotide reductase by altering the quaternary structure of its large subunit[J].
Proc Natl Acad Sci USA, 2011, 108(24): 9815-20.
DOI: 10.1073/pnas.1013274108. |
| [30] |
Fu Y, Lin H, Wisitpitthaya S, et al. A fluorimetric readout reporting the kinetics of nucleotide-induced human ribonucleotide reductase oligomerization[J].
Chembiochem, 2014, 15(17): 2598-604.
DOI: 10.1002/cbic.201402368. |
| [31] |
Tassone P, Di Martino MT, Ventura M, et al. Loss of BRCA1 function increases the antitumor activity of cisplatin against human breast cancer xenografts in vivo[J].
Cancer Biol Ther, 2009, 8(7): 648-53.
DOI: 10.4161/cbt.8.7.7968. |
| [32] |
Rosell R, Danenberg KD, Alberola V, et al. Ribonucleotide reductase messenger RNA expression and survival in gemcitabine/cisplatintreated advanced non-small cell lung cancer patients[J].
Clin Cancer Res, 2004, 10(4): 1318-25.
DOI: 10.1158/1078-0432.CCR-03-0156. |
| [33] |
Gautam A, Li ZR, Bepler G. RRM1-induced metastasis suppression through PTEN-regulated pathways[J].
Oncogene, 2003, 22(14): 2135-42.
DOI: 10.1038/sj.onc.1206232. |
 2019, Vol. 39
2019, Vol. 39

