2. 南方医科大学医学放射图像及检测技术重点实验室,广东 广州 510515
2. Guangzhou Key Laboratory of Medical Radiation Imaging and Detection Technology, Southern Medical University, Guangzhou 510515, China
准确的良恶性肿瘤鉴别诊断对于乳腺癌后续治疗方案的确定至关重要[1],全数字乳腺成像(FFDM)提供常规投影的头尾位(CC)和内外侧斜位(MLO)的高空间分辨率视图是诊断癌症的重要依据,然而,FFDM图像存在组织重叠,使得人眼主观识别图像的假阳性高、诊断的能效存在争议,但它对于降低乳腺癌的死亡率发挥着重要作用[2-3]。关键在于制定合适的FFDM图像筛查策略,提高诊断的准确性。计算机辅助诊断对FFDM图像自动分析,可提高乳腺肿瘤诊断效率,更客观、准确的判断病情,具有重要的临床研究及应用价值。计算机辅助诊断乳腺肿瘤分类主要通过放射组学实现[4]。近年来,许多放射组学的相关研究都是基于有效的传统图像特征(HCF)来实现乳腺肿瘤分类,表明HCF能够反映医学病理的专业领域知识,且能获得较好的乳腺肿瘤分类效果[5-8]。但是,要在高维的特征空间中提取出有区分性的高质量特征往往存在一定的困难,需要时间和经验,并且这些传统特征大部分局限于当前图像数据的分类任务,算法的稳定性不能保证,严重制约了算法模型在临床中的实际推广和应用。
深度学习作为自动的学习特征方法,避免了传统HCF提取的复杂性和局限性,卷积神经网络(CNN)是常见的深度学习模型,在多种领域获得广泛应用[9-10]。研究表明深度学习特征(DF)能够获取更多有用的高层次肿瘤特性信息,并且在乳腺图像识别任务中能获得更好的结果[11-13]。虽然DF代表图像整体的、更高层次的特征,对提升肿瘤分类的准确率有所帮助,但不能全面的包含肿瘤特性信息。因此,本文采用结合深度学习与放射组学这两种研究方法,将高层次的DF和低层次的HCF特征信息同时应用于乳腺肿瘤分类任务中,使得诊断信息更加完整,以提高分类精度。目前这方面的研究还很少,尤其未见在FFDM的乳腺肿瘤分类研究成果。
本文以106例乳腺肿瘤患者的FFDM图像作为研究对象,提出一个基于深度学习网络特征的乳腺肿瘤分类模型。该模型结合两个投影视图的DF和HCF,采用多分类器融合的模型进行训练,以实现乳腺良恶性肿瘤分类。
1 资料和方法 1.1 实验资料本研究回顾性分析了2015年12月~2016年12月在南方医院确诊的106个乳腺肿瘤病例,年龄43.7±8.4岁。每个病例均包含CC位和MLO位方向投影的FFDM图像,共212幅FFDM图像。其中恶性乳腺癌51例由相应的活检结果证实,良性纤维腺瘤55例由长期的随访结果证实。病例信息见表 1。所有的病例数据FFDM扫描采用Selenia Diemnsions System(Hologic, Bedford, MA, USA)。图片分辨率为3328 × 2560或4096 × 3328,灰度级别为4096(12 bit),像素大小为0.07×0.07 mm2。
| 表 1 病人信息 Tab.1 Patient characteristics |
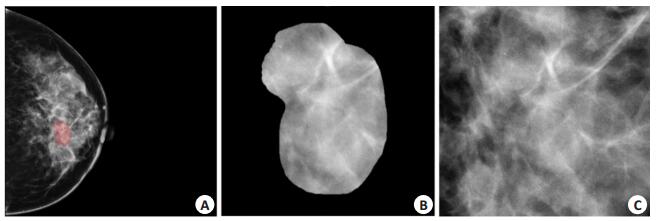
首先由经验丰富的影像专家应用ITK-SNAP软件手动勾画肿瘤区域,然后分割出感兴趣区域(ROI,图 1)。由于图像噪声会造成图像质量下降,为提高图像诊断的准确率,对ROI使用σ=1的高斯滤波来降低图像噪声。最后再将图像离散化到512(9 bit)。分割、滤波及离散化均在Matlab R2015b平台上操作。本研究的统计显著性设置为0.05。
|
图 1 FFDM图像数据 Fig.1 FFDM images showing the breast tumor (A, red area), the segmentation of the ROI (B, Original magnification: ×10), and the tumor image patch (C, ×10). |
HCF能够定量的描素肿瘤低层次的表型特征,是疾病诊断和良恶性分类的基础。本研究提取的HCF主要为2类:形态特征13个和GLGLM纹理特征11个。其中,形态特征可以量化肿瘤的形态特性,提供了良恶性肿瘤不同的几何形态信息,包括面积、长度、直径、离心率、固牢程度、扩展范围程度等。灰度间隙矩阵是统计具有相同灰度级别的像素点在某方向上的相隔距离[14]。纹理特征可以量化肿瘤内部的异质差异,由于在不同方向上计算特征,提供了图像中不同灰度级别的相对位置信息,包括间隙长度的不均匀性度量、低灰度间隙优势、高灰度间隙优势、长间隙优势、短间隙优势、间隙总数百分比等。本研究中使用4个方向(0°、45°、90°、135°)分别提取灰度间隙矩阵,取4个矩阵特征的平均值。为了降低HCF之间的冗余性,采用T检验选择出最优的HCF子集。
1.3 DF提取CNN模型通过构建含有多个隐藏层的深度非线性神经网络,将低层次特征进行逐层抽取和组合,形成更高层次的抽象特征,避免了传统机器学习中HCF复杂的提取过程。本研究对比目前常用的深度CNN模型AlexNet [15],VGG16 [16]和GoogLeNet [17],研究不同深度模型所提取的DF在乳腺肿瘤分类上的性能差异,以确定合适的结构模型。其中,AlexNet共有8层,是浅层结构模型;VGG16共有16层,是基于AlexNet架构的深度结构模型;GoogLeNet共有22层,通过构造Inception结构达到最优的局部稀疏结构。本研究将所有病人数据输入到已训练好的模型中,提取输出分类层之前的最后一层全连接层作为该模型的DF,特征的维度分别是AlexNet为1×4096,VGG16为1×4096和GoogLeNet为1×1024。
由于收集的FFDM数据不足以达到训练完整、有效的深度CNN模型,容易造成过拟合问题。因此本研究采用2种方法解决该问题:(1)数据增强[18]。主要通过-10°~+10°旋转、水平和垂直方向作镜像和-5~+5个像素平移,将原始数据扩充了10倍;(2)迁移学习[19]。采用基于大规模自然图像集ImageNet(包含140万多幅自然图像和1000种类别)预训练的CNN模型,将模型的初始化参数和权重迁移到FFDM数据集上进行微调。网络的训练集为800张,测试集260张。图像缩放到224× 224(像素),并对图像像素值进行归一化操作(图 1C)。训练过程采用五折交叉验证,取性能最佳的模型。所有网络框架运行的平台为linux-Centos7.0系统,深度学习工具Caffe。硬件配置为:显存:Titan Xp 12G,显卡:4张,服务器内存:64 G。
1.4 特征结合与分类模型构建本研究将HCF与不同网络框架的DF进行结合,分别在3种不同的传统机器学习分类器:SVM [20],随机森林[21]、逻辑回归[22]上训练和测试,并采取一种自动的融合策略,将3种不同分类器的预测结果融合成为一个结果,构建一个多分类器的乳腺肿瘤分类模型,模型结构如下:
p(l)=w1·pc1·l+w2·pc2·l+w3·pc3·l
其中p(l)为第l个病人的最终预测结果,l= 1, 2, …, 106。c1代表支持向量机分类器,c2代表随机森林分类器,c3代表逻辑回归分类器。pc1·l、pc2·l、pc3·l分别代表第1个病人在c1、c2、c3的预测结果。w1、w2、w3分别代表 3种分类器的权重,且Σiwi = 1。本研究中3种分类器权重是根据各自的AUC值大小分配,从大到小分别设置为0.5、0.3和0.2。分类模型的阈值设置为0.7,当p(l) < 0.7时,预测结果为良性;p(l)≥0.7时,预测结果为恶性。所有实验均执行20次留一交叉验证,评价结果为20次验证的平均值。训练过程中采用AUC、准确率、敏感性、特异性[31]和Yonden's index(Sensitivity+Specificity-1)[24]来评估分类模型的性能。
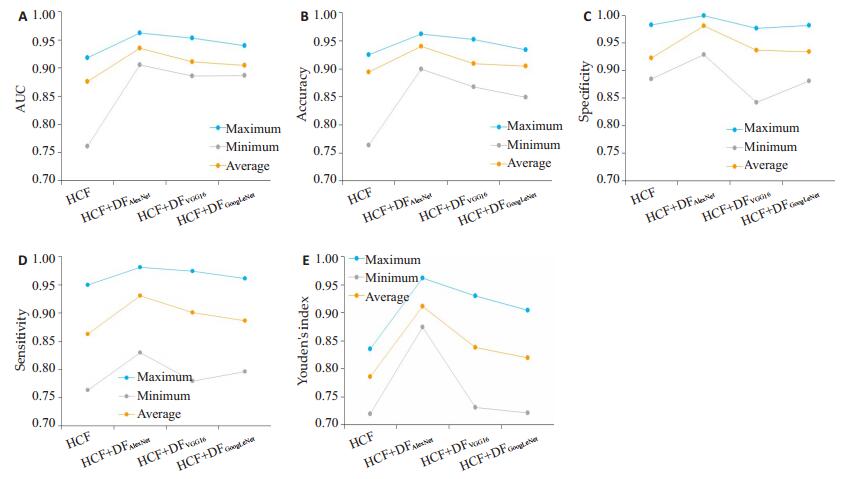
2 结果为验证DF和HCF的结合在乳腺肿瘤分类中的有效性,并且评估不同网络特征的分类性能,将4种特征组HCF、DFAlexNet+HCF、DFVGG16+HCF和DFGoogLeNet+ HCF的分类结果进行比较(表 2)。不同的特征组对应的AUC、准确率、特异性、敏感性和Youden's index的曲线见图 2。结果显示,无论采用哪种网络框架,采用DF+ HCF的分类模型分类性能均高于HCF;在AUC值方面,DFAlexNet+HCF明显优于其它网络框架,而DFVGG16+ HCF和DFGoogLeNet+HCF基本一致,但DFVGG16+HCF在最大值和平均值上表现出较好的性能。在准确率方面,DFAlexNet+HCF的分类准确率最大值、最小值和平均值明显优于其它深度学习网络架构;对比分类准确率的最小值,DFAlexNet + HCF最小值为0.94,而HCF为0.894,DFVGG16+HCF为0.909,DFGoogLeNet+HCF为0.905。在特异性方面,DFAlexNet+HCF明显优于其他网络框架,DFVGG16+ HCF的最大值和最小值低于DFGoogLeNet+HCF,但平均值表现出较好的性能。在敏感性方面,DFAlexNet+HCF优于其它网络框架,DFVGG16+HCF在最大值和平均值上优于DFGoogLeNet+HCF。在Youden's index方面,DFAlexNet+HCF明显优于其它网络框架,而DFVGG16 + HCF优于DFGoogLeNet+HCF。
| 表 2 不同的特征组在乳腺肿瘤分类中的实验结果 Tab.2 Experimental results of different feature set for breast tumor classification |
|
图 2 不同的特征组的AUC (A),准确率(B),特异性(C),敏感性(D)和Youden's index (E)曲线 Fig.2 The area under curve (A), accuracy (B), specificity (C), sensitivity (D) and Youden's index (E) of the classification model using different feature sets. |
另外,AlexNet提取的DF与HCF的结合在乳腺肿瘤分类任务上显著优于其他深度卷积神经网络框架,而VGG16略优于GoogLeNet。
3 讨论深度学习作为一个新的特征学习方法,使得特征提取的方法发生改变,尤其是CNN模型,它能让计算机从数据中自动学习高层次的特征,得到更易于分类的特征表达输出[10]。DF能提高图像分类识别的精准度[15-17],且对图像转化和扭曲有较好的鲁棒性[23]。目前CNN模型应用于FFDM图像的研究并不多[25],原因在于模型的训练需要大量的数据,才能较好的防止训练过程出现过拟合,但符合要求的医学图像数据通常比较难收集。首先,本研究通过对基于ImageNet数据集的预训练模型进行迁移学习,将预训练模型在ImageNet上学习到的一些特征和权重迁移到FFDM图像上,模拟人脑对图像的识别抽取过程,提取出DF。第二,本研究结合CC视图和MLO视图的图像信息,同时将HCF和DF结合应用于良恶性乳腺肿瘤分类。这是因为HCF和DF能够表达不同层次的图像特性[25-27]并且不同视图能够提供更加完整的目标肿瘤信息。这种方式能获得丰富的肿瘤内部异质性和形态、纹理信息,这些特征有助于乳腺肿瘤的分类任务,因而具有更高的识别率。第三,我们采用多权重融合的分类器模型对乳腺肿瘤进行分类,目的在于提高弱学习算法的泛化能力,降低整体模型的学习错误率,从而使模型更加稳定。这种方式比单分类器能产生更加稳定的分类结果[28]。实验证明本文方法提高了分类性能,具有较好的稳定性和泛化性,在一定程度上能够满足更高要求的临床需求。
本文针对目前计算机辅助诊断乳腺肿瘤分类中特征提取单纯依靠HCF,存在分类结果不稳定的问题,提出了一种基于深度学习网络特征的乳腺肿瘤分类模型。该模型结合DF和HCF,并利用多分类器加权融合的方法进行分类。在HCF中,11个GLGLM特征和12个形态特征能够反映低层次的、直观的FFDM图像纹理模式。在DF中,不同的深度网络模型特征能够反映高层次的图像纹理。相对于传统使用的HCF,高层次的DF与低层次的HCF特征结合,能够得到更具区分能力的肿瘤特性信息,获得更好的分类效果,从而大大降低了FFDM的假阳性(1-特异性)。本研究表明,不同神经网络的DF和HCF的结合在AUC值、准确率、特异性、敏感性和Youden's index上均比HCF有所提高,低层次的HCF和高层次的DF能够相互补充,因此这种特征结合的方法对于实现精准医疗具有非常重要的意义;另外,本研究AlexNet提取的DF与HCF的结合在乳腺肿瘤分类任务上显著优于其他深度卷积神经网络框架,而VGG16略优于GoogLeNet。这表明,相较于深层结构的网络,浅层结构的网络更适合用于乳腺肿瘤分类任务,网络越深,分类性能反而有下降趋势。AlexNet这浅层的网络框架的DF更适合于乳腺肿瘤的医疗诊断,能够获得高区分性的肿瘤内部特性信息,实现良恶性肿瘤的高精度分类。为检验本实验方法的分类性能,选择基于传统图像特征的方法进行对比,采用二折交叉验证,最终选用对应文献的最优结果进行对比。第1个对比实验提出多尺度的灰度特征和纹理特征[5],结果为采用不同尺度图像的最优分类结果;第2个对比实验是融合CC位和MLO位的径向局部三值模式纹理特征[6],分别使用3种分类器方法SVM,随机森林和人工神经网络(ANN)进行分类,结果显示本研究方法的分类准确率和AUC值均明显高于其他方法。
本研究存在一些局限性。第一,研究的初衷在于初步探索DF与HCF的结合在乳腺肿瘤分类中的潜能,但仅对原始图像进行分析,未来将结合多尺度图像的特征表达,以实现图像特征多元化提取。第二,由于深度学习网络中存在的“黑箱”问题[29],本文没有深入研究DF与乳腺良恶性肿瘤的异质性之间的内在联系,未来应该开展更多的工作来探索深度学习解释性与肿瘤异质性的相关问题。第三,所提出的基于深度学习特征的模型方法未尝试应用到其他方向,例如肿瘤风险评估[30]、生存预测等,希望未来的研究可扩展到这些领域。
| [1] |
Siegel RL, Miller KD, Jemal A. Cancer statistics, 2018[J].
CA Cancer J Clin, 2018, 68(1): 7-30.
DOI: 10.3322/caac.21442. |
| [2] |
Lehman CD, Arao RF, Sprague BL, et al. National performance benchmarks for modern screening digital mammography: update from the breast cancer surveillance consortium[J].
Radiology, 2017, 283(1): 49-58.
|
| [3] |
Coldman A, Phillips N, Wilson C, et al. Pan-canadian study of mammography screening and mortality from breast cancer[J].
J Natl Cancer Inst, 2014, 106(11): 1-7.
|
| [4] |
Kumar V, Gu Y, Basu S, et al. Radiomics: the process and the challenges[J].
Magn Reson Imaging, 2012, 30(9): 1234-48.
DOI: 10.1016/j.mri.2012.06.010. |
| [5] |
Xie WY, Li YS, Ma YD. Breast mass classification in digital mammography based on extreme learning machine[J].
Neurocomputing, 2016, 173(p3): 930-41.
|
| [6] |
Muramatsu C, Hara T, Endo T, et al. Breast mass classification on mammograms using radial local ternary patterns[J].
Comput Biol Med, 2016, 72(1): 43-53.
|
| [7] |
Velikova M, Samulski M, Lucas PJ. Improved mammographic CAD performance using multi-view information: a bayesian network framework[J].
Phys Med Biol, 2009, 54(5): 1131-47.
DOI: 10.1088/0031-9155/54/5/003. |
| [8] |
Tan MX, Pu JT, Zheng B. Reduction of false-positive recalls using a computerized mammographic image feature analysis scheme[J].
Phys Med Biol, 2014, 59(15): 4357-73.
DOI: 10.1088/0031-9155/59/15/4357. |
| [9] |
Schmidhuber J. Deep learning in neural networks: an overview[J].
Neural Networks, 2015, 61(1): 85-117.
|
| [10] |
He KM, Zhang XY, Ren SQ, et al. Deep residual learning for image recognition[C] //2016 IEEE Conference on Computer Vision and Pattern Recognition (CPVR), 2016: 770-8.
https://ieeexplore.ieee.org/document/7780459
|
| [11] |
Spanhol FA, Oliveira LS, Cavalin PR, et al. Deep features for breast cancer histopathological image classification [C] //2017 IEEE International Conference on Systems, Man and Cybemetics, 2017: 1868-73.
https://www.researchgate.net/publication/321411653_Deep_features_for_breast_cancer_histopathological_image_classification
|
| [12] |
Dhungel N, Carneiro G, Bradley AP. The automated learning of deep features for breast mass classification from mammograms[C] // Lecture Notes in Computer Science, 2016: 106-14.
https://link.springer.com/chapter/10.1007%2F978-3-319-46723-8_13
|
| [13] |
Jiao ZC, Gao XB, Wang Y, et al. A deep feature based framework for breast masses classification[J].
Neurocomputing, 2016, 197(2): 221-31.
|
| [14] |
Wang X, Albregtsen F, Foyn B. Texture features from gray level gap length matrix[C] // Iapr Workshop on Machine Vision Applications, 2009: 375-8.
|
| [15] |
Krizhevsky A, Sutskever I, Hinton G. ImageNet classification with deep convolutional neural networks[C] // International Conference on Neural Information Processing Systems, 2012: 1097-105.
https://www.researchgate.net/publication/267960550_ImageNet_Classification_with_Deep_Convolutional_Neural_Networks
|
| [16] |
Simonyan K, Zisserman A. Very deep convolutional networks for large-scale image recognition[J].
Comput Sci, 2014, 23(14): 1409-556.
|
| [17] |
Szegedy C, Liu W, Jia YQ, et al. Going deeper with convolutions [C] // 2015 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), 2015: 1-9.
https://www.researchgate.net/publication/265787949_Going_Deeper_with_Convolutions
|
| [18] |
Shen D, Wu G, Suk HI. Deep learning in medical image analysis[J].
Annu Rev Biomed Eng, 2017, 19(1): 221-48.
DOI: 10.1146/annurev-bioeng-071516-044442. |
| [19] |
Shin HC, Roth HR, Gao M, et al. Deep convolutional neural networks for computer-aided detection: CNN architectures, dataset characteristics and transfer learning[J].
IEEE Trans Med Imaging, 2016, 35(5): 1285-98.
DOI: 10.1109/TMI.2016.2528162. |
| [20] |
Srikant R, Agrawal R. Mining generalized association rules[C] // International Conference on Very Large Data Bases, 1995: 407-19.
https://www.sciencedirect.com/science/article/pii/S0167739X97000198
|
| [21] |
Breiman L. Random forest[J].
Mach Learn, 2001, 45(1): 5-32.
DOI: 10.1023/A:1010933404324. |
| [22] |
Conklin JD. Applied logistic regression[J].
Technometrics, 2013, 44(1): 81-2.
|
| [23] |
Zou WY, Ng AY, Zhu S, et al. Deep learning of invariant features via simulated fixations in video[J].
Adv Neural Inf Process Syst, 2012, 25(12): 3212-20.
|
| [24] |
Hilden J, Glasziou P. Regret graphs, diagnostic uncertainty and Youden's Index[J].
Stat Med, 2015, 15(10): 969-86.
|
| [25] |
Li H, Mendel KR, Lee JH, et al. Deep learning in breast cancer risk assessment: evaluation of fine-tuned convolutional neural networks on a clinical dataset of FFDMs[C] //Medical Imanging 2018: Computer-aided Diagnosis, 2018: 10575-8.
https://www.researchgate.net/publication/323434520_Deep_learning_in_breast_cancer_risk_assessment_evaluation_of_fine-tuned_convolutional_neural_networks_on_a_clinical_dataset_of_FFDMs
|
| [26] |
Zhou B, Garcia AL, Xiao J, et al. Learning deep features for scene recognition using places database[C] // International Conference on Neural Information Processing Systems: mit press, 2014: 358-63.
https://www.researchgate.net/publication/279839496_Learning_Deep_Features_for_Scene_Recognition_using_Places_Database
|
| [27] |
Zhang Q, Xiao Y, Dai W, et al. Deep learning based classification of breast tumors with shear-wave elastography[J].
Ultrasonics, 2016, 72(2): 150-7.
|
| [28] |
Kuncheva L.
Combining pattern classifiers: methods and algorithms[M]. New York: Wiley-Interscience, 2004.
|
| [29] |
Marcus G. Deep learning: a critical appraisal[J].
CoRR, 2018, 476(13): 1-26.
|
| [30] |
Kayla M, Hui L, Deepa S, et al. Transfer learning from convolutional neural networks for computer-aided diagnosis: a comparison of digital breast tomosynthesis and full-field digital mammography[J].
Acad Radiol, 2018, 25(10): 1-9.
|
| [31] |
周志华.
机器学习[M]. 北京: 清华大学出版社, 2016.
|
 2019, Vol. 39
2019, Vol. 39

