2. 广州医科大学附属第三医院妇产科,广东 广州 510515;
3. 南方医院妇产科,广东 广州 510515;
4. 汕头大学医学院,广东 汕头 515041
2. Department of Gynecology and Obstetrics, Third Affiliated Hospital of Guangzhou Medical University, Guangzhou 510150, China;
3. Department of Gynecology and Obstetrics, Nanfang Hospital, Southern Medical University, Guangzhou 510515, China;
4. Shantou University Medical College, Shantou 515041, China
临床试验中, 随着研究者对生物学、基因组学的了解愈发深入, 更多的证据表明, 即使是同样的疾病, 因为患者之间存在的特征差异, 药物疗效也不尽相同, 特定的人群对某一个治疗有效, 而另一特定人群对该治疗无效或比传统治疗效果差[1-2]。于是在获得了总体人群中的疗效后, 研究者期待了解在不同特征人群中疗效是否和总体一致。这些不同的人群就是我们所说的亚组[3-4]。
此外, 随着人类基因组测序技术和生物医学分析技术的进步以及大数据分析工具的出现等, 精准医疗的概念逐渐被提出以及重视[5]。实现精准医疗的一个重要的手段就是进行亚组识别, 通过将个体分为不同亚群可以使预防或治疗性的干预集中在那些获益更多、节约费用、不产生副作用的人群中[6]。
目前, 许多学者已经提出了不同亚组识别方法。Foster等[7]提出的"virtualtwins"方法是针对二分类结局资料, 通过随机森林和回归树建模, 分别计算每个人在试验组和对照组中的阳性概率并计算差值, 根据设定的规则获得试验组中的亚组人群, 进而进行判别和预测; Diagonal Linear Discriminant Analysis(DLDA)"方法[8]和"Adaptive Signature Design(ASD)"方法[9]用以识别对治疗或药物敏感的亚组人群; 利用参数得分系统, 根据每一个患者的得分将他们分为不同的亚组[10-11]; 还有基于结构化"Logistic-normal"的混合模型, 对亚组进行识别[12]等。
前述亚组识别方法主要是设法在试验组中找到获益亚组, 适用于对照组为安慰剂的情况[7]。而目前很多疾病已经存在具有一定疗效或者疗效较好的治疗方法, 因此, 出于伦理上的考虑, 目前很多临床试验采用的是阳性对照, 临床试验结果常显示试验组疗效与对照组无差异或仅显示轻微的疗效改善。针对这种情形, 本文在virtualtwins"方法[7]基础上, 提出一种新的方法, 拟判断试验组、阳性对照组是否分别有相应的获益亚组, 并通过建立多分类判别模型以识别试验组获益人群、对照组获益人群以及在两组获益相当的人群, 为个体化治疗提供参考。
1 方法简介 1.1 Logistic回归方法简介假设协变量为X1、X2、…、Xk, 二分类Logistic回归模型如下所示[13]:
| $ \mathit{log}it\left( P \right) = \mathit{ln}\left( {\frac{P}{{1{\rm{ - }}P}}} \right) = {\beta _0} + {\beta _1}{X_\mathit{1}} + \ldots + {\beta _k}{X_k} $ | (1) |
其中P为阳性结果发生的概率; βj(j=0, 1, 2, …, k)为模型的回归系数, 参数估计采用最大似然法进行估计, 其估计值记为bj, 显著性检验采用似然比检验。
1.2 亚组识别方法介绍目前大多学者认为亚组人群产生的原因是由于亚组人群的特征协变量与不同的处理产生了交互作用, 则Logistic模型形式为logit(P(Y=1|TX))=α+βT+yh(X)+θTw(X)[7, 14]。其中, Y表示反应变量; T表示处理组别; h(X)表示与Y相关的协变量; Tw(X)表示不同处理与亚组人群的特征协变量的交互作用。w(X)即与亚组人群相关的协变量, 所以我们希望能够确定w(X), 从而找到亚组的特征值。
假定Y=0表示失败, Y=1表示成功; Xp代表p个协变量; T=1表示A组, T=0表示B组。
第1步:根据组别T分层, 分别对Y(成功与否)做Logistic回归, 得到两个Logistic模型。即当T=1时得到模型A, 当T=0时得到模型B。
第2步:将每个患者的协变量分别带入这两个Logistic模型, 获得每个患者i分别在两个处理组内的成功概率P0i=P(Yi=1|Ti=0, Xi)和P1i=P(Yi=1|Ti=1, Xi)。
第3步:令Zi=P1i-P0i, 计算
第4步:以多分类变量newsubgroup为反应变量, Xi为协变量建立多分类Logistic回归模型, 寻找亚组相关协变量并用于亚组判别。
本研究采用Monte-Carlo技术进行模拟研究[15-16], 然后进行实例分析, 所有模拟及计算均通过R3.4.0[17]和SPSS19.0[18]软件实现。
2 模拟实验 2.1 模拟错判率假定有两个协变量X1, X2, 其中X1为连续型变量, X2为二分类变量, X1~N(10, 32), X2~B(n, 0.5)。模拟过程如下:
(1) 根据模型Logistic(P)=5+10*X1+10*X2, P=exp(logit(P))/(1+exp(logit(P)))产生样本量为2n例患者, 每个患者的二分类结局变量用Yi=rbinom(1, 1, Pi)获得。并将这2n例患者随机等分为A, B两组, 每组有n例患者。
(2) 分别以Y为因变量, A, B两组分别建立两个Logistic回归模型(modelA, modelB)。将所有患者的协变量信息分别带入这两个模型中, 每个患者均获得两个治愈概率:PiA, PiB(i=1, 2, 3, …, 2n), 共2n对。令Zi=PiA-PiB, 求
(3) 重复前述步骤共1000次, 这1000次错误率的平均值即为错判率。
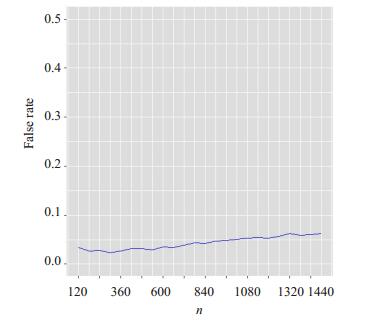
由图 1可见, 当样本量较小时错判率略不稳定, 但均小于0.05, 随着样本量n的逐渐增加, 错判率有增大的趋势, 但在所设定的样本量1500范围内, 错判率都在0.07以下。
|
图 1 错判率与样本量n的关系 Fig.1 Relationship between false rate and the sample size n. |
本研究在产生含亚组的数据时, 暂考虑两个亚组相关协变量的情形。假定两个协变量X1和X2, X1为分类变量, X2为连续型变量, modelA中X1~N(n, 0.5), X2~B(2, 22); modelB中X1~N(n, 0.5), X2~B(8, 32)。
(1) 分别根据所设置的modelA:Logistic(A_P)=-2+6*X1+0.5*X2, A_P=exp(logit(A_P))/(1+exp(logit(A_P)))和modelB:Logistic(B_P)=-4+10*X1+2.5*X2, B_P=exp(logit(B_P))/(1+exp(logit(B_P)))产生样本量足够(例如50000例)数据集, 每个患者有三个变量值, 即Xi1, Xi2, Pi。
(2) 从modelA所产生的数据中, 挑选治愈概率较大的(Pi>0.5), 同时满足将其协变量带入modelB所得治愈概率较小(Pi < 0.5)的患者n/3例, 获得数据集dataA+B-, 这些是对处理组A敏感的亚组, Subgroup=A。产生二分类结局变量Yi=rbinom(1, 1, A_Pi)。
(3) 从modelA所产生的数据中, 挑选治愈概率较小的(Pi < 0.5), 同时满足将其协变量带入模型B所得治愈概率较大(Pi>0.5)的患者n/3例, 获得数据集dataA-B+, 这些是对处理组B敏感的亚组, Subgroup=B。产生二分类结局变量Yi=rbinom(1, 1, A_Pi)。
(4) 从modelA所产生的数据中, 挑选治愈概率较小的(Pi < 0.5), 同时满足将其协变量带入模型B所得治愈概率也较小(Pi < 0.5)的患者, 或者挑选治愈概率较大的(Pi>0.5), 同时满足将其协变量带入模型B所得治愈概率较大(Pi>0.5)的患者, 共n/3例, 获得数据集dataAB, 这些是对处理组A, B都敏感或对处理组A, B都不敏感的亚组, Subgroup=C。产生结局变量Yi=rbinom(1, 1, A_Pi)。
(5) 合并步骤(2)、(3)、(4)产生的数据, 获得数据集dataA, 相当于实际接受A处理的患者, 共n例。其中对A药敏感、对B药敏感、对A和B药都敏感或都不敏感的患者, 各占约1/3。
(6) 以Yi为因变量进行Logistic回归, 获得的modelA2, 相当于原始总体modelA的样本估计模型。
(7) 用同样方法产生实际接受B处理的数据集dataB以及modelB2。
(8) 合并dataA和dataB为data_total, 共2n例患者。用Sub_ori表示数据的来源, 即Sub_ori=1表示数据来源于dataA, Sub_ori=2表示数据来源于dataB。
(9) 将所有患者的协变量信息分别带入两个Logistic回归模型modelA2和modelB2中, 每个患者均获得两个治愈概率:PiA, PiB(i=1, 2, 3, …, 2n), 共2n对。令Zi=PiA-PiB, 计算2n个Z值的均值
(10) 定义亚组:若Zi>0, 且大于
(11) 以多分类变量newsubgroup为反应变量, 对各协变量建立多分类Logistic回归模型, 并计算该模型判对率。
(12) 前瞻性考核:将步骤(1)~(8)所产生的数据按7:3随机分为两部分, 一部分作为训练集用于建立判定亚组的模型, 另一部分作为考核数据集用于前瞻性考核, 按步骤(10)~(11)分别计算出符合率和模型判对率。
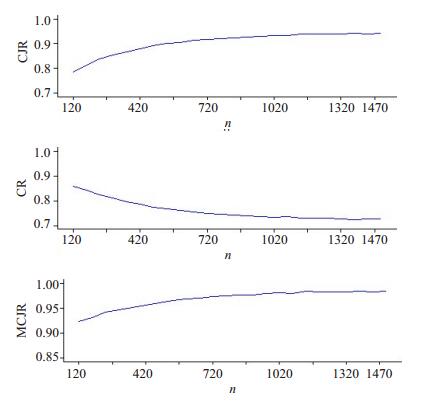
图 2可见, 在本研究所设置的样本量范围内(1500), 正确判断率均在0.75以上, 随着n增大, 正确判断率逐渐增大, 最终稳定在0.94附近; 金标准符合率逐渐降低, 最终稳定在0.72以上; 多分类Logistic模型判对率均在0.92以上, 随着n逐渐增大, 判对率逐渐增大, 最终稳定在0.98附近。
|
图 2 正确判断率、符合率和模型判对率与样本量n的关系 Fig.2 Relationship of correct judgment rate (CJR), coincidence rate (CR), and model correct judgment rate (MCJR) with the sample size n. |
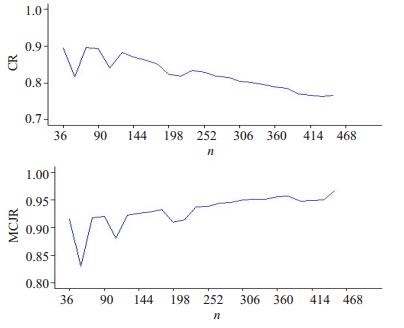
图 3可见, 考核数据集分析结果显示随着样本量增加, 符合率逐渐降低, 最终稳定均在0.75以上; 多分类Logistic模型判对率均在0.82以上。
|
图 3 考核数据集符合率和判对率与样本量n的关系 Fig.3 Relationship of the coincidence rate (CR) and model correct judgment rate (MCJR) with the sample size n. |
目前临床上常有妊娠足月的孕妇具备引产指征需适时终止妊娠。足月妊娠引产可以提高阴道自然分娩率, 减少剖宫产的发生, 对母婴预后有积极的作用。尤其是妊娠达到41周的孕妇, 胎儿发育成熟, 宫内出现不利于胎儿生存的因素, 应该立即采取引产措施[19-20]。根据美国FDA批准的引产药物中, 临床使用率较高的为缩宫素和前列腺素制剂, 根据临床多项研究[21-22], 含有PEG2的前列腺素制剂对于宫颈条件不成熟的孕妇, 具有促进宫颈成熟、缩短产程、引产成功率高的临床效果。地诺前列酮栓是被认为较为安全和有效的药物之一, 其主要成分是前列腺素E2(PEG2), 在中晚期妊娠方面取得了较为满意的疗效[19, 22-26]。
由于个体的差异性, 不同人群可能对药物的敏感性不同, 本例的重点是应用本研究提出的亚组识别的方法, 判断患者所适用的药物。
3.1 数据介绍选取2010年1月~2014年12月在10家医院收治的1080例用地诺前列酮栓晚孕引产孕妇和同期使用缩宫素晚孕引产的963例孕妇[27]。
3.2 数据格式数据变量格式见表 1。
| 表 1 数据变量格式 Tab.1 Variables in dataset |
以是否经阴道成功分娩为因变量, 以年龄、BMI、是否初产、引产前宫颈评分、孕周、胎膜早破、羊水过多、羊水过少、延期妊娠、妊娠高血糖、妊娠高血压、胎儿窘迫和胎盘功能减退为自变量, 分别以地诺前列酮栓组和缩宫素组数据各建立一个二分类Logistic回归模型, 用本研究所提方法判断患者所属亚组。进一步, 以所获亚组作为因变量建立多分类Logistic回归模型, 有统计学意义的变量即为亚组相关协变量并用于亚组识别。
3.4 分析结果影响地诺前列酮栓组和缩宫素组经阴道分娩率的因素如表 2和表 3所示, 拟合的方程分别为
| 表 2 地诺前列酮栓组经阴道分娩成功率的影响因素分析 Tab.2 Factors influencing the success rate of delivery in the Dinoprostone Suppositories group (n=1080)* |
| 表 3 缩宫素组经阴道分娩成功率的影响因素分析 Tab.3 Factors influencing the success rate of delivery in the Oxytocin group (n=963)* |
logit(P1)=3.119-2.353*是否初产-0.487*延期妊娠-0.526*妊娠高血糖-1.144*妊娠高血压+0.238*引产前宫颈评分
logit(P2)=6.107-0.081*年龄-0.083*BMI-1.598*是否初产+0.350*胎膜早破-1.571*妊娠高血压+0.179*引产前宫颈评分。
进行亚组判别的多分类Logistic回归分析结果见表 4。可见影响亚组判别的变量有年龄、BMI、引产前宫颈评分、胎膜早破、延期妊娠和妊娠高血糖。模型的总判对率为84.09%((717+198+803)/2043), 根据该模型可对孕妇引产时选择药物提供参考。
| 表 4 多分类Logistic回归分析结果 Tab.4 Results of multinomial Logistic regression analysis (n=2043)* |
在进行亚组辨识时, 基于Logistic回归, 模型通常需要引入交互项[7]。当与亚组相关的协变量较多时, 即使只考虑一维或者二维的交互作用, 交互项也会很多, 按照此方法通常需要很大的样本量才能比较准确的找到亚组人群。而其他亚组识别方法, 比如基于树形结构模型的方法[28-30], 在临床实际运用中过于复杂, 需要掌握高级的编程技术。
本研究在亚组识别方法[7]基础之上, 直接按照处理组分层, 通过分别建立的二分类Logistic模型算出每个患者在不同的处理组下的概率而将患者归为不同的亚组之中, 分别找到在两处理组中的获益人群, 以及在两组中获益相当的人群, 避免了在模型中加入复杂的交互项, 计算和操作起来更加简便。当存在亚组相关协变量时, 模拟结果显示本研究所提出的方法均保持较为理想的水平(错判率 < 0.07, 正确判断率>0.75, 金标准符合率>72%, 多分类模型判对率>92%)。
在实例分析中, 通过分别建立地诺前列酮栓组和缩宫素组经阴道分娩成功率的模型, 获得患者的亚组身份, 进一步以患者所属亚组作为因变量建立的多分类Logistic回归模型显示亚组相关协变量分别为年龄、BMI、引产前宫颈评分、胎膜早破、延期妊娠和妊娠高血糖。模型判对率为84.09%。该模型可对孕妇引产时选择药物提供参考。因此本研究所提出的亚组判别方法简单、可靠, 适用于双臂、阳性对照设计下的亚组识别。
本研究尚存在以下不足:所提亚组识别方法仅适用于两个处理组的临床试验; 尚未考虑二分类结局比例以及目标人群的亚组比例对正确判断率的影响。上述所存在的问题有待于进一步的研究。
| [1] |
Lagarto L, Cerejeira J. Identification of sub-groups in acutely ill elderly patients with delirium:a cluster analysis[J].
Int Psychogeriatrics, 2016, 28(8): 1283-92.
DOI: 10.1017/S1041610216000302. |
| [2] |
Wu RF, Zheng M, Yu W. Subgroup analysis with time-to-event data under a logistic-cox mixture model[J].
Scandinavian J Statistics, 2016, 43(3): 863-78.
DOI: 10.1111/sjos.v43.3. |
| [3] |
Alosh M, Huque MF, Bretz F, et al. Tutorial on statistical considerations on subgroup analysis in confirmatory clinical trials[J].
Stat Med, 2017, 36(8): 1334-60.
DOI: 10.1002/sim.v36.8. |
| [4] |
Rosenkranz GK. Exploratory subgroup analysis in clinical trials by model selection[J].
Biom J, 2016, 58(5): 1217-28.
DOI: 10.1002/bimj.v58.5. |
| [5] |
杨咪, 杨小丽. 理性审视精准医疗发展中的问题及其对策探讨[J].
中国全科医学, 2017, 20(7): 886-90.
DOI: 10.3969/j.issn.1007-9572.2017.07.024. |
| [6] |
史越, 张华. 精准医疗发展面临的挑战与问题分析[J].
卫生政策与管理, 2018, 35(4): 327-9.
|
| [7] |
Foster JC, Taylor JM, Ruberg SJ. Subgroup identification from randomized clinical trial data[J].
Stat Med, 2011, 30(24): 2867-80.
DOI: 10.1002/sim.v30.24. |
| [8] |
Lu TP, Chen JJ. Subgroup identification for treatmentselection in biomarker adaptive design[J].
BMC Med Res Methodol, 2015, 15: 105-15.
DOI: 10.1186/s12874-015-0098-7. |
| [9] |
Freidlin B, Jiang WY, Simon R. The cross-validated adaptive signature design[J].
Clinical Cancer Res, 2010, 16(2): 691-8.
DOI: 10.1158/1078-0432.CCR-09-1357. |
| [10] |
Shen YY, Cai TX. Identifying predictive markers for personalized treatment selection[J].
Biometrics, 2016, 72(4): 1017-25.
DOI: 10.1111/biom.12511. |
| [11] |
Kang S, Lu WB, Song R. Subgroup detection and sample size calculation with proportional hazards regression for survival data[J].
Stat Med, 2017, 36(29): 4646-59.
DOI: 10.1002/sim.v36.29. |
| [12] |
Shen J, He XM. Inference for subgroup analysis with a structured Logistic-Normal mixture model[J].
J Am Stat Assoc, 2015, 110(59): 303-12.
|
| [13] |
孙振球.
医学统计学[M]. 2版. 北京: 人民卫生出版社, 2008: 333-9.
|
| [14] |
Hou JE, Seneviratne C, Su XG, et al. Subgroup identification in personalized treatment of alcohol dependence[J].
Alcohol Clin Exp Res, 2015, 39(7): 1253-9.
DOI: 10.1111/acer.2015.39.issue-7. |
| [15] |
Kerns LX. Simultaneous confidence bands for log-logistic regression with applications in risk assessment[J].
Biom J, 2017, 59(3): 420-9.
DOI: 10.1002/bimj.v59.3. |
| [16] |
Finch WH, Bolin JH, Kelley K. Group membership prediction when known groups consist of unknown subgroups:a monte carlo comparison of methods[J].
Front Psychol, 2014, 5(337): 1-12.
|
| [17] |
Rteam R, Team R, Team R, et al. R:a language and environment for statistical computing[J].
Computing, 2014, 14(1): 12-21.
|
| [18] |
陈平雁, 黄浙明.
IBM SPSS 19统计软件应用教程[M]. 2版. 北京: 人民卫生出版社, 2012: 205-25.
|
| [19] |
张琦, 赵晓兰. 地诺前列酮栓和缩宫素对促宫颈成熟及分娩结局的影响[J].
中国生化药物杂志, 2017, 37(2): 180-2.
|
| [20] |
Alfirevic Z, Keeney E, Dowswell T, et al. Which method is best for the induction of labour?A systematic review, network meta-analysis and cost-effectiveness analysis[J].
Health Tech Assess, 2016, 20(65): 1-584.
DOI: 10.3310/hta20650. |
| [21] |
Mayer RB, Oppelt P, Shebl O, et al. Initial clinical experience with a misoprostol vaginal insert in comparison with a dinoprostone insert for inducing labor[J].
Eur J Obstet Gynecol Reprod Biol, 2016, 200(5): 89-93.
|
| [22] |
李艳, 李妘. 地诺前列酮栓与催产素用于足月妊娠促宫颈成熟及引产的临床效果观察[J].
世界最新医学信息文摘, 2016, 16(89): 94.
DOI: 10.3969/j.issn.1671-3141.2016.89.085. |
| [23] |
刘宝霞. 地诺前列酮栓在足月妊娠促宫颈成熟及引产中的临床效果观察[J].
实用妇科内分泌杂志:电子版, 2017, 4(28): 44-5.
|
| [24] |
景联红. 催产素与地诺前列酮栓用于产科足月妊娠促宫颈成熟及引产中的效果[J].
中外医学研究, 2018, 16(8): 161-2.
|
| [25] |
Shirley M. Dinoprostone vaginal insert:a review in cervical ripening[J].
Drugs, 2018, 78(15): 1615-24.
DOI: 10.1007/s40265-018-0995-2. |
| [26] |
Pierce S, Bakker R, Myers D, et al. Clinical insights for cervical ripening and labor induction using prostaglandins[J].
AJP Rep, 2018, 8(4): e307-14.
DOI: 10.1055/s-0038-1675351. |
| [27] |
李雪媛, 郭跃文, 许燕文, 等. 孕晚期地诺前列酮栓引产的临床疗效及安全性:多中心回顾性分析[J].
南方医科大学学报, 2017, 37(1): 18-23.
|
| [28] |
Fokkema M, Smits N, Zeileis A, et al. Detecting treatment-subgroup interactions in clustered data with generalized linear mixed-effects model trees[J].
Behav Res Methods, 2018, 50(5): 2016-34.
DOI: 10.3758/s13428-017-0971-x. |
| [29] |
Zhao Y, Zheng W, Zhuo DY, et al. Bayesian additive decision trees of biomarker by treatment interactions for predictive biomarker detection and subgroup identification[J].
J Biopharmaceutical Statistics, 2018, 28(3): 534-49.
DOI: 10.1080/10543406.2017.1372770. |
| [30] |
Chen YC, Chen JJ. Ensemble survival trees for identifying subpopulations in personalized medicine[J].
Biom J, 2016, 58(5): 1151-63.
DOI: 10.1002/bimj.201500075. |
 2018, Vol. 38
2018, Vol. 38

