2. 广东省东莞市妇幼保健院,广东 东莞 523122;
3. 南方医科大学南方医院惠侨医疗中心,广东 广州 510515
2. Dongguan Maternal and Children's Healthcare Hospital, Dongguan 523122, China;
3. Huiqiao Medical Center, Nanfang Hospital, Southern Medical University, Guangzhou 510515, China
α-地中海贫血(简称α-地贫)是由于α-珠蛋白链缺乏或合成减少而导致的一种常染色体隐性遗传病,其中我国南方广东、广西地区的α-地贫基因人群携带率分别高达8.53%和17.55% [1-2]。当夫妇双方同为--SEA/αα基因型时,胎儿有1/4的几率为重型α-地贫(Bart's水肿胎),导致胎儿严重缺氧而死于腹中或出生不久后死亡。此病的有效防控措施主要是对高风险胎儿进行产前诊断,但创伤性穿刺采样手术可导致宫内感染、流产、胎儿死亡等并发症,并且淘汰重症患儿的引产或流产手术,给孕妇带来严重的身心创伤和社会伦理问题,还有可能导致不孕不育和习惯性流产等严重后果。胚胎植入前遗传学诊断(PGD)是对配子或移入宫腔之前的胚胎进行遗传学诊断,选择无遗传学疾患的胚胎植入子宫而实现优生优育[3-5]。α-地贫PGD已有相关报道及临床实践[6-7],但普遍存在基于单细胞模板的扩增失败、等位基因脱扣(ADO)等诸多问题[8];且操作繁琐而检测时间长,技术流程和质量控制的完整性尚不理想。研发适用于临床实践的PGD技术与方法,是当前生殖医学领域的研究热点之一,更是我国南方地区α-地贫防控的现实需求。本研究即采用MDA全基因组扩增(WGA)技术,以囊胚活检细胞为检测对象,建立了一种α-地贫SEA缺失突变快速植入前遗传学诊断方法,报道如下。
1 材料和方法 1.1 样本来源收集2017年6月在本实验室进行地贫基因检测,并确诊基因型为--SEA/αα的乙二胺四乙酸(EDTA)抗凝外周全血4份(男女各2份,年龄18~28岁),其中2份用于淋巴细胞分离,另2份采用传统酚氯仿法抽提基因组DNA(gDNA),灭菌双蒸水溶解并调整浓度≈50 ng/μL,用于检测体系建立与优化、方法评价。2016年3~12月间,募集6对拟进行卵胞浆内单精子显微注射(ICSI)辅助生殖的夫妇,其中4对单方基因型为--SEA/αα,2对双方基因型为--SEA/αα,待其分娩正常胎儿后,收集自愿捐献的废弃胚胎12个(每对夫妇2个),均为第5天(D5)囊胚期玻璃化冷冻保存,用于方法学应用评价。以上样本均由样本提供者和捐献者签署知情同意书。
1.2 方法 1.2.1 方法学策略及引物探针设计本研究的技术策略是对单细胞gDNA进行全基因组扩增(WGA),通过质检确定WGA成功后,即进行目标序列DNA分析以实现胚胎的快速基因分型。据此策略,采用多重置换扩增(MDA)作为WGA的前提下,本研究需设计并建立WGA质检及α-地贫SEA缺失突变基因分型两个体系。质检体系是以管家基因GAPDH和β-actin为靶基因,分别设计特异性引物和TaqMan探针,建立双重TaqMan Real-time荧光PCR体系,对WGA产物中的目标基因进行相对定量检测,以确定WGA是否成功及扩增产量。α-地贫SEA缺失突变基因分型体系是根据基突变断裂点及截短DNA序列,于断裂点两端及缺失区域范围内分别设计突变及正常对照上下游引物,建立双重PCR扩增体系,根据特异性扩增产物的有无而实现基因分型。引物和探针序列见表 1所列,均由生工生物工程(上海)有限公司合成。
| 表 1 引物和探针序列 Table 1 Sequences of primers and probes |
以基因型--SEA/αα的外周全血gDNA为模板,分别建立并优化WGA质检体系和α-地贫SEA缺失突变基因分型体系。WGA质检检测体系总体积为20.0 μL,包含10×TaKaRa Ex Buffer(宝生物工程)2.4 μL、dNTPs(2.5 mmol/L each)1.5 μL、5.0 U/μL TaKaRa ExTaq 0.3 μL、10 pmol/μL的GAPDH和β-actin的引物及探针各0.2 μL、DNA模板≈100 ng;检测条件为Bio-Rad CFX96荧光PCR仪(Bio-Rad),95 ℃预变性3 min,94 ℃ 30 s+60 ℃ 1 min共45个循环,于60 ℃末采集FAM和HEX通道荧光信号。α-地贫SEA缺失突变基因分型体系总体积为25.0 μL,包含10× TaKaRa LA Buffer 2.5 μL、5.0 mol/L Betaine 2.5 μL、DMSO 1.0 μL、dNTPs(2.5 mmol/L each)1.5 μL、5.0 U/μL TaKaRa LATaq 0.3 μL、10 pmol/μL的α-Control(正常对照)和SEA deletion(阳性目标序列)上下游引物各0.2 μL、DNA模板≈100 ng;检测条件为Bio-Rad C1000 PCR仪(Bio-Rad),95 ℃预变性7 min;95 ℃ 45 s+68 ℃ 45 s+72 ℃ 30 s共35个循环,72 ℃再延伸5 min;PCR产物经1.5%琼脂糖凝胶电泳分析并观察目的条带。
1.2.3 基于全基因组扩增的检测方法验证于洁净10 mL离心管中加入1.0 mL人外周血淋巴细胞分离液(TBD,天津市灏洋生物科技有限公司);同时,将基因型为--SEA/αα的抗凝外周全血1.0 mL与等体积PBS缓冲液(pH 7.2~ 7.4)颠倒混匀,用吸管缓慢加于分离液液面之上,置于4 ℃预冷离心机400 g离心30 min,吸取淋巴细胞层并用pH 7.2~7.4的PBS缓冲液洗涤3次,充分稀释后滴于直径60 mm细胞培养皿中,倒置显微镜下分别吸取2、3、4、5、6个淋巴细胞转入含4.0 μL PBS(pH 7.2~7.4)的200 μL EP管中,每细胞数各2管,2样本共20管。然后,按试剂盒使用说明,用REPLI-g Single Cell kit(cat. # 150343, Qiagen, Germany),对此20管细胞样本进行全基因组扩增(WGA)。
用灭菌双蒸水将WGA产物稀释20倍,同时以≈ 50 ng/μL外周全血gDNA为对照样本,分别取2.0 μL为检样,用上述建立的WGA质检检测体系进行检测。每样本平行检测2次,根据GAPDH和β-actin基因对应的FAM与HEX荧光通道信号的有无判断WGA是否成功;同时分别以WGA产物和对照样本的各荧光通道Ct值,参照2-ΔΔCt相对定量规则[9-11],根据2-ΔCt公式(ΔCt= Ct_WGA产物-Ct_gDNA对照样本,相对浓度ng/μL=2-ΔCt×gDNA对照样本浓度ng/μL),计算各WGA产物的gDNA相对浓度,每样本取两荧光通道计算值均值。然后,据其相对浓度,各取≈100 ng WGA产物为检材,用上述所建立的α-地贫SEA缺失突变基因分型体系进行检测,每样本平行检测2次。根据基因分型结果对检测方法的准确性、灵敏度、WGA成功率及等位基因脱扣率进行评价。
1.2.4 基于囊胚滋养层细胞活检的检测方法应用评价复苏所收集的玻璃化冷冻胚胎样本,采用激光切割结合抽吸法采集4~6个囊胚滋养层细胞,并将活检后的完整囊胚胚胎采用0.5%链蛋白酶去除透明带,分别置于含4.0 μL PBS(pH7.2~7.4)的200 μL EP管中,瞬时离心后置-20 ℃保存。将这些样本转移至实验室,采用上述已经建立的方法进行WGA、WGA质检、α-地贫SEA缺失突变基因分型全流程检测分析,以囊胚活检的检测结果与完整囊胚胚胎的检测结果者对比分析而进行应用评价。
2 结果 2.1 检测体系的灵敏度与准确性参照相关文献[12-13],对PCR体系中缓冲液、引物和探针等组份进行优化。所确定的WGA质检体系为双重TaqMan实时荧光PCR反应体系,体系中gDNA模板量为10.0~500.0 ng时FAM和HEX荧光通道均为典型“S”形扩增曲线(如图 1B所示),Ct值≤32.0。所确定的α-地贫SEA缺失突变基因分型体系为双重PCR反应体系,SEA缺失和正常内参目的片段长度分别为698 bp和385 bp,体系中gDNA模板量为20.0~500.0 ng时,目的条带清晰,能准确实现SEA缺失基因分型(图 1C)。
|
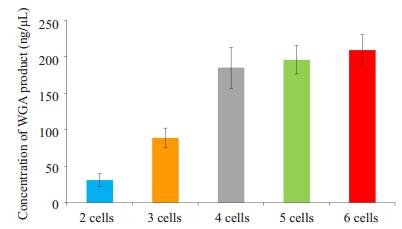
图 1 不同细胞水平WGA产物gDNA相对浓度 Figure 1 gDNA concentration of WGA products for different cell numbers. |
从2份--SEA/αα抗凝外周血中,分别制备2、3、4、5、6个淋巴细胞的样本各4管,共20管。MDA全基因组扩增总体积为50.0 μL/样本,质检结果显示所有样本管WGA成功,扩增产物的gDNA相对浓度分别为31.0± 8.6 ng/μL(2细胞)、89.0±13.5 ng/μL(3细胞)、185.0± 28.0 ng/μL(4细胞)、196.0±19.4 ng/μL(5细胞)、209.0± 21.5 ng/μL(6细胞)(图 1)。以WGA产物为模板的α-地贫SEA缺失突变基因分型结果中,有1例2细胞样本出现等位基因脱扣(--SEA/--SEA),其它19例样本的基因分型结果正确(--SEA/αα)。
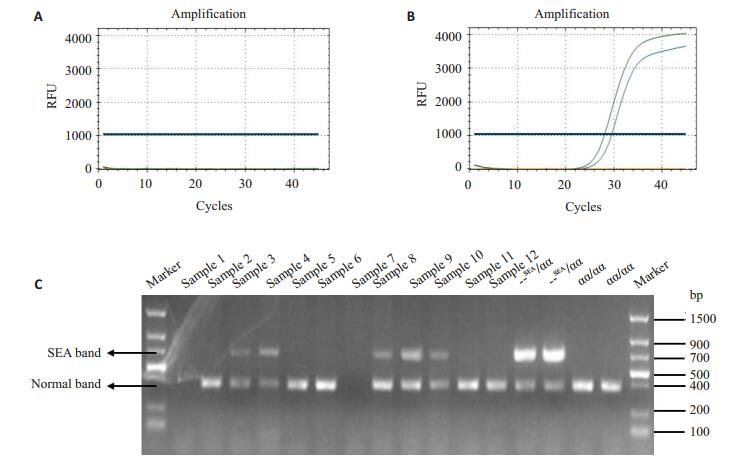
2.3 基于囊胚滋养层细胞活检的检测方法应用评价12例囊胚活检样本中,有2例WGA质检检测结果无荧光信号(图 2A),判定为WGA失败;其它样本均为双荧光信号(图 2B),WGA成功。所有囊胚活检样本的α-地贫SEA缺失突变基因分型结果见图 2C。以全胚胎的基因分型结果为目标基因型,10例囊胚活检样本的检测结果(表 2)与目标基因型相符。
|
图 2 WGA产物质检结果示意图及12例囊胚活检样本SEA基因分型电泳结果图 Figure 2 Result of WGA quality testing and electrophoresis of 12 blastocyst biopsy α-Thalassemia SEA deletion genotypes. A: Quality testing showing WGA failure; B: Quality testing showing successful WGA; C: Genotyping results of the 12 blastocyst biopsy samples and 4 control samples. |
| 表 2 12例囊胚活检样本检测结果 Table 2 Results of 12 blastocyst biopsy cell samples |
α-地贫SEA缺失为东南亚及我国南方地区最常见的地贫基因突变类型,每年约有2.6万名孕妇面临α-地贫Bart's水肿胎高风险[14-16]。采用植入前遗传学诊断(PGD)优选不致病胚胎进行植入,能从源头上实现优生优育。目前α-地贫SEA缺失突变PGD的方法主要有多重荧光PCR[17],巢式PCR结合数字PCR的拷贝数变异分析[6],MDA结合短串联重复序列多态性分析[18-20]等方法。这些方法均需要测序仪或数字PCR仪等特殊设备,有的还需进行家系分析,操作繁琐且检测时间长。鉴于这些技术难点与局限,目前能独立完成α-地贫SEA缺失突变PGD的医疗机构很少,多采用“活检标本外送检测”,增加了工作流程及费用,急需建立可操作性强、具有完整流程、能够应用于临床实践的PGD技术平台[21]。
PGD流程中,最关键步骤是对卵裂球或囊胚细胞等有限细胞检材进行全基因组扩增,为后续目标基因分型提供足够DNA模板,目前最常用的是MDA技术[22]。据Qiagen公司的MDA试剂盒说明,由于引物二聚体的随机延伸,无模板空白对照样本的DNA产物同样能达到40 μg/反应(REPLI-g Single Cell Handbook,Qiagen, Germany)。据此,以OD260确定的WGA后DNA浓度,不能准确判断WGA是否成功,也不能准确反映目标基因组扩增后的具体浓度。所以,本研究建立的WGA质检体系,是以人类基因组管家基因GAPDH和β-Actin为靶基因,以≈50 ng/μL外周全血gDNA为参照样本,通过对WGA产物的质检,可获取两个重要信息:①如果这两个靶基因均有典型扩增曲线则表明人类基因组WGA成功;②可获得gDNA的相对浓度,而有效把握下一步基因分型检测的模板量。本研究采用的2-ΔCt公式计算简单,即于同一荧光通道,如果WGA产物的Ct值与对照样本的Ct值相同,则表明WGA产物中的目标基因模板与对照样本一致,也就是WGA产物的gDNA相对浓度与对照样本的浓度一致(20×50 ng/μL);如果WGA产物与对照样本的Ct值差异为1,则说明WGA产物的gDNA相对浓度为对照样本的1/2(2-1×50 ng/μL)。本研究中淋巴细胞WGA质检结果说明,虽然MDA的DNA产量普遍能达到40 μg,但此产物中目标gDNA只是其中一部分,结果显示检材为2、3细胞时WGA的gDNA相对浓度较低,当达到4细胞则趋于稳定。另外,PGD所面临的等位基因脱扣(ADO),也源于单细胞WGA过程中扩增不平衡或gDNA模板断裂。通过增加检材的细胞数可以大大降低gDNA模板断裂的影响,本研究中4例2淋巴细胞样本中,就有一例出现等位基因脱扣,与已报道的情况相似[23-24, 26]。综合本研究中基于淋巴细胞全基因组扩增检测方法验证结果的gDNA相对浓度和ADO发生率,可以认为囊胚活检≥4细胞即可达到理想的WGA效果。
基于单细胞或少数几个细胞的WGA,MDA技术的扩增产物最长(平均长度12 kb),但兼并碱基与基因组DNA的结合起点是随机的,如果后续目标基因分型所涵盖的DNA序列越长,WGA产物中包含的完整模板也就越少。据此,本研究建立的α-地贫SEA缺失突变基因分型体系,采用了短片段Gap-PCR方案,不但保证了检测体系的特异性与敏感性,也大大缩短了检测时间,实现快速基因分型。
本研究的12例囊胚活检样本,10例WGA成功样本的基因分型结果与全胚胎的基因分型结果相符。其中2例WGA不成功,很可能是囊胚活检细胞取样后,经过近2 h的远距离运输至本实验室,样本解冻及样本管颠倒而造成了细胞丢失。由此也说明“活检标本外送检测”模式[21]具有一定的风险,检测流程的完整性与连贯性是检测成功的关键之一,应尽可能于同一实验室完成囊胚活检、WGA、目标基因分型等全部操作;如确需活检取样后送检,样本必须无解冻、无颠倒而保证细胞无丢失。
已有研究表明,人类胚胎单个卵裂球的ADO率高达25% [25],且存在细胞嵌合情况。随后的研究表明,当使用4个以上细胞作为起始模板进行WGA时ADO显著降低[26]。如果以囊胚滋养层细胞为受检样本,不仅可提供更多受检细胞,极大地降低等位基因脱扣情况的发生,且基本不影响胚胎后续着床发育[27-28]。本研究建立的方法为一个完整检测流程,即采用MDA技术,4-6个囊胚细胞为检材,经WGA产物质检及相对浓度评估后,以短片段Gap-PCR体系,实现α-地贫SEA缺失突变的快速基因分型。经评价验证,此方法结果准确、操作简单、检测成本低、无需特殊的仪器设备,能在12 h内完成α-地贫SEA缺失突变的快速植入前遗传诊断,为临床上α-地贫SEA缺失突变PGD提供一种新方法,也为缺失型α-地贫基因其它突变类型(-α3.7,-α4.2等),或其他单基因遗传病致病基因突变的PGD方法学研究提供参考。
| [1] |
Shang X, Xu X. Update in the genetics of thalassemia: What clinicians need to know[J].
Best Pract Res Clin Obstet Gynaecol, 2017, 39(3): 3-15.
|
| [2] |
Xiong F, Sun M, Zhang X, et al. Molecular epidemiological survey of haemoglobinopathies in the guangxi zhuang autonomous region of southern china[J].
Clin Genet, 2010, 78(2): 139-48.
DOI: 10.1111/cge.2010.78.issue-2. |
| [3] |
唐永梅, 韦立红, 秦祖兴, 等. 不同活检时机对地中海贫血PGD胚胎发育及诊断结果的影响[J].
中国优生与遗传杂志, 2017, 25(4): 122-3, 136.
|
| [4] |
Monni G, Peddes C, Iuculano A, et al. From prenatal to preimplantation genetic diagnosis of beta-thalassemia. prevention model in 8748 cases: 40 years of single center experience[J].
J Clin Med, 2018, 7(2): E35.
DOI: 10.3390/jcm7020035. |
| [5] |
Rund D. Thalassemia 2016: modern medicine battles an ancient disease[J].
Am J Hematol, 2016, 91(1): 15-21.
DOI: 10.1002/ajh.24231. |
| [6] |
Lee TH, Hsu YC, Chang CL. Detection of SEA-type alphathalassemia in embryo biopsies by digital PCR[J].
Taiwan J Obstet Gynecol, 2017, 56(4): 487-94.
DOI: 10.1016/j.tjog.2017.03.002. |
| [7] |
Chen LJ, Diao ZY, Xu ZP, et al. The clinical application of NGSbased SNP haplotyping for PGD of Hb H disease[J].
Syst Biol Reprod Med, 2017, 63(3): 212-7.
DOI: 10.1080/19396368.2017.1296501. |
| [8] |
李荣, 邓明芬, 徐艳文, 等. 应用全基因组扩增技术对α地中海贫血进行植入前遗传学诊断的效果分析[J].
生殖医学杂志, 2018, 27(2): 166-70.
DOI: 10.3969/j.issn.1004-3845.2018.02.013. |
| [9] |
Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) method[J].
Methods, 2001, 25(4): 402-8.
DOI: 10.1006/meth.2001.1262. |
| [10] |
Zhou WJ, Wang G, Zhao XE, et al. A multiplex qPCR gene dosage assay for rapid genotyping and Large-Scale population screening for deletional alpha-Thalassemia[J].
J Mol Diagn, 2013, 15(5): 642-51.
DOI: 10.1016/j.jmoldx.2013.05.007. |
| [11] |
Hu XW, Zhang LJ, Nan SZ, et al. Selection and validation of reference genes for quantitative real-time PCR in artemisia sphaerocephala based on transcriptome sequence data[J].
Gene, 2018, 657(4): 39-49.
|
| [12] |
Henegariu O, N a, Dlouhy SR, et al. Multiplex PCR:critical parameters and step-by-step protocol[J].
Biotechniques, 1997, 23(3): 504-11.
DOI: 10.2144/97233rr01. |
| [13] |
Standish I, Leis E, Schmitz N, et al. Optimizing, validating, and field testing a multiplex qPCR for the detection of amphibian pathogens[J].
Dis Aquat Organ, 2018, 129(1): 1-13.
DOI: 10.3354/dao03230. |
| [14] |
Liu JZ, Jia XY, Tang N, et al. A comprehensive, simple molecular assay of common deletions and mutations causing alphathalassemia in southeast asia and southern China[J].
Am J Hematol, 2010, 85(5): 370-2.
|
| [15] |
Songdej D, Babbs C, Higgs DR. An international registry of survivors with Hb Bart's HydropsFetalis Syndrome[J].
Blood, 2017, 129(10): 1251-9.
DOI: 10.1182/blood-2016-08-697110. |
| [16] |
Farashi S, Harteveld CL. Molecular basis of α-thalassemia[J].
Blood Cells Mol Dis, 2017, 70(5): 43-53.
|
| [17] |
Piyamongkol W, Vutyavanich T, Sanguansermsri T. Preimplantation genetic diagnosis of alpha-thalassemia(-SEA) using novel multiplex fluorescent PCR[J].
J Assist Reprod Genet, 2012, 29(1): 95-102.
DOI: 10.1007/s10815-011-9641-6. |
| [18] |
He F, Zhou WJ, Cai R, et al. Systematic assessment of the performance of whole-genome amplification for SNP/CNV detection and beta-thalassemia genotyping[J].
J Hum Genet, 2018, 63(4): 407-16.
DOI: 10.1038/s10038-018-0411-5. |
| [19] |
沈晓婷, 徐艳文, 钟依平, 等. 多重置换扩增结合短串联重复序列在植入前遗传学诊断中的应用[J].
北京大学学报:医学版, 2013, 45(6): 852-8.
|
| [20] |
Chen M, Chan JK, Nadarajah S, et al. Single-tube nonaplex microsatellite PCR panel for preimplantation genetic diagnosis of Hb Bart's hydrops fetalis syndrome[J].
Prenat Diagn, 2015, 35(6): 534-43.
DOI: 10.1002/pd.v35.6. |
| [21] |
范莉.广西β地中海贫血胚胎植入前遗传学诊断技术平台建立及临床应用研究[D].南宁: 广西医科大学, 2017.
http://cdmd.cnki.com.cn/Article/CDMD-10598-1017204609.htm
|
| [22] |
Huang L, Ma F, Chapman A, et al. Single-Cell Whole-Genome amplification and sequencing: methodology and applications[J].
Annu Rev Genomics Hum Genet, 2015, 16(2): 79-102.
|
| [23] |
Durmaz B, Ozkinay F, Onay H, et al. Genotyping of beta-globin gene mutations in single lymphocytes: a preliminary study for preimplantation genetic diagnosis of monogenic disorders[J].
Hemoglobin, 2012, 36(3): 230-43.
DOI: 10.3109/03630269.2012.675891. |
| [24] |
Collins SC. Preimplantation genetic diagnosis: technical advances and expanding applications[J].
Curr Opin Obstet Gynecol, 2013, 25(3): 201-6.
DOI: 10.1097/GCO.0b013e32835faafe. |
| [25] |
Moutou C, Gardes N, Viville S. Duplex, triplex and quadruplex PCR for the preimplantation genetic diagnosis (PGD) of cystic fibrosis (CF), an exhaustive approach[J].
Prenat Diagn, 2004, 24(7): 562-9.
DOI: 10.1002/(ISSN)1097-0223. |
| [26] |
Liu WQ, Zhang HM, Hu D, et al. The performance of MALBAC and MDA methods in the identification of concurrent mutations and aneuploidy screening to diagnose beta-thalassaemia disorders at the single- and multiple-cell levels[J].
J Clin Lab Anal, 2018, 32(2): e22267.
DOI: 10.1002/jcla.2018.32.issue-2. |
| [27] |
Scott J, Upham KM, Forman EJ, et al. Cleavage-stage biopsy significantly impairs human embryonic implantation potential while blastocyst biopsy does not: a randomized and paired clinical trial[J].
Fertil Steril, 2013, 100(3): 624-30.
DOI: 10.1016/j.fertnstert.2013.04.039. |
| [28] |
Chang LJ, Huang CC, Tsai YY, et al. Blastocyst biopsy and vitrification are effective for preimplantation genetic diagnosis of monogenic diseases[J].
Hum Reprod, 2013, 28(5): 1435-44.
DOI: 10.1093/humrep/det048. |
 2018, Vol. 38
2018, Vol. 38

