2. 陕西省结核病防治院,陕西 西安 710100
2. Shaanxi Tuberculosis Hospital, Xi'an 710100, China
胃癌是最常见的恶性肿瘤之一,进展期胃癌预后差、术后复发率高。因为缺乏可靠的早期诊断标志物及治疗靶点,胃癌的诊断和治疗目前仍然面临重大挑战,因此,筛选胃癌肿瘤标志物仍然是目前的研究热点。
研究人员已经发现了一些在胃癌发生、发展中有重要作用的标志物,其中,KIAA0101,也称为PCLAF (PCNA-associate factor),由于其在众多肿瘤进展、转移中起作用,受到越来越多的重视。在人体许多肿瘤中,包括乳腺癌[1]、食管癌[2]及肝癌等[3],都检测出了KIAA0101表达水平的升高。我们前期的研究已经发现,KIAA0101基因可能在胃癌进展和转移过程中起重要作用,最终导致复发和死亡[4-5]。
KIAA0101的高表达可以显著影响人体细胞周期的调控,通过siRNA下调KIAA0101表达可以显著减少胃癌细胞增殖。研究表明敲除KIAA0101基因可能通过上调p21WAF1和p21WAF1-PCNA复合体数量,导致细胞在G1/S期的过渡受到阻滞[6-8]。因此,高表达KIAA0101通过影响细胞周期的某些基因或通路,促进了胃癌细胞的增殖,可能是导致胃癌进展和复发的重要原因。
尽管如此,KIAA0101在胃癌中影响细胞周期的作用机制还不是完全的清楚,对KIAA0101通过调控哪些基因影响胃癌细胞周期,仍未有系统的研究结果。如果能够筛选出KIAA0101下游基因,从而阐明其影响胃癌细胞周期的机制,可能找到更加可靠的肿瘤标志物,为临床医师提供新型的胃癌治疗靶点。因此,本部分实验将研究胃癌中KIAA0101基因表达量,利用分子生物学方法和生物信息学工具,研究KIAA0101基因对细胞周期相关基因的可能影响,探讨KIAA0101在胃癌细胞周期调控中的作用意义。
1 材料和方法 1.1 材料收集2016年10月~2017年12月西安交通大学医学院第一附属医院肿瘤外科收治的80例经病理诊断的胃腺癌患者癌组织和癌旁组织手术切除标本,其中男性53例,女性37例,年龄43~72岁。所有患者手术前均未接受过化学治疗或放射治疗。病理学诊断依据世界卫生组标准,所有新鲜标本从体腔移除后迅速冷冻储存于-80 ℃液氮罐内以备进一步实验。本研究经西安交通大学医学院第一附属医院伦理委员会批准,所有患者均签署知情同意书。
1.2 方法 1.2.1 主要试剂及仪器Trizol试剂(Invitrogen Cat. No.15596-026)、Nanodrop核酸/蛋白质定量仪(Thermo),SensiCapture凝胶图像分析系统(培清),梯度PCR仪(Bio-Rad),iQTM5多重实时荧光定量PCR仪(Bio-Rad),稳压稳流型电泳仪(六一仪器),Milli-Q纯水器(Millipore),Bio-Rad荧光定量PCR仪(Bio-Rad CFX96)、DAVID Bioinformatics Resources6.8。
1.2.2 RNA提取用液氮将研钵预冷后,从液氮罐中取出组织样本进行研磨,边研磨边补充液氮,待组织被磨成粉末状后,加入1 mL Trizol试剂混匀、裂解组织,再把溶解液移至无菌无酶的1.5 mL EP管中备用。将EP管中的Trizol混合液中加入0.2 mL氯仿,振摇混匀15 s,室温放置10 min,吸取上层水相至新的EP管中,加入0.5 mL异丙醇,振摇混匀,室温放置10 min。4 ℃,12 000 r/min离心10 min,可于EP管底部看见白色胶状RNA沉淀,弃去上清。加入75%乙醇,振摇充分,洗涤RNA沉淀。4 ℃,7500 r/min离心5 min,弃上清,开盖干燥RNA沉淀30 min,然后加入20~40 μL DEPC水溶解后,进入下一步总RNA的鉴定。
1.2.3 RNA的鉴定将RNA溶液适当稀释后取10 μL样本测定核酸A值并计算RNA浓度,读取A260/A280的比值,纯的RNA样品A260/A280的比值应为1.7-2.0。取RNA稀释液10 μL行1%琼脂糖凝胶电泳检测。
逆转录反应以5 μg总RNA为模板,在逆转录酶催化下逆转录为cDNA。按照Fermentas公司生产的RevertAidTM First Strand cDNA Synthesis Kit操作说明,在普通PCR仪上进行逆转录反应。引物信息如下KIAA0101, F,5′-TCCTGAAGAGGCAGGAAGCAG T-3′,R,5′-TTGTGTGATCAGGTTGCAAAGGA-3′;GAPDH,F,5′-GGAGCGAGATCCCTCCAAAAT-3′,R,5′-GGCTGTTGTCATACTTCTCATGG-3′。
1.2.4 生物信息学分析使用TCGA(The Cancer Genome Atlas)中的cBioPortal数据库(http://www.cbioportal.org/)寻找胃腺癌中KIAA0101的相关基因,根据数据库返回的信息,计算所有标本中上调mRNA水平的百分比,使用KEGG(Kyoto Encyclopedia of Genes and Genomes)绘制通路图,并将相应基因在通路中的相关位置用红色圆圈加以标注。
1.2.5 数据统计使用SPSS19.0软件进行数据统计,组间比较采用配对样本的t检验,P<0.05认为差异有统计学意义。
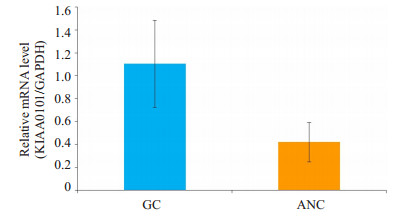
2 结果 2.1 KIAA0101癌组织中的表达水平显著高于癌旁组织胃腺癌组织中KIAA0101 mRNA表达水平显著高于配对癌旁组织(P<0.05,图 1)。
|
图 1 胃腺癌组织中KIAA0101 mRNA表达水平与配对癌旁组织之间的对比结果 Figure 1 Comparison of KIAA0101 mRNA expression levels between gastric cancer tissues (GC) and paired adjacent noncancerous tissues (ANC). P < 0.05. |
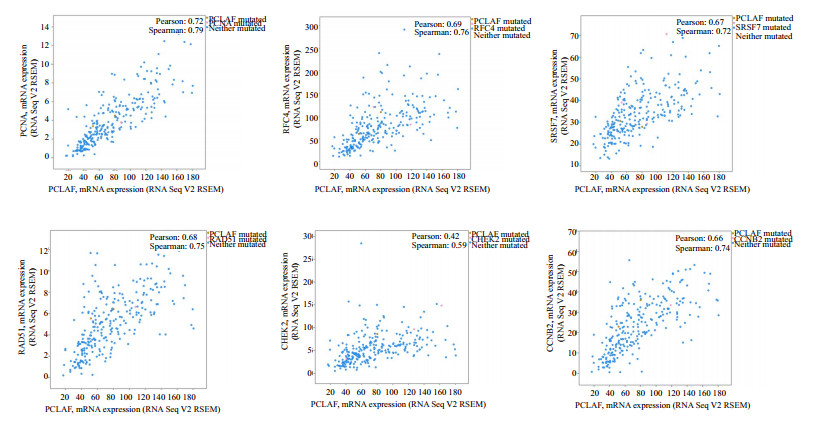
cBioPortal分析平台通过汇总分析来源于478例位腺癌的全部15400多个基因探针的表达强度,将与KIAA0101基因在胃腺癌中表达水平具有相关关系的基因进行了筛选。我们选取前20位加以展示(表 1)。表达水平相关关系的评判标准主要以Pearson Score和Spearman Score进行评价,分数越高,代表两基因表达水平在所有478例肿瘤组织中越一致。与KIAA0101表达和突变相关联前6位的基因在各标本中的相对表达情况如图 2。
| 表 1 TCGA胃腺癌组织数据中与KIAA0101表达相关的基因(前20位) Table 1 KIAA0101-related genes in TCGA gastric adenocarcinoma database |
|
图 2 TCGA胃腺癌数据库中与KIAA0101表达和突变相关性最高的6个基因 Figure 2 Top 6 genes associated with KIAA0101 expression and mutation in TGCA. |
采用DAVID 6.8对差异基因进行GO功能富集分析,发现KIAA0101表达改变后表达变化的上下游基因主要在以下方面发挥作用:蛋白磷酸化、RNA加工、细胞周期、DNA代谢过程、蛋白质转运、乙酰化、细胞凋亡、蛋白质水解、氧化还原等(表 2)。
| 表 2 Gene ontology analysis分析结果 Table 2 Results of gene ontology analysis |
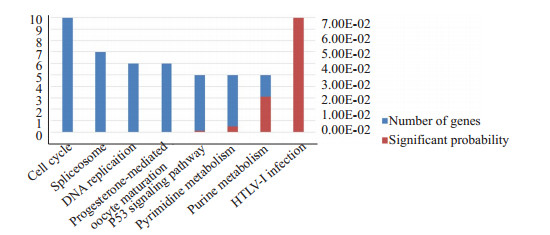
我们根据KEGG与BIOCARTA中所有pathway的基因信息,将差异基因所在的通路P进行排序,选取前8位加以展示(表 3,图 3)。结果显示KIAA0101表达水平改变主要影响的胃癌相关通路有:细胞周期、剪接体、DNA复制、p53信号转导通路等。
| 表 3 前8位差异基因所在的通路 Table 3 Top 8 pathways with differential gene expressions |
|
图 3 KIAA0101表达主要水平改变主要影响的胃癌相关通路 Figure 3 Changes of KIAA0101 expression mainly affected gastric cancer-related pathways. |
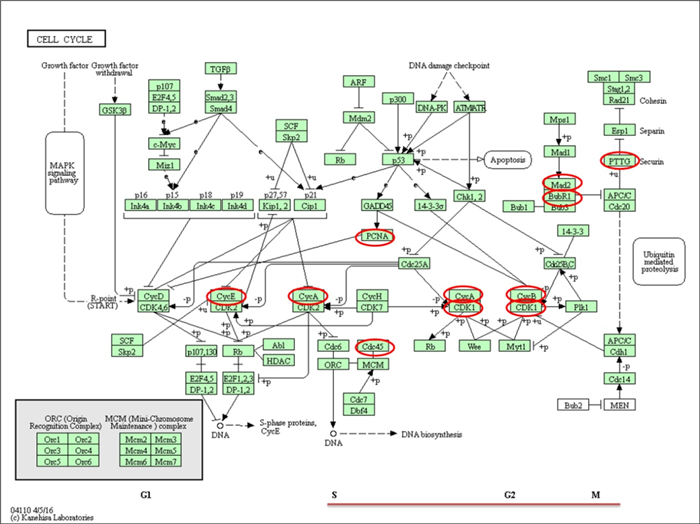
我们观察到,与KIAA0101表达相关性较好的基因大多涉及DNA周期调控方面,故我们将通路中的各个基因带入KEGG通路相关网站生成通路图(图 4),并将相应基因在通路中的相关位置用红色圆圈加以标注。结果发现,BUB1B (budding uninhibited by benzimidazoles 1 homolog beta)、MAD2L1 (MAD2 mitotic arrest deficientlike 1)、CDC45(cell division cycle 45)、CDK1(cyclindependent kinase 1)等基因处于重要位置,这些基因主要位于细胞周期的S期或G2/M期,而调控G1期的基因很少见。
|
图 4 KIAA0101相关基因在细胞周期中的位置关系图 Figure 4 Positional relationship of KIAA0101-associated genes in cell cycle. |
最后,我们将相应与KIAA0101表达模式相类似的基因列表回带入TCGA cBioPortal进行基因之间相互网络作用的关系研究,并借由系统生成基因拓扑关系图(图 5)。我们可以发现,BUB1B、MAD2L1、CDC45、CDK1、CCNE1(Cyclin E1)、CCNB2(Cyclin B2)、PCNA (proliferating cell nuclear antigen)等基因都位于网络之中,并处于重要位置。
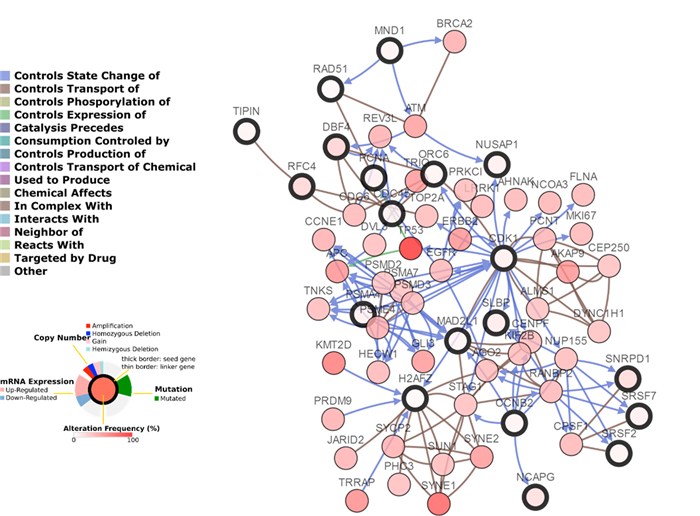
|
图 5 KIAA0101相关基因在拓扑关系图中的位置 Figure 5 Location of KIAA0101-associated genes in the topology map. |
综合几项生物信息学分析结果及候选基因的研究现状,我们选择BUB1B、MAD2L1、CDC45、CDK1、CCNE1、CCNB2六种基因进行mRNA水平验证,结果发现,80例癌组织中BUB1B、MAD2L、CDK1、CCNE1、CCNB2 mRNA表达水平显著高于配对癌旁组织(P<0.05),CDC45 mRNA表达水平无显著性差异(P>0.05,图 6)。
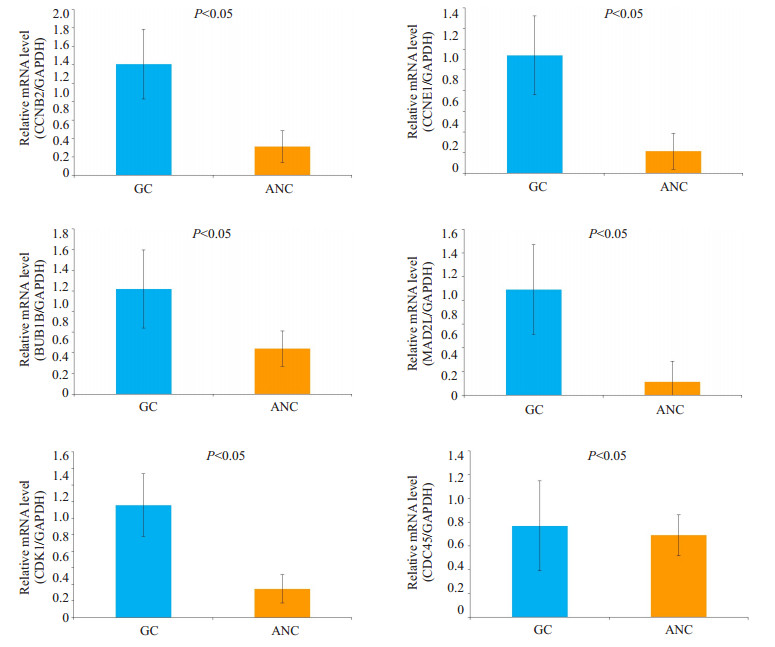
|
图 6 候选基因mRNA在胃癌组织及配对癌旁组织的对比 Figure 6 Comparison of mRNA expression of the candidate genes between gastric cancer and paired adjacent tissues. BUB1B, MAD2L, CDK1, CCNE1 and CCNB2 mRNA expressions were significantly higher in gastric cancer tissues than in the adjacent tissues (P < 0.05). No significant differential expression of CDC45 mRNA was found in gastric cancer tissues (P > 0.05). |
本研究发现,BUB1B、MAD2L1、CDK1、CCNE1、CCNB2等基因与KIAA0101之间存在明显的相互调控关系,或者它们在细胞中形成复合物或多聚体,共同调控细胞周期或参与其他重要的生物学功能。
在前期的研究中,我们应用RT-PCR、免疫组化染色和Western blot等方法证实,KIAA0101 mRNA和蛋白在胃癌组织和细胞中的表达上调,且KIAA0101蛋白在胃癌组织中的表达与肿瘤的pTNM分期和生存预后呈显著相关。因此,我们在既往研究的基础上,采用生物信息学方法,筛选相关下游基因并进行验证,研究该基因对胃腺癌细胞周期的影响。
在有关KIAA0101与胃癌细胞周期的关系研究中,降低细胞中KIAA0101表达可以使细胞周期中G2/M期和细胞凋亡增加,表明KIAA0101可能参与了细胞周期和细胞凋亡调控进程[1-4]。我们进一步通过生物信息学研究发现,KIAA0101表达变化主要影响了与肿瘤相关的以下通路:细胞周期、剪接体、DNA复制、p53信号转导通路等。调整KIAA0101表达后表达量有所改变的上下游基因主要有以下功能作用:磷酸化、剪接变异体、选择性剪接、乙酰化作用等。与KIAA0101在胃腺癌中表达情况相一致的基因主要有BUB1B、MAD2L1、CDC45、CDK1、CCNE1、CCNB2及PCNA等。
一些研究虽然发现BUB1B在胃癌中过表达, 但其表达水平对胃癌细胞增殖和预后的影响尚有争议。有学者发现BUB1B与肿瘤细胞增殖呈正相关[1],BUB1B高表达与胃癌浸润、淋巴结转移、肝转移及不良预后相关[2];然而,另有学者却发现BUB1B高表达的胃癌患者无病生存率显著高于正常表达或低表达的患者[2]。
CDK1全称是细胞周期蛋白依赖性激酶,属于丝氨酸/苏氨酸蛋白激酶家族中的成员,在细胞周期进程中尤其是在G2/M期中具有重要调控作用,有报道认为CDK1可以作为一种评估预后的肿瘤标志物[1]。我们进行的生物信息系分析表明,CDK1可能参与了KIAA0101介导的细胞周期变化,这为CDK1与胃癌预后的相关机制提供了新的研究方向。
MAD2L1的功能是在有丝分裂染色体与纺锤体解离时维持染色体的分离状态,并在有丝分裂时的关卡点发挥作用[2-3]。失去正常调控的MAD2L1与染色体不稳定性和大量非整倍体出现有关,可导致肿瘤发生[1]。有研究发现,MAD2L1在肺腺癌组织过表达,并且,MAD2L1的过表达可能提示患者预后不良及肿瘤复发风险增加,可以作为判断肺腺癌预后的标志物[2]。在我们的生物信息学分析和RT-PCR实验中提示,MAD2L1可能与KIAA0101对胃癌细胞周期的调控有关,我们将对其具体机制进行进一步研究。
在癌细胞暴露在致癌物苯并[a]芘二氢化环氧化物的实验中,研究人员首次发现了CHK1对CDC45负载的独立调控作用[2]。有报道认为CHK1对Treslin,一种刺激CDC45负载的复制因子有负向调控作用[1]。因此,在KIAA0101引起的细胞周期变化中,可能同样存在CHK1对CDC45的调控作用,从而导致肿瘤细胞增殖、侵袭性的改变。但本研究未能发现癌组织中CDC45 mRNA水平显著高于配对癌旁组织,具体原因我们将在后续的实验中继续分析。
CCNE1编码细胞周期蛋白E1, 与肿瘤的进展有关。CCNE1在多种肿瘤中扩增或过表达,导致染色体不稳定,对其调控的丧失可能导致肿瘤的发生和发展[1-3]。CCNE1在细胞周期中的作用逐渐的得到重视,但在胃癌中的研究尚少,本研究发现癌组织中CCNE1水平显著升高,说明其可能与胃癌发病有密切关系,我们将继续探讨其在胃癌细胞周期改变中的作用。
CCNB2是启动有丝分裂的关键因素,可与CDK1结合形成复合物调控G2/M期,在多种肿瘤中,CCNB2均被检测出过表达并可以继发肿瘤细胞增殖失控。最终导致肿瘤进展及不良预后[1-2]。本研究结果与这些文献报道一致,本研究还发现CCNB2可能受到KIAA0101的调控,在胃癌细胞周期中发挥作用。这种作用关系可能是一种未知的调控机制,为KIAA0101对胃癌细胞周期的调控作用提供了新的研究方向。
在细胞周期方面,之前的研究在KIAA0101影响细胞周期的分布规律方面意见并不一致。一项研究认为,KIAA0101蛋白在S期和G2期表达量最丰富,主要作用于DNA损伤反应[1]。另一项研究则报道,抑制KIAA0101可以降低p21蛋白结合PCNA的比例和减少S期细胞数,诱导细胞G1期阻滞[4]。还有研究发现,KIAA0101 cDNA体外转染可抑制肝癌细胞生长并把细胞周期阻滞于G1-S期[5]。因此,KIAA0101对细胞周期的具体调控机制仍缺乏有说服力的研究。本研究利用生物信息学工具和分子生物学实验方法,筛选出了BUB1B、MAD2L1、CDK1、CCNE1、CCNB2等KIAA0101对细胞周期调控的可能相关基因,为KIAA0101影响细胞周期的作用机制、肿瘤标志物的筛选和药物靶点的选择提供了参考。
| [1] |
Kato T, Daigo Y, Aragaki M, et al. Overexpression of KIAA0101 predicts poor prognosis in primary lung cancer patients[J].
Lung Cancer, 2012, 75(1): 110-8.
DOI: 10.1016/j.lungcan.2011.05.024. |
| [2] |
Cheng Y, Li K, Diao D, et al. Expression of KIAA0101 protein is associated with poor survival of esophageal cancer patients and resistance to cisplatintreatment in vitro[J].
Lab Invest, 2013, 93(12): 1276-87.
DOI: 10.1038/labinvest.2013.124. |
| [3] |
Liu L1, Chen X, Xie S, et al. Variant 1 of KIAA0101, overexpressed in hepatocellular carcinoma, prevents doxorubicin-inducedapoptosis by inhibiting p53 activation[J].
Hepatology, 2012, 56(5): 1760-9.
DOI: 10.1002/hep.25834. |
| [4] |
Zhu K, Diao D, Dang C, et al. Elevated KIAA0101 expression is a marker of recurrence in human gastric cancer[J].
Cancer Sci, 2013, 104(3): 353-9.
DOI: 10.1111/cas.12083. |
| [5] |
Yuan D, Zhu K, Dang C, et al. NS5ATP9 mRNA levels in peripheral blood mononuclear cells predict prognosis in patients with gastric cancer[J].
Med Oncol, 2014, 31(8): 1-8.
|
| [6] |
Xie C, Yao M, Dong Q. Proliferating cell unclear antigen-associated factor (PAF15): a novel oncogene[J].
Int J Biochem Cell Biol, 2014, 50: 127-31.
DOI: 10.1016/j.biocel.2014.02.024. |
| [7] |
Povlsen LK, Beli P, Wagner SA, et al. Systems-wide analysis of ubiquitylation dynamics reveals a key role for PAF15 ubiquitylation in DNA-damage bypass[J].
Nat Cell Biol, 2012, 14(10): 1089-98.
DOI: 10.1038/ncb2579. |
| [8] |
Turchi L, Fareh M, Aberdam E, et al. ATF3 and p15PAF are novel gatekeepers of genomic integrity upon UV stress[J].
Cell Death Differ, 2009, 16(5): 728-37.
DOI: 10.1038/cdd.2009.2. |
| [9] |
Emanuele MJ, Ciccia A, Elia AE, et al. Proliferating cell nuclear antigen (PCNA)-associated KIAA0101/PAF15 protein is a cell cycle-regulated anaphase-promoting complex/cyclosome substrate[J].
Proc Natl Acad Sci USA, 2011, 108(24): 9845-50.
DOI: 10.1073/pnas.1106136108. |
| [10] |
Björck E, Ek S, Landgren O, J, et al. High expression of cyclin B1 predicts a favorable outcome in patients with follicular lymphoma[J].
Blood, 2005, 105(7): 2908-15.
DOI: 10.1182/blood-2004-07-2721. |
| [11] |
Lv W, Su B, Li Y, et al. KIAA0101 inhibition suppresses cell proliferation and cell cycle progression by promoting the interaction between p53 and Sp1 in breast cancer[J].
Biochem Biophys Res Commun, 2018, 503(2): 600-6.
DOI: 10.1016/j.bbrc.2018.06.046. |
| [12] |
张彩凤, 夏永华, 郑庆芬, 等. KIAA0101在胃癌细胞中的表达及其对胃癌MKN-45细胞增殖和侵袭力的影响[J].
中华病理学杂志, 2012, 41(8): 553-7.
DOI: 10.3760/cma.j.issn.0529-5807.2012.08.010. |
| [13] |
Grabsch H, Takeno S, Parsons WJ, et al. Overexpression of the mitotic checkpoint genes BUB1, BUBR1, and BUB3 in gastric cancer--association with tumour cell proliferation[J].
J Pathol, 2003, 200(1): 16-22.
|
| [14] |
Ando K, Kakeji Y, Kitao H, et al. High expression of BUBR1 is one of the factors for inducing DNA aneuploidy and progression in gastric cancer[J]. Cancer Sci, 2010(3), 101: 639-45.
http://www.ncbi.nlm.nih.gov/pubmed/20132214
|
| [15] |
Enjoji M, Iida S, Sugita H, et al. BubR1 and AURKB overexpression are associated with a favorable prognosis in gastric cancer[J].
Mol Med Report, 2009, 2(4): 589-96.
|
| [16] |
Roskoski R Jr. Cyclin-dependent protein kinase inhibitors includingpalbociclib as anticancer drugs[J].
Pharmacol Res, 2016, 107: 249-75.
DOI: 10.1016/j.phrs.2016.03.012. |
| [17] |
Wang ZW, Katsaros D, Shen Y, et al. Biological and clinical significance of MAD2L1 and BUB1, genes frequently appearing in expression signatures for breast cancer prognosis[J].
PLoS One, 2015, 10(8): e0136246.
DOI: 10.1371/journal.pone.0136246. |
| [18] |
Guo Y, Zhang X, Yang M, et al. Functional evaluation of missense variations in the human[J].
J Med Genet, 2010, 47(9): 616-22.
DOI: 10.1136/jmg.2009.074252. |
| [19] |
Li Y, Benezra R. Identification of a human mitotic checkpoint gene: hsMAD2[J].
Science, 1996, 274(5285): 246-8.
DOI: 10.1126/science.274.5285.246. |
| [20] |
Shi YX, Zhu T, Zou T, et al. Prognostic and predictive values of CDK1 and MAD2L1 in lung adenocarcinoma[J].
Oncotarget, 2016, 7(51): 85235-43.
|
| [21] |
Liu P, Barkley LR, Day T, et al. The Chk1- mediated S-phase checkpoint targets initiation factor Cdc45 via a Cdc25A/Cdk2-independent mechanism[J].
J Biol Chem, 2006, 281(41): 30631-44.
DOI: 10.1074/jbc.M602982200. |
| [22] |
Guo C, Kumagai A, Schlacher K, et al. Interaction of Chk1 with Treslin negatively regulates the initiation of chromosomal DNA replication[J].
Mol Cell, 2015, 57(3): 492-505.
DOI: 10.1016/j.molcel.2014.12.003. |
| [23] |
Lockwood WW, Stack D, Morris T, et al. Cyclin E1 is amplifi ed and overexpressed in osteosarcoma[J].
J Mol Diagn, 2011, 13(3): 289-96.
DOI: 10.1016/j.jmoldx.2010.11.020. |
| [24] |
Kuhn E, Bahadirli-Talbott A, Shih Ie M. Frequent CCNE1 amplifi cation in endometrial intraepithelial carcinoma and uterine serous carcinoma[J].
Mod Pathol, 2014, 27(7): 1014-9.
DOI: 10.1038/modpathol.2013.209. |
| [25] |
Wu S, Chen J, Dong P, et al. Global gene expression profiling identi fies ALDH2, CCNE1 and SMAD3 as potential prognostic markers in upper tract urothelial carcinoma[J].
BMC Cancer, 2014, 14: 836.
DOI: 10.1186/1471-2407-14-836. |
| [26] |
Lei CY, Wang W, Zhu YT, et al. The decrease of cyclin B2 expression inhibits invasion and metastasis of bladder cancer[J].
Urol Oncol, 2016, 34(5): 237.e1-10.
DOI: 10.1016/j.urolonc.2015.11.011. |
| [27] |
Nam HJ, van DeursenJM. Cyclin B2 and p53 control proper timing of centrosome separation[J].
Nat Cell Biol, 2014, 16(6): 538-49.
|
| [28] |
Kais Z, Barsky S H, Mathsyaraja H, et al. KIAA0101 interacts with BRCA1 and regulates centrosome number[J].
Mol Cancer Res, 2011, 9(8): 1091-9.
DOI: 10.1158/1541-7786.MCR-10-0503. |
| [29] |
Jain M, Zhang L, Patterson E E, et al. KIAA0101 is overexpressed, and promotes growth and invasion in adrenal cancer[J].
PLoS One, 2011, 6(11): e26866-26866.
DOI: 10.1371/journal.pone.0026866. |
| [30] |
Guo M, Li J, Wan D, et al. KIAA0101 (OEACT-1), an expressionally down-regulated and growth-inhibitory gene in human hepatocellular carcinoma[J].
BMC Cancer, 2006, 6: 109.
DOI: 10.1186/1471-2407-6-109. |
 2018, Vol. 38
2018, Vol. 38

