2. 新疆喀什地区第一人民医院肿瘤中心,新疆 喀什 844000
2. Cancer Center of the First People's Hospital of Kashi, Kashi 844000, China
据世界卫生组织统计,癌症是全球死亡的主因,而肺癌是癌症相关性死亡的最主要原因[1]。肺癌的发生发展基于多种遗传学和表观遗传学方面的变化,受到多个基因、因子的调控[2-3]。研究发现泛素-蛋白酶体系统(UPS)在肿瘤的发生、发展中发挥重要作用[4-6]。去泛素化酶是泛素化修饰过程中一种能逆转泛素化修饰的关键酶,它在肿瘤进展过程中的新功能、新机制一直是肿瘤研究中的重点。USP33是泛素特异性蛋白酶的一种,我们实验室采用免疫组化、Real time-PCR以及Western blot检测其在肺癌组织中表达情况,发现,在肺腺癌组织中USP33表达明显下调,对抑制肺癌的增殖和迁移侵袭的机制未进行研究,国内外均未见报道。本实验室以肺腺癌细胞系为研究对象,把USP33沉默以后,研究其对细胞增殖、迁移侵袭的影响以及通过SLIT2/ROBO1信号通路抑制肺腺癌细胞增殖和侵袭转移。
1 材料和方法 1.1 材料A549细胞购自美国典型培养物保藏中心(ATCC),SPC-A-1、H1299、PC9、人肺支气管正常上皮细胞16HB细胞由南方医院中心实验室馈赠。RPMI 1640培养基(Hyclone),四季青胎牛血清(Gibco),TRIzol Kit (Takara),SYBR Green PCR master mix (Roche);Lipofectamine 2000 reagents (Invitrogen),Opti-MEM(Invitrogen);PVDF膜(Bio-rad)一抗anti-USP33(Abcan);一抗anti-SLIT2 and ROBLO1(Cell Signaling Technolog),β- tubulin (Proteintech Group);二抗goat anti-rabbit(invitrogen);Transwell inserts(Corning),USP33的沉默序列(锐博生物),IL6刺激因子(selleck)。
1.2 方法 1.2.1 组织收集与提取2017年3月~6月在南方医院胸外科收集20对原发性肺腺癌Ⅰ期和Ⅱ期癌组织及其相应的癌旁组织标本。所有患者均未接受过放疗或化疗,本研究经南方医科大学南方医院伦理审查委员会批准。手术中取出所有标本,立即储存于-80 ℃中。
1.2.2 细胞培养所有细胞由10%胎牛血清的RPMI 1640培养基中培养。保持在37 ℃、饱和湿度及5% CO2的培养箱中传代培养。
1.2.3 USP33 RNA干扰序列的设计以及转染USP333条靶序列,3条靶序列si-h-USP33-001 CCTCAGAACATTTGGGATA si-USP33-002 CAGCTCAAATTGTGACATA si-h-USP33-003 GAGAAGATGTGCAATAAGA,阴性对照siRNA序列为锐博公司专有。将A549、SPC-A-1细胞按照20万/孔接种于6孔板上,过夜细胞贴壁后换液,参照Lipofectamine2000说明书的方法将各组siRNA序列分别转染至A549、SPC-A-1细胞中,终浓度达到100 nmol,同时以非特异性siRNA作为阴性对照组(NC组),每组设3个复孔,72 h后提取全蛋白,采用Western blot检测USP33的表达。
1.2.4 提取RNA和qPCR使用Trizol试剂盒根据制造商的说明从细胞系或组织提取总RNA。使用Takara RT试剂合成cDNA。使用SYBR Green PCR master mix试剂盒在Light Cycler 480系统上进行qRT-PCR实验。我们使用甘油醛-3-磷酸脱氢酶(GAPDH)作为内参。
1.2.5 Western blotting通过10% SDS-PAGE分离等量的蛋白质并印迹到用一抗兔抗人SLIT2、ROBO1、β- tubulin、兔抗人USP33和荧光山羊抗兔二抗的PVDF膜(Millipore)上。一抗4 ℃孵育过夜,荧光二抗室温下孵育1 h,Western blot检测系统测定目的蛋白的表达水平。
1.2.6 细胞划痕实验当细胞长到约90%汇合(48 h后)时,用10 μL移液器尖端产生人造伤口。将细胞在不含血清的新鲜培养基中培养。在0 h和36 h拍摄图像以观察伤口愈合。使用以下公式计算伤口愈合的相对百分比:(伤口宽度0 h-伤口宽度36 h)/伤口宽度0 h。
1.2.7 细胞迁移和侵袭检测使用transwell小室在24孔板中评估细胞的迁移和侵袭能力。对于侵袭测定,将每组细胞(5×104/100 μL)重悬浮于不含血清的RPMI 1640培养基中,并接种至含有基质胶涂覆的膜的上室中。在培养箱中孵育24 h后,用棉签从培养基的上表面除去孵育培养基和非侵入细胞。将粘附于腔室下表面的侵入细胞在4%多聚甲醛中固定20 min,并用0.1%结晶紫染色30 min。使用显微镜以200倍放大率对浸染细胞进行照相并手动计数。对于transwell迁移测定,过程是相同的。每个测定至少独立进行3次。
1.2.8 免疫组化按照烤片、脱蜡、切片、水化、抗原修复等步骤操作进行免疫组化,结果判定USP33阳性反应均为细胞和(或)胞浆深黄色或棕黄色染色,在400倍镜下随机挑取5个视野,采用半定量结果判断,分别对镜下阳性细胞的百分比和染色强度给予评分。免疫组化评分标准:染色深度共分为0分(无色)、1分(淡黄色)、2分(黄色)、3分(棕黄色),每种染色所占面积则依照0分(无细胞染色)、1分(25%染色)、2分(50%染色)、3分(75%染色)及4分(100%染色)。然后染色深度评分和对应的染色面积评分相乘后相加,既得到了最后的染色评分。其中0~2分定义为阴性表达,3~7分定义为弱阳性表达,8~12分定义为强阳性表达。
1.3 统计学分析采用SPSS 22.0统计软件进行数据分析,实验数据中正态分布的计量资料以均数±标准差表示,组间比较使用t检验,P < 0.05认为差异具有统计学意义。
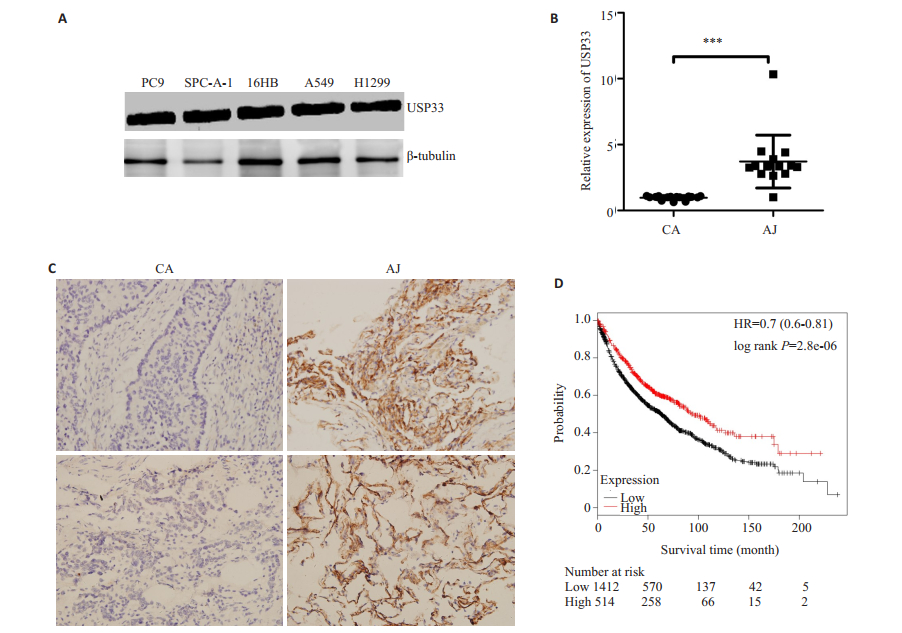
2 结果 2.1 USP33在非小细胞肺癌组织和细胞株中的表达,并与生存预后相关与16HBE正常肺上皮细胞相比,USP33在A549,SPC-A-1,H1299和PC9肺癌细胞系中呈不同程度表达下调(图 1A),USP33在SCP-A-1细胞系中表达下调最明显而在A549细胞系下调程度最小。qRT-PCR结果显示癌旁组织中USP33的平均表达水平比相对应的癌组织高(P < 0.001,图 1B)。利用免疫组化分析了10例肺腺癌患者癌组织和匹配的癌旁组织USP33的表达,试验结果与qPCR结果一致,10例癌组织中USP33都被证明是低表达。无倾向任意选取其中两例作为代表,癌组织0分,为阴性表达,而癌旁为强阳性表达(图 1C)。同时,这与我们通过TCGA数据库分析USP33生存预后相一致,USP33高表达生存预后较好(图 1D)。
|
图 1 USP33在非小细胞肺癌组织和细胞株中的表达与生存预后的关系 Figure 1 Expression of USP33 in NSCLC tissues and cell lines and its association with the survival and prognosis of the patients. A: The expression levels of USP33 were analyzed in various lung cancer cell lines. B: The expression of miR-365 in lung adenocarcinoma tissues. C: Immunohistochemistry showed that USP33 was lowly expressed in lung adenocarcinoma (Original magnification: ×400). D: Survival analysis. ***P < 0.001. |
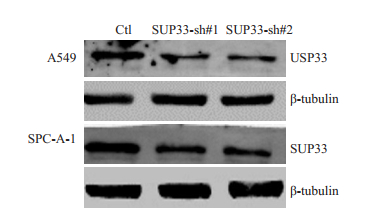
USP33-si-1、USP33-si-2转染可显著下调A549、SPC-A-1细胞中USP33蛋白的表达水平(P < 0.05),说明沉默序列可以抑制USP33的表达(图 2)。
|
图 2 沉默序列转染A549、SPC-A-1细胞系后USP33的表达 Figure 2 Expression of USP33 after transfection in A549 and SPC-A-1 cells. |
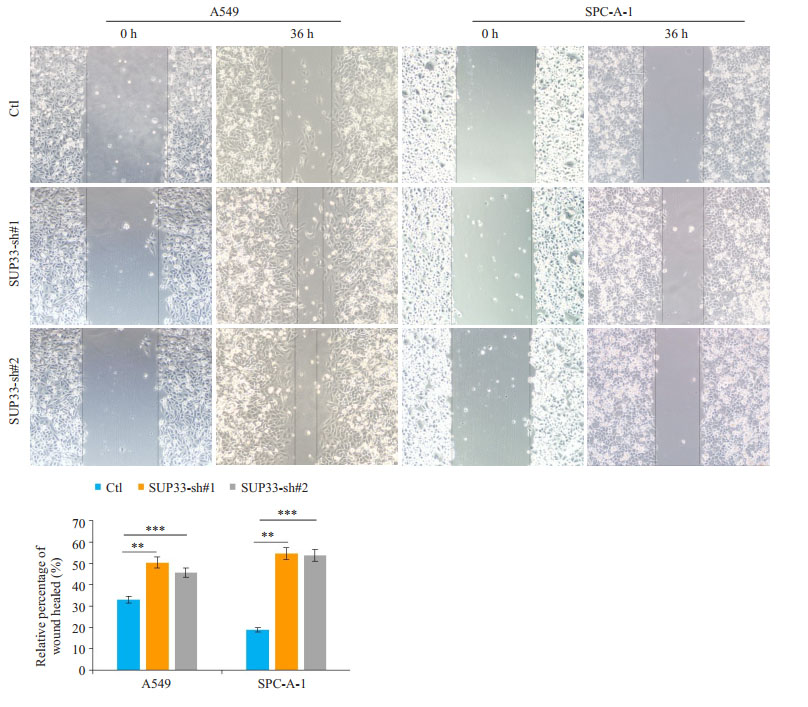
细胞划痕实验结果显示,USP33沉默后可明显促进A549、SPC-A-1细胞的迁移,相较于阴性对照组,差异具有统计学意义(P < 0.05,图 3)。
|
图 3 USP33沉默后对肺癌细胞迁移的影响 Figure 3 Wound healing assay of A549 and SPC-A-1 cells transfected with USP33-si-1, USP33-si-2 or the control siRNA. All experiments were performed in triplicate and repeated for 3 times. **P < 0.01, ***P < 0.001 vs control. |
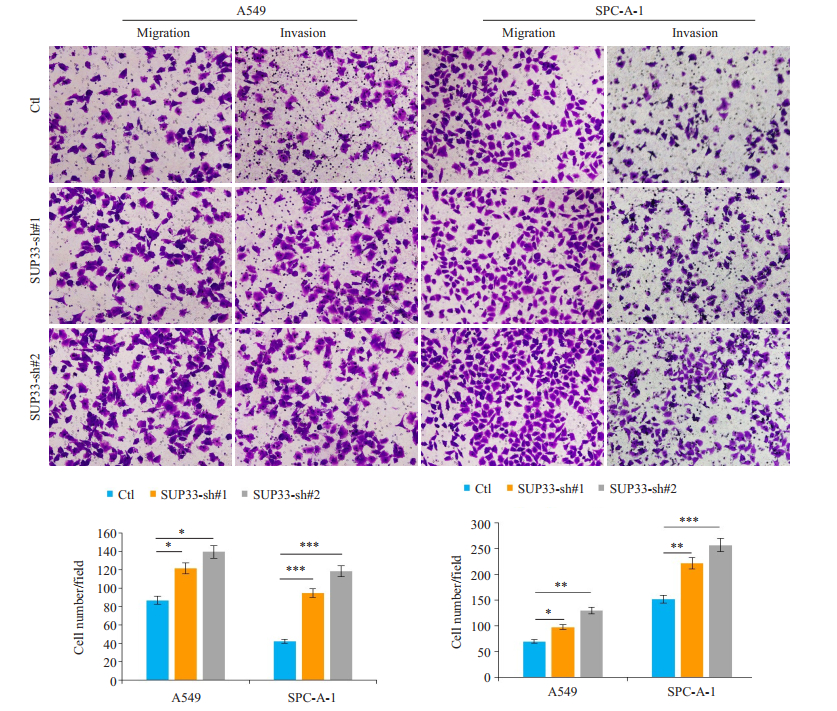
Transwell迁移和侵袭实验结果显示,USP33沉默后A549和SPC-A-1细胞迁移和侵袭力明显增加,与阴性对照组相比,结果均具有显说明USP33可抑制肺癌细胞的迁移和侵袭(图 4)。
|
图 4 USP33沉默后对肺癌细胞迁移侵袭的影响 Figure 4 Transwell migration and invasion assay of A549 and SPC-A-1 cells transfected with USP33-si-1, USP33-si-2 or the control siRNA. *P < 0.05, **P < 0.01, ***P < 0.001 vs control. |
与阴性对照组相比,在A549和SPC-A-1细胞中沉默USP33后SLIT2和ROBO1蛋白表达均下调(图 5)。

|
图 5 Western blot检测SLIT2/ROBO1通路蛋白的变化 Figure 5 Western blotting for detecting SLIT2 and ROBO1 expressions in A549 and SPC-A-1 cells following USP33 silencing. |
侵袭转移是肺癌难以根治的主要原因,探讨肺癌转移机理并找到阻断转移的方法,是目前国际医学界研究的热点[7-8]。去泛素化酶在肿瘤进展中起到的作用逐步受到重视,其调控的分子机制也逐步明确,已有超过40种去泛素化酶被发现与肿瘤的发生、发展有关[9-16]。如USP22可以与目标底物相互结合,水解相应的多聚或单泛素标签,从而稳定目标底物蛋白或是影响底物蛋白对下游分子的调节作用。目前已经在不同的细胞中发现了并证实了数种能与USP22直接相互作用的目标底物蛋白,包括Sirt1、TRF1、FUBP1、RCAN1、NFATc2以及AR等[17-22]。在临床应用方面,硼替佐米是第一个被临床认可的UPS抑制剂,用于治疗B细胞淋巴瘤及多发性骨髓瘤,是UPS作为治疗靶点的重要范例[23]。另一个蛋白酶体抑制剂卡非佐米在2012年被FDA批准用于多发性骨髓瘤的治疗[24]。而对于USP33的研究,目前研究较少,USP33可通过与CP110在细胞周期S/G2及G2/M期的相互作用,维持CP110的稳定,拮抗cyclin-F的作用,维持中心粒的稳态,保证细胞有丝分裂的正常进行和基因组稳定性,减少肿瘤发生[9]。
我们通过免疫组化发现USP33在肺腺癌组织和细胞中低表达,TCGA数据库分析与肺癌患者生存预后紧密相关,通过功能试验验证了USP33可抑制肺癌细胞的迁移侵袭的能力,进而,我们又提出了针对USP33通过什么机制抑制肺腺细胞迁移侵袭进行了解答。
SLIT/ROBO信号通路较早是在中枢神经系统中进行研究的,其在中枢神经系统中有抑制神经细胞迁移,阻止合缝处神经轴突穿过神经管中线,控制神经轴突在神经系统中准确定位的作用。Prasad等[25]在几种乳腺癌细胞系中发现有ROBO1、ROBO2的表达,并且SLIT2可抑制CXCL12/CXCR 4诱导的乳腺癌细胞趋化、侵袭、粘附作用以及MMP-2和MMP-9的分泌, 表明SLIT/ROBO信号能够抑制乳腺癌细胞迁移。SLIT/ ROBO在卵巢癌细胞系可通过激活半胱天冬酶-3诱导程序性细胞凋亡,SLIT2、SLIT3或ROBO1蛋白在小鼠乳腺癌模型中的缺失导致组织损伤修复增加,细胞增殖数量增加,刺激上皮中SDF1-CXCR4轴,表明SLIT/ROBO信号能够抑制卵巢癌细胞、乳腺癌细胞增殖[26-29]。以上说明SLIT/ROBO信号通路可抑制肿瘤细胞迁移侵袭。我们发现沉默USP33后SLIT2、ROBO1表达下调,结合以上体外试验结果,我们得出结论,USP33在肺腺癌组织和肺癌细胞系中表达下调,抑制USP33可促进肺癌细胞增迁移侵袭的能力。USP33作为抑癌基因很大可能参与到SLIT2/ROBO1信号通路,至于如何参与到SLIT2/ROBO1信号通路对肺癌的发生发展产生的影响,有待后续研究证明。同时,为了验证USP33的功能,在实验中我们用多种刺激因子刺激USP33,利用WB和qPCR实验,观察A549和SPC-A-1细胞,发现IL6刺激48 h后,IL6刺激组USP33蛋白的表达量明显增加。以上说明IL6在肺癌的发生发展中与USP33密切相关。已知在NSCLC细胞增殖、侵袭、迁移、血管形成中炎症因子IL-6均发挥作用[30-32]。作为功能因子IL-6与USP33的是如何相互发挥作用,需更为完整的机制进一步证实。
| [1] |
World Health Organization (WHO). GLOBOCAN 2012: estimated cancer incidence, mortality and prevalence world wide in 2012[C], 2018.
https://www.mendeley.com/research-papers/globocan-2012-estimated-cancer-incidence-mortality-prevalence-worldwide-2012/
|
| [2] |
Lagos-Quintana M, Rauhut R, Lendeckel W, et al. Identification of novel genes coding for small expressed RNAs[J].
Science, 2001, 294(5543): 853-8.
DOI: 10.1126/science.1064921. |
| [3] |
Siegel R, Naishadham D, Jemal A. Cancer statistics, 2013[J].
CA Cancer J Clin, 2013, 63(1): 11-30.
DOI: 10.3322/caac.v63.1. |
| [4] |
Ashley EA. The precision medicine initiative: a new National effort[J].
JAMA, 2015, 313(21): 2119-20.
DOI: 10.1001/jama.2015.3595. |
| [5] |
Chen SC, Lin MC, Chang JW, et al. Phase Ⅱ study of regimen of gemcitabine and cisplatin in advanced non-small cell lung cancer[J].
Jpn J Clin Oncol, 2000, 30(11): 494-8.
DOI: 10.1093/jjco/hyd123. |
| [6] |
Yuasa-Kawada J, Kinoshita-Kawada M, Rao Y, et al. Deubiquitinating enzyme USP33/VDU1 is required for Slit signaling in inhibiting breast cancer cell migration[J].
Proc Natl Acad Sci USA, 2009, 106(34): 14530-5.
DOI: 10.1073/pnas.0801262106. |
| [7] |
Werbowetski-Ogilvie TE, Seyed Sadr M, Jabado N, et al. Inhibition of medulloblastoma cell invasion by Slit[J].
Oncogene, 2006, 25(37): 5103-12.
DOI: 10.1038/sj.onc.1209524. |
| [8] |
Sozzi G, Pastorino U, Croce CM. MicroRNAs and lung cancer: from markers to targets[J].
Cell Cycle, 2011, 10(13): 2045-6.
DOI: 10.4161/cc.10.13.15712. |
| [9] |
Simicek M, Lievens S, Laga M, et al. The deubiquitylase USP33 discriminates between RALB functions in autophagy and innate immune response[J].
Nat Cell Biol, 2013, 15(10): 1220-30.
DOI: 10.1038/ncb2847. |
| [10] |
Li J, D'angiolella V, Seeley ES, et al. USP33 regulates centrosome biogenesis via deubiquitination of the centriolar protein CP110[J].
Nature, 2013, 495(7440): 255-9.
DOI: 10.1038/nature11941. |
| [11] |
Fraile JM, Manchado E, Lujambio A, et al. USP39 deubiquitinase is essential for KRAS oncogene-driven cancer[J].
J Biol Chem, 2017, 292(10): 4164-75.
DOI: 10.1074/jbc.M116.762757. |
| [12] |
Fraile JM, Campos-Iglesias D, Rodríguez F, et al. The deubiquitinase USP54 is overexpressed in colorectal cancer stem cells and promotes intestinal tumorigenesis[J].
Oncotarget, 2016, 7(46): 74427-34.
|
| [13] |
Wu Y, Wang Y, Yang XH, et al. The deubiquitinase USP28 stabilizes LSD1 and confers stem-cell-like traits to breast cancer cells[J].
Cell Rep, 2013, 5(1): 224-36.
DOI: 10.1016/j.celrep.2013.08.030. |
| [14] |
He M, Zhou Z, Shah AA, et al. The emerging role of deubiquitinating enzymes in genomic integrity, diseases, and therapeutics[J].
Cell Biosci, 2016, 6: 62.
DOI: 10.1186/s13578-016-0127-1. |
| [15] |
Fraile JM, Quesada V, Rodriguez D, et al. Deubiquitinases in cancer: new functions and therapeutic options[J].
Oncogene, 2012, 31(19): 2373-88.
DOI: 10.1038/onc.2011.443. |
| [16] |
Suresh B, Lee J, Kim KS, et al. The importance of ubiquitination and deubiquitination in cellular reprogramming[J].
Stem Cells Int, 2016, 2016: 6705927.
|
| [17] |
Armour SM, Bennett EJ, Braun CR, et al. A high-confidence interaction map identifies SIRT1 as a mediator of acetylation of USP22 and the SAGA coactivator complex[J].
Mol Cell Biol, 2013, 33(8): 1487-502.
DOI: 10.1128/MCB.00971-12. |
| [18] |
Atanassov BS, Evrard YA, Multani AS, et al. Gcn5 and SAGA regulate shelterin protein turnover and telomere maintenance[J].
Mol Cell, 2009, 35(3): 352-64.
DOI: 10.1016/j.molcel.2009.06.015. |
| [19] |
Atanassov BS, Dent SY. USP22 regulates cell proliferation by deubiquitinating the transcriptional regulator FBP1[J].
EMBO Rep, 2011, 12(9): 924-30.
DOI: 10.1038/embor.2011.140. |
| [20] |
Hong A, Lee JE, Chung KC. Ubiquitin-specific protease 22 (USP22) positively regulates RCAN1 protein levels through RCAN1 de-ubiquitination[J].
J Cell Physiol, 2015, 230(7): 1651-60.
DOI: 10.1002/jcp.24917. |
| [21] |
Gao Y, Lin F, Xu P, et al. USP22 is a positive regulator of NFATc2 on promoting IL2 expression[J].
FEBS Lett, 2014, 588(6): 878-83.
DOI: 10.1016/j.febslet.2014.02.016. |
| [22] |
Schrecengost RS, Dean JL, Goodwin JF, et al. USP22 regulates oncogenic signaling pathways to drive lethal cancer progression[J].
Cancer Res, 2014, 74(1): 272-86.
DOI: 10.1158/0008-5472.CAN-13-1954. |
| [23] |
Bold R. "Development of the proteasome inhibitor Velcade (Bortezomib) " by Julian Adams, Ph.D, and Michael Kauffman, M.D., Ph.D[J].
Cancer Invest, 2004, 22(2): 328-9.
DOI: 10.1081/CNV-120030223. |
| [24] |
Steele JM. Carfilzomib: a new proteasome inhibitor for relapsed or refractory multiple myeloma[J].
J Oncol Pharm Pract, 2013, 19(4): 348-54.
DOI: 10.1177/1078155212470388. |
| [25] |
Prasad A, Fernandis AZ, Rao Y, et al. Slit protein-mediated inhibition of CXCR4-induced chemotactic and chemoinvasive signaling pathways in breast cancer cells[J].
J Biol Chem, 2004, 279(10): 9115-24.
DOI: 10.1074/jbc.M308083200. |
| [26] |
Marlow R, Strickland P, Lee JS, et al. SLITs suppress tumor growth in vivo by silencing Sdf1/Cxcr4 within breast epithelium[J].
Cancer Res, 2008, 68(19): 7819-27.
DOI: 10.1158/0008-5472.CAN-08-1357. |
| [27] |
Wang LJ, Zhao Y, Han B, et al. Targeting Slit-Roundabout signaling inhibits tumor angiogenesis in chemical-induced squamous cell carcinogenesis[J].
Cancer Sci, 2008, 99(3): 510-7.
DOI: 10.1111/cas.2008.99.issue-3. |
| [28] |
Mertsch S, Schmitz N, Jeibmann A, et al. Senner V. Slit2 involvement in glioma cell migration is mediated by Robo1 receptor[J].
J Neurooncol, 2008, 87(1): 1-7.
DOI: 10.1007/s11060-007-9484-2. |
| [29] |
Huang Z, Wen P, Kong R, et al. USP33 mediates Slit-Robo signaling in inhibiting colorectal cancer cell migration[J].
Int J Cancer, 2015, 136(8): 1792-802.
DOI: 10.1002/ijc.29226. |
| [30] |
Kris MG, Johnson BE, Berry LD, et al. Using multiplexed assays of oncogenic drivers in lung cancers to select targeted drugs[J].
JAMA, 2014, 311(19): 1998-2006.
DOI: 10.1001/jama.2014.3741. |
| [31] |
Zarogoulidis P, Yarmus L, Darwiche K, et al. Interleukin-6 cytokine: a multifunctional glycoprotein for cancer[J].
Immunome Res, 2013, 9(62): 16535.
|
| [32] |
De Pittà C, Tombolan L, Campo Dell'orto M, et al. A leukemia[J].
Haematologica, 2005, 90(7): 890-8.
|
 2018, Vol. 38
2018, Vol. 38

