2. 南方医科大学 南方医院妇产科,广东 广州 510515;
3. 南方医科大学 基础医学院生物化学与分子生物学教研室,广东 广州 510515
2. Department of Gynecology and Obstetrics, Nanfang Hospital, Southern Medical University, Guangzhou 510515, China;
3. Department of Biochemistry and Molecular Biology, School of Basic Medical Sciences, Southern Medical University, Guangzhou 510515, China
宫颈癌是常见的女性生殖系统恶性肿瘤,其发病率和死亡率在全球范围内分列第3位和第4位[1]。尽管宫颈癌筛査的普及和针对性疫苗的推广应用使其发生率在发达地区显著下降;但在欠发达地区,由于筛査的不规范、疫苗价格昂贵,其发生率仍居高不下,且近年来呈发病年轻化的趋向[2]。微小RNA (miRNA)通常在转录后水平上调控基因表达,其与肿瘤的关系备受关注。因此,揭示miRNA在宫颈癌发生发展中的作用及机制,将有助于我们更准确地认识、理解和完善宫颈癌的致癌机理,并有望为宫颈癌的分子诊断、靶向治疗以及预后评价提供科学依据。
miR-519d定位于人第19号染色体19q13.41区,是最大的miRNA基因簇C19MC所在位置,最早发现它能引起脂肪酸代谢紊乱,与肥胖有关[3]。我们在前期研究中发现,miR-519d与宫颈癌细胞周期调控相关[4],还能通过影响TGF-β信号通路的关键分子Smad7,调控宫颈癌侵袭和转移[5]。研究发现,miR-519d参与调控肝癌、乳腺癌、肺癌、卵巢癌、结直肠癌、黑色素瘤等[6-16]肿瘤的发生发展,但其在肿瘤中究竟是发挥癌基因,还是抑癌基因的作用,报道并不一致。且最新研究提示miR-519d在宫颈癌中具有抑癌效应,与我们前期结果相反[17]。为此,本研究利用基因芯片技术分析miR-519d过表达前后引起宫颈癌SiHa细胞基因表达谱的变化,为更深入了探讨miR-519d在宫颈癌发生中的作用及机制提供了重要的实验依据。
1 材料和方法 1.1 试剂宫颈癌SiHa细胞(中国科学院上海细胞库),Lipofectamine 2000、DMEM培养基(高糖)、胎牛血清、Opti-MEM培养基、SuperscriptⅡ逆转录试剂盒、双链cDNA合成试剂盒(Invitrogen),Roche NimbleGen 12× 135K人全基因组表达谱芯片、单色DNA标记试剂盒、芯片杂交试剂盒、洗涤缓冲试剂盒(NimbleGen),RNeasy mini试剂盒(Qiagen),荧光定量PCR试剂盒(Takara)。miR-519d mimics及negative control(NC)均为购自上海吉玛制药技术有限公司,其序列分别为:NC:5'-UUCUCCGAACGUGUCACGUTT-3'(forward),5'- ACGUGACACGUUCGGAGAATT-3'(reverse);miR- 519d mimics:5'-CAAAGUGCCUCCCUUUAGAGUG- 3'(forward),5'-CUCUAAAGGGAGGCACUUUGUU 3'(reverse)。荧光定量PCR引物由华大基因合成。
1.2 仪器ND-1000紫外分光光度计、琼脂糖凝胶电泳系统(NanoDrop),NimbleGen芯片杂交系统(NimbleGen),GenePix 4000B扫描仪、GenePix Pro V6.0(Axon公司),ABI 7500荧光定量PCR仪(ABI)。
1.3 细胞培养与转染宫颈癌SiHa细胞培养于10%胎牛血清的高糖DMEM培养液,于37 ℃、5% CO2条件下培养。每2~3d换液传代培养使细胞维持对数生长,至70%~80%时进行转染实验。细胞分2组:实验组(miR-519d mimic)、阴性对照组(NC)。转染过程如下:将小分子RNA稀释于200 μL Opti-MEM中,轻敲管壁混匀;每管加入5 μL转染试剂,轻敲管壁混匀,室温放置20 min,将RNA/ Lipofectamine复合物加入2 mL的细胞稀释液,混匀后加入细胞培养板(使细胞密度在转染后24 h大约为30% ~50%),培养48 h后收集细胞。
1.4 芯片检测与分析转染48 h收集实验组和对照组细胞,由上海康成生物公司进行基因芯片检测和数据分析。具体如下:(1)总RNA提取和质控:Trizol法抽提样本总RNA,NanoDrop ND-1000检测其浓度与纯度,甲醛变性凝胶电泳评估总RNA的完整性;(2)基因表达谱芯片检测:采用包含45033个基因的Roche NimbleGen 12×135K人全基因组表达谱芯片;(3)荧光标记和芯片杂交:将总RNA逆转录成双链cDNA,用配套试剂纯化后NimbleGen One-Color DNA labelling kit进行Cy3荧光标记,进一步与芯片杂交,杂交后用NimbleGen Wash Buffer kit洗涤,通过Axon GenePix 4000B芯片扫描仪采集信号,GenePix Pro 6.0软件进行图像分析、数据采集,采用RMA法对实验数据进行标准化;(4)数据分析:校正后的数据通过Agilent GeneSpring GX software(version 11.5.1)进一步分析,通过Volcano Plot筛选出显著差异表达基因(上调或下调倍数≥2.0,P < 0.05),Hierarchical clustering比较样本间差异基因表达水平。
1.5 候选靶基因的筛选及生物信息学分析将芯片筛选得到的下调基因与TargetScan 6.2软件预测的miR-519d的潜在靶基因取交集,获得miR-519d的候选靶基因。对候选靶基因进行生物信息学分析,基于Gene Ontology数据库、KEGG数据库进行GO和Pathway分析基因相关功能及生物学通路。进一步通过STRING数据库[18](http://string.embl.de)分析候选靶蛋白间的可能作用关系,构建相互作用网络,并找出重要节点蛋白;pSTIING[19](http://pstiing.licr.org)对候选基因进行转录相关和物理相关关系预测,找出重要节点基因。
1.6 实时荧光定量PCR将实验组、阴性对照组细胞提取总RNA并逆转录为cDNA,以U6和β-actin为内参分别进行qRT-PCR,验证miR-519d及差异基因的表达情况。引物见表 1,反应体系如下:Mix 10 μL,上下游引物各0.5 μL,RNase-free water 7 μL,cDNA 2 μL(共计20 μL)。扩增条件为:95 ℃ 10 min;95 ℃ 15 s,60 ℃ 1 min,40个循环。结果采用2-∆∆Ct法计算相对表达量。
| 表 1 荧光定量PCR引物序列 Table 1 Sequence of the primers for qRT-PCR |
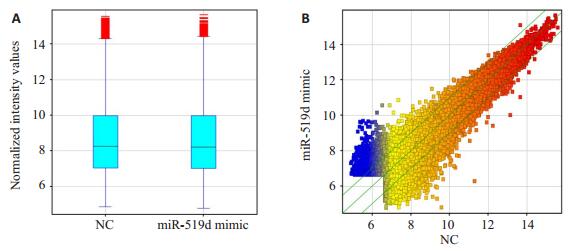
使用Axon GenePix 4000B芯片扫描仪对miR-519d转染组和NC对照组表达谱芯片的荧光强度进行扫描。其荧光信号扫描图结果显示,芯片杂交信号强度高且均一,基因点清晰、均衡,说明该基因表达谱芯片实验总体结果理想。此外,芯片数据标准化后作Box-Plot图提示两组样本的标准化信号值(log2形式)比率分布基本上是相同的,未见明显差异(图 1A)。如图 1B所示,图中点的分布大部分集中在上下两条绿色线之间及周围,但仍有部分点的分布远离这两条绿色线。
|
图 1 两组表达谱芯片的箱形图和散点图 Figure 1 Box plot (A) and scatter plot (B) for gene expression profiles derived from the microarray data of cells transfected with miR-519d mimic and negative control (NC). |
应用Roche NimbleGen芯片比较了miR-519d转染前后基因表达谱的变化。结果发现,与NC组相比,共筛选到差异表达基因5172条,其中mimc组中表达上调基因2476条,表达下调基因2796条。随后,将芯片筛查得到的显著下调基因与TargetScan 6.2软件预测的miR-519d潜在靶基因做交集,发现了164个候选靶基因,作为miR-519d功能研究的候选分子。
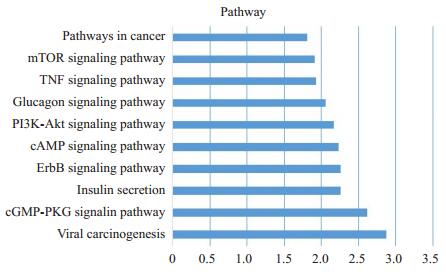
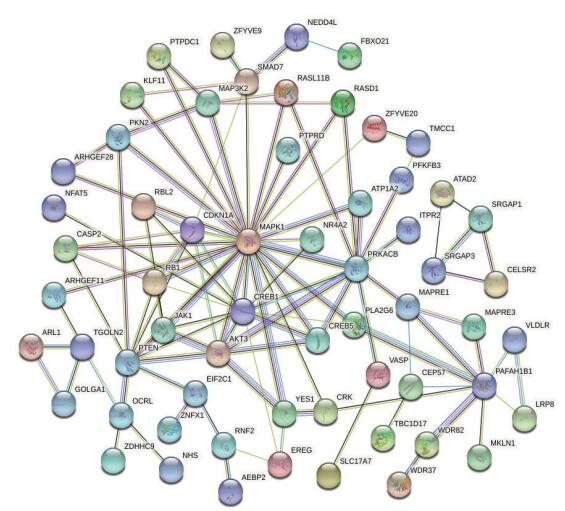
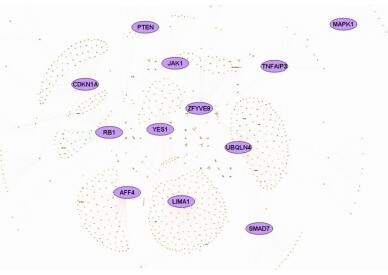
2.3 候选基因的生物信息学分析结果KEGG信号通路分析提示miR-519d的功能可能与病毒致癌、肿瘤发生、PI3K-AKT等信号通路密切相关(图 2)。进一步通过STRING软件构建候选靶蛋白间的相作关系,发现了一个复杂的多中心差异表达基因互作网络,处于网络中心位置的一些节点,除了与网络周边节点间的相互作用外,与其他网络中心节点间也存在广泛的互作关系。蛋白相互作用网络主要集中于CDKN1A、RB1、RBL2、PTEN、MAPK1、YES1、JAK1、AKT3等重要节点蛋白(图 3)。采用pSTIING对候选基因进行转录相关和物理相关关系预测,共涉及979边,915个结点。其中的重要节点包括CDKN1A、RB1、AFF4、LIMA1、YES1、PTEN、JAK1等,与STRING软件结果较为一致(图 4)。图中蓝色的线条表达基因之间的相互作用关系已经有实验证实;红色的线条代表两基因间存在相关但尚未有实验证实;虚线表示两基因间存在转录相关作用。
|
图 2 候选靶基因的信号通路分析结果 Figure 2 Results of KEGG pathway analysis (Top ten pathways are listed according to -lgP). |
|
图 3 STRING软件构建候选基因编码蛋白间的互作关系 Figure 3 Networks of the protein-protein interactions encoded by these differential genes using STRING database. |
|
图 4 pSTIING对候选基因进行转录相关和物理相关关系预测 Figure 4 Interaction and transcriptional regulatory networks in cancer predicted using pSTIING. |
我们采用qRT-PCR技术在宫颈癌SiHa细胞中验证了14个候选靶基因的表达情况,其中12个与芯片结果基本一致(图 5、表 2)。
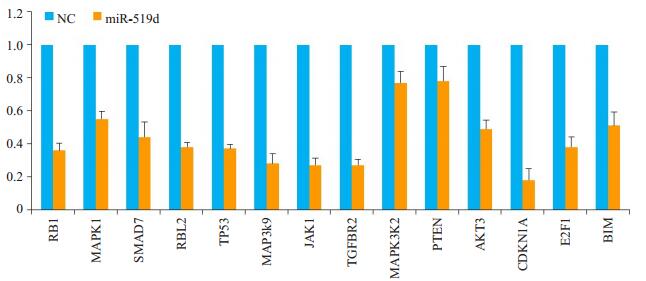
|
图 5 荧光定量PCR验证部分芯片结果 Figure 5 Part of the results for microarray was validated by qRT-PCR. |
| 表 2 荧光定量PCR结果与芯片结果列表 Table 2 Results for microarray and qRT-PCR experiments |
宫颈癌的发生、发展和转归是一个极其复杂的多阶段、多基因调控异常的过程,从上皮增生到化生、不典型增生、原位癌、浸润癌和最后转移,涉及到多种编码基因与非编码基因结构与表达异常[20-21]。尽管高危HPV感染是宫颈癌发生的必要条件,但是单纯的HPV感染并不足以诱导宫颈癌的发生,提示必然有其他未知因素的协同作用[22]。miRNAs作为一类非编码RNA分子,它们不仅参与了细胞凋亡、代谢、分化、激素分泌等生理过程,也在肿瘤发生发展等病理过程发挥重要作用,并在感染性疾病中与病毒的感染、清除、复制等过程密切相关[23-24]。研究人员采用高通量检测技术证明了某些miRNA在宫颈癌发生发展过程中发挥着十分重要的作用,且其中部分miRNA与HPV感染之间存在着密切的关系[25-29]。前期的研究中,我们已经报道了miR-519d的表达在HeLa细胞中具有周期依赖性,其表达水平的周期性的波动很可能与细胞周期调控有关[1]。临床标本中,我们发现miR-519d在60%左右宫颈癌临床组织中呈高表达。然而,当前有关miR-519d在肿瘤中究竟是发挥癌基因,还是抑癌基因的作用,以及其具体的分子机制仍需进一步探讨。Hou等[7]研究发现,miR-519d在肝癌组织中的表达较癌旁正常组织降低,它能通过靶向MKi67而抑制肝癌细胞的生长与增殖,具有抑癌基因的作用。Fornari等[8]发现miR-519d在肝癌组织中表达升高,通过靶向p21、PTEN、AKT3、TIMP2而促进肝癌细胞的增殖、侵袭,抑制抗癌药物诱导的细胞凋亡。肺癌A549中,miR-519d通过抑制TGF-β信号通路而促进细胞增殖[9]。近年来的不少研究分别报道了miR-519d在胃癌[10]、结肠癌[11]、乳腺癌[30]及肺腺癌[31]中的抑癌基因功能,然而它又被证实能够促进黑色素瘤的进展,发挥癌基因的作用[16]。因此,深入研究其在不同肿瘤发生发展中的作用机理,将有助于我们更准确地认识、理解和完善相关肿瘤的致病机理,并有望为肿瘤的分子诊断、靶向治疗以及预后评价提供新的线索和科学依据。
在本研究中,我们采用包含45033个人类基因的Roche NimbleGen全基因组表达谱芯片对miR-519d转染前后宫颈癌SiHa细胞的基因表达谱进行了分析,共筛选到差异表达基因5172条,其中表达上调基因2476条,表达下调基因2796条。上述基因的发现以及对差异表达基因的生物信息学分析,将有助于进一步了解miR-519d在宫颈癌中的具体功能。众所周知,miRNA在生物体内发挥作用主要是通过其下游靶基因而实现,因此,对于miRNA分子机制研究的主要工作之一便是揭示其具体作用的直接靶基因。然而,每个miRNA可能靶向的下游分子至少包括100个以上,而生物信息学预测的靶基因往往数量众多,给后续研究工作造成了一定的困扰。为此,我们将高通量的表达谱芯片技术和生物信息学软件预测相结合,将芯片筛查得到的显著下调基因与TargetScan 6.2软件预测的miR-519d潜在靶基因做交集,发现了164个候选靶基因,作为miR-519d功能研究的候选分子,这在一定程度上缩小了靶基因研究的范围。进一步我们对于上述候选靶基因进行了生物信息学分析,以便更好地理解miR-519d作用的分子机制。KEGG信号通路分析提示其功能可能涉及病毒致癌、肿瘤发生、PI3K-AKT等信号通路。随后,我们采用STRING软件构建候选靶蛋白间的互作关系,pSTIING对候选基因进行转录相关和物理相关关系预测,发现了包括CDKN1A、RB1、RBL2、SMAD7、PTEN、MAPK1、YES1、JAK1等重要节点,这为后续开展miR-519d的功能研究及靶基因的筛选奠定了重要的基础。此外,我们还通过qRT-PCR技术对部分重要节点基因及随机选择的14个基因的表达水平进行了验证,与芯片结果进行了比较,结果显示其中12个基因的表达情况基本一致,充分说明了芯片结果具有较好的稳定性。
miRNA可调控与细胞增殖、凋亡和分化等重要的生物学过程相关的靶基因,而在肿瘤发生发展过程中发挥至关重要的作用。然而,miRNA参与基因表达调控的方式十分复杂,往往是多基因共同调控的最终结果。当前对于miRNA功能机制研究的重要工作之一仍是对靶基因的鉴定。我们通过高通量的表达谱芯片技术筛选出2796条下调基因,它们可能是miR-519d直接或间接调控的靶分子,是其在转录及转录后水平上调控基因表达的重要基础。传统研究模式通过一次实验只能确定单个或几个靶基因,但本研究将miR-519d模拟物转染宫颈癌SiHa细胞,并从全基因组水平上分析其对于真核生物细胞基因表达的影响,同时综合生物信息学软件预测结果,获得了164个候选靶基因,这为下一步深入了解miR-519d的功能及其具体的作用机制提供了重要的线索和依据。这种将高通量的表达谱分析与生物信息学预测相结合的研究模式将有助于提升靶基因鉴定的效率,同时也为系统、全面地开展miRNA靶基因研究提供了一种新思路。
| [1] |
Torre LA, Bray F, Siegel RL, et al. Global cancer statistics, 2012[J].
CA Cancer J Clin, 2015, 65(2): 87-108.
DOI: 10.3322/caac.21262. |
| [2] |
Small W, Bacon MA, Bajaj A, et al. Cervical cancer: A global health crisis[J].
Cancer, 2017, 123(13): 2404-12.
DOI: 10.1002/cncr.v123.13. |
| [3] |
Martinelli R, Nardelli C, Pilone V, et al. miR-519d overexpression is associated with human obesity[J].
Obesity (Silver Spring), 2010, 18(11): 2170-6.
DOI: 10.1038/oby.2009.474. |
| [4] |
Zhou JY, Ma WL, Liang S, et al. Analysis of microRNA expression profiles during the cell cycle in synchronized HeLa cells[J].
BMB Rep, 2009, 42(9): 593-8.
DOI: 10.5483/BMBRep.2009.42.9.593. |
| [5] |
Zhou JY, Zheng SR, Liu J, et al. MiR-519d facilitates the progression and metastasis of cervical cancer through direct targeting Smad7[J].
Cancer Cell Int, 2016, 16: 21.
DOI: 10.1186/s12935-016-0298-1. |
| [6] |
Lehmann U, Hasemeier B, Christgen MA, et al. Identification of differentially expressed microRNAs in human male breast cancer[J].
Cancer Res, 2009, 69(9): 109.
|
| [7] |
Hou YY, Cao WW, Li L, et al. MicroRNA-519d targets MKi67 and suppresses cell growth in the hepatocellular carcinoma cell line QGY- 7703[J].
Cancer Lett, 2011, 307(2): 182-90.
DOI: 10.1016/j.canlet.2011.04.002. |
| [8] |
Fornari F, Milazzo M, Chieco PA, et al. In hepatocellular carcinoma miR-519d is up-regulated by p53 and DNA hypomethylation and targets CDKN1A/p21, PTEN, AKT3 and TIMP2[J].
J Pathol, 2012, 227(3): 275-85.
DOI: 10.1002/path.3995. |
| [9] |
Gennarino VA, D'angelo G, Dharmalingam G, et al. Identification of microRNA-regulated gene networks by expression analysis of target genes[J].
Genome Res, 2012, 22(6): 1163-72.
DOI: 10.1101/gr.130435.111. |
| [10] |
Yue H, Tang B, Zhao Y, et al. MIR-519d suppresses the gastric cancer epithelial-mesenchymal transition via Twist1 and inhibits Wnt/β- catenin signaling pathway[J].
Am J Transl Res, 2017, 9(8): 3654-64.
|
| [11] |
Yang X, Hu YQ, Liu YK, et al. C14orf28 downregulated by miR- 519d contributes to oncogenicity and regulates apoptosis and EMT in colorectal cancer[J].
Mol Cell Biochem, 2017, 434(1/2): 197-208.
|
| [12] |
Pang YX, Mao HL, Shen L, et al. MiR-519d represses ovarian cancer cell proliferation and enhances cisplatin-mediated cytotoxicity in vitro by targeting XIAP[J].
Onco Targets Ther, 2014, 7: 587-97.
|
| [13] |
Li YY, Shao JP, Zhang SP, et al. miR-519d-3p inhibits cell proliferation and invasion of gastric cancer by downregulating BCell lymphoma 6[J].
Cytogenet Genome Res, 2018, 154(1): 12-9.
DOI: 10.1159/000487372. |
| [14] |
Chu C, Liu X, Bai X, et al. MiR-519d suppresses breast cancer tumorigenesis and metastasis via targeting MMP3[J].
Int J Biol Sci, 2018, 14(2): 228-36.
DOI: 10.7150/ijbs.22849. |
| [15] |
Li D, Song H, Wu T, et al. MiR-519d-3p suppresses breast cancer cell growth and motility via targeting LIM domain kinase 1[J].
Mol Cell Biochem, 2017, 29: 10.
|
| [16] |
Hua KT, Hong JB, Sheen YS, et al. miR-519d promotes melanoma progression by downregulating EphA4[J].
Cancer Res, 2018, 78(1): 216-29.
DOI: 10.1158/0008-5472.CAN-17-1933. |
| [17] |
Jiang L, Shi S, Shi Q, et al. MicroRNA-519d-3p inhibits proliferation and promotes apoptosis by targeting HIF-2α in cervical cancer under hypoxic conditions [J]. Oncol Res, 2018, [Epub ahead of print].
|
| [18] |
Szklarczyk D, Franceschini A, Wyder S, et al. STRING v10: proteinprotein interaction networks, integrated over the tree of Life[J].
Nucleic Acids Res, 2015, 43(Database issue): D447-52.
|
| [19] |
Ng A, Bursteinas B, Gao Q, et al. pSTIING: a 'systems' approach towards integrating signalling pathways, interaction and transcriptional regulatory networks in inflammation and cancer[J].
Nucleic Acids Res, 2006, 34(Database issue): D527-34.
|
| [20] |
Melsheimer P, Vinokurova S, Wentzensen N, et al. DNA aneuploidy and integration of human papillomavirus type 16 E6/E7 oncogenes in Intraepithelial neoplasia and invasive squamous cell carcinoma of the cervix uteri[J].
Clin Cancer Res, 2004, 10(9): 3059-63.
DOI: 10.1158/1078-0432.CCR-03-0565. |
| [21] |
Snijders PJ, Steenbergen RD, Heideman DA, et al. HPV-mediated cervical carcinogenesis: concepts and clinical implications[J].
J Pathol, 2006, 208(2): 152-64.
DOI: 10.1002/(ISSN)1096-9896. |
| [22] |
Martin CM, Astbury K, O'leary JJ. Molecular profiling of cervical neoplasia[J].
Expert Rev Mol Diagn, 2006, 6(2): 217-29.
DOI: 10.1586/14737159.6.2.217. |
| [23] |
Hayes J, Peruzzi PP, Lawler S. MicroRNAs in cancer: biomarkers, functions and therapy[J].
Trends Mol Med, 2014, 20(8): 460-9.
DOI: 10.1016/j.molmed.2014.06.005. |
| [24] |
Swaminathan G, Martin-Garcia J, Navas-Martin S. RNA viruses and microRNAs: challenging discoveries for the 21st century[J].
Physiol Genomics, 2013, 45(22): 1035-48.
DOI: 10.1152/physiolgenomics.00112.2013. |
| [25] |
Park S, Eom K, Kim J, et al. MiR-9, miR-21, and miR-155 as potential biomarkers for HPV positive and negative cervical cancer[J].
BMC Cancer, 2017, 17(1): 658.
DOI: 10.1186/s12885-017-3642-5. |
| [26] |
Harden ME, Munger K. Human papillomavirus 16 E6 and E7 oncoprotein expression alters microRNA expression in extracellular vesicles[J].
Virology, 2017, 508: 63-9.
DOI: 10.1016/j.virol.2017.05.005. |
| [27] |
Xia YF, Pei GH, Wang N, et al. miR-3156-3p is downregulated in HPV- positive cervical cancer and performs as a tumor-suppressive miRNA[J].
Virol J, 2017, 14(1): 20.
DOI: 10.1186/s12985-017-0695-7. |
| [28] |
Gómez-Gómez Y, Organista-Nava J, Ocadiz-Delgado R, et al. The expression of miR-21 and miR-143 is deregulated by the HPV16 E7 oncoprotein and 17β-estradiol[J].
Int J Oncol, 2016, 49(2): 549-58.
DOI: 10.3892/ijo.2016.3575. |
| [29] |
Zhang R, Su J, Xue SL, et al. HPV E6/p53 mediated down-regulation of miR-34a inhibits Warburg effect through targeting LDHA in cervical cancer[J].
Am J Cancer Res, 2016, 6(2): 312-20.
|
| [30] |
Xie Q, Wang S, Zhao Y, et al. MiR-519d impedes cisplatin-resistance in breast cancer stem cells by down-regulating the expression of MCL-1[J].
Oncotarget, 2017, 8(13): 22003-13.
|
| [31] |
Bai Y, Lu C, Zhang G, et al. Overexpression of miR-519d in lung adenocarcinoma inhibits cell proliferation and invasion via the association of eIF4H[J].
Tumour Biol, 2017, 39(3): 1010428317694566.
|
 2018, Vol. 38
2018, Vol. 38

