2. 南方医科大学 南方医院影像中心,广东 广州 510515;
3. 汕头市中心医院肿瘤放疗科,广东 汕头 515000
2. Imaging Center, Nanfang Hospital, Southern Medical University, Guangzhou 510515, China;
3. Department of Radiation Oncology, Shantou Central Hospital, Shantou 515000, China
肝细胞癌(HCC)作为一种肝脏的主要恶性病变,是当今世界范围内3大癌症杀手之一[1]。肝血管瘤(HHE)则是一种常见的肝脏良性肿瘤[2]。两者的治疗方法完全不同,肝细胞癌的最佳治疗方法为手术切除、肝移植或进行介入治疗;而血管瘤通常并不需要特殊干预,仅当出现明显的症状时才需处理[3-4],因此两者间的准确的鉴别诊断对于后续治疗方案的确定具有重大的临床指导意义。
目前,肝细胞癌与血管瘤的鉴别诊断主要依靠超声、计算机断层成像增强扫描、磁共振成像(MRI)等常规影像学方法[5-8]。虽然肝细胞癌与血管瘤有着完全不同的病理结构,但却常常有相似的影像学表现[9]。肝硬化背景下的血管瘤更是容易表现出非典型的影像征象,如快速填充型的血管瘤可表现出与肝细胞癌相类似的“快进快出”强化特点,玻璃样变性的血管瘤会丧失T2WI图像上的“灯泡征”,与肝细胞癌的信号特征更为相似[10-12]。这使得传统依靠人眼主观视觉进行诊断的方式准确性与一致性有限。近年来,一些非侵入性的量化MRI方法被提出来并应用于肝脏良恶性病变的诊断。其中,有学者采用T2弛豫时间来鉴别肝脏良恶性占位[6],但由于将肝囊肿等病变纳入良性病变组来增大组间差距,且没有进行各病灶间的鉴别,临床应用仍然受到限制。有研究表明T2*弛豫时间[13]、弥散加权成像的表观扩散系数(ADC)[14]以及基于普美显增强MRI的T1弛豫时间[15]用来鉴别肝细胞癌及海绵状血管瘤均有较好的效能,但由于需要增加额外扫描时间且对于不典型血管瘤的鉴别能力有限而没有得到广泛应用。
影像组学是医学影像领域新兴的发展研究方向[16]。目前尚无关于影像组学方法用于鉴别诊断肝细胞癌与血管瘤的相关研究。普美显增强MRI扫描在肝脏疾病诊断的广泛应用[17-18],因此,本文提出一种基于普美显增强MR扫描的肝胆期图像,利用影像组学分析来无创、快速、准确辨别肝细胞癌与肝血管瘤的方法。
1 资料和方法 1.1 资料来源本研究回顾性收集了2014年8月~2016年7月南方医科大学南方医院92例患者共135个病灶(75个HCC,60个HHE)。患者统计信息见表 1。所有患者均行普美显MRI增强扫描,并于MR检查后4周(中位数)进行穿刺活检或手术,得出每个病灶的病理结果。MRI增强扫描方法:采用普美显(Gd-EOB-DTPA, Primovist; Bayer Schering Pharma, Osaka, Japan)作为对比剂,所用机器为Philips Achieva 3.0TX(Philips Healthcare,Best,Netherland)磁共振扫描仪,采用8通道相控阵体部线圈。Gd-EOB-DTPA通过人工静脉高压注射筒推注,注射剂量为0.1 mL/kg,流率为2 mL/s。注射对比剂后立即用20 mL生理盐水进行冲洗,冲洗流率为2 mL/s。动态增强扫描序列(包括动脉早期、晚期、门脉期、延迟期)及肝胆特异期(注射后20 min),使用eThrive(T1高分辨力各向同性容积激发,TR/TE:3.1/ 1.51 ms,FOV:375 mm×298 mm×200 mm,层厚:2.5 mm,层间距0,反转角:10°)。
| 表 1 人口资料统计 Table 1 Summary of the study population |
对于每个患者的普美显MRI增强扫描数据,从中选择肝胆特异期图像进行分析,图像大小中位数为320×320,像素大小中位数为1.17 mm×1.17 mm。
1.2 肿瘤勾画与图像预处理肿瘤区域由一名经验丰富的影像科医生逐层手动勾画,并交由另一名影像科医生进行检查。所使用的勾画软件为3Dslicer(http://www.slicer.org)。考虑到3维的影像组学特征提取受层厚影响较大,最终特征提取与分析在病灶面积最大的层面进行。为了减少电子噪声以及图像噪声对特征稳定性的影响,在提取特征前需要对图像进行归一化。对层面内所有灰度值使用minmax方法按1%分位点到99%分位点归一化到[0 1]之间。最后再将图像量化为8 bit。归一化与图像量化均在MATLAB 2014b 64位平台上完成。
1.3 影像组学特征提取本研究主要采用纹理分析方法对肿瘤区域提取影像组学特征。作为医学图像后处理的重要手段,纹理分析能够定量分析图像像素的细微差异与变化,是当前影像组学分析中主要的特征提取方法[19-20]。基于MaZda图像纹理分析工具,本研究共提取243个影像特征(http://www.eletel.eu/mazda/),包括直方图特征10个、游程矩阵(Gray Level Run Length Matrix,GRLM)特征20个、灰度共生矩阵(Gray-Level Co-Occurrence Matrix,GLCM)特征132个、灰度梯度特征(Gradient Matrix,GRM)5个、小波特征(wavelet)4个以及几何结构(geometry)特征72个[21-22]。其中,直方图特征为最基本图像描述,包括:图像均值、方差、丰度、偏度以及百分位数等。游程矩阵是指连续的、共线的并具有相同灰度级的像素点,对于一幅图像可以计算指定方向(一般为0°、45°、90°、135° 4个方向)对应的游程矩阵,特征参量包括:行程长度的不均匀性度量、灰度不均匀性度量、长行程优势、短行程优势、行程总数百分率[23]。灰度共生矩阵建立在估计图像二阶组合条件密度基函数上,是当前人们公认的一种重要的纹理分析方法。本研究中使用3个距离(1、2、3)4个方向(0°、45°、90°、135°)分别提取灰度共生矩阵特征[24]。灰度梯度特征指灰度区域的梯度描述,包括绝对梯度的均值、方差、偏度、丰度等[25]。几何特征主要定量描述肿瘤的外观形态,如最大直径、重心坐标等。关于特征的详细信息可以通过MaZda用户手册以及特征列表查看。所有特征不做其他处理直接用于单特征分析。
1.4 单特征分析对每个特征,执行两样本t检验(两组均满足正态分布时)或Mann Whitney U检验(任何一组不满足正态分布时),判断特征对于两种疾病是否有显著性差异。之后对具有显著性差异的特征执行ROC分析。由于单一分类性能指标(如准确率)并不能很好地反映特征的分类能力,本文依次根据AUC值、Yoden-index、准确率对特征进行排序,分类性能较好的特征对应的以上三个分类性能指标相对较大,排序也靠前。所有统计分析过程在MATLAB 2014b 64位平台上进行。P < 0.05认为差异具有统计学意义。
1.5 多特征分析高通量影像组学特征往往存在冗余性、重复性、噪声。另外,高维特征直接用于训练分类容易过拟合,使得模型泛化能力差。因此,在进行分类器训练之前需要进行特征选择,从高通量特征中选择出最优特征子集。本研究分别使用最小冗余-最大相关(mRmR)算法、近邻成分分析(NCA)算法以及序列前向选择(SFS)算法进行特征选择[26-28]。结合支持向量机分类器,比较3种算法的平均性能,以确定合适的特征选择方法。最终特征选择的结果在3种不同的分类器:支持向量机RBFSVM、线性判别分析(LDA)、逻辑回归上训练、测试[29-30]。所有实验均执行重复5次10折的交叉验证,即随机将数据集平均分为10份,依次取其中一份作为测试集测试分类器性能,余下作为训练集训练分类器,直至完成单次交叉验证实验,整个过程重复执行5次[31]。最后的评价结果由50次的测试结果(包括准确率、AUC值、敏感性以及特异性)进行平均得到。在进行多特征分析前,对所有特征进行归一化,以减少训练时间。上述分析过程在MATLAB 2014b 64位平台上完成。
2 结果 2.1 单特征分析结果T检验或Mann Whitney U检验的结果显示,超过50%的特征对于两种疾病存在显著性差异。如图 1特征P值的累积分布曲线所示。
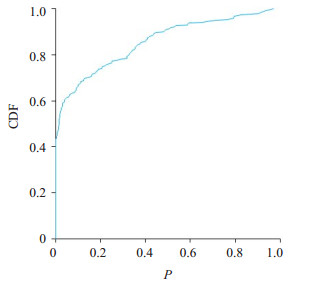
|
图 1 P值累计分布曲线 Figure 1 Empirical cumulative distribution function (CDF) curve of the P-values of the features |
根据ROC分析的结果(AUC值、Yoden-index及准确率)对特征排序,前5个特征的鉴别诊断性能如表 2所示。图 2为前5个特征的ROC曲线。
| 表 2 根据ROC分析结果排序,前5个特征的鉴别诊断性能统计 Table 2 Summary of the performance metric estimates of the top 5 texture features with significant differences |
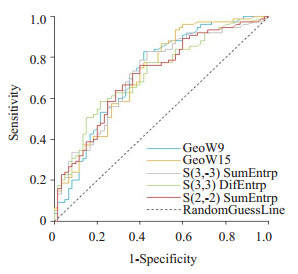
|
图 2 根据ROC分析结果排序,前5个特征的ROC曲线 Figure 2 Receiver operating characteristic (ROC) curve of the top 5 features with significant differences. All the features were sorted according to the AUC value and Yoden index |
排序靠前的特征主要是反映肿瘤区域不均质性的GLCM熵特征,以及反映肿瘤形态的几何结构特征。
2.2 多特征分析结果图 3显示了不同特征选择方法下,平均测试准确率(即重复5次10折交叉验证实验结果的平均)随着选择特征个数增加的变化曲线。结果显示,SFS算法所得到的结果明显优于另外两种算法。之后综合考虑平均敏感度、特异性、AUC值等指标确定最优特征子集的大小。图 4显示了SFS算法下平均分类性能(AUC、敏感性、特异性)随着选择特征个数增加的变化曲线。根据性能变化的趋势最终确定选择15个特征。之后将最优特征子集分别在线性以及非线性分类器上进行训练与测试。表 2为3种分类器:RBF-SVM、LDA、logistic回归的平均分类性能,其中RBF-SVM:AUC达到0.86,敏感性为0.88,特异性为0.76,测试准确率:0.82。
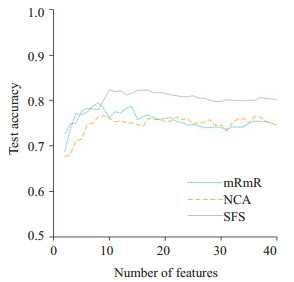
|
图 3 不同特征选择算法的性能比较 Figure 3 Average test accuracy changes as the number of features increases under 3 different feature selection algorithms (mRmR, NCA and SFS) |
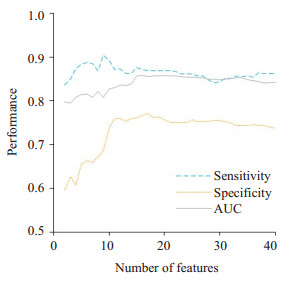
|
图 4 SFS算法下的平均分类性能的变化曲线 Figure 4 Average performance (sensitivity, specificity, and AUC value) with increasing number of features under SFS feature selection algorithm |
随着精准医疗的提出与完善,影像组学方法越来越得到人们的关注。与传统的蛋白组学和基因组学手段相比,影像组学能够无创地评估肿瘤及其微环境,并且不受采样随机性影响,能够在不同的时间点重复进行,量化肿瘤的时空异质性[32]。
| 表 3 使用SFS算法选择的15个特征,不同分类器上的性能比较 Table 3 Comparison of the performance metrics of the 3 classifiers (Mean±SD) |
本研究利用影像组学方法,对92例肝细胞癌与血管瘤患者(共135个病灶)的普美显增强磁共振图像提取影像纹理特征,其中包括反映区域像素强度分布的一阶直方图特征、反映肿瘤异质性的二阶特征(如灰度共生矩阵等)以及反映肿瘤形态的几何特征。单特征分析的结果显示超过50%的特征具有一定的鉴别诊断能力,其中GLCM的熵特征S(3, -3)SumEntrp的分类结果:AUC为0.72,AUC P值远小于0.01,敏感度为0.83,特异性为0.57。这一结果表明,影像组学纹理特征能够捕捉肿瘤微结构环境的差异,为肝细胞癌与血管瘤的鉴别诊断提供有价值的信息。进一步的多特征分析结果显示,当使用序列前向选择算法筛选出15个特征后建立SVM分类器,准确率达0.82,AUC达0.86,敏感性0.88,特异性0.76。
近年来,MR弥散加权成像、MR增强成像等技术被广泛用于肝脏局灶性病变的良恶性鉴别诊断研究。袁灼彬等人的研究均表明ADC值在肝脏良恶性病变组间的差异具有统计学意义[33-35]。然而实际上,MR弥散加权成像中b值的选择对于ADC值影响很大,已有的关于ADC值用于肝脏良恶性肿瘤鉴别的研究在b值的选择上并没有统一的标准,因此目前也未有统一的ADC值标准用来区分肝脏良恶性病变。此外,在这些研究中,良性病变组往往纳入肝囊肿等容易鉴别的病变来增大组间差距,且缺乏专门针对肝细胞癌与血管瘤鉴别诊断的研究,临床应用有限。同样基于普美显增强磁共振扫描,通过T1弛豫时间鉴别肝细胞癌、肝血管瘤以及局灶性结节增生等3种肝脏局灶性病变,准确率为0.88,但该方法需要额外序列,增加了扫描时间,实际诊断效能有限[15]。本研究基于临床常用于肝脏疾病检查的MR增强扫描序列,在不要求额外增加扫描时间、采集更多数据的基础上,进行肝细胞癌与血管瘤的鉴别诊断,研究结果表明仅利用普美显增强磁共振肝胆期图像提取影像组学特征进行分析建模能够有效鉴别诊断肝细胞癌与血管瘤,这有望为肝细胞癌与血管瘤的临床诊断提供有利的辅助手段,对于肝脏疾病患者精准医疗的实现具有较大意义。
由于图像数据的轴向分辨率有限,本研究未进行影像组学的三维特征提取。未来需要获取高质量的图像,以完成三维特征提取,进一步提高分类性能。本研究作为探索性研究,仅将普美显MR增强扫描的肝胆期图像进行影像组学分析,未来将加入多模态、多参数图像数据,以期获得更佳的诊断与鉴别诊断。此外,还需要进行多中心数据的收集,完成影像组学特征的可重复性研究。
综上所述,基于普美显MR增强图像提取的影像组学特征对于肝细胞与血管瘤的鉴别诊断具有较好的性能,有望为临床诊断提供辅助支持,帮助实现肝脏疾病患者的精准医疗。
| [1] |
Ferlay J SH. Estimates of worldwide burden of cancer in 2008:GLOBOCAN 2008[J].
Int J Cancer, 2010, 127(12): 2893-917.
DOI: 10.1002/ijc.v127:12. |
| [2] |
Werner M, Flejou JF, Hainaut P, et al. World Health Organization classification of tumors: Pathology and genetics: tumors of the digestive system[J].
Adenocarcinoma of Esophagus, IARC Press, Lyon, 2000: 20-26.
|
| [3] |
Little SA, Fong Y. Hepatocellular carcinoma: current surgical management[J].
Gastroenterology, 2001, 28(5): 474-86.
|
| [4] |
Vilgrain V, Boulos L, Vullierme MP, et al. Imaging of atypical hemangiomas of the liver with pathologic correlation[J].
Radiographics, 2000, 20(2): 379-97.
DOI: 10.1148/radiographics.20.2.g00mc01379. |
| [5] |
Soyer P, Corno L, Boudiaf M, et al. Differentiation between cavernous hemangiomas and untreated malignant neoplasms of the liver with free-breathing diffusion-weighted Mr imaging:comparison with T2-weighted fast spin-echo Mr imaging[J].
Eur J Radiol, 2011, 80(2): 316-24.
DOI: 10.1016/j.ejrad.2010.08.011. |
| [6] |
Farraher SW, Jara H, Chang KJ, et al. Differentiation of hepatocellular carcinoma and hepatic metastasis from cysts and hemangiomas with calculated T2 relaxation times and the T1/T2 relaxation times ratio[J].
J Magn Reson Imaging, 2006, 24(6): 1333-41.
DOI: 10.1002/(ISSN)1522-2586. |
| [7] |
Lee JH, Lee JM, Kim SJ, et al. Enhancement patterns of hepatocellular carcinomas on multiphasicmultidetector row CT:comparison with pathological differentiation[J].
Br J Radiol, 2012, 85(117): e573-83.
|
| [8] |
Santoro L, Grazioli L, Filippone A, et al. Resovist enhanced Mr imaging of the liver: does quantitative assessment help in focal lesion classification and characterization[J].
J Magn Reson Imaging, 2009, 30(5): 1012-20.
DOI: 10.1002/jmri.v30:5. |
| [9] |
Bajenaru N, Balaban V, Săvulescu F, et al. Hepatic hemangiomareview[J].
J Med Life, 2015, 8 Spec Issue(4): 4-11.
|
| [10] |
Tateyama A, Fukukura Y, Takumi K, et al. Hepatic hemangiomas:factors associated with pseudo washout sign on Gd-EOB-DTPAenhanced Mr imaging[J].
Magn Reson Med Sci, 2016, 15(1): 73-82.
DOI: 10.2463/mrms.2014-0125. |
| [11] |
Tamada T, Ito K, Ueki A, et al. Peripheral low intensity sign in hepatic hemangioma: diagnostic pitfall in hepatobiliary phase of Gd-EOB-DTPA-enhanced MRI of the liver[J].
J Magn Reson Imaging, 2012, 35(4): 852-8.
DOI: 10.1002/jmri.v35.4. |
| [12] |
Becker-Weidman DJ, Hope TA, Doshi PH, et al. Transient washout of hepatic hemangiomas: Potential pitfall mimicking malignancy[J].
Radiol Case Rep, 2016, 11(2): 62-6.
DOI: 10.1016/j.radcr.2016.02.010. |
| [13] |
Sun M, Wang S, Song Q, et al. Utility of R2* obtained from T2*-weighted imaging in differentiating hepatocellular carcinomas from cavernous hemangiomas of the liver[J].
PLoS One, 2014, 9(3): e91751.
DOI: 10.1371/journal.pone.0091751. |
| [14] |
Quan XY, Sun XJ, Yu ZJ, et al. Evaluation of diffusion weighted imaging of magnetic resonance imaging in small focal hepatic lesions:a quantitative study in 56 cases[J].
Hepatobiliary Pancreat Dis Int, 2005, 4(3): 406-9.
|
| [15] |
Peng Z, Li C, Chan T, et al. Quantitative evaluation of Gd-EOBDTPA uptake in focal liver lesions by using T1 mapping:differences between hepatocellular carcinoma, hepatic focal nodular hyperplasia and cavernous hemangioma[J].
Oncotarget, 2017, 8(39): 65435-44.
|
| [16] |
Lambin P, Rios-Velazquez E, Leijenaar R, et al. Radiomics:extracting more information from medical images using advanced feature analysis[J].
Eur J Cancer, 2012, 48(4): 441-6.
DOI: 10.1016/j.ejca.2011.11.036. |
| [17] |
Campos JT, Sirlin CB, Choi JY. Focal hepatic lesions in Gd-EOBDTPA enhanced MRI: the Atlas[J].
Insights Imaging, 2012, 3(5): 451-74.
DOI: 10.1007/s13244-012-0179-7. |
| [18] |
Agarwal S, Grajo JR, Fuentes-Orrego JM, et al. Distinguishing hemangiomas from metastases on liver MRI performed with gadoxetate disodium: Value of the extended washout sign[J].
Eur J Radiol, 2016, 85(3): 635-40.
DOI: 10.1016/j.ejrad.2015.12.028. |
| [19] |
Gillies RJ, Kinahan PE, Hricak H. Radiomics: images are more than pictures, they are data[J].
Radiology, 2016, 278(2): 563-77.
DOI: 10.1148/radiol.2015151169. |
| [20] |
Davnall F, Yip CS, Ljungqvist G, et al. Assessment of tumor heterogeneity: an emerging imaging tool for clinical practice?[J].
Insights Imaging, 2012, 3(6): 573-89.
DOI: 10.1007/s13244-012-0196-6. |
| [21] |
Szczypinski PM, Strzelecki M, Materka A. A software for texture analysis. in: international [C]//Symposium on Information Technology Convergence, 2007: 245-9.
|
| [22] |
Szczypiński PM, Strzelecki M, Materka A, et al. MaZda--a software package for image texture analysis[J].
Comput Methods Programs Biomed, 2009, 94(1): 66-76.
DOI: 10.1016/j.cmpb.2008.08.005. |
| [23] |
Galloway MM. Texture analysis using gray level run lengths[J].
Compu Grap Image Processing, 1975, 4(2): 172-9.
DOI: 10.1016/S0146-664X(75)80008-6. |
| [24] |
Haralick RM, Shanmugam K, Dinstein IH. Textural features for image classification[J].
Systems Man & Cybernetics IEEE Transactions, 1973, 3(6): 610-21.
|
| [25] |
Haralick RM. Statistical and structural approaches to texture[J].
Proceedings IEEE, 1979, 67(5): 786-804.
DOI: 10.1109/PROC.1979.11328. |
| [26] |
Peng H, Long F, Ding C. Feature selection based on mutual information: criteria of max-dependency, max-relevance, and minredundancy[J].
IEEE Trans Pattern Anal Mach Intell, 2005, 27(8): 1226-38.
DOI: 10.1109/TPAMI.2005.159. |
| [27] |
Yang W, Wang K, Zuo W. Neighborhood component feature selection for High-Dimensional data[J].
J Comput (Taipei), 2012, 7(1): 161-8.
|
| [28] |
Rückstie T, Osendorfer C, Smagt P. Sequential feature selection for classification[J].
Ai Advances Artificial Intelligence, 2011, 7106(7): 132-41.
|
| [29] |
Chang CC, Lin C. A library for support vector machines[J].
ACM Transactions on Intelligent Systems and Technology (TIST), 2011, 2(3): 27.
|
| [30] |
Xanthopoulos P, Pardalos PM, Trafalis TB. Linear discriminant analysis[J].
Chicago, 2001, 3(6): 27-33.
|
| [31] |
Kohavi R. A study of cross-validation and bootstrap for accuracy estimation and model selection. In: International [C]//Joint Conference on Artificial Intelligence: 1995, 1995: 1137-43.
|
| [32] |
Limkin EJ, Sun R, Dercle L, et al. Promises and challenges for the implementation of computational medical imaging (radiomics) in oncology[J].
Ann Oncol, 2017, 28(6): 1191-206.
DOI: 10.1093/annonc/mdx034. |
| [33] |
袁灼彬, 郑晓林, 王惠媚, 等. DWI定量分析在鉴别肝脏良恶性病变的价值[J].
影像诊断与介入放射学, 2014, 45(1): 70-4.
|
| [34] |
李晓娟, 刘志兰, 何冰峰, 等. ADC值对肝脏良恶性肿瘤鉴别诊断的研究[J].
哈尔滨医科大学学报, 2009, 43(3): 265-7.
|
| [35] |
戚喜勋, 李祥胜, 张莹莹, 等. 磁共振弥散加权成像对肝局灶性病变良恶性的鉴别诊断[J].
中国医科大学学报, 2010, 39(4): 311-3.
|
 2018, Vol. 38
2018, Vol. 38

