急性T淋巴细胞性白血病(T-ALL)起源于胸腺非成熟T淋巴细胞是一种血液系统恶性肿瘤[1-2],分别占儿童急性淋巴细胞白血病的15%和成人急性淋巴细胞白血病的25%左右[3],主要表现为循环白细胞增多,肝脾肿大,出血倾向以及中枢神经浸润等[4-5]。T-ALL的治疗主要有化疗,干细胞移植以及支持治疗,随着加强化疗的使用,T-ALL病人的预后有所改善,但5年无病生存率仍仅为60%~70%,许多T-ALL病人不能得到完全缓解最终导致复发[6]。目前针对T-ALL的治疗仍不容乐观,缺乏对T-ALL发生发展的分子机制的认知限制了对疾病的治疗能力。
T-ALL的发生发展是一个多基因多步骤协同过程, 存在多种分子及信号通路异常,仅关注单个或某几个基因的研究方法已经不能满足当前研究需要。基于高通量平台的基因表达谱芯片可以检测上万种基因mRNA水平变化,基因芯片技术联合生物信息学使得研究多个基因表达情况成为可能,现已用于分子诊断,癌症分型,预后预测以及新药研究等[7-9]。目前,针对T-ALL的基因芯片研究相对较少,在本文中,我们利用多个数据库对T-ALL病人基因表达谱芯片GSE14317进行全面综合分析,针对差异表达基因进行生物信息学分析并利用实时荧光定量PCR验证,旨在为临床筛选T-ALL的相关分子标志物及药物靶点提供理论基础。
1 资料和方法 1.1 表达谱芯片本文自公共数据库基因表达数据库(GEO)中下载T-ALL基因表达谱芯片GSE14317[10],该芯片基于GPL571平台([HG-U133A_2]Affymetrix Human Genome U133A 2.0 Array),共有26例样本,包括19例T-ALL病人骨髓标本以及7例正常人胸腺CD4+T细胞对照标本。
1.2 筛选差异表达基因芯片数据进行数据过滤、标准化和对数化处理,将处理后的数据转化为基因表达矩阵,使用稳健多芯片平均标准化分析(RMA)方法对数据进行归一化处理。利用R语言limma包对T-ALL实验组与对照组比较分析,筛选差异表达基因。
1.3 芯片数据生物信息学分析利用DAVID(https://david.ncifcrf.gov/)在线数据库对差异基因进行GO功能富集分析以及KEGG通路富集分析。利用STRING等在线工具筛选交互作用评分大于0.9(高度可信)的蛋白质构建蛋白互作网络图,并用Cytoscape软件构建蛋白互作子网络,并对子网络基因进行通路富集分析。
1.4 RT-PCR验证基因表达收集我院5例T-ALL病人骨髓标本以及3例正常人胸腺细胞作为对照,Trizol法提取细胞总RNA并逆转录为cDNA,利用SYBR荧光定量PCR法检测枢纽基因的mRNA表达水平。各基因的引物序列见表 1。最后根据2-△△Ct值计算基因相对表达量。
| 表 1 引物信息 Table 1 Sequences of the primers for RT-PCR |
在UCSC数据库中下载核心枢纽基因启动子上游1000 bp序列,利用JASPAR数据库下载人类全基因转录因子序列,进行转录因子预测,将得到的阈值设为90%(高度可信)。筛选有差异表达的转录因子,将转录因子和枢纽基因导入Cytoscape软件构建转录因子-靶基因网络。
1.6 统计学方法实验结果采用SPSS19.0软件进行统计学分析,数据均以均数±标准差表示,采用单因素方差分析。P < 0.05为差异有统计学意义。
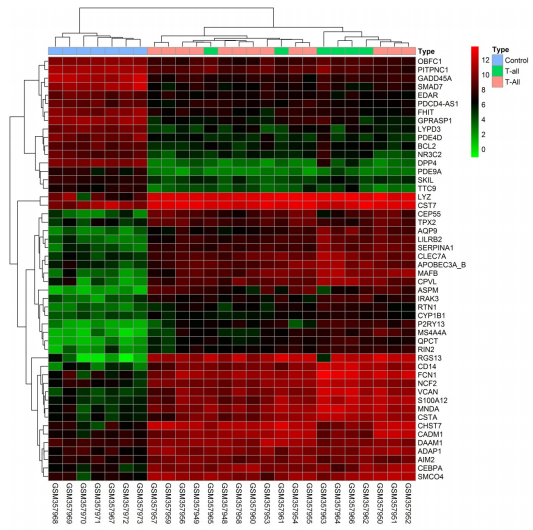
2 结果 2.1 基因表达谱芯片数据结果样本均一化结果显示每个样本表达量的中位数均一致,表明样本已均一化处理(图 1)。本研究对样本进行贝叶斯检验,按照差异倍数 > 2,P < 0.05作为筛选条件,筛选T-ALL病人表达谱芯片中差异基因。结果发现共有1443个差异表达基因,其中上调的有800个,下调的有463个,选取最有差异的50个基因做热点图反应基因表达情况(图 2)。
|
图 1 标本均一化处理 Figure 1 The expression spectrum data of GSE14317 profiles before or after normalization. A: Box plots of the expression spectrum data before normalization; B: Box plots of the expression spectrum data after normalization. |
|
图 2 最有差异的50个基因热点图 Figure 2 Heat map of the top 50 differentially expressed genes. Red: up-regulation; green: down-regulation. |
利用DAVID数据库对差异基因进行GO功能富集分析与KEGG通路分析。GO结果(P < 0.05和FDR < 0.05)显示上调的基因富集在细胞周期(GO:0007049)等,下调基因主要富集在蛋白刺激反应(GO:0051789)(表 2)。KEGG结果(P < 0.05和FDR < 0.05)显示上调基因主要富集于造血细胞系(hsa04640)和细胞周期(hsa04110)等,下调基因主要富集于细胞因子间相互作用(hsa04060)和T细胞受体信号通路(hsa04630)(表 3)。
| 表 2 上调基因功能和通路富集分析 Table 2 Function and KEGG pathway analysis of the up-regulated genes. |
| 表 3 上调基因功能和通路富集分析 Table 3 Function and KEGG pathway analysis of the down-regulated genes |
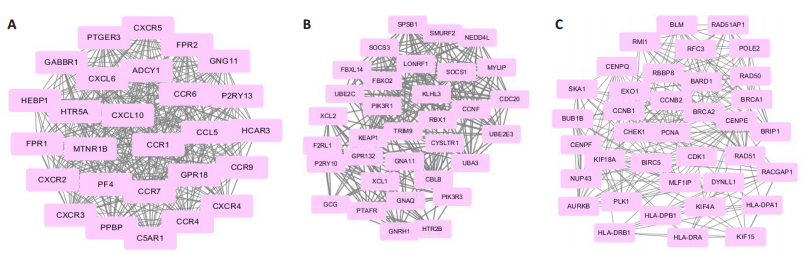
对蛋白互作网络图分析发现,满足条件的共有694个节点,3348条关系,其中CDK1,PIK3R1,CCNB1,CCNA2,CDC20,JUN,GNG11,PLK1,PCNA和CCNB2基因具有最大互作关系。利用MCODE [11]构建高度蛋白互作子网络,以发现潜在的功能模块,筛选出最有意义的3个模块并进行信号通路富集分析(P < 0.05和FDR < 0.05),结果显示模块1主要富集于趋化因子信号通路,模块2主要富集于泛素介导的蛋白降解,模块3主要富集于细胞周期(图 3)。
|
图 3 差异基因蛋白互作网络 Figure 3 Top 3 modules screened from the protein-protein network. A: Module 1; B: Module 2; C: Module 3. |
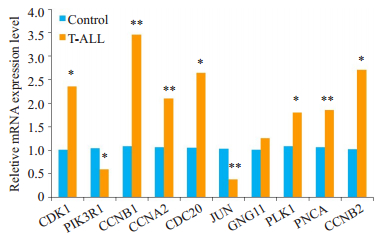
利用RT-PCR验证CDK1,PIK3R1,CCNB1,CCNA2,CDC20,JUN,GNG11,PLK1,PCNA和CCNB2在T-ALL病人中的mRNA表达情况。结果显示CDK1,CCNB2,CDC20,PLK1,CCNB2的mRNA表达量较正常对照组增加(P < 0.05),CNB1,CCNA2,PCNA的mRNA表达量较正常对照组明显增加(P < 0.01)。PIK3R1的mRNA表达量较正常对照组降低(P < 0.05),JUN的mRNA表达量较正常对照组明显降低(P < 0.01),与芯片结果数据分析一致。但GNG11的mRNA表达量与正常对照组相比无统计学意义(P > 0.05,图 4)。
|
图 4 各基因在T-ALL病人中mRNA表达水平的变化 Figure 4 Changes in mRNA expressions of the identified DEGs in T-ALL patients. *P < 0.05 vs normal control group, **P < 0.01 vs normal control group. |
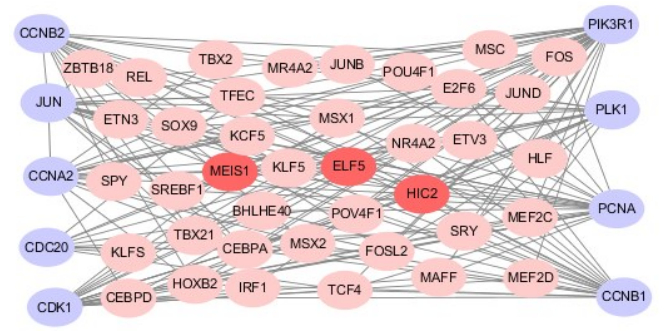
利用JASPAR数据库预测9个关键基因启动子上游转录因子结合位点,筛选出42个差异表达转录因子,其中ELF5,HIC2和MEIS1与候选的9个关键基因启动子上游均有结合位点。将转录因子和关键基因导入Cytoscape软件构建转录因子-靶基因网络(图 5)。
|
图 5 转录因子-靶基因网络 Figure 5 The TFs-target genes regulatory network of T-ALL; the grey nodes represent hub genes, the pink nodes represent transcription factors and the croci nodes represent the three most significant TFs. |
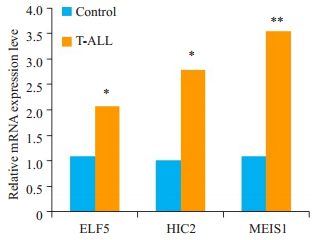
用RT-PCR验证ELF5,HIC2和MEIS1在T-ALL病人中的mRNA表达情况。结果显示ELF5和HIC2 MEIS1的mRNA表达量较正常对照组增加(P < 0.05)MEIS1的mRNA表达量较正常对照组明显增加(P < 0.01,图 6),与芯片结果一致。
|
图 6 转录因子在T-ALL病人中mRNA表达水平的变化 Figure 6 Changes in mRNA expressions of the identified TFs in T-ALL patients. *P < 0.05 vs normal control group, **P < 0.01 vs normal control group. |
急性T淋巴细胞性白血病(T-ALL)是一种血液系统急进性恶性肿瘤,占急性淋巴细胞白血病(ALL)的15%~25%左右。目前T-ALL的治疗主要有化疗,干细胞移植以及支持治疗,近年来T-ALL的治疗取得了较大改善,但仍有25%的患者对T-ALL的治疗耐受[12]或出现严重副作用,探讨研究T-ALL发生发展的具体机制仍是当务之急。基因芯片技术联合生物信息学方法突破传统的小规模研究方式为全面综合分析多基因调控事件在T-ALL发生发展的作用提供了可能。通过对差异表达基因构建蛋白互作网络分析显示节点最多的10个基因:CDK1,PIK3R1,CCNB1,CCNA2,CDC20,JUN,GNG11,PLK1,PCNA和CCNB1。利用RT-PCR验证基因表达情况发现除GNG11外,其余9个关键基因表达情况与芯片结果一致。
CDK1是丝氨酸/苏氨酸蛋白激酶家族的一员,通过催化不同底物磷酸化实现对细胞周期不同时相的推进和转化作用。此外Lu等[13]研究发现,在人类黑色素瘤中,CDK1可以使P53凋亡刺激蛋白抑制因子(IASPP)磷酸化,促进IASPP单体进入核,并暴露其P53结合位点,导致P53抑制增加。在T-ALL中上调的CDK1可能通过促进细胞周期并抑制P53途径加重疾病进程。细胞周期蛋白CCNB1,CCNA2和CCNB2与细胞周期密切相关, 在控制细胞周期G1/S向G1/M转换中起着必不可少的作用,它们参与多种肿瘤细胞周期调控,如结直肠癌,乳腺癌,肺癌等。细胞周期相关蛋白CDC20是纺锤体组装检查点的靶向物和有丝分裂后期促进复合体的正调控因子,参与细胞周期中某些蛋白质的泛素化降解,使细胞周期以单向的方式推进[14]。Bie等[15]发现在胶质母细胞瘤中CDC20的表达受BRCA1调控与疾病的分级有关。BRCA1是多种肿瘤的易感基因,在本文中BRCA1与CDC20表达均升高,据此可以推测BRCA1与T-ALL的分级有关,提示研究抗BRCA1的药物可以为T-ALL的治疗提供靶向。PLK1属于PLKs家族成员,是一种广泛存在于真核生物中的丝氨酸/苏氨酸蛋白激酶。PLK1在多种肿瘤中高表达并与预后有关,Shao等[16]研究发现PLK1可以介导Numb的磷酸化,抑制P53的转录活性,P53失去抑癌功能最终导致肿瘤的发生。PCNA与细胞DNA合成密切相关,在细胞增殖的启动上起重要作用,目前PCNA作为诊断肿瘤增殖的指标,随疾病的进程逐渐增多[17]。PIK3R1在多种肿瘤中有较高的突变率[18],参与PI3K-AKT信号通路。Remke [19]发现异常的PI3K-AKT信号通路与白血病的生成以及T-ALL早期治疗效果差有关。JUN属于转录因子AP-1家族成员之一,JUN和FOS共同组成异源二聚体复合物AP-1,AP-1在肿瘤中扮演重要的作用参与多种细胞生物过程,如细胞增殖,细胞死亡,细胞周期以及细胞分化等。Scupoli等[20]报道在T-ALL中CXCL12可以激活JNK/AP-1信号通路导致IL-8增多影响T-ALL进程。
| 表 4 模块信号通路分析 Table 4 KEGG pathway analysis of top 3 modules |
转录因子通过与靶基因启动子上游结合调控基因表达。因此基于JASPAR数据库,我们对筛选出的9个关键基因进行转录因子预测,共有42个差异表达转录因子,其中ELF5,HIC2和MEIS1与9个关键基因启动子上游均有结合位点。ELF5参与细胞增殖和肿瘤形成,在急性髓性白血病中ELF5抑制P53/P21信号通路导致白血病的发生[21]。MEIS1属于TALE转录因子家族,在正常的胚胎造血中发挥重要作用,在儿童急性白血病中MEIS1与HOXA9共表达加速白血病进程[22]。肿瘤高甲基化基因2(HIC2)是HIC1家族一员,研究发现启动子高甲基化与白血病的发生密切相关,去甲基化治疗是治疗白血病的又一重要途径。目前针对HIC2在T-ALL中的具体作用机制报道较少仍需进一步研究。
综上所述,应用生物信息学方法综合系统的分析了T-ALL差异表达基因,筛选并验证11个关键基因,从分子水平为探讨T-ALL发生发展机制提供了理论依据。筛选出的关键基因为T-ALL的早期诊断及治疗靶点提供了新的方向,为下一步研究T-ALL的发生机制提供了有意义的信息。
| [1] |
Girardi T, Vicente C, Cools J, et al. The genetics and molecular biology of T-ALL[J].
Blood, 2017, 129(9): 1113-23.
DOI: 10.1182/blood-2016-10-706465. |
| [2] |
Karrman K, Johansson B. Pediatric T-cell acute lymphoblastic leukemia[J].
Genes Chromosomes Cancer, 2017, 56(2): 89-116.
DOI: 10.1002/gcc.v56.2. |
| [3] |
Kramer AC, Kothari A, Wilson WC, et al. Dnmt3a regulates T-cell development and suppresses T-ALL transformation[J].
Leukemia, 2017, 31(11): 2479-90.
DOI: 10.1038/leu.2017.89. |
| [4] |
Donnellan W, Mineishi S, Wicker J, et al. Gamma-Delta T cell acute lymphoblastic leukemia: a Single-Center experience[J].
Glob J Cancer Ther, 2016, 2(1): 26-9.
DOI: 10.17352/gjct. |
| [5] |
Jost TR, Borga C, Radaelli EA, et al. Role of CXCR4-mediated bone marrow colonization in CNS infiltration by T cell acute lymphoblastic leukemia[J].
J Leukoc Biol, 2016, 99(6): 1077-87.
DOI: 10.1189/jlb.5MA0915-394R. |
| [6] |
Sutton R, Shaw PJ, Venn NC, et al. Persistent MRD before and after allogeneic BMT predicts relapse in children with acute lymphoblastic leukaemia[J].
Br J Haematol, 2015, 168(3): 395-404.
DOI: 10.1111/bjh.13142. |
| [7] |
Hoelzer D, Thiel E, Arnold R, et al. Successful subtype oriented treatment strategies in adult T-All; results of 744 patients treated in three consecutive GMALL studies[J].
Blood, 2009, 114(22): 137.
|
| [8] |
Damavandi MD, Braconi C, Cascione LA, et al. Global gene expression profiling of mice tumor-derived organoids identifies key microRNAs and metabolic genes involved in CRC progression[J].
Cancer Res, 2015, 75(15): 3038.
|
| [9] |
Kulasingam V, Diamandis EP. Strategies for discovering novel cancer biomarkers through utilization of emerging technologies[J].
Nat Clin Pract Oncol, 2008, 5(10): 588-99.
DOI: 10.1038/ncponc1187. |
| [10] |
Pise-Masison CA, Radonovich M, Dohoney K, et al. Gene expression profiling of ATL patients: compilation of disease-related genes and evidence for TCF4 involvement in BIRC5 gene expression and cell viability[J].
Blood, 2009, 113(17): 4016-26.
DOI: 10.1182/blood-2008-08-175901. |
| [11] |
Bader GD, Hogue CW. An automated method for finding molecular complexes in large protein interaction networks[J].
BMC Bioinformatics, 2003, 4(1): 2.
DOI: 10.1186/1471-2105-4-2. |
| [12] |
El Zawily A, Mcewen E, Toosi B, et al. The EphB6 receptor is overexpressed in pediatric T cell acute lymphoblastic leukemia and increases its sensitivity to doxorubicin treatment[J].
Sci Rep, 2017, 7(1): 14767.
DOI: 10.1038/s41598-017-15200-3. |
| [13] |
Lu M, Breyssens H, Salter V, et al. Restoring p53 function in human melanoma cells by inhibiting MDM2 and cyclin B1/CDK1-phosphorylated nuclear iASPP[J].
Cancer Cell, 2013, 23(5): 618-33.
DOI: 10.1016/j.ccr.2013.03.013. |
| [14] |
Wang Z, Wan L, Zhong J, et al. Cdc20: a potential novel therapeutic target for cancer treatment[J].
Curr Pharm Des, 2013, 19(18): 3210-4.
DOI: 10.2174/1381612811319180005. |
| [15] |
Bie L, Zhao G, Cheng P, et al. The accuracy of survival time prediction for patients with glioma is improved by measuring mitotic spindle checkpoint gene expression[J].
PLoS One, 2011, 6(10): e25631.
DOI: 10.1371/journal.pone.0025631. |
| [16] |
Shao C, Chien SJ, Farah E, et al. Plk1 phosphorylation of Numb leads to impaired DNA damage response[J].
Oncogene, 2018, 37(6): 810-20.
DOI: 10.1038/onc.2017.379. |
| [17] |
Juríková M, Danihel L', Polák Š, et al. Ki67, PCNA, and MCM proteins: Markers of proliferation in the diagnosis of breast cancer[J].
Acta Histochem, 2016, 118(5): 544-52.
DOI: 10.1016/j.acthis.2016.05.002. |
| [18] |
Park SW, Kang MR, Eom HS, et al. Somatic mutation of PIK3R1 gene is rare in common human cancers[J].
Acta Oncol (Madr), 2010, 49(1): 128-30.
DOI: 10.3109/02841860903107890. |
| [19] |
Remke M, Pfister S, Kox C, et al. High-resolution genomic profiling of childhood T-ALL reveals frequent copy-number alterations affecting the TGF-beta and PI3K-AKT pathways and deletions at 6q15-16.1 as a genomic marker for unfavorable early treatment response[J].
Blood, 2009, 114(5): 1053-62.
DOI: 10.1182/blood-2008-10-186536. |
| [20] |
Scupoli MT, Donadelli M, Cioffi F, et al. Bone marrow stromal cells and the upregulation of interleukin-8 production in human Tcell acute lymphoblastic leukemia through the CXCL12/CXCR4 axis and the NF-kappaB and JNK/AP-1 pathways[J].
Haematologica, 2008, 93(4): 524-32.
DOI: 10.3324/haematol.12098. |
| [21] |
Endo A, Tomizawa D, Aoki Y, et al. EWSR1/ELF5 induces acute myeloid leukemia by inhibiting p53/p21 pathway[J].
Cancer Sci, 2016, 107(12): 1745-54.
DOI: 10.1111/cas.2016.107.issue-12. |
| [22] |
Adamaki M, Lambrou GI, Athanasiadou AA, et al. HOXA9 and MEIS1 gene overexpression in the diagnosis of childhood acute leukemias: Significant correlation with relapse and overall survival[J].
Leuk Res, 2015, 39(8): 874-82.
DOI: 10.1016/j.leukres.2015.04.012. |
 2018, Vol. 38
2018, Vol. 38

