2. 南方医科大学珠江医院检验医学部器官衰竭 研究国家重点实验室,广东 广州 510515;
3. 南方医科大学 南方医科大学珠江医院老年病科,广东 广州 510515;
4. 南方医科大学珠江医院感染科,广东 广州 510282
2. State Key Laboratory of Organ Failure Research, Division of Laboratory Medicine, Guangzhou 510515, China;
3. Department of Geriatrics, Guangzhou 510515, China;
4. Department of Infectious Diseases4, Zhujiang Hospital, Southern Medical University, Guangzhou 510282, China
研究表明,人体微生物与人体细胞数量相近[1],数量庞大的微生物与宿主相互作用影响着宿主的健康。肠道菌群失衡与众多疾病的发生发展密切相关,如肥胖[2]、心血管疾病[3]、糖尿病[4]等代谢性疾病,并可能扮演病因的角色[5]。维持肠道菌群的健康与稳态对预防和控制慢性代谢性病的发生与发展有重大的意义。
调节肠道菌群方式主要有功能食品干预、抗生素治疗以及粪菌移植等途径。菊粉类果聚糖(ITF)包括菊粉与低聚果糖(FOS),与低聚半乳糖(GOS)是重要的功能食品,是两类经典益生元,已经广泛应用在食品添加剂、功能食品等领域。非测序技术研究表明,FOS与GOS可通过增加肠道中的双歧杆菌从而促进健康[6]。随着测序技术的发展,基于肠道微生态研究益生元效应显得尤为重要。Jeroen等[7]报道第一个基于高通量测序的随机对照研究来探索益生元对于便秘人群肠道菌群的影响,发现益生元干预4周未改变肠道菌群的多样性,但特异性增殖了双歧杆菌。Ellen等[8]报道12周的GOS干预特异性增殖双歧杆菌但未影响宿主的血糖等代谢指标。此外,有研究报道短期的膳食干预可快速并可逆地改变肠道菌群的多样性[9]。但目前尚未有关于短期FOS或GOS补充对人体肠道菌群结构与功能的影响的研究。同时这种影响是否受自身菌群的影响也尚未有定论。
因此,我们选用FOS与GOS两种经典益生元进行短期干预,观察两种益生元对健康人肠道微生态的影响,并通过机器学习方法进一步探索益生元的个体化响应,为通过膳食靶向调整肠道菌群、实现精准膳食提供一定的参考依据。
1 资料和方法 1.1 对象招募志愿者纳入标准为:①BMI≥18 kg/m2,②无严重胃肠道疾病,③无严重心脑血管系统疾病,④无严重内分泌疾病;排除标准为:①妊娠期哺乳期妇女,②近1月内服用过益生元或益生菌或合生元制剂,③近3月内服用过抗生素。纳入的志愿者中,所有志愿者均为健康人。最终35名志愿者入组并完成研究。
1.2 伦理审查以及临床试验注册本研究经中国临床试验中心伦理审查委员会批准,伦理审查批号为ChiECRCT-20160021,临床试验注册号为ChiCTR-IPR-16008460。所有志愿者均签署了书面版知情同意书。
1.3 研究设计本研究是一项双盲随机自身对照试验,受试者被随机分到FOS组与GOS组,随机方法为随机数字表法。两个干预组分别每天接受16 g FOS或GOS干预,干预时间为9 d。低聚果糖与低聚半乳糖的成分与来源见表 1。分别第0、5、9天采集粪便标本,FOS干预3个时间点定义为FD0、FD5与FD9,GOS干预3个时间点定义为GD0、GD5与GD9。在干预开始2 d,我们建议志愿者每天服用8 g,以适应益生元并减少胃肠道的不适反应。益生元用不透明的牛皮纸袋包装并分发给志愿者。志愿者可将益生元添加到日常饮品,如牛奶、茶、咖啡或水中。在整个研究期间,志愿者保持原有的饮食习惯与生活方式。
| 表 1 FOS的结构与来源 Table 1 Characteristics of FOS and GOS used in the study |
用欧姆龙人体体成分分析仪(Omron HBF-701)测量体质量、体脂率、基础代谢率(BMR)指标,并计算BMI。
1.5 肠道菌群DNA提取与PCR粪便标本总DNA提取:用深圳易瑞磁珠法试剂盒在DNA自动提取仪(杭州奥盛仪器有限公司)进行粪便总DNA提取。再以获得的细菌DNA为模板进行细菌16S rRNA基因V4可变区PCR扩增。V4上游引物F-5'GTGTGCCAGCMGCCGCGGTAA3'和V4下游引物R-5'CCGGACTACHVGGGTWTCTAAT3',本研究使用带有barcode的Ion torrent引物。进行30个PCR循环扩增DNA。
1.6 菌群数据测序与结构分析采用Ion Torrent PCM测序仪、Hi-QTM测序试剂以及318 V2芯片进行测序。测序得到的原始数据和reads进行去除接头序列、低复杂度序列和低质量序列的处理,得到高通量原始碱基序列,采用BIPES分析流程[10]进行质量控制。用QIIME1.9.1(Quantitative Insights Into Microbial Ecology)[11]进行菌群多样性分析,将相似性大于97%的序列划分为一个分类操作单元(OTUs),OTU是指满足一定距离关系的序列,视为同一个分类级别的微生物。菌群多样性分析包括菌群α多样性分析、β多样性分析与组间差异菌线性判别分析[12](LEfSe)。肠道菌群α多样性用于反映肠道菌群丰富度,其中包括Shannon指数、Chao 1、Phylogenetic diversity whole tree(PD_whole_tree)与Observed OTUs指标,Shannon指数用于计算群落中的所有物种数及平均度;Chao1以及Observed OTUs用来描述物种的丰富度,而PD whole tree用于比较进化关系上组间种属的多样性。β多样性通过Unweighted unifrac距离与Weighted unifrac距离展示。
1.7 肠道菌群功能预测分析运用PICRUSt[13](Phylogenetic Investigation of Communities by Reconstruction of Unobserved States)通过16sRNA的基因序列预测宏基因组的功能。通过与京都基因和基因组百科全书(KEGG)数据库比对,基于预测基因组,计算KEGG基因、通路或者模块,再进行统计学分析。
1.8 机器学习建立预测模型将双歧杆菌的变化差值(△Bifidobacterium)按照四分位间距区分,定义上、下四分位分别为升高组和降低组,①用随机森林的方法建模区分升高组与降低组,并找出对双歧杆菌改变的贡献OTU,②判定OTU性质:若某个OTU在increase组的数量均值大于decrease组的数量均值则判定为positive OTU,若某个OTU在increase组数量均值小于decrease组的数量均值则判定为negative OTU,③通过贡献OTU构建连续性指数(index),构建指数的方法为:Index=∑ OTU数量(positive)-∑OTU数量(negtive),如:随机森林模型的贡献OTU为OTU 3294726 Faecalibacterium. prausnitzii与OTU 187219 Lachnospiraceae,根据判定规则将OTU 3294726判定为positive OTU,将OTU 187219判定为阴性OTU,在A样品中,OTU 3294726数目为173条,OTU 187219数目为30条,则index=173-30。依次类推,计算所有人的index,并与双歧杆菌差值做关联性分析,并用GOS干预第9天的微生物数据验证模型的准确性。
1.9 统计方法身体测量指标数据用均数±标准差表示,采用R 3.0.2软件与SPSS20.0作数据统计。参数资料运用配对t检验进行前后对比。大于两组的非参数资料用Fridman检验,而后用wilcoxon配对检验进行两两比较。用pearson进行关联性分析。R软件作图。P < 0.05,有统计学差异。
2 结果 2.1 一般临床资料描述本研究共35名健康人参加,其中男性10人,女性25人,18人接受FOS干预,17人接受GOS干预,所有志愿者完成研究(试验设计见图 1)。身体测量指标结果见表 2,结果显示,经过9 d的干预,FOS与GOS对志愿者体质量、BMI、体脂率以及基础代谢率均无显著影响(P > 0.05)。
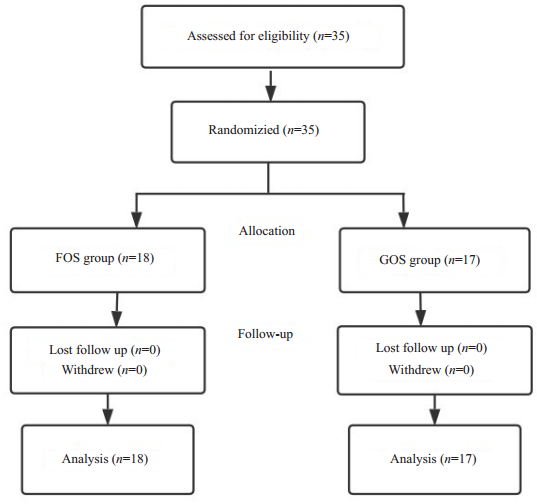
|
图 1 研究设计图 Figure 1 Flow chart of subject recruitment, allocation, follow-up and analysis. |
| 表 2 志愿者身体测量指标情况 Table 2 Characteristics of the study participants |
通过门水平与属水平的堆积图发现:在门水平上(图 2A、C),FOS与GOS干预前(FD0与GD0)的肠道菌群主要以拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)为主。在FOS与GOS干预第5、9天(FD5与GD5、FD9与GD9),肠道菌群主要以拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)以及放线菌门(Actinobacteria)为主;在属水平上(图 2B、D),FOS与GOS干预前(FD0与GD0)的肠道菌群主要以拟杆菌属(Bacteroides)、普氏菌属(Prevotella)、柔嫩梭菌(Faecalibacterium)以及瘤胃球菌(Ruminococcaceae)为主,在FOS与GOS干预第5、9天(FD5与GD5、FD9与GD9),肠道菌群以拟杆菌属(Bacteroides)、普氏菌属(Prevotella)、柔嫩梭菌(Faecalibacterium)以及瘤胃球菌(Ruminococcaceae)与双歧杆菌属(Bifidobacterium)等为主。
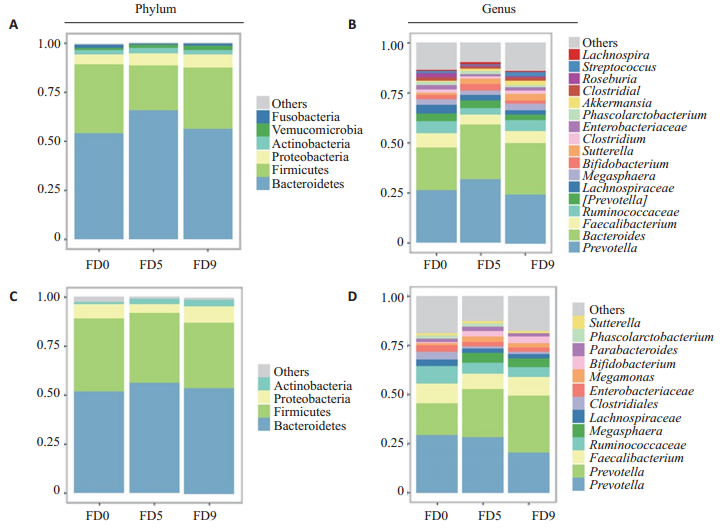
|
图 2 肠道菌群构成 Figure 2 Taxonomic summary of the gut microbiota at phylum and genus level. A: Phylum level in FOS group; B: Genus level in FOS group; C: Phylum level in GOS group; D: Genus level in GOS group. |
肠道菌群α多样性分析显示:两种益生元干预期间,均出现α多样性下降。与干预前相比,FOS干预第5天即出现Shannon指数(P < 0.01,图 3A)与PD_whole_tree(P < 0.01,图 3B)下降。在GOS干预组中,干预第9天与干预前相比,Shannon指数(P < 0.05,图 3E)、PD_whole_tree(P < 0.05,图 3F)、Chao1(P < 0.05,图 3G)与Observed OTUs(P < 0.05,图 3H)出现下降,而且这种现象在第5天时已经发生。
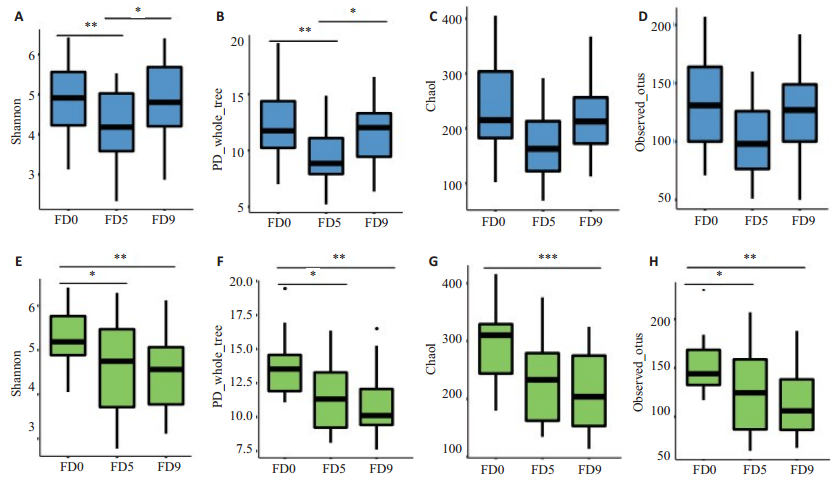
|
图 3 肠道菌群α多样性比较 Figure 3 Comparison of α-diversity in FOS and GOS group. A: Comparison Shannon index among FD0, FD5 and FD9; B: Comparison Chao1 among FD0, FD5 and FD9; C: Comparison PD whole tree among FD0, FD5 and FD9; D: Comparison observed otus among FD0, FD5 and FD9; E: Comparison Shannon index among GD0, GD5 and GD9; F: Comparison Chao1 among GD0, GD5 and GD9, G: Comparison PD whole tree among GD0, GD5 and GD9; H: Comparison observed_otus among GD0, GD5 and GD9. Data are median (IQR). (*P < 0.05; **P < 0.01, ***P < 0.005, Wilcoxon matched-pairs signed rank test). |
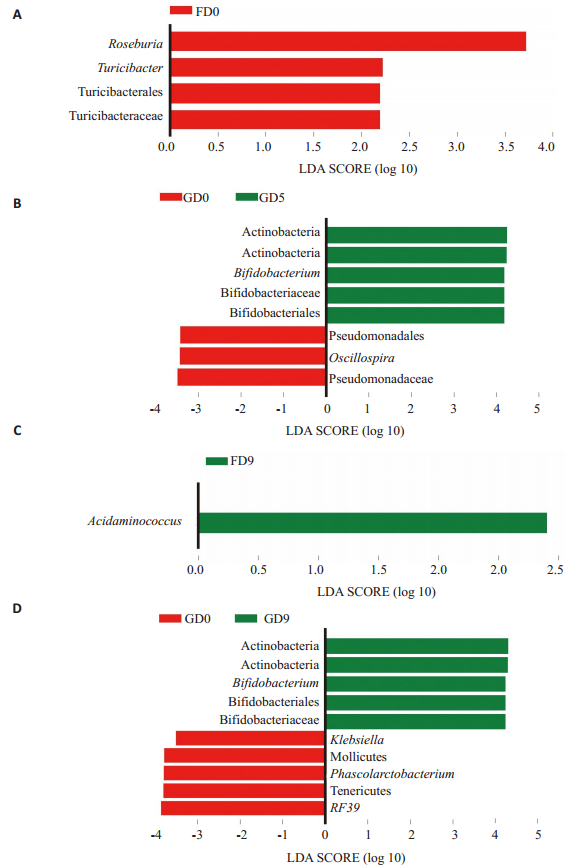
基于unweighted unifrac与weighted unifrac距离的主坐标分析(PCoA)发现:两种益生元干预对肠道菌群整体结构无显著影响(adonis,P > 0.05,图 4A~D)。进一步,我们对菌群进行LEfSe(LDA > 2)分析发现:与干预前相比,FOS干预第5天,罗斯氏菌属(Roseburia)以及Turicibacter下的Turicibacteraceae下降(图 5A),GOS干预第5天,双歧杆菌属(Bifidobacterium)显著升高,同时伴随着考拉杆菌属(Phascolarctobacterium),颤螺菌属(Oscillosira)与假单胞菌科(Pseudomonadaceae)的下降(图 5B);在第9天时,FOS干预只升高氨基酸球菌属(Acidaminococcus)(图 5C),但在GOS干预组中发现双歧杆菌属(Bifidobacterium)显著升高的同时伴随着考拉杆菌属(Phascolarctobacterium)、克雷伯氏菌属(Klebsiella)、柔膜菌纲(Mollicutes),柔膜菌门(Tenericutes)和RF39的下降(图 5D)。
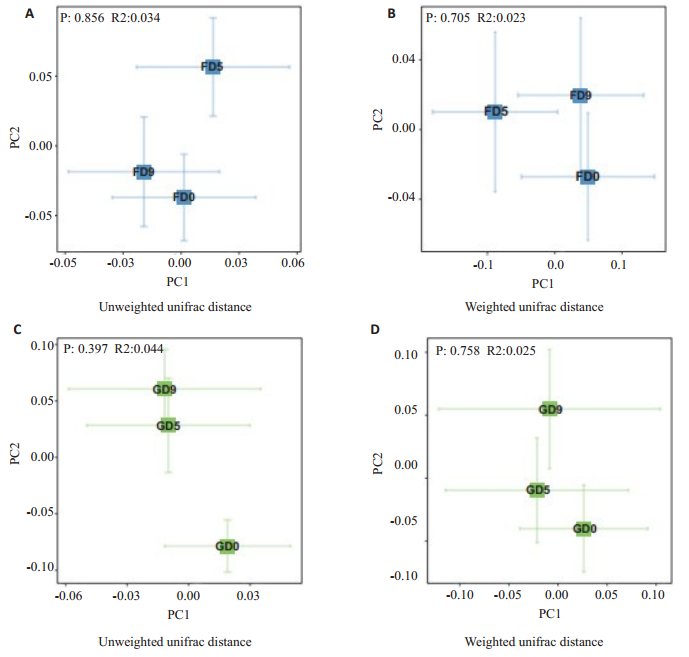
|
图 4 肠道菌群β多样性比较 Figure 4 Principal coordinate analysis (PCoA) based on unweighted and weighted UniFrac distances. A: Unweighted UniFrac distances in FOS group; B: Weighted UniFrac distances in FOS group; C: Unweighted UniFrac distances in GOS group; D: Weighted UniFrac distances in FOS group. Data are mean ± SEM (Adonis test). |
|
图 5 LEfSe统计比较各组间差异菌属 Figure 5 Differential gut bacterial determined by linear discriminant analysis effect size (LEfSe). |
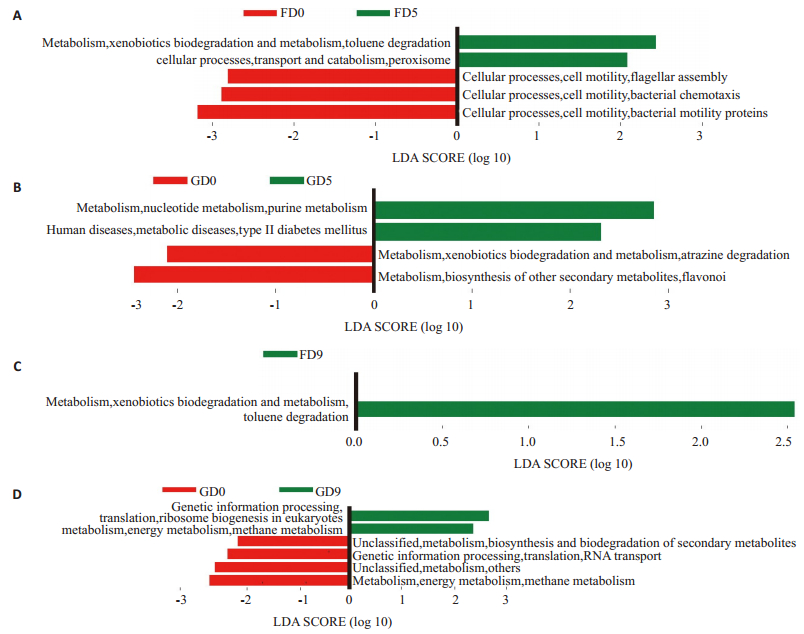
通过PICRUSt(LDA > 2)分析发现,在益生元干预5 d后,FOS组表现为分解甲苯的能力以及分解代谢过氧化物酶的能力增强,细菌的运动能力减弱,包括细菌的鞭毛组装能力、趋向性与细菌运动型蛋白(图 6A);GOS干预5 d后,嘌呤代谢能力增强,阿拉特津分解能力与类黄酮合成能力减弱(图 6B);在益生元干预9 d后,FOS组细菌功能的改变只体现在增强分解甲苯的能力(图 6C),在GOS组,益生元干预增强氨基酸代谢能力与真核生物的核糖体生物起源的翻译能力,降低次生代谢物的生物合成和生物降解的能力、RNA转运能力与甲烷代谢能力(图 6D)。
|
图 6 PICRUSt分析肠道菌群功能差异性 Figure 6 KEGG pathways analysis by PICRUSt in FOS and GOS groups. |
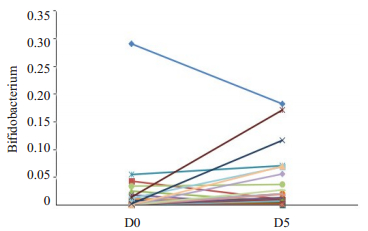
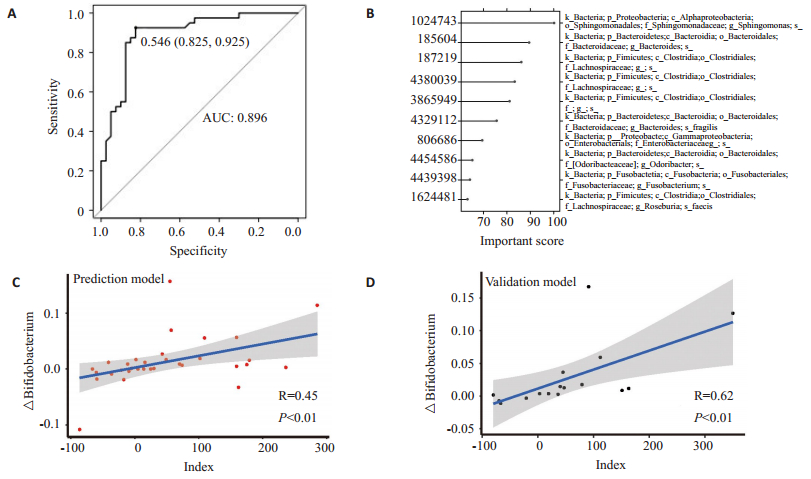
在FOS与GOS干预过程中,结果显示肠道菌群α多样性在干预5 d时已显著改变,并且通过LEfSe分析显示益生元干预5 d主要升高了双歧杆菌,因此我们进一步观察双歧杆菌的变化情况,发现,在5 d的益生元干预后,双歧杆菌的变化呈现升降不一的现象(图 7),共有20人双歧杆菌属升高,其中GOS组占12人,FOS组占8人;共有8人双歧杆菌属降低,其中GOS组占5人,FOS组占3人,有4人双歧杆菌改变量为0,均属FOS组。进一步,我们将益生元干预5 d后双歧杆菌变化量按照四分位间距划分,将上、下四分位间距分别定义为升高响应组和降低响应组。共有8人为升高响应组,双歧杆菌改变量均为正值,8人为降低响应组,双歧杆菌改变量均为负值,16个人为响应平衡组,用初始的肠道菌群(OTU水平)通过随机森林的方法对两组实现建模区分,结果显示:ROC曲线下面积为89.6%(图 8A),并且,通过贡献因素排序可以发现,对模型贡献的前10个OTU为:鞘氨醇单胞菌(OTU 1024743 Sphingomonas)、拟杆菌属(OTU 185604 Bacteroides)、毛螺旋菌科(OTU 187219 Lachnospiraceae)、毛螺旋菌科(OTU 4380039 Lachnospiraceae)、梭菌目(OTU 3865949 Clostridiales)、脆弱拟杆菌(OTU 4329112 Bacteroides fragilis)、肠杆菌科(OTU 806686 Enterobacteriaceae)、OTU4454586 Odoribacter、梭杆菌属(OTU 4439398 Fusobacterium)与罗斯氏菌(OTU 1624481 Roseburia faecis)(图 8B)。进一步,用所有贡献OTU建立连续型index,并计算每个人的index,将index与双歧杆菌变化量做关联,结果显示:建立的指数与双歧杆菌变化量的呈正相关,r值为0.45(P=0.01)(图 8C)。进一步,为了验证规律的准确性,首先对GOS干预9 d后的双歧杆菌改变量按四分位间距划分,结果显示分组情况与第5天双歧杆菌改变分组显著不同,因此可选用GOS干预9 d时双歧杆菌的变化量作为验证样本,样本量为16人,同理,用建模所用的贡献菌变化GD9d双歧杆菌的index,并用index跟实际的差值做关联,实现验证,最终验证模型r值为0.62(P=0.01,图 8D)。该结果支持上述模型的可靠性。
|
图 7 双歧杆菌改变情况 Figure 7 Change of Bifidobacterium among subjects after 5 days of prebiotics intervention. |
|
图 8 随机森林方法建立模型与模型验证 Figure 8 Prediction model and the validation model random forest. A: Receiver operating characteristic curve (ROC) of classification of the Bifidobacterium changes based only on initial microbiome data (AUC=89.6%); B: Important factors attributed to changes of Bifidobacterium; C: Correlation between index and changes of Bifidobacterium after 5 days FOS and GOS supplement in prediction model; D: Correlation between index and changes of Bifidobacterium after 9 days GOS supplement in validation model. |
本研究通过两种不同的益生元对健康人进行短期的干预,发现益生元干预5 d即可改变肠道菌群结构,降低肠道菌群的α多样性,FOS组在干预5 d后α多样性呈现急剧下降,第9天已有回升。GOS干预组在5 d时α多样性亦下降,9 d时下降仍在持续。GOS干预5 d后即可显著升高双歧杆菌。同时,用干预前的肠道菌群通过随机森林方法建模可区分益生元干预后双歧杆菌的变化效应,通过模型构建连续型指数可成功预测益生元干预后双歧杆菌的变化情况。
早期关于益生元对肠道菌群影响的研究是基于非测序技术进行,这可能不能全面地揭示益生元对肠道微生态的影响。而基于高通量测序技术研究可全面揭示益生元对肠道菌群结构的影响,这符合新的益生元定义:益生元是一类可被宿主微生物选择性利用并产生健康效应的物质[14]。益生元研究不应该只关注于双歧杆菌或乳酸菌增殖效应,同时应该着眼于肠道微生态结构与功能[15]。并且有研究表明,7 d的短期膳食干预即可显著影响肠道菌群多样性[9]。因此,本研究设定一个较短的干预周期来进行干预。在研究过程中,FOS与GOS干预均呈现降低肠道菌群α多样性的结果。目前尚未有直接的证据论证菌群α多样性下降对健康的影响。日本学者报道一项2周的GOS干预显著升高双歧杆菌,未出现副作用的同时降低健康婴儿肠道菌群α多样性的研究,但未阐明原因[16]。Metere等[17]研究表明,肥胖的孕妇肠道菌群α多样性出现下降。益生元降低肠道菌群多样性可能是由于选择性地增殖双歧杆菌抑制其他条件致病菌生长造成[18]。我们进一步深入探索益生元干预对健康成年人肠道微生物产生的影响,通过LEfSe分析发现,9 d的FOS干预对微生物几乎影响不大。而GOS干预显著升高双歧杆菌,这与大多数益生元研究报道相似[19-21],近期Ellen等研究者的研究表明,12周的GOS干预可显著升高糖尿病前期病人的双歧杆菌丰度,且不影响其他代谢指标[8]。同时,我们的结果显示:GOS干预在升高双歧杆菌的同时,降低考拉菌属,考拉菌属是一类产丙酸、丁酸菌[22],在一项糖尿病患者的研究中发现产丁酸菌显著降低[4],同时,也有文献报道产丁酸的菌降低,可能不利于维护肠屏障稳定,导致内毒素入血[23-24],引发肥胖、糖尿病等一系列疾病的发生[25]。由于我们的研究未检测血糖、血脂等其他指标,因此,FOS与GOS干预降低的产丁酸菌对宿主产生了何种影响仍旧未知,值得进一步探索。服用FOS可特异性增殖双歧杆菌,4周的益生元干预显著升高双歧杆菌,降低嗜胆菌属[7]。而本次研究发现FOS干预9 d未显著改变双歧菌属与嗜胆菌属,这可能与本次研究干预较短不足以体现低聚果糖的效应有关,也可能是由于不同地区人群肠道菌群存在差异导致。
双歧杆菌是重要的益生菌,婴儿时期的肠道菌群主要以双歧杆菌构成为主,补充双歧杆菌,能促进母乳低聚糖的利用[26],缺乏双歧杆菌,婴儿发生过敏的风险提高[27]。在成人中,双歧杆菌也发挥着重要的功能,双歧杆菌利用多糖产生短链脂肪酸(主要为乙酸、丙酸与丁酸)以及乳酸,短链脂肪酸可为肠上皮提供能量[28],同时双歧杆菌产生的短链脂肪酸与乳酸可降低肠道pH值,抑制致病菌的增长[29],有文献报道,补充益生菌可以增强免疫功能,增强肿瘤治疗效果[18]。补充益生元被认为是增殖双歧杆菌的有效方式,但在本次研究中,发现益生元干预5 d后,双歧杆菌显著增加,但并非统一增加,存在个体化差异。揭示双歧杆菌变化的个体化差异可能有助于靶向调整双歧杆菌,实现益生元干预精准化。有研究显示,肠道菌群的本底值可能会影响宿主对膳食干预的响应[30],这可能说明双歧杆菌的变化受肠道菌群本底值或初始肠道微生态环境影响,为用基线的肠道菌群研究机体对益生元的效应提供线索。因此,用初始的肠道菌群OTU数据,通过随机森林方法建模预测双歧杆菌的变化量,结果显示ROC曲线下面积达89.6%。黄适等通过随机森林的模型,通过口腔微生物数据建立index预测牙龈炎发展以及预后[31]。在随机森林模型中,我们发现区分双歧杆菌变化的贡献的细菌并非双歧杆菌,益生元种类对双歧杆菌改变量无显著贡献。在所有贡献的OTU中,OTU 187219 Lachnospiraceae、OTU 4380039 Lachnospiraceae、OTU 3865949 Clostridiales与OTU 1624481 Roseburia faecis属梭菌目下的细菌,这类细菌是常见的产丁酸菌[32],而双歧杆菌与一些产丁酸菌存在互养关系,体现在双歧杆菌分解多糖产生乙酸与乳酸,罗斯氏菌、柔嫩梭菌等可以利用双歧杆菌的代谢产物产生丁酸[33-34]。在模型中,梭菌目下的OTU产生贡献的原因可能是与双歧杆菌产生互养关系引起。利用贡献OTU对GOS干预第9天双歧杆菌变化量进行验证,结果显示验证效果良好。这说明初始的肠道菌群能准确预测服用益生元后的双歧杆菌的变化。此前,Petia根据摄入膳食纤维后血糖变化量分组,发现不同血糖变化量的个体其初始肠道菌群存在差异[35],此项研究为我们定义双歧杆菌变化分组提供了参考。通过初始的微生物数据预测机体对不同膳食的代谢情况已经应用与许多研究中David [36]和Eran [37]均发现宿主对膳食的响应受初始的肠道菌群的影响较大,用初始的肠道菌群预测效果良好。基于初始肠道菌群建模预测的方法可能为未来实现精准膳食提供了较好的策略。同时,本研究提供的模型,为未来开展大规模人群肠道菌群评估,以及在人群水平开展益生元干预的效能预测提供依据。
本研究通过低聚果糖与低聚半乳糖两种益生元对健康人进行短期干预,评估益生元干预9 d对肠道菌群结构与功能的影响并挖掘双歧杆菌的变化影响因素,通过机器学习方法建立预测模型可有效预测双歧杆菌的变化,可用于靶向与精准调节双歧杆菌,实现益生元精准干预。同时,基于肠道微生物的机器学习建模方式,为实现精准膳食提供了新视角。
| [1] |
Sender R, Fuchs S, Milo R. Are we really vastly outnumbered? revisiting the ratio of bacterial to host cells in humans[J].
Cell, 2016, 164(3): 337-40.
DOI: 10.1016/j.cell.2016.01.013. |
| [2] |
Ridaura VK, Faith JJ, Rey FE, et al. Gut microbiota from twins discordant for obesity modulate metabolism in mice[J].
Science, 2013, 341(6150): 1241214.
DOI: 10.1126/science.1241214. |
| [3] |
Wang Z, Klipfell E, Bennett BJ, et al. Gut flora metabolism of phosphatidylcholine promotes cardiovascular disease[J].
Nature, 2011, 472(7341): 57-63.
DOI: 10.1038/nature09922. |
| [4] |
Karlsson FH, Tremaroli V, Nookaew I, et al. Gut metagenome in European women with normal, impaired and diabetic glucose control[J].
Nature, 2013, 498(7452): 99-103.
DOI: 10.1038/nature12198. |
| [5] |
Walker AW, Parkhill J. Fighting obesity with bacteria[J].
Science, 2013, 341(6150): 1069-70.
DOI: 10.1126/science.1243787. |
| [6] |
Roberfroid M, Gibson GR, Hoyles L, et al. Prebiotic effects: metabolic and health benefits[J].
Br J Nutr, 2010, 104(Suppl 2): S1-63.
|
| [7] |
Vandeputte D, Falony G, Vieira-Silva S, et al. Prebiotic inulin-type fructans induce specific changes in the human gut microbiota[J].
Gut, 2017, 66(11): 1968-74.
DOI: 10.1136/gutjnl-2016-313271. |
| [8] |
Canfora EE, Van Der Beek CM, Hermes G, et al. Supplementation of Diet with galacto-oligosaccharides increases bifidobacteria, but not insulin sensitivity, in obese prediabetic individuals[J].
Gastroenterology, 2017, 153(1): 87-97.
DOI: 10.1053/j.gastro.2017.03.051. |
| [9] |
David LA, Maurice CF, Carmody RN, et al. Diet rapidly and reproducibly alters the human gut microbiome[J].
Nature, 2014, 505(7484): 559-63.
DOI: 10.1038/nature12820. |
| [10] |
Zhou HW, Li DF, Tam NF, et al. BIPES, a cost-effective highthroughput method for assessing microbial diversity[J].
ISME Journal, 2011, 5(4): 741-9.
DOI: 10.1038/ismej.2010.160. |
| [11] |
Caporaso JG, Kuczynski J, Stombaugh J, et al. QⅡME allows analysis of high-throughput community sequencing data[J].
Nat Methods, 2010, 7(5): 335-6.
DOI: 10.1038/nmeth.f.303. |
| [12] |
Segata N, Izard J, Waldron L, et al. Metagenomic biomarker discovery and explanation[J].
Genome Biol, 2011, 12(6): R60.
DOI: 10.1186/gb-2011-12-6-r60. |
| [13] |
Langille MG, Zaneveld J, Caporaso J, et al. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences[J].
Nat Biotechnol, 2013, 31(9): 814.
DOI: 10.1038/nbt.2676. |
| [14] |
Gibson GR, Hutkins R, Sanders ME, et al. The international scientific association for probiotics and prebiotics (ISAPP) consensus statement on the definition and scope of prebiotics[J].
Nat Rev Gastroenterol Hepatol, 2017, 14(8): 491-502.
|
| [15] |
Bindels LB, Delzenne NM, Cani PD. Towards a more comprehensive concept for prebiotics[J].
Nat Rev Gastroenterol Hepatol, 2015, 12(5): 303-10.
DOI: 10.1038/nrgastro.2015.47. |
| [16] |
Matsuki T, Tajima S, Hara T, et al. Infant formula with galactooligosaccharides(OM55N) stimulates the growth of indigenous bifidobacteria in healthy term infants[J].
Benef Microbes, 2016, 7(4): 453-61.
DOI: 10.3920/BM2015.0168. |
| [17] |
Stanislawski MA, Dabelea D, Wagner BD, et al. Pre-pregnancy weight, gestational weight gain, and the gut microbiota of mothers and their infants[J].
Microbiome, 2017, 5(1): 113.
DOI: 10.1186/s40168-017-0332-0. |
| [18] |
Slavin J. Fiber and prebiotics: mechanisms and health benefits[J].
Nutrients, 2013, 5(4): 1417-35.
DOI: 10.3390/nu5041417. |
| [19] |
Meyer D, Stasse-Wolthuis M. The bifidogenic effect of inulin and oligofructose and its Consequences for gut health[J].
Eur J Clin Nutr, 2009, 63(11): 1277-89.
DOI: 10.1038/ejcn.2009.64. |
| [20] |
Wang LL, Hu LJ, Yan S, et al. Effects of different oligosaccharides at various dosages on the composition of gut microbiota and shortchain fatty acids in mice with constipation[J].
Food Funct, 2017, 8(5): 1966-78.
DOI: 10.1039/C7FO00031F. |
| [21] |
Azcarate-Peril MA, Ritter A, Savaiano DA. Impact of Short-Chain galactooligosaccharides on the gut microbiome of lactose intolerant individuals[J].
Gastroenterology, 2016, 150(4, 1): S424.
|
| [22] |
Zhang X, Fang Z, Zhang C, et al. Effects of acarbose on the gut microbiota of prediabetic patients: a randomized, double-blind, controlled crossover trial[J].
Diabetes Ther, 2017, 8(2): 293-307.
DOI: 10.1007/s13300-017-0226-y. |
| [23] |
Flint HJ, Duncan SH, Scott KP, et al. Links between diet, gut microbiota composition and gut metabolism[J].
Proc Nutr Soc, 2015, 74(1): 13-22.
DOI: 10.1017/S0029665114001463. |
| [24] |
Canfora EE, Jocken JW, Blaak EE. Short-chain fatty acids in control of body weight and insulin sensitivity[J].
Nat Rev Endocrinol, 2015, 11(10): 577-91.
DOI: 10.1038/nrendo.2015.128. |
| [25] |
Bron PA, Kleerebezem M, Brummer R, et al. Can probiotics modulate human disease by impacting intestinal barrier function? J][J].
Br J Nutr, 2017, 117(1): 93-107.
DOI: 10.1017/S0007114516004037. |
| [26] |
Da SL, Chapman J, Adeuya A, et al. The genome sequence of Bifidobacterium longum subsp.infantis reveals adaptations for milk utilization within the infant microbiome[J].
Proc Natl Acad Sci U S A, 2008, 105(48): 18964-9.
DOI: 10.1073/pnas.0809584105. |
| [27] |
Fujimura KE, Sitarik AR, Haystad S, et al. Neonatal gut microbiota associates with childhood multisensitized atopy and T cell differentiation[J].
Nat Med, 2016, 22(10): 1187-91.
DOI: 10.1038/nm.4176. |
| [28] |
Donohoe DR, Garge N, Zhang X, et al. The microbiome and butyrate regulate energy metabolism and autophagy in the mammalian colon[J].
Cell Metab, 2011, 13(5): 517-26.
DOI: 10.1016/j.cmet.2011.02.018. |
| [29] |
Sivan A, Corrales L, Hubert N, et al. Commensal bifidobacterium promotes antitumor immunity and facilitates anti-PD-L1 efficacy[J].
Science, 2015, 350(6264): 1084-9.
DOI: 10.1126/science.aac4255. |
| [30] |
Venkataraman A, Sieber JR, Schmidt AW, et al. Variable responses of human microbiomes to dietary supplementation with resistant starch[J].
Microbiome, 2016, 4(1): 33.
DOI: 10.1186/s40168-016-0178-x. |
| [31] |
Huang S, Li R, Zeng XW, et al. Predictive modeling of gingivitis severity and susceptibility via oral microbiota[J].
ISME J, 2014, 8(9): 1768-80.
DOI: 10.1038/ismej.2014.32. |
| [32] |
Stearns JC, Zulyniak MA, De Souza RJ, et al. Ethnic and dietrelated differences in the healthy infant microbiome[J].
Genome Med, 2017, 9(1): 32.
DOI: 10.1186/s13073-017-0421-5. |
| [33] |
Bottacini F, Van Sinderen D, Ventura M. Omics of bifidobacteria: research and insights into their health-promoting activities[J].
Biochem J, 2017, 474(24): 4137-52.
DOI: 10.1042/BCJ20160756. |
| [34] |
Turroni F, Milani C, Duranti SA, et al. Bifidobacteria and the infant gut: an example of co-evolution and natural selection[J].
Cell Mol Life Sci, 2018, 75(1): 103-18.
DOI: 10.1007/s00018-017-2672-0. |
| [35] |
Kovatcheva-Datchary P, Nilsson A, Akrami R, et al. Dietary FiberInduced improvement in glucose metabolism is associated with increased abundance of prevotella[J].
Cell Metab, 2015, 22(6): 971-82.
DOI: 10.1016/j.cmet.2015.10.001. |
| [36] |
Zeevi D, Korem T, Zmora N, et al. Personalized nutrition by prediction of glycemic responses[J].
Cell, 2015, 163(5): 1079-94.
DOI: 10.1016/j.cell.2015.11.001. |
| [37] |
Korem T, Zeevi D, Zmora N, et al. Bread affects clinical parameters and induces gut microbiome-associated personal glycemic responses[J].
Cell Metab, 2017, 25(6): 1243-53.
DOI: 10.1016/j.cmet.2017.05.002. |
 2018, Vol. 38
2018, Vol. 38

