2. 中国人民解放军广州总医院检验科,广东 广州 510010
2. Department of Laboratory Medicine, General Hospital of Guangzhou Military Command of PLA, Guangzhou 510010, China
艰难梭菌(CD)是广泛存在于自然界的革兰阳性专性厌氧芽孢杆菌[1]。正常情况下CD并不致病,当大量使用抗菌药物或人体免疫力低下时,栖生在人体肠道或通过粪-口途径传播的CD可引起轻至腹泻、重至中毒性巨结肠、结肠穿孔等临床症状,甚至可能导致死亡[2-3]。
近年来,艰难梭菌感染的准确快速诊断成为临床预防和治疗面临的问题之一。2010年美国医疗保健流行病学学会和传染病学会推出的临床实践指南推荐艰难梭菌感染(CDI)的实验室诊断标准为:粪便中检出产毒型CD或其毒素,或组织病理学证实为假膜性肠炎[4]。目前用于诊断CDI的实验室方法有培养法、酶免疫法及分子生物学方法[5-6]。我国仅少数医院开展CDI检测,方法主要为毒素A/B(CDAB)的酶免疫法检测[7]。但CDAB敏感性较低,无法满足临床准确诊断的需求。2001年国内就有关于普通PCR方法检测CD的文献报道,阳性率可高达46.7%,而当时的培养率仅为19.2%(7/36)[8]。随着技术的发展,q-PCR因其较高的灵敏度和特异性,已经开始用于结核分枝杆菌等的临床检测[9-10],而关于艰难梭菌感染的检测尚未普及。因此,本研究旨在以产毒株的培养为诊断标准,评价CDAB检测、GDH检测以及q-PCR检测艰难梭菌特异性基因tpi以及毒素基因(tcdA/tcdB)3种艰难梭菌感染检测方法的诊断效能,并提出灵敏度高、特异度强、快速经济的检测方案。
1 资料和方法 1.1 标本来源收集中国人民解放军广州总医院2016年5月~12月住院期间单日发生大于或等于3次水样便,临床诊断疑似艰难梭菌感染的住院患者粪便标本70例,采用酶联荧光分析检测CDAB和GDH、q-PCR法检测特异性基因tpi和毒素相关基因,以产毒株培养作为判断艰难梭菌感染的金标准。
1.2 试剂与仪器VIDAS CDAB/GDH试剂盒及VIDAS系列荧光免疫分析仪、艰难梭菌显色培养基ChromID、MALDITOF MS Vitek-MS(生物梅里埃);粪便基因组DNA提取试剂盒(天根生化);厌氧培养系统(尤德);PCR扩增仪(BIO.RAD);哥伦比亚血琼脂平板(环凯微生物);艰难梭菌ATCC-BAA-2155(ATCC菌种库),PCR试剂(Takara)。
1.3 粪便培养艰难梭菌适量粪便标本与无水乙醇1: 1体积混合,静置1 h后分别接种哥伦比亚血平板和艰难梭菌显色平板。置于厌氧罐中填充混合气体至0.1%含氧量,35 ℃温箱中培养48 h。根据血平板以及显色平板上菌落形态分离纯化可疑菌落,并进行耐氧实验筛选。
1.4 培养阳性菌株鉴定根据革兰染色镜下形态以及特殊马厩性气味进行初步筛选;利用基质辅助激光解吸电离飞行时间质谱(MADLI-TOF MS)对筛选出的可疑菌落进行菌种鉴定,并采用热激法提取细菌DNA扩增艰难梭菌特异性片段tpi和毒素基因(tcdA/tcdB)加以鉴定。PCR扩增体系如下:上下游引物各1 μL(10 μmol/L),模板1 μL,5× PCR mix 10 μL,去离子水7 μL。反应条件:94 ℃预变性30 s,94 ℃变性30 s,55 ℃退火30 s,72℃延伸1 min,30个循环之后72 ℃彻底延伸5 min。所用引物见表 1。
| 表 1 培养阳性菌株PCR艰难梭菌tpi、毒素基因(tcdA/tcdB)所用引物 Table 1 Primers for PCR of tpi and toxin gene (tcdA/tcdB) in Clostridium difficile (CD) |
取新鲜粪便标本200 g,分别加入CDAB和GDH标本稀释液1 mL,吹打混匀后离心取300 μL上清液于相应反应试剂条,上机测定。
CDAB结果检测值< 0.13为阴性;0.13≤检测值< 0.37为灰区,无法确诊,需结合患者的临床表征进行判断;≥0.37为阳性。GDH结果检测值< 0.1为阴性,>0.1为阳性。二者均按试剂说明书要求做定标和质控。
1.6 q-PCR法检测粪便艰难梭菌和毒素基因取180~220 mg粪便标本,参照粪便基因组DNA提取试剂盒说明书操作步骤,提取基因组DNA(gDNA)作为模板。采用TaqMan探针法定性三重qPCR检测tpi、tcdA和tcdB的存在。10 μL反应体系如下:2 × Master Mix 5.0 μL,上下游引物(10 μmol)各0.3 μL,模板(1 ng/μL)1.0 μL,去离子水3.4 μL。反应条件:95 ℃预变性5 min,95 ℃变性15 s,60 ℃退火30 s,72 ℃延伸15 s,40个循环后95 ℃ 15 s,60 ℃ 1 min,95 ℃ 15 s。结果判定标准:(1)tpi和tcdB的Ct≤40,为产毒型艰难梭菌阳性;(2)tpi≤38,tcdB无扩增,为不产毒型艰难梭菌阳性;(3)tpi的38 < Ct值≤40,tcdB无扩增,需加大浓度复测;(4)tpi和tcdB均无扩增,内标正常扩增,为艰难梭菌阴性;(5)tpi和tcdB均无扩增,内标Ct值>36或No Ct,为结果无效。所用引物见表 2。
| 表 2 粪便q-PCR艰难梭菌tpi、毒素基因(tcdA/tcdB)所用引物 Table 2 Primers for q-PCR of CD tpi and toxin gene (tcdA/tcdB) in stool specimens |
实验数据用SPSS 20.0进行数据分析,3种检测方法的阳性率比较采用配对卡方检验,以P < 0.05认为差异有统计学意义,并计算灵敏度、特异度、阳性预测值和阴性预测值。
2 结果 2.1 3种检测方法与参考标准检测结果本实验共收集疑似艰难梭菌感染腹泻粪便标本70例,经过培养法分离获得13株艰难梭菌,其中6例携带毒素基因,阴性标本57例(表 3);CDAB检测结果6例为阳性,3例灰区,61例为阴性(表 4);70例标本中20例GDH为阳性,50例阴性(表 3)。根据荧光定量PCR检测结果,tpi基因阳性标本共计17例(表 3),其中毒素基因tcdA/tcdB双阳性7例(表 4)。
| 表 3 GDH及q-PCR检测tpi与培养法结果的比较 Table 3 Comparison of GDH and q-PCR detection for tpi with culture method |
| 表 4 CDAB,q-PCR检测tcdA/tcdB与产毒株培养结果的比较 Table 4 Comparison of CDAB and q-PCR detection for tcdA/tcdB with culture of toxin-producing strains |
q-PCR(tpi)联合CDAB与GDH联合CDAB分别将产毒株培养作为检测标准进行比较,至少一种方法检测为阳性认为结果判断为阳性(表 5)。
| 表 5 q-PCR与GDH联合CDAB检测结果与产毒株培养比较 Table 5 Comparison of q-PCR and detection of both GDH and CDAB with culture of the toxin-producing strains |
以表 3~5的数据计算各检测方法的灵敏度,特异度,阳性预测值,阴性预测值及诊断符合率,各诊断指标最优并有统计学意义的加粗下划线标注(表 6、7)。
| 表 6 各实验室方法单独及联合检测的诊断学指标 Table 6 Diagnostic efficacy of the laboratory methods alone and in combination |
| 表 7 q-PCR与GDH和CDAB检测数据及统计学分析 Table 7 Statistical analysis of the results q-PCR, GDH and CDAB detection |
艰难梭菌在哥伦比亚血平板上面呈灰白色,边缘不规则菌落,随着暴露于空气中的时间增加,菌落颜色变得更灰,边缘深染(图 1A)。在显色平板上,艰难梭菌为黑色有光泽菌落,边缘不整齐(图 1B)。
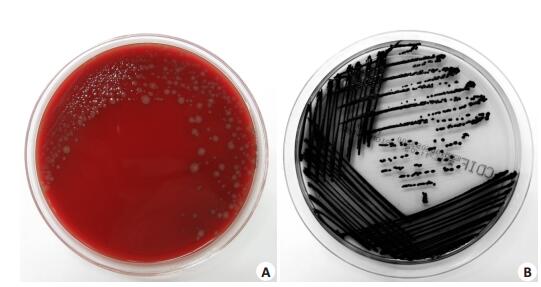
|
图 1 艰难梭菌在血平板上及显色平板上的菌落形态 Figure 1 Colony morphology of CD on blood plate (A) and chromogenic plate (B). |
艰难梭菌特异性基因tpi能辅助质谱确证分离的菌株为艰难梭菌,tcdA/B是艰难梭菌的毒素编码基因,提取13株MALDI-TOF MS鉴定为艰难梭菌的DNA,PCR扩增tpi和tcdA/tcdB(图 2)。13株均含有tpi基因,确为艰难梭菌,6株产毒株中有5株为A+B+型菌株,1株为A-B+型,其余均为不产毒菌株。
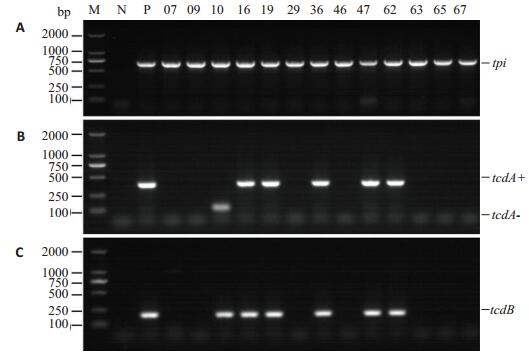
|
图 2 艰难梭菌tpi及毒素基因(tcdA/tcdB)扩增电泳图 Figure 2 Amplification and electrophoresis of CD tpi and toxin gene (tcdA/tcdB). M: DL2000 DNA Marker N: Negative control, H2O; P: Positive control, Clostridium difficile strain ATCC-BAA-2155. |
艰难梭菌感染(CDI)被认为是引起抗生素相关性腹泻的主要原因。尽管各级卫生防疫部门实行了一系列预防感染和降低细菌传播的措施,但艰难梭菌的感染率仍居高不下[11-13]。目前CDI的准确诊断是临床预防和治疗所面对的问题之一。
为此,本研究同时采用哥伦比亚血琼脂平板和艰难梭菌显色平板进行艰难梭菌的分离培养,对两种平板上的疑似菌落均采用MALDI-TOF MS进行准确鉴定作为判断其他方法的参考标准。在本研究收集的70例粪便标本中,我们以培养法鉴定艰难梭菌13株,包括6株产毒株和7株非产毒株,分离率达到18.57%。参考国内同类研究,2008年上海华山医院[14]从587份住院病人的粪便标本中检出56份艰难梭菌阳性的标本,分离率为9.4%;2009年北京的程颖等[14]从112份腹泻标本中检测出12株艰难梭菌,阳性率为10.7%;2013年广州医学院[16]从467例腹泻患者粪便标本中分离到艰难梭菌29例,阳性检出率6.2%;本课题组2014年至2015年在本院收集成人腹泻标本675份,培养出艰难梭菌57株,分离率为8.44% [17]。以上研究仅使用了常规血平板培养法,而本研究在联合血平板及显色平板后,以及MALDI-TOF MS的使用,使菌株分离率显著提高。
目前艰难梭菌感染检测最常见的方法是酶联荧光分析检测细菌毒素CDAB。本研究中粪便样本的CDAB检出率为8.57%(6/70),与国内同类研究的毒素检出率8.70%(8/92)[18]较为一致,显著低于国外报道的15.86%(88/555)[19]。另3例灰区标本需结合临床判断是否为艰难梭菌感染。6例阳性标本中有1例经培养未得到艰难梭菌,但分离出气荚膜梭菌。据文献报道,产气荚膜梭菌毒素TpeL的氨基酸序列与艰难梭菌毒素A(TcdA)和B(TcdB)具有30%~39%的同源性,且艰难梭菌和产气荚膜梭菌在肠道疾病中可能具有协同作用[20],因此我们将该粪便标本采用哥伦比亚血平板和艰难梭菌显色平板再次培养,依然仅获得产气荚膜梭菌。将获得的产气荚膜梭菌菌株培养48 h后检测细菌CDAB,测试值为0.27,判断为灰区。因此我们认为产气荚膜梭菌可能造成CDAB检测的假阳性结果。另外由于样本的保存与运送条件不当等容易造成蛋白降解或抗原变异,也可能造成检出率的偏差。
与此同时,谷氨酸脱氢酶是艰难梭菌表面大量存在的保守抗原,其稳定性和灵敏度较高,最大优势在于有很高的阴性预测值,通过Meta分析发现,GDH检测的阴性预测值在94.6%~100% [20]。本实验中GDH检测灵敏度和特异度分别为84.62%和84.21%,阴性预测值96.00%,与现有研究较为一致[23-24]。
最后,本实验采用q-PCR方法同步扩增艰难梭菌特异性基因tpi、毒素基因tcdA和tcdB,其中tpi阳性标本17例,毒素基因阳性7例,产毒型艰难梭菌阳性率为10%。本实验中扩增特异性基因tpi和毒素基因tcdA/ tcdB与标准方法比较一致性分别达到91.43%和92.86%,tpi检测的阴性预测值(98.11%)高于GDH(96.00%)检测,扩增毒素基因tcdA/tcdB的诊断价值显著优于CDAB。上海交通大学医学院附属瑞金医院2014年报道采用Xpert Clostridium difficile检测试剂盒检测艰难梭菌毒素基因tcdB,与培养法相比较敏感性、特异性、阳性预测值和阴性预测值分别为87.2%、92.2%、81.0%和94.9%,与该研究相比,二者在特异度上差异并不大,q-PCR的高特异性得以论证。我们同时扩增毒素基因tcdA和tcdB,可以用于筛查临床少见但不能忽略的A-B+型菌株[1]。艰难梭菌特异性基因tpi也有助于筛选携带非产毒艰难梭菌的标本,进行分离培养后可用于后续的科学研究。Etienne-Mesmin等[25]在研究小鼠艰难梭菌定殖与发病时发现q-PCR检测到的粪便中毒素基因水平与细胞毒性实验检测到的粪便毒素水平是相关的。综合二者,开展q-PCR方法检测艰难梭菌特异性基因tpi、毒素基因可满足临床快速准确诊断的需要。在本研究中,q-PCR方法仍然具有一定的假阳性出现,我们将进一步加大样本量进行验证,以及寻求特异性更高的引物序列方案。
我们还通过回顾性查阅病例发现:所有艰难梭菌培养阳性患者住院期间均长期使用质子泵抑制剂联合亚胺培南和替加环素等抗生素。大量研究也证实CDI发病与患者年龄(≥65岁)、住院时间、炎性反应肠病、使用免疫抑制剂、质子泵抑制剂及广谱抗生素等多种危险因素密切相关[26-27]。因此对具备以上诱发CDI因素的患者应加强对CDI的监测。对于疑似CDI患者,可利用qPCR法进行初筛。在满足临床快速诊断的前提下,可结合艰难梭菌显色平板的菌培养进行确诊。
总之,与其他检测方法相比,q-PCR检测粪便标本中的艰难梭菌具有时效性强、灵敏度高、特异性好等优点,适用于临床推广。
| [1] | Chen LD, Li LH, Liao Y, et al. Molecular characterization and antimicrobial susceptibility of tcdA-negative Clostridium difficile isolates from Guangzhou, China[J]. Diagn Microbiol Infect Dis, 2016, 84(4): 361-5. DOI: 10.1016/j.diagmicrobio.2015.12.015. |
| [2] | Telekes A. Oncologic aspects of Clostridium difficile[J]. Orv Hetil, 2016, 157(28): 1110-6. DOI: 10.1556/650.2016.30463. |
| [3] | Martin JS, Monaghan TM, Wilcox MH. Clostridium difficile infection: epidemiology, diagnosis and understanding transmission[J]. Nat Rev Gastroenterol Hepatol, 2016, 13(4): 206-16. DOI: 10.1038/nrgastro.2016.25. |
| [4] | Cohen SH, Gerding DN, Johnson S. Clinical practice guidelines for clostridium difficile infection in adults: 2010 update by the society for healthcare epidemiology of America(SHEA)and the infectious diseases society of America (IDSA)[J]. Infect Control Hosp Epidemiol, 2010, 31(5): 431-55. DOI: 10.1086/651677. |
| [5] | 章黎华, 李贞, 江岑, 等. 3种检测艰难梭菌方法的临床应用评估[J]. 微生物与感染, 2016, 11(1): 24-7. |
| [6] | Chen SY, Gu HW, Sun CL, et al. Rapid detection of Clostridium difficile toxins and laboratory diagnosis of Clostridium difficile infections[J]. Infection, 2017, 45(3): 255-62. DOI: 10.1007/s15010-016-0940-9. |
| [7] | 郭晓月, 黄磊. 艰难梭菌感染实验室诊断的研究进展[J]. 检验医学与临床, 2013, 20(10): 2763-5. |
| [8] | 关宏, 钱家鸣, 陆星华, 等. 用PCR方法检测粪便难辨梭状芽胞杆菌[J]. 临床消化病杂志, 2001, 13(04): 147-8. DOI: 10.3870/j.issn.1005-541X.2001.04.001. |
| [9] | 祖冬梅. 耐多药结核分枝杆菌多重Q-PCR快速检测方法的建立[D]. 广州: 华南理工大学, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10561-1013319353.htm |
| [10] | 王宇, 靳伟东, 兰凯, 等. 荧光定量PCR技术在临床微生物检测中的应用[J]. 中国卫生标准管理, 2015, 30(6): 151-2. |
| [11] | Rodriquez C, Van Broeck J, Taminiau B, et al. Clostridium difficile infection: Early history, diagnosis and molecular strain typing methods[J]. Microb Pathog, 2016, 97: 59-78. DOI: 10.1016/j.micpath.2016.05.018. |
| [12] | Nicholas A, Kim YK, Lee WK, et al. Molecular epidemiology and antimicrobial susceptibility of Clostridium difficile isolates from two Korean hospitals[J]. PLoS One, 2017, 12(3): e174716. |
| [13] | Shea KM, Hobbs ALV, Jaso TC, et al. Effect of a healthcare-system respiratory fluoroquinolone restriction program to alter utilization and impact rates of Clostridium difficile infection[J]. Antimicrob Agents Chemother, 2017, 61(6): e00125-17. |
| [14] | Huang H, Wu S, Wang M, et al. Molecular and clinical characteristics of Clostridium difficile infection in a University Hospital in Shanghai, China[J]. Clin Infect Dis, 2008, 47(12): 1606-8. |
| [15] | 程颖, 卢金星, 鄢盛恺, 等. 临床分离艰难梭菌毒素携带特征研究[J]. 疾病监测, 2009, 24(03): 193-5. DOI: 10.3784/j.issn.1003-9961.2009.03.15. |
| [16] | 唐海先. 腹泻患者艰难梭菌检测及分析[D]. 广州医科大学, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10570-1014076237.htm |
| [17] | 陈丽丹. 不同来源的艰难梭菌其毒素, MLST分型及药敏情况的分析[D]. 广州: 南方医科大学, 2016. http://d.g.wanfangdata.com.cn/Thesis_Y3116659.aspx |
| [18] | 李艳明, 王伟琦, 简子娟, 等. 住院腹泻患者艰难梭菌的检测和分析[C]. 中华医学会第十二次全国临床微生物学术年会暨第十一次全球华人临床微生物学与感染症学术年会, 甘肃兰州, 2015. http://d.wanfangdata.com.cn/Periodical/zggrkzzz201204014 |
| [19] | Shin BM, Lee EJ, Kuak EY, et al. Comparison of VIDAS CDAB and CDA immunoassay for the detection of Clostridium difficile in a tcdA(-) tcdB(+) C. difficile prevalent area[J]. Anaerobe, 2009, 15(6, SI): 266-9. DOI: 10.1016/j.anaerobe.2009.09.008. |
| [20] | Amimoto K, Noro T, Oishi E, et al. A novel toxin homologous to large clostridial cytotoxins found in culture supernatant of Clostridium perfringens type C[J]. Microbiology, 2007, 153(Pt 4): 1198-206. |
| [21] | Diniz AN, Silva ROS, Oliveria Junior CA, et al. Clostridium perfringens type A netF and netE positive and Clostridium difficile co-infection in two adult dogs[J]. Anaerobe, 2016, 38: 94-6. DOI: 10.1016/j.anaerobe.2015.12.013. |
| [22] | Uzal FA, Diab SS, Blanchard P, et al. Clostridium perfringens type C and Clostridium difficile co-infection in foals[J]. Vet Microbiol, 2012, 156(3/4): 395-402. |
| [23] | Shetty N, Wren M, Coen PG. The role of glutamate dehydrogenase for the detection of Clostridium difficile in faecal samples: a metaanalysis[J]. J Hosp Infect, 2011, 77(1): 1-6. DOI: 10.1016/j.jhin.2010.07.024. |
| [24] | Crobach M, Dekkers OM, Wilcox MH, et al. European society of clinical microbiology and infectious diseases (ESCMID): data review and recommendations for diagnosing clostridium difficileinfection (CDI)[J]. Clin Microbiol Infect, 2009, 15(12): 1053-66. DOI: 10.1111/j.1469-0691.2009.03098.x. |
| [25] | Etienne-Mesmin L, Chassaing B, Adekunle OA, et al. Toxinpositive Clostridium difficile latently infect mouse colonies and protect against highly pathogenic C. difficile[J]. Gut, 2017, pii: 313510. |
| [26] | Lo Vecchio A, Zacur GM. Clostridium difficile infection: an update on epidemiology, risk factors, and therapeutic options[J]. Curr Opin Gastroenterol, 2012, 28(1): 1-9. DOI: 10.1097/MOG.0b013e32834bc9a9. |
| [27] | 朱苗苗, 李慧兰, 罗佳, 等. 质子泵抑制剂的使用与艰难梭菌感染风险相关性的Meta分析[J]. 中国循证医学杂志, 2016, 16(03): 278-85. |
 2017, Vol. 37
2017, Vol. 37

