神经病理性疼痛(NP)是由躯体感觉系统的损害或疾病导致的疼痛,临床实践中,周围神经损伤是NP的常见病因之一[1]。然而,目前仍无针对NP的特效方法和根治手段[2],因此NP的病理机制研究对于探寻新的治疗方法和手段至关重要。背根神经节(DRG)是疼痛信息传入的第一级神经元,许多研究表明,DRG介导的外周机制在外周神经损伤NP的发病机制中起着尤为重要的作用[3]。NP的外周机制主要是由外周神经损伤后,引起DRG疼痛相关基因(如阿片、γ氨基丁酸、嘌呤等受体基因及Na+、K+离子通道受体基因)表达改变,导致神经元兴奋性异常增高,从而引起皮肤痛觉过敏和痛觉超敏[4]。组蛋白精氨酸甲基化酶(PRMT)通过特异性催化组蛋白或非组蛋白精氨酸位点甲基化等蛋白质翻译后修饰,参与调控基因转录表达、RNA加工、信号转导和DNA损伤修复等重要细胞活动,广泛分布于神经系统中[5]。然而,PRMTs是否参与调控外周神经损伤后DRG疼痛相关基因表达,目前尚不清楚。目前发现PRMTs由PRMT1-3、CARM1、和PRMT5-9共9个蛋白组成,分别由Prmt1-3、Carm1和Prmt5-9基因编码[5-6],外周神经损伤究竟引起DRG哪些PRMTs转录表达改变?为回答这个问题,本研究运用RNA-Seq检测脊神经结扎(SNL)疼痛模型对应受损DRG,全面分析上述PRMT在DRG的组织分布和神经损伤对其转录表达的影响,筛选差异表达基因,使用RT-qPCR在不同神经损伤疼痛模型上进一步验证,探讨外周神经损伤对PRMT转录表达的影响,为后续深入探讨PRMT介导外周神经损伤引起的神经病理性疼痛分子生物学机制提供基础。
1 材料和方法 1.1 小鼠外周神经损伤疼痛模型制作实验动物清洁级8周大健康成年雄性C57BL6小鼠,体质量25~30 g,RNA-Seq检测和RT-qPCR验证小鼠则由南方医科大学实验动物中心提供,经动物伦理委员会许可,小鼠疼痛实验符合国际疼痛研究会(IASP)的准则。小鼠腰4(L4)脊神经SNL模型方法为,沿小鼠背部切开皮肤并钝性分离一侧竖脊肌,暴露并咬断L5横突显露L4脊神经,7-0丝线结扎并离断L4脊神经后逐层缝合,对应Sham组仅暴露L4神经不予结扎。而慢性坐骨神经结扎模型(CCI),方法是暴露出左侧坐骨神经干,用4-0铬制肠线环绕神经干分别做4个轻度结扎环,间距1 mm,结扎强度以引起小腿肌肉轻度颤动反应为宜。对应Sham组为仅分离左侧坐骨神经不做任何处理。术后根据实验需要检测动物疼痛行为学,观察有无伤肢自噬现象。
1.2 痛阈的测定 1.2.1 机械触诱发痛行为检测检测方法同我们既往研究[7],即采用校正后的0.4 g von Frey纤毛(Stoelting Co.,Wood Dale,IL,USA)垂直刺激小鼠后爪足掌中部,在纤毛适度弯曲后持续刺激3 s,连续刺激10次,每次刺激间隔5 s。纤毛刺激时,若动物产生了缩爪反应,则记为有效反应,计数10次刺激中出现有效反应的频数,计算缩爪反应频率(PWF)=(有效反应次数/10)×100%,以此来反应小鼠机械痛敏情况。
1.2.2 热痛觉过敏行为检测检测方法同我们既往研究[7],即采用热痛仪(Model 336,IITC Inc./Life Science Instruments,Woodland Hills,CA,USA)的辐射热刺激小鼠后爪足掌中部,通过热缩爪反应潜伏期(PWL)来判定小鼠热痛敏情况。为避免热刺激灼伤小鼠皮肤,设定热刺激强度使TWL基础值为10 s左右(50%的光刺激强度),切断时间为20 s。每只大鼠测定5次,间隔5 min,取中间3个数值求平均值作为该侧肢体的PWL。
1.3 总RNA提取疼痛行为学检测结束后,根据实验需要断头处死小鼠,快速收集SNL和Sham组L4患侧和对侧DRG组织并浸泡在RNAlater保存液中备用。为获得足量的L4 DRG组织提取总RNA做RNA-Seq,将SNL或Sham术后7 d(疼痛症状最明显)自同组的8个L4 DRG汇成一个样本,PCR验证则按SNL或Sham术前、术后3、7和14 d不同时点的2个同组DRG汇成一个样本,所有实验3个或3个以上生物学重复。上述样本按miRNeasy试剂盒(QIAGEN,Valencia,CA)的操作步骤进行滤柱式抽提总RNA,使用NanoDrop 2000分光光度计测量RNA浓度,A260 nm/A280 nm的比率在1.97和2.08之间。Agilent 2100生物分析仪(Agilent technologies, Santa Clara, CA)评估RNA完整性数字在7.5和8.4之间。
1.4 RNA-Seq测序获得总RNA后,使用Ribo-Zero rRNA Removal(Human/Mouse/Rat)Kit(Illumina,San Diego,CA)去除样本内核糖体RNA(rRNA),使用没有多聚腺苷酸选择性的TruSeq Stranded Total RNA Sample Preparation Kit试剂盒作为RNA文库制备,上述操作按试剂盒操作说明进行。RNA-Seq测序采用Illumina公司HiSeq 2500平台高通量输出模式,2×100 bp模式双末端测序,每通道有超过190 M的总读取量(每样本至少60 M的读取量),使用RPKM(Reads Per Kilobases per Million reads,每百万reads中来自某一基因每千碱基长度的reads数)来表示基因的表达量,其具体方法详见我们既往文章[8]。
1.5 PRMT基因差异基因RT-qPCR验证使用TaKaRa Prime-Script@RT reagent kit with DNA eraser逆转录试剂盒将总RNA逆转录合成cDNA,10 µL PCR反应体系:mRNA模板2 µL,Premix Taq Version 2.0 2 µL,ddH2O 6 µL。PCR反应条件:37 ℃ 15 min,85 ℃ 5 s,随后采用SYBR Green方法进行实时定量PCR反应,所需引物用Primer 3.0在线软件设计,由上海生工生物工程股份有限公司合成,引物信息详见表 1,10 µL反应体系:cDNA模板1 µL,SYBR Green Master 5 µL,ddH2O 3.6 µL,上下游引物各0.2 µL。PCR反应条件:95 ℃ 3 min预变性;95 ℃变性10 s,60 ℃退火30 s,72 ℃延伸1 min,共40循环,溶解曲线65~95 ℃。反应结束后,Bio-Rad Prime PCR软件分析荧光信号数据,计算各反应管Ct值,以Tubala(Tubulin alpha 1A gene)为内参并与对侧DRG比较,采用2-ddCt法计算各目的基因的相对表达量。
| 表 1 引物信息表 Table 1 All primers used |
采用SigmaPlot 12.0统计学软件进行分析,正态分布的计量资料以均数±标准差表示,组间比较采用成组t检验,组内比较采用单因素方差分析,多重比较采用双因素方差分析,P < 0.05为差异有统计学意义。
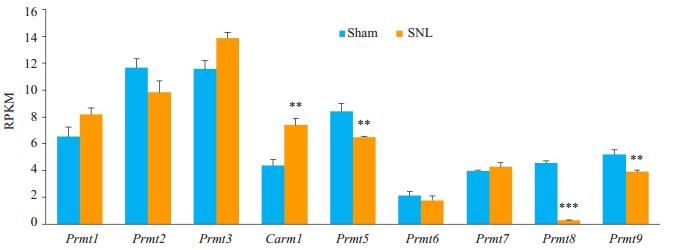
2 结果 2.1 SNL外周神经损伤DRG PRMT转录表达改变目前研究发现组蛋白精氨酸甲基化酶有9个,分别是Prmt1-3、Carm1、Prmt5-9基因编码的基因[5]。由Sham组可知,上述9蛋白精氨酸甲基化酶个基因均表达于DRG中,其基础表达量最多的是Prmt2和Prmt3两个基因,最少的是Prmt6。与Sham组比较,SNL促进Carm1基因表达上调,而Prmt5、Prmt8和Prmt9基因表达抑制,其余基因表达无明显变化(图 1)。
|
图 1 SNL术后7 d DRG PRMT转录表达改变 Figure 1 Changes in transcriptional levels of protein arginine methylation enzyme genes in injured DRG following spinal nerve ligation injury. **P < 0.01, ***P < 0.001 vs Sham group. RPKM: Reads per kilobases per million reads. SNL: Spinal nerve ligation. |
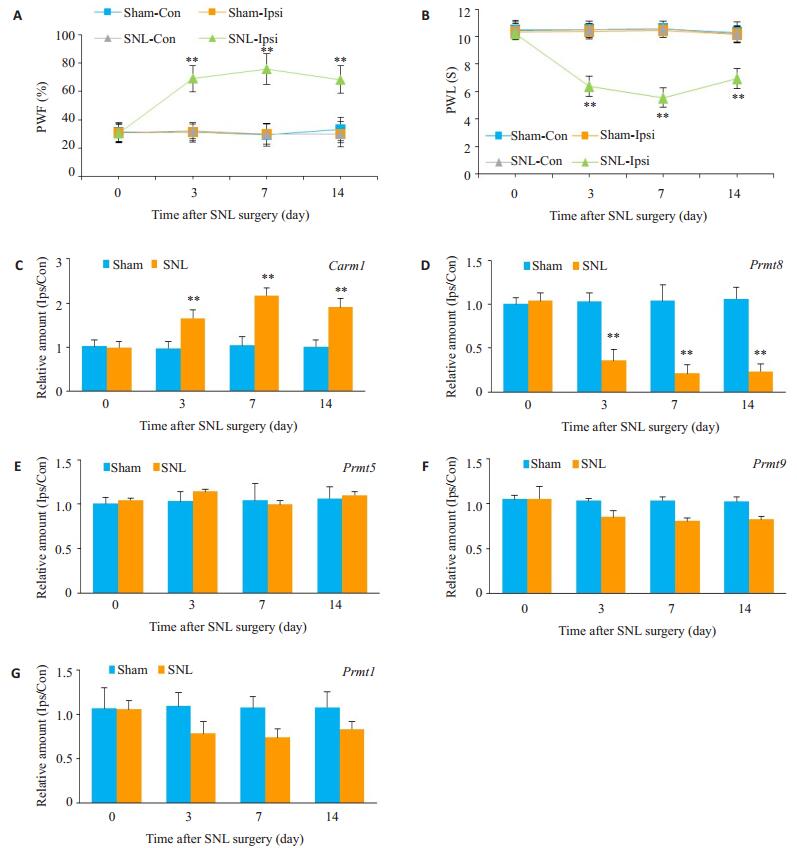
高通量测序在准确获取大量转录信息的同时会因为变异系数过大,而导致信息的误读[9]。因此,我们建立SNL模型并设计了Prmt1、Carm1、Prmt5、Prmt8和Prmt9特异性引物(表 1),RT-qPCR进一步验证SNL术后不同时点的表达。行为学测量分别于SNL术前(0 d)、术后第3、7和14天进行。与Sham组比较,SNL组在术后3、7和14 d小鼠患侧后爪PWF明显增加(P < 0.01),PWL明显缩短(P < 0.01),而Sham组患侧和两组健侧后爪PWF和PWL无明显改变(P>0.05,n=6,图 2AB)。我们使用RT-qPCR检测上述时点差异基因表达,结果表明,较Sham组术前,SNL上调了受损侧L4 DRG Carm1在术后3、7和14 d mRNA表达(P < 0.01,n=4,图 2C),相反,下调Prmt8基因表达(P < 0.01,n=3,图 2D),而Prmt5、Prmt9和Prmt1则无明显改变(P>0.05,n=6,图 2E-G)。
|
图 2 SNL神经损伤后DRG PRMT差异基因RT-qPCR验证 Figure 2 Validation of differentially expressed protein arginine methylation enzyme genes by RT-qPCR. Paw withdrawal responses to mechanical (A) and thermal (B) stimuli on the ipsilateral (Ipsi) and contralateral (Con) sides were tested at different time points after spinal nerve ligation (SNL) in the mice. **P < 0.01, n=6, vs the corresponding Sham group on the ipsilateral side (Sham-Ipsi). The ipsilateral-to-contralateral ratios are shown for Carm1, Prmt8, Prmt5, Prmt9 and Prmt1 mRNA expressions in the L4 DRGs after SNL in mice. **P < 0.01 vs the corresponding Sham group. PWF: Paw withdrawal frequency. PWL: Paw withdrawal latency |
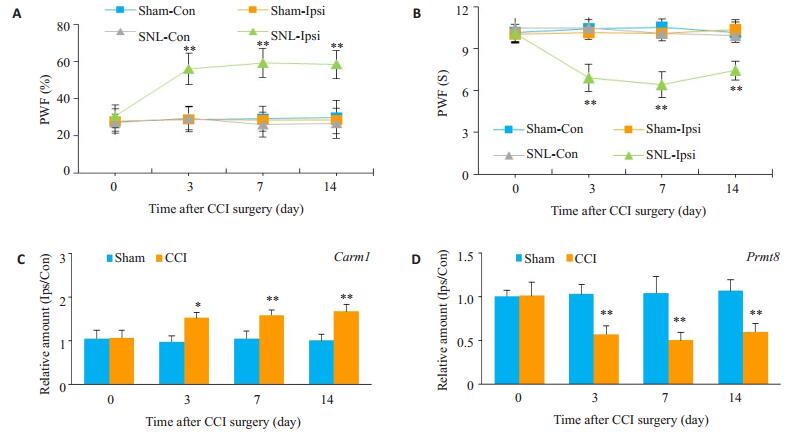
为排除SNL单一神经损伤模型对DRG PRMT基因表达的影响,我们采用另一个常用的外周神经损伤模型CCI模型进一步验证。CCI行为学测量方法和时点与SNL一致,与Sham组比较,CCI组患侧后爪在术后3、7和14 d PWF明显增加(P < 0.05),PWL明显缩短(P < 0.05),而Sham组患侧和两组健侧后爪PWF和PWL无明显改变(P>0.05,图 3A、B)。RT-qPCR验证结果表明与SNL一致,即:较Sham组,CCI术后3、7和14 d时间依赖性上调患侧L4 DRG Carm1表达(图 3C),而抑制Prmt8表达(图 3D)。
|
图 3 DRG Carm1和Prmt8基因在CCI术后0、3、7和14 d表达改变 Figure 3 Carm1 and Prmt8 mRNA expressions in the ipsilateral L3 and L4 DRGs on days 0, 3, 7 and 14 after CCI or sham surgery. Paw withdrawal responses to mechanical (A) and thermal (B) stimulation on the ipsilateral (Ipsi) and contralateral (Con) sides on day 0, 3, 7 and 14 after CCI or sham surgery in mice. **P < 0.01, n=6, vs the corresponding Sham group on the ipsilateral side (Sham-Ipsi). The Carm1 and Prmt8 gene expressions at time points as shown after Sham or SNL surgery on L3 and 4 DRGs. *P < 0.05, **P < 0.01, n=3-6, vs the corresponding Sham group. CCI: Chronic constriction injury. |
本研究首次揭示了外周神经损伤DRG PTMT转录表达规律,发现组蛋白精氨酸甲基化酶基因Carm1转录表达明显上调,而Prmt5、Prmt8和Prmt9转录抑制,Prmt8抑制最明显,进一步RT-qPCR验证发现,外周神经损伤时间依赖性上调受损侧L4 DRG Carm1表达,仅抑制Prmt8转录表达,而Prmt5和Prmt9无明显改变,上述表达改变与NP行为密切相关,为后续进一步探讨Carm1或Prmt8介导外周神经损伤引起的NP外周分子生物学机制提供了基础。
DRG神经元不仅含有传递伤害性刺激的神经递质和调质,如速激肽、兴奋性氨基酸等,也含有起突触前调制作用的受体,如γ氨基丁酸、阿片和嘌呤等受体,以及钠、钾、钙等离子通道[7, 10-12]。当外周神经损伤后,引起DRG上述物质表达改变,导致神经元兴奋性异常增高,是NP产生和维持重要机制[4, 13]。因此,明确神经损伤上述物质在DRG内变化的调控机制,可进一步明确NP的发生外周机制,进而为疼痛治疗提供潜在靶点[14]。而RNA-Seq能够很好地显示处于表达状态的基因数量和活跃程度,进行多方向多维度的研究不同病理状态的基因表达差异,分析疾病相关基因等[9, 15]。本研究基于Hiseq2500高通量测序平台2×100 bp模式完成小鼠SNL模型DRG PRMT转录组的深度测序,分析结果显示,PRMT基础表达量最多的是Prmt2和Prmt3两个基因,表达最少的是Prmt6。在差异基因筛查中发现,SNL促进Carm1基因表达上调,Prmt5、Prmt8和Prmt9则转录抑制,其余基因表达无明显变化。我们进一步使用RT-qPCR验证上述差异基因在不同疼痛模型(SNL和CCI)的表达,结果发现外周神经损伤上调受损侧L4 DRG Carm1表达,仅抑制Prmt8转录表达,而Prmt5和Prmt9无明显改变,这可能是由于RNA-Seq高通量测序时Prmt5和Prmt9的变异系数过大造成的[9]。
组蛋白精氨酸甲基化修饰是组蛋白质翻译后修饰的重要方式之一,由PRMT家族催化产生,在胞浆和胞核内参与诸多细胞反应,如:转录调控、RNA剪切、信号转导、DNA损伤修复和蛋白和蛋白间相互作用[5, 16]。PRMT家族根据功能的不同分为两类(Ⅰ和Ⅱ类),由PRMT1-3、CARM1、和PRMT5-9共9个蛋白组成,分别由Prmt1-3、Carm1和Prmt5-9基因编码,催化组蛋白和非组蛋白的精氨酸位点的甲基化,参与细胞诸多活动[5-6]。本研究全面分析了上述9个PRMTs的基因神经损伤后DRG表达改变,发现SNL和CCI模型均上调DRG Carm1的转录表达,而抑制Prmt8转录,它们的转录表达改变在神经损伤过程中发挥怎样的功能呢?目前仍然不清楚。
研究表明,CARM1在DNA包装,转录调控和未成熟mRNA剪切和RNA稳定性方面发挥重要作用[19]。其中,CARM1可催化组蛋白H3第17和26号精氨酸位点二甲基化(H3R17me2和H3R26me2),激活组蛋白上结合的该段DNA转录表达[20-21]。另有研究表明,CARM1参与JAK/STAT信号通路的调控[22],而JAK/STAT信号通路是趋化因子、生长因子和白介素等细胞因子介导DRG和脊髓背角神经元兴奋性和胶质细胞活化致神经病理性疼痛和骨癌痛等慢性疼痛的关键机制[23-24]。那么,外周神经损伤上调DRG Carm1转录表达,可能通过组蛋白H3R17me2修饰或JAK/STAT信号通路参与神经病理性疼痛的产生和维持,我们需要进一步研究。
与此同时,神经损伤下调的DRG Prmt8基因是否参与疼痛的产生或维持,至今未见报道。细胞膜上PRMT8可与磷脂酰肌醇-3-羟激酶(PI3K)调控蛋白P85相互作用,激活PI3K,导致三磷酸磷脂酰肌醇((PIP3)合成增加,激活ATK,形成PRMT8/PI3K/AKT轴,在维持人类多能干细胞多能性和调控中胚层分化发挥重要作用[25]。而PI3K/AKT信号通路在DRG和脊髓等中枢系统介导慢性疼痛发挥重要作用[26],DRG Prmt8是否通过PI3K/AKT通路影响神经病理性疼痛的产生和发展,仍需进一步研究。
综上所述,本研究初步探讨PRMT在外周神经损伤DRG的转录改变,发现神经损伤上调DRG Carm1表达,抑制Prmt8表达,为后续深入探讨Carm1或Prmt8介导外周神经损伤引起的NP分子生物学机制提供基础,以期待为NP的防治提供新的潜在靶点。
| [1] | Campbell JN, Meyer RA. Mechanisms of neuropathic pain[J]. Neuron, 2006, 52(1): 77-92. DOI: 10.1016/j.neuron.2006.09.021. |
| [2] | Yan YY, Li CY, Zhou L, et al. Research progress of mechanisms and drug therapy for neuropathic pain[J]. Life Sci, 2017, 190: 68-77. DOI: 10.1016/j.lfs.2017.09.033. |
| [3] | Basbaum AI, Bautista DM, Scherrer GA. Cellular and molecular mechanisms of pain[J]. Cell, 2009, 139(2): 267-84. DOI: 10.1016/j.cell.2009.09.028. |
| [4] | Krames ES. The role of the dorsal root ganglion in the development of neuropathic pain[J]. Pain Medicine, 2014, 15(10): 1669-85. DOI: 10.1111/pme.12413. |
| [5] | Bedford MT, Richard S. Arginine methylation: An emerging regulator of protein function[J]. Mol Cell, 2005, 18(3): 263-72. DOI: 10.1016/j.molcel.2005.04.003. |
| [6] | Morales Y, Cáceres T, May K, et al. Biochemistry and regulation of the protein arginine methyltransferases (PRMTs)[J]. Arch Biochem Biophys, 2016, 590: 138-52. DOI: 10.1016/j.abb.2015.11.030. |
| [7] | Zhao JY, Liang LL, Gu XY, et al. DNA methyltransferase DNMT3a contributes to neuropathic pain by repressing Kcna2 in primary afferent neurons[J]. Nat Commun, 2017, 8: 14712. DOI: 10.1038/ncomms14712. |
| [8] | Wu S, Marie Lutz B, Miao X, et al. Dorsal root ganglion transcriptome analysis following peripheral nerve injury in mice[J]. Mol Pain, 2016, 12: 1744806916629048. |
| [9] | Wang Z, Gerstein M, Snyder M. RNA-Seq: a revolutionary tool for transcriptomics[J]. Nat Rev Genet, 2009, 10(1): 57-63. DOI: 10.1038/nrg2484. |
| [10] | 冼培凤, 陈莹, 杨路, 等. 蜂毒对胶原诱导性关节炎炎性痛大鼠背根神经节TrkA、TRPV1的影响[J]. 南方医科大学学报, 2016, 36(6): 838-41. |
| [11] | 蒋群, 屠伟峰. 舒芬太尼预处理对大鼠急性胃粘膜病变的保护作用及与酸敏感离子通道的关系[J]. 南方医科大学学报, 2010, 30(5): 1099-102. |
| [12] | Liang LL, Gu XY, Zhao JY, et al. G9a participates in nerve injuryinduced Kcna2 downregulation in primary sensory neurons[J]. Sci Rep, 2016, 6: 37704. DOI: 10.1038/srep37704. |
| [13] | Krames ES. The dorsal root ganglion in chronic pain and as a target for neuromodulation: a review[J]. Neuromodulation, 2015, 18(1): 24-32. DOI: 10.1111/ner.2015.18.issue-1. |
| [14] | Coutaux A, Adam F, Willer JC, et al. Hyperalgesia and allodynia: peripheral mechanisms[J]. Joint Bone Spine, 2005, 72(5): 359-71. DOI: 10.1016/j.jbspin.2004.01.010. |
| [15] | Yi F, Yang F, Liu XQ, et al. RNA-seq identified a super-long intergenic transcript functioning in adipogenesis[J]. RNA Biol, 2013, 10(6): 991-1002. |
| [16] | Wesche J, Kuehn S, Kessler BM, et al. Protein arginine methylation: a prominent modification and its demethylation[J]. Cell Mol Life Sci, 2017, 74(18): 3305-15. DOI: 10.1007/s00018-017-2515-z. |
| [17] | Simandi Z, Czipa E, Horvath A, et al. PRMT1 and PRMT8 regulate retinoic Acid-Dependent neuronal differentiation with implications to neuropathology[J]. Stem Cells, 2015, 33(3): 726-41. DOI: 10.1002/stem.1894. |
| [18] | Shin HJ, Kim H, Oh S, et al. AMPK-SKP2-CARM1 signalling cascade in transcriptional regulation of autophagy[J]. Nature, 2016, 534(7608): 553-7. DOI: 10.1038/nature18014. |
| [19] | Cheng D, Côté J, Shaaban S, et al. The arginine methyltransferase CARM1 regulates the coupling of transcription and mRNA processing[J]. Mol Cell, 2007, 25(1): 71-83. DOI: 10.1016/j.molcel.2006.11.019. |
| [20] | Jacques SL, Aquino KP, Gureasko JA, et al. CARM1 preferentially methylates H3R17 over H3R26 through a random kinetic mechanism[J]. Cancer Res, 2015, 75(15): 1635-44. |
| [21] | Selvi BR, Batta K, Kishore A, et al. Identification of a novel inhibitor of coactivator-associated arginine methyltransferase 1(CARM1)-mediated methylation of histone H3 Arg-17[J]. J Biol Chem, 2010, 285(10): 7143-52. DOI: 10.1074/jbc.M109.063933. |
| [22] | Coughlan N, Thillainadesan G, Andrews J, et al. β-Estradioldependent activation of the JAK/STAT pathway requires p/CIP and CARM1[J]. Biochim Biophys Acta, 2013, 1833(6): 1463-75. DOI: 10.1016/j.bbamcr.2013.02.009. |
| [23] | Fang D, Kong LY, Cai J, et al. Interleukin-6-mediated functional upregulation of TRPV1 receptors in dorsal root ganglion neurons through the activation of JAK/PI3K signaling pathway: roles in the development of bone cancer pain in a rat model[J]. Pain, 2015, 156(6): 1124-44. |
| [24] | Dominguez E, Rivat C, Pommier B, et al. JAK/STAT3 pathway is activated in spinal cord microglia after peripheral nerve injury and contributes to neuropathic pain development in rat[J]. J Neurochem, 2008, 107(1): 50-60. DOI: 10.1111/jnc.2008.107.issue-1. |
| [25] | Jeong HC, Park SJ, Choi JJ, et al. PRMT8 controls the pluripotency and mesodermal fate of human embryonic stem cells by enhancing the PI3K/AKT/SOX2 axis[J]. Stem Cells, 2017, 35(9): 2037-49. DOI: 10.1002/stem.v35.9. |
| [26] | Chen SP, Zhou YQ, Liu DQ, et al. PI3K/Akt pathway: a potential therapeutic target for chronic pain[J]. Curr Pharm Des, 2017, 23(12): 1860-8. DOI: 10.2174/1381612823666170210150147. |
 2017, Vol. 37
2017, Vol. 37

