蛋白4.1R是一类高度保守的细胞膜骨架蛋白,属于蛋白4.1家族,此家族还包括4.1B、4.1N和4.1G[1-4]。研究发现,蛋白4.1R在T细胞、B细胞、巨噬细胞等免疫细胞中高表达[5],CD4+T细胞中,蛋白4.1R通过结合T细胞活化连接蛋白(LAT)并抑制ZAP-70对LAT的磷酸化负调控CD4+T细胞增殖及分化[6];并且,蛋白4.1R通过抑制CD4+T细胞的活化从而减弱小鼠实验性变态反应性脑脊髓炎(EAE)的病症[7]。随着研究的深入,4.1R在获得性免疫中的调节作用逐渐被发现,但4.1R在天然免疫尤其是在巨噬细胞中的功能尚不明确。对于其功能的研究,过去传统上依赖于RNA干扰技术[8],但RNA干扰技术存在的瞬时性和不彻底性的弊端会影响对基因功能的正确判断[9]。而近几年迅速发展起来的CRISPR/Cas9基因编辑技术能够在基因组水平上特异性剪切目的基因[10],是研究基因功能的有力的工具。本研究拟利用CRISPR/Cas9技术定点敲除巨噬细胞系RAW264.7细胞中4.1R基因,得到稳定敲除4.1R基因的RAW264.7细胞株,为深入研究4.1R在巨噬细胞中的作用机制提供有效工具。
1 材料和方法 1.1 材料与试剂鼠源巨噬细胞系RAW264.7细胞、293T细胞、lentiCRISPRv2质粒(实验室保存);PEG6000慢病毒纯化试剂、慢病毒包装辅助载体pH1、pH2质粒(英茂盛业生物科技);质粒小提试剂盒、胶回收试剂盒、细胞基因组DNA提取试剂盒(Axygen);DH5α感受态细胞(鼎国昌盛生物技术);T4多聚核酸激酶、限制性内切酶BsmBI、T4连接酶(TaKaRa);DMEM高糖培养基、胎牛血清、青霉素和链霉素(Hyclone);jetPRIME®转染试剂(Polyplus Transfection);Polybrene(聚凝胺,翊圣生物科技);RIPA裂解液(索莱宝)4.1R抗体(Proteintech);GADPH抗体(Abways)、辣根过氧化物酶(HRP)偶联山羊抗兔IgG(鼎国昌盛生物技术);ECL化学发光液(Thermo Scientific),小向导RNA(sgRNA)序列合成及质粒测序由苏州金唯智生物科技有限公司完成。
1.2 方法 1.2.1 sgRNA的设计和寡核苷酸链的合成根据CRISPR/Cas9靶向原理,利用CRISPR在线设计工具(http://crispr.mit.edu/),选择3条分数较高的针对4.1R外显子的sgRNA序列,在正义链模板的5′端添加CACC,反义链模板的5′端添加AAAC,与BsmBⅠ酶切后形成的黏性末端互补。同时根据靶位点设计CRISPR/Cas9敲除鉴定引物4.1R-F:5'-AGATTCTGAAACGAAG-3',4.1R-R:5'-TCTTCTGTTAATTGTGCTG-3'。
1.2.2 重组真核表达质粒lentiCRISPRv2-sgRNA构建使用BsmBⅠ限制性内切酶酶切质粒lentiCRISPRv2质粒并切胶回收。合成的寡核苷酸单链sgRNA用T4多聚核苷酸激酶对退火形成二聚体,用T4连接酶将退火后的sgRNA与胶回收的lentiCRISPRv2质粒连接,然后将连接产物体系转化到DH5α感受态细胞中,涂于氨苄抗性的LB培养基平板上,挑取单菌落并摇菌培养,小提质粒并测序鉴定。
1.2.3 293T细胞培养与慢病毒制备将293T细胞用DMEM培养基常规培养,转染前以细胞密度生长至60%~80%。将构建好的sgRNA-lentiCRISPRv2质粒和慢病毒包装辅助载体pH1、pH2质粒按照sgRNAlentiCRISPR: pH1: pH2为9: 3: 1的比例混合,将混合好的慢病毒表达系统按照jetPRIME®转染试剂说明书转入293T细胞内,转染48 h后收集细胞上清,按照PEG6000慢病毒纯化试剂说明书纯化慢病毒。
1.2.4 RAW264.7细胞培养与慢病毒侵染RAW264.7细胞DMEM培养基常规培养,侵染前将细胞接种至12孔板,当细胞密度生长为80%左右,每孔加入sgRNACas9慢病毒50~200 μL、Polybrene(聚凝胺)6 μg/mL,加入DMEM培养基补足至1 mL,侵染24 h后改换为DMEM培养基继续培养。
1.2.5 敲除4.1R基因的RAW264.7细胞株的单克隆筛选sgRNA-Cas9慢病毒侵染RAW264.7细胞48 h后,更换含2.5 μg/mL嘌呤霉素的DMEM培养基,1周后,将带有抗性的细胞采用有限稀释法接种至96孔板,待细胞形成单克隆集落后,用枪头吸取吹散于24孔板内,长满后接种至6孔板培养。
1.2.6 敲除4.1R基因的RAW264.7细胞株的鉴定6孔板内长满的细胞,部分细胞保种,余下细胞继续培养长满6孔板时加入RIPA裂解液提取蛋白,提取蛋白加入SDS缓冲液沸水浴10 min,进行SDS-PAGE电泳电泳,结束后转移至PVDF膜,5%脱脂奶粉室温封闭2 h,内源性4.1R抗体4 ℃过夜孵育,TBST洗膜,HRP偶联的羊抗兔IgG室温孵育1 h,TBST洗膜,ECL化学发光液进行显影、曝光。经Western blotting印记检测的蛋白4.1R完全没有表达的RAW264.7细胞提取基因组DNA,经PCR扩增后将产物交付苏州金唯智公司测序,测序结果与4.1R基因进行比对。
1.2.7 CCK8细胞增殖实验将处于对数生长期的细胞以密度为2×104细胞/mL,传至96孔板,每孔100 μL,每组实验设计5复孔,在37 ℃,5% CO2细胞培养箱中培养24、48、72 h,每孔加入10 μLCCK8试剂,在孵箱培养1~ 4 h,在450 nm处读取吸光度(A)。
1.2.8 统计学处理计数数据以均数±标准差表示,采用GraphPadPrism 5.0软件进行分析,两组间采用student's T test比较,以P < 0.05表示差异具有统计学意义。
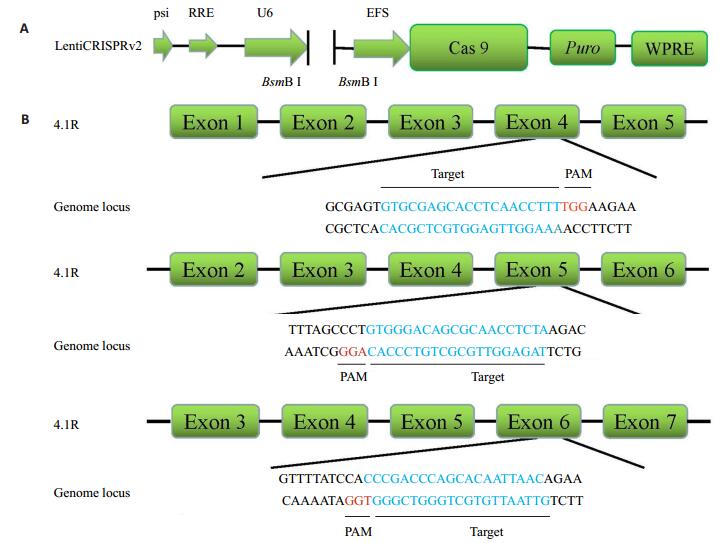
2 结果 2.1 sgRNA靶点的选择及寡核苷酸链设计4.1R基因上有两个起始密码子,分别编码大小为135 000和80 000的两个亚基,这两个密码子分别位于4.1R基因的1号外显子和4号外显子上,因此,为了能够彻底干扰4.1R基因的表达,我们分别针对4.1R基因的外显子4、5、6设计sgRNA(图 1),并在5′端添加相应碱基(表 1),送苏州金唯智公司进行寡核苷酸链合成。
|
图 1 靶向4.1R基因的sgRNA的设计 Figure 1 Design of sgRNA targeting 4.1R gene. A: Structure of lentiCRISPRv2 plasmid; B: Recognition sequence of sgRNA targeting Exons 4, 5, and 6. |
| 表 1 4.1R-sgRNA寡核苷酸序列 Table 1 Oligonucleotide sequence of 4.1R-sgRNA |
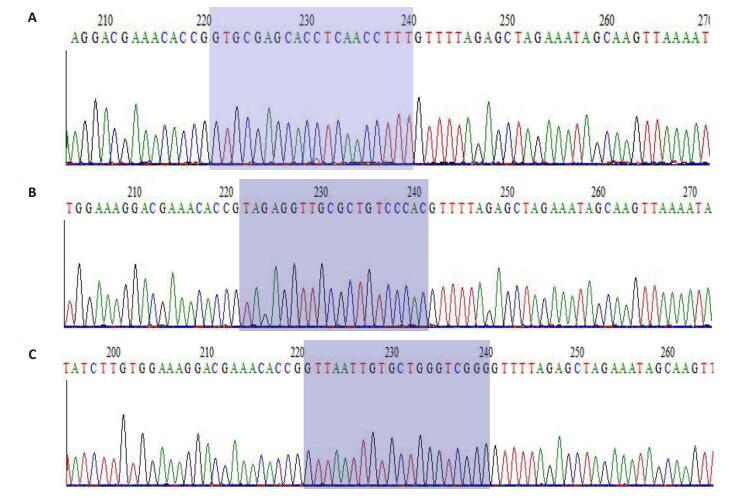
将设计好sgRNA进行程序退火形成双链,用BsmBI酶切lentiCRISPRv2质粒,T4连接酶将sgRNA与酶切后的质粒连接。连接产物转化经转化、挑菌、摇菌、小提质粒后,送往金唯智公司进行测序,结果显示4.1R-sgRNA1、4.1R-sgRNA2、4.1R-sgRNA3序列均正确插入lentiCRISPRv2,插入序列的位置、方向及序列与预期相符(图 2),证明lentiCRISPRv2-sgRNA表达载体构建完全正确。
|
图 2 LentiCRISPRv2-sgRNA测序图 Figure 2 Sequence map of lentiCRISPRv2-sgRNA. A: Sequencing result of 4.1R-sgRNA1-lentiCRISPRv2; B: Sequencing result of 4.1R-sgRNA2-lentiCRISPRv2; C: Sequencing result of 4.1R-sgRNA3-lentiCRISPRv2. |
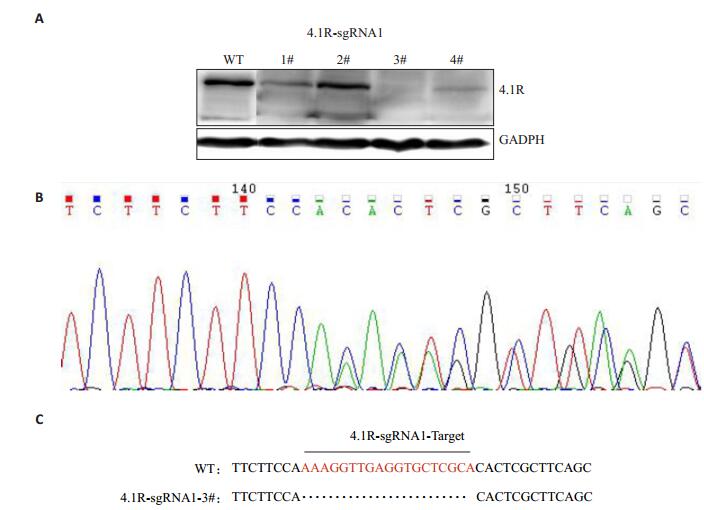
分别用sgRNA1-Cas9、sgRNA2-Cas9、sgRNA3-Cas9慢病毒侵染RAW264.7细胞后,加入2.5 μg/mL嘌呤霉素筛选1周,结果只有在用sgRNA1-Cas9慢病毒侵染的RAW264.7细胞中出现抗嘌呤霉素的阳性细胞,将阳性细胞稀释至单克隆,取其中4个单克隆细胞,用Western blotting印迹法检测其蛋白4.1R的表达,其中sgRNA1-Cas9慢病毒侵染后筛选的单克隆中3#细胞株表现为蛋白4.1R的表达完全缺失(图 3)。将3#细胞株的细胞提取基因组DNA,进行鉴定PCR,PCR产物测序,通过与4.1R基因原序列对比发现,4.1R-sgRNA1-3#出现了19个碱基的缺失突变,此突变能改变4.1R基因编码的开放阅读框,进而终止蛋白4.1R的翻译。结果说明稳定敲除4.1R基因的RAW264.7细胞株构建成功。
|
图 3 Western blotting印迹鉴定及DNA测序结果 Figure 3 Western blotting and DNA sequencing for verifying stable 4.1R gene knockout in the cells. A: Detection of 4.1R protein expression in the selected monoclonal cells using Western blotting; B: Sequencing result of 4.1RsgRNA1-3#; C: Comparison of 4.1R sequence in 4.1R-sgRNA1-3# and the original 4.1R sequence. |
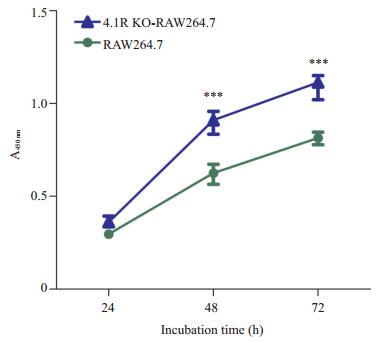
利用CCK8法检测筛选出的稳定敲除4.1R基因的RAW264.7细胞(4.1RKO-RAW264.7)的增殖。结果显示,4.1R基因敲除后,RAW264.7细胞增殖能力显著增加(图 4),提示4.1R参与调节巨噬细胞系RAW264.7细胞的增殖。
|
图 4 4.1R基因敲除对巨噬细胞系RAW264.7细胞增殖的影响 Figure 4 Effect of 4.1R gene knockout on the proliferation of murine macrophage line RAW264.7. ***P < 0.001 vs RAW264.7. |
本实验利用CRISPR/Cas9系统成功地敲除巨噬细胞系RAW264.7细胞中4.1R基因,得到稳定敲除4.1R基因的RAW264.7细胞株,DNA测序结果表明该细胞株中4.1R基因发生了19 bp的缺失突变,发生突变的位点正是sgRNA1的识别序列,这说明sgRNA1-Cas9慢病毒入侵RAW264.7细胞后,sgRNA1准确地靶向了4.1R基因的4号外显子,随后Cas9核酸酶在靶位点产生DNA双链断裂[11],RAW264.7细胞DNA在修复过程中,产生了缺失突变[12],此突变改变了4.1R基因编码的开放阅读框,提前终止蛋白4.1R的翻译,western blotting鉴定结果也证明了该细胞株中蛋白4.1R的表达完全缺失。
CRISPR/Cas9技术出现之前,细胞水平研究基因功能的常用技术是RNA干扰技术。RNA干扰技术作为一种转录后沉默基因表达的有力工具为分析基因的功能和机制作出了重要贡献,但随着技术的发展,其显现出瞬时性和不彻底性的弊端[13-14]。CRISPR/Cas9系统是利用sgRNA引导Cas9核酸酶在靶位点处进行DNA特异性剪切[15],从而达到敲除基因的目的。本研究利用CRISPR/Cas9系统在RAW264.7细胞基因组水平上造成了4.1R基因的缺失突变从而干扰4.1R基因的表达,消除了已往应用RNA干扰技术中存在的瞬时性和不彻底性的弊端,因此,相比RNA干扰技术,CRISPR/ Cas9系统是更适用于基因功能研究的有效工具。
蛋白4.1R是最初在红细胞中发现的细胞膜骨架蛋白[16],在维持红细胞的形状、脆性及正常生理功能中发挥着重要的作用[17],例如,人类4.1R的表达缺失会导致遗传性椭圆型红细胞增多症[18]。随着研究的深入发现,蛋白4.1R在许多哺乳动物组织和不同类型的细胞中都有广泛的表达[19-20],并且所表达的蛋白4.1R都含有完整的FERM结构域[21],红细胞中4.1R的FERM结构域结合多种跨膜蛋白[22];CD4+T细胞中4.1R的FERM结构域与LAT结合[6];角质细胞中与β1-整合素结合[23];小肠上皮细胞中与质膜钙离子ATP酶(PMCA)结合[24];胃上皮细胞中与β-连环蛋白结合[25],通过这些相互作用,4.1R调节细胞增殖、粘附和迁移。本实验中利用敲除4.1R基因的巨噬细胞系RAW264.7细胞检测4.1R对巨噬细胞增殖的影响,结果表明4.1R基因敲除后,RAW264.7细胞的增殖能力显著增加,提示4.1R负调控巨噬细胞的增殖,这一实验结果也与研究报道的4.1R作为细胞增殖的负调控分子相吻合。接下来,4.1R调控巨噬细胞增殖的作用机制以及4.1R在巨噬细胞其他功能中的作用还需做进一步深入的研究。
综上所述,CRISPR/Cas9系统作为一种新型的基因编辑技术,是研究基因功能的强有力的工具,我们利用CRISPR/Cas9系统成功的敲除了巨噬细胞系RAW264.7细胞中的4.1R基因,构建了稳定敲除4.1R基因的RAW264.7细胞株,为研究4.1R在巨噬细胞中的功能了奠定基础。
| [1] | Peters LL, Weier HU, Walensky LD, et al. Four paralogous protein 4.1 genes map to distinct chromosomes in mouse and man[J]. Genomics, 1998, 54(2): 348-50. DOI: 10.1006/geno.1998.5537. |
| [2] | Parra M, Gascard P, Walensky LD, et al. Molecular and functional characterization of protein 4.1B, a novel member of the protein 4.1 family with high level, focal expression in brain[J]. J BiolChem, 2000, 275(5): 3247-55. |
| [3] | Walensky LD, Blackshaw S, Liao D, et al. A novel neuron-enriched homolog of the erythrocyte membrane cytoskeletal protein 4.1[J]. J Neurosci, 1999, 19(15): 6457-67. |
| [4] | Parra M, Gascard P, Walensky LD, et al. Cloning and characterization of 4.1G (EPB41L2), a new member of the skeletal protein 4.1(EPB41) gene family[J]. Genomics, 1998, 49(2): 298-306. DOI: 10.1006/geno.1998.5265. |
| [5] | Tepass U. FERM proteins in animal morphogenesis[J]. CurrOpin Genet Dev, 2009, 19(4): 357-67. DOI: 10.1016/j.gde.2009.05.006. |
| [6] | Jung Y, Mccarty JH. Band 4.1 proteins regulate integrin-dependent cell spreading[J]. BiochemBiophys Res Commun, 2012, 426(4): 578-84. DOI: 10.1016/j.bbrc.2012.08.129. |
| [7] | Ward RE, Schweizer L, Lamb RS, et al. The protein 4.1, ezrin, radixin, moesin (FERM) domain of Drosophila Coracle, a cytoplasmic component of the septate junction, provides functions essential for embryonic development and imaginal cell proliferation[J]. Genetics, 2001, 159(1): 219-28. |
| [8] | Wang H, Liu C, Debnath G, et al. Comprehensive characterization of expression patterns of protein 4.1 family members in mouse adrenal gland: implications for functions[J]. Histochem Cell Biol, 2010, 134(4): 411-20. DOI: 10.1007/s00418-010-0749-z. |
| [9] | Kang Q, Yu Y, Pei X, et al. Cytoskeletal protein 4.1R negatively regulates T-cell activation by inhibiting the phosphorylation of LAT[J]. Blood, 2009, 113(24): 6128-37. DOI: 10.1182/blood-2008-10-182329. |
| [10] | Liu X, Zhou Q, Ji Z, et al. Protein 4.1R attenuates autoreactivity in experimental autoimmune encephalomyelitis by suppressing CD4(+) T cell activation[J]. Cell Immunol, 1900, 292(1/2): 19-24. |
| [11] | Gasiunas G, Barrangou R, Horvath PA. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria[J]. ProcNatlAcadSci USA, 2012, 109(39): E2579-86. DOI: 10.1073/pnas.1208507109. |
| [12] | Gratz SJ, Cummings AM, Nguyen JN, et al. Genome engineering of Drosophila with the CRISPR RNA-guided Cas9 nuclease[J]. Genetics, 2013, 194(4): 1029-35. DOI: 10.1534/genetics.113.152710. |
| [13] | Sontheimer RJ. Origins and mechanisms of miRNAs and siRNAs[J]. Cell, 2009, 136(4): 642. DOI: 10.1016/j.cell.2009.01.035. |
| [14] | Wilson RC, Doudna JA. Molecular mechanisms of RNA interference[J]. Annu Rev Biophys, 2013, 42(42): 217-39. |
| [15] | Zhang F, Wen Y, Guo X. CRISPR/Cas9 for genome editing: progress, implications and challenges[J]. Hum Mol Genet, 2014, 23(R1): R40-6. DOI: 10.1093/hmg/ddu125. |
| [16] | Correas I, Speicher DW, Marchesi VT. Structure of the spectrinactin binding site of erythrocyte protein 4.1[J]. J BiolChem, 1986, 261(28): 13362-6. |
| [17] | An X, Mohandas N. Disorders of red cell membrane[J]. Br J Haematol, 2008, 141(3): 367-75. |
| [18] | Tchernia G, Mohandas N, Shohet SB. Deficiency of skeletal membrane protein band 4.1 in homozygous hereditary elliptocytosis[J]. J Clin Invest, 1981, 68(2): 454-60. DOI: 10.1172/JCI110275. |
| [19] | Ramez M, Blot-Chabaud M, Cluzeaud F, et al. Distinct distribution of specific members of protein 4.1 gene family in the mouse nephron[J]. Kidney Int, 2003, 63(4): 1321-37. DOI: 10.1046/j.1523-1755.2003.00870.x. |
| [20] | Baines AJ, Lu HC, Bennett PM. The protein 4.1 family: hub proteins in animals for organizing membrane proteins[J]. Biochim Biophys Acta, 2014, 1838(2): 605-19. DOI: 10.1016/j.bbamem.2013.05.030. |
| [21] | Zhang J, Yang S, An C, et al. Comprehensive characterization of protein 4.1 expression in epithelium of large intestine[J]. Histochem Cell Biol, 2014, 142(5): 529-39. DOI: 10.1007/s00418-014-1224-z. |
| [22] | Salomao M, Zhang X, Yang Y, et al. Protein 4.1R-dependent multiprotein complex: new insights into the structural organization of the red blood cell membrane[J]. Proc Natl Acad Sci USA, 2008, 105(23): 8026-31. DOI: 10.1073/pnas.0803225105. |
| [23] | Chen L, Hughes RA, Baines AJ, et al. Protein 4.1R regulates cell adhesion, spreading, migration and motility of mouse keratinocytes by modulating surface expression of beta1 integrin[J]. J Cell Sci, 2011, 124(Pt 14): 2478-87. |
| [24] | Liu C, Weng H, Chen L, et al. Impaired intestinal Calcium absorption in protein 4.1R-deficient mice due to altered expression of plasma membrane Calcium ATPase 1b (PMCA1b)[J]. J Biol Chem, 2013, 288(16): 11407-15. DOI: 10.1074/jbc.M112.436659. |
| [25] | Yang S, Guo X, Debnath G, et al. Protein 4.1R links E-cadherin/ beta-catenin complex to the cytoskeleton through its direct interaction with beta-catenin and modulates adherens junction integrity[J]. Biochim Biophys Acta, 2009, 1788(7): 1458-65. DOI: 10.1016/j.bbamem.2009.03.022. |
 2017, Vol. 37
2017, Vol. 37

