2. 中南大学湘雅三医院皮肤科,湖南 长沙 410013;
3. 中南大学湘雅二医院急诊科,湖南 长沙 410011
2. Department of Dermatology, Third Xiangya Hospital, Central South University, Changsha 410013, China;
3. Department of Emergency Medicine, Second Xiangya Hospital, Central South University, Changsha 410011, China
系统性红斑狼疮(SLE)是一种慢性自身免疫性疾病,涉及到多重致病机制[1-2]。近年来,越来越多的研究证明了T细胞某些基因表观遗传学的改变在SLE的发病机制中起到了关键的作用[3-4]。表观遗传学指的是不涉及DNA序列变化的稳定且可遗传的基因表达改变,其机制主要包括DNA甲基化,组蛋白修饰,非编码RNA调控,以及染色质重塑[5-6]。而在这些表观遗传学调控机制中,作为基因沉默标志的组蛋白H3赖氨酸27三甲基化(H3K27me3)一直备受关注[6-7]。已知H3K27me3的水平由组蛋白去甲基化酶JMJD3 [8-9]、UTX [10-11]和组蛋白甲基转移酶EZH2 [12]共同参与调控。
研究发现cAMP反应元件调控因子α (CREMα)在SLE的发病机制中起到关键作用。CREMα水平在SLE患者的T细胞中显著升高,且CREMα启动子活性与SLE疾病活动指数(SLEDAI)呈正相关[13-15]。升高的CREMα可从多个方面促使SLE的发生与发展:首先,CREMα水平升高可导致IL-2减少,进而致使机体对细胞毒素反应的减弱,Treg细胞数目和功能的降低,以及活化诱导的细胞死亡(AICD)的缺陷[16-17];其次,CREMα水平升高还可导致IL-17A增加,而增加的IL-17A则会与多种趋化因子和细胞因子相互作用从而引发多重炎症反应[18];IL-17A也能刺激B细胞增殖,从而产生更多的自身抗体[15, 19-20];此外,CREMα的过表达能抑制TCR/CD3ζ链的转录从而阻碍其终止T细胞反应,导致T细胞持续活化;它还能抑制转录因子c-fos、抗原提呈细胞分子CD86、Notch信号通路分子Notch-1等而参与SLE的发病[21-24]。那么SLE患者T细胞CREMα水平升高的原因又是什么呢?
通过染色质免疫沉淀(ChIP)微阵列,我们发现SLE患者CD4+ T细胞CREMα启动子区的H3K27me3水平显著低于正常对照。以此为线索,我们进一步探讨SLE CD4+ T细胞CREMα表达升高的原因,为揭示SLE的发病机制提供新的思路。
1 资料和方法 1.1 研究对象30名SLE患者来自中南大学湘雅二医院皮肤科门诊及住院部。所有的患者均符合1997年美国风湿协会制订的SLE诊断标准[25]。SLE患者相关的临床信息见表 1。其中女性27例,男性3例,年龄20~42(28.567± 6.558)岁,SLEDAI评分0~16(7.567±4.384)分。30名正常对照均为中南大学湘雅二医院健康职工和研究生。其中女性27例,男性3例;年龄20~41(27.133± 6.067)岁。患者及正常对照年龄性别均无统计学差异(P>0.05),并均签署了知情同意书。本次研究获得了中南大学湘雅二医院伦理委员会的批准。
| 表 1 患者资料表 Table 1 Patient profiles |
淋巴细胞分离液购自瑞典GE Healthcare公司;CD4+ T细胞阳性分选试剂盒购自德国Miltenyi公司;ChIP试剂盒购自美国Millipore公司;TRIzol试剂购自美国Invitrogen公司;SYBR® Premix Ex TaqTM(Tli RNaseH Plus)和One Step SYBR PrimeScriptTM RT-PCR试剂盒购自日本Takara公司;抗H3K27me3抗体购自美国Millipore公司;抗JMJD3抗体、抗UTX抗体和抗EZH2抗体购自美国Abcam公司;PCR引物由上海铂尚生物有限公司合成。
1.3 细胞分离抽取实验对象外周静脉血60 mL (用20 U/mL肝素抗凝),加入淋巴细胞分离液,采用密度梯度离心法分离外周血单个核细胞(PBMC)。所得PBMC加入PBS洗涤,随后使用免疫磁珠进行阳性分选获得CD4+ T细胞。
1.4 ChIP微阵列使用1%的甲醛对5名SLE患者和5名年龄、性别均匹配的正常对照的CD4+ T细胞进行固定,随后使用裂解缓冲液对细胞进行裂解。SLE患者和正常对照细胞的裂解液分别进行混合,随后送至北京博奥生物有限公司。ChIP微阵列的质控、标记、杂交、扫描以及统计分析由博奥公司进行。抗H3K27me3抗体沉淀的DNA和总DNA (input)分别采用Cy5 (红色)和Cy3 (绿色)进行标记。标本随后杂交于微阵列板中,最后得到Cy3/Cy5比例图像。在这些图像中,不同的颜色强度代表各种基因启动子区相对的H3K27me3水平。与正常对照CD4+ T细胞相比,SLE CD4+ T细胞启动子区H3K27me3水平增加至2倍以上或减少至0.5倍以下被认为具有显著意义。
1.5 ChIP结合实时定量PCR按照厂家说明书,采用ChIP试剂盒进行ChIP分析。简而言之,CD4+ T细胞使用1%甲醛固定10 min,随后使用裂解缓冲液进行裂解,并用超声波剪切细胞裂解液中的DNA,离心后取上清液。使用蛋白G琼脂糖珠去除非特异性背景后,加入抗体并在4 ℃中涡旋孵育过夜。次日,加入蛋白G琼脂糖珠并在4 ℃中涡旋孵育1 h以结合免疫复合物。琼脂糖珠-DNA-蛋白复合物经清洗后,再使用洗脱缓冲液将DNA-蛋白复合物洗脱出来,置于65 ℃中加热4 h以解除DNA和蛋白质之间的交联,随后将DNA进行纯化。使用SYBR® Premix Ex TaqTM (Tli RNaseH Plus)试剂盒,通过标准曲线相对定量法进行实时定量PCR检测DNA水平。具体方法如下:以获得的DNA为模板进行扩增,同时取一份DNA样本,将其对倍稀释成5个梯度作为标准品,2/4/8/16/32倍稀释,以此产生标准曲线用于计算每一份样本的相对浓度,同时以input作为内参照。目的蛋白结合的DNA浓度相对于input DNA浓度的倍数即为相对定量的结果。所有实验重复3次。引物序列如下:CREMα启动子区上游引物:5'-TGGGGAGATAGAGGTTGCAG-3',下游引物5'-CGCCAGAAATCCAATGACTT-3'。反应条件为:95 ℃,30 s;95 ℃,10 s,60 ℃,15 s,72 ℃,20 s,共40次循环。
1.6 RNA抽提与实时定量一步法RT-PCR按照厂家说明书,采用TRIzol对分离的CD4+ T细胞总RNA进行抽提,紫外/可见光分光光度计测定总RNA浓度及A260/A280比值。A260/A280比值均在1.8~2.0之间。所得RNA分装冻存于-80 ℃中。使用One Step SYBR PrimeScriptTM RT-PCR试剂盒,通过标准曲线相对定量法进行实时定量一步法RT-PCR检测mRNA水平。方法与前述的实时定量PCR类似,以CD4+ T细胞的RNA为模板,同时扩增β-actin作为内参照。同一标本目的基因的浓度相对于其β-actin的浓度的倍数即为相对定量的结果。所有实验重复3次。引物序列如下:CREMα上游引物5'-GAAACAGTTGAATCCCAGCATG ATGGAAGT-3',下游引物5'-TGCCCCGTGCTAGTC TGATATATG-3';β-actin上游引物5'-CGCGAGAAGAT TGACCCAGAT-3',下游引物5'-GCACTGTGTTGGCG TACAGG-3'。反应条件为:42 ℃,5 min;95 ℃,10 s;95 ℃,10 s,60 ℃,20 s,共40次循环。
1.7 统计分析采用SPSS 16.0 for windows统计软件储存和分析数据。计量资料以均数±标准差表示。两组独立样本均数之间进行比较采用两样本t检验,部分实验指标间作单因素直线相关分析,计算Pearson相关系数。P<0.05认为差异有统计学意义。
2 结果 2.1 ChIP微阵列结果在ChIP微阵列中,共筛查了20832个不同的基因启动子,其中552个基因启动子区H3K27me3水平在两组中差异达到2倍以上。在这些基因中,SLE CD4+ T细胞CREMα启动子区H3K27me3水平是正常对照CD4+ T细胞的0.23倍。
2.2 ChIP微阵列结果验证为了证实ChIP微阵列的结果,我们采用ChIP结合实时定量PCR检测了30名正常对照和30名SLE患者的CD4+ T细胞CREMα启动子区H3K27me3的水平。相对于正常对照,SLE患者CD4+ T细胞CREMα启动子区H3K27me3水平显著降低(正常对照vs SLE患者:2.723±0.659 vs 0.489±0.146,P<0.001),这与我们的ChIP微阵列结果相符。我们进一步检测了SLE患者CD4+ T细胞CREMα mRNA水平,结果证实了在SLE患者的CD4+ T细胞中,CREMα启动子区的H3K27me3与其mRNA水平呈负相关(r=-0.796,P<0.001,图 1)。
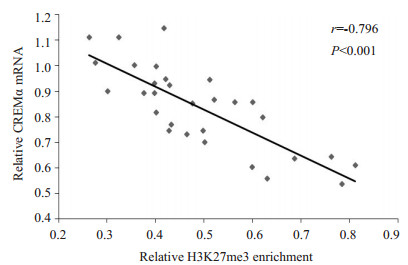
|
图 1 SLE CD4+ T细胞CREMα启动子区H3K27me3的水平与CREMα mRNA表达的相关 Figure 1 Correlation between H3K27me3 enrichment within the CREMα promoter in SLE CD4+ T cells and the levels of CREMα mRNA. |
ChIP结合实时定量PCR结果显示,相对于正常对照,SLE患者CD4+ T细胞CREMα启动子区JMJD3水平明显增加(P<0.001,图 2A)。且在SLE患者的CD4+ T细胞中,此区域的JMJD3水平与H3K27me3呈负相关(r=-0.803,P<0.001,图 2B),而与CREMα mRNA水平呈正相关(r=0.697,P<0.001,图 2C)。然而,SLE患者和正常对照CD4+ T细胞的CREMα启动子区UTX(P= 0.172)及EZH2 (P=0.281)水平并无明显差异(图 2A)。
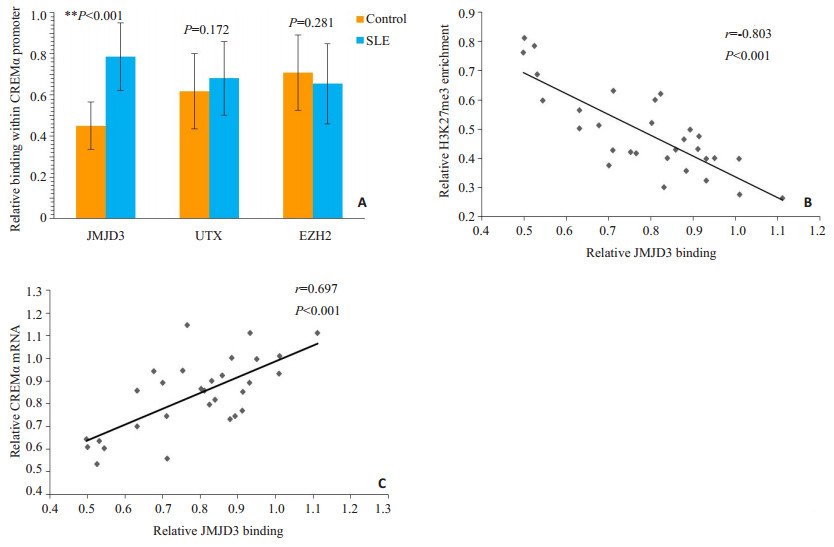
|
图 2 正常对照和SLE CD4+ T细胞CREMα启动子区JMJD3、UTX和EZHZ的水平及JMJD3与H3K27me3、CREMα水平的相关 Figure 2 Levels of JMJD3, UTX and EZH2 binding within the CREMα promoter region in CD4+ T cells from healthy controls and SLE patients and the correlations of JMJD3 with H3K27me3 and CREMα. A: Relative levels of JMJD3, UTX, and EZH2 within the HPK1 promoter region in healthy and SLE CD4+ T cells. B: Correlation between JMJD3 promoter binding and H3K27me3 level in SLE CD4+ T cells. C: Correlation between JMJD3 promoter binding and CREMα mRNA level in SLE CD4+ T cells. |
SLE患者发生自身免疫的关键在于CD4+ T细胞过度活化,进而刺激B细胞,结果导致各种自身抗体过度产生。而CD4+ T细胞某些免疫相关基因启动子区的表观遗传学改变则是CD4+ T细胞过度活化的重要原因。但目前的研究大多集中于DNA甲基化上[26-27],而对SLE CD4+ T细胞组蛋白修饰的探讨则非常有限。
已知H3K27me3能抑制基因的转录。它可与PRC1中的Pc蛋白结合,从而募集PRC1到染色质。PRC1可阻断转录活化因子及染色质重塑因子与DNA结合,并阻碍RNA聚合酶Ⅱ发动的转录;此外,PRC1还能与组蛋白去乙酰化酶相联,后者能抑制基因的转录;而且,PRC1和H3K27me3还能阻碍正性活化标志,例如H3K4的甲基化[28-29]。因此,H3K27me3一直是表观遗传学的研究热点之一。为了探讨SLE患者CD4+ T细胞的基因启动子区H3K27me3水平与正常对照有无差异,我们通过ChIP微阵列对正常对照和SLE患者的CD4+ T细胞各种基因启动子区的H3K27me3水平进行了检测和筛选,结果我们发现,SLE患者CD4+ T细胞CREMα启动子区H3K27me3的水平较低,这与SLE患者CD4+ T细胞CREMα表达水平升高正相吻合。
近年来,CREMα在SLE中所起到的作用已得到了充分的研究和验证,然而,引起SLE T细胞CREMα增加的分子机制至今仍不清楚。ChIP微阵列的结果提示了我们,可能正是由于SLE患者CD4+ T细胞CREMα启动子区H3K27me3水平降低导致了CREMα水平升高。为此,我们首先通过ChIP结合实时定量PCR对ChIP微阵列的结果进行了验证,结果正如所料,SLE患者CD4+ T细胞CREMα启动子区H3K27me3的水平显著低于正常对照。而且,我们发现H3K27me3与CREMα mRNA的水平呈负相关。这些结果表明SLE患者CD4+ T细胞CREMα水平升高的原因可能是因为其启动子区H3K27me3水平较低所致。
那么,SLE患者CD4+ T细胞CREMα启动子区H3K27me3水平降低的原因又是什么呢?前文已述,H3K27me3的水平由组蛋白去甲基化酶JMJD3、UTX和组蛋白甲基转移酶EZH2共同参与调控。于是我们采用ChIP结合实时定量PCR对这3种H3K27甲基化调控酶在CREMα启动子区的表达进行了检测,结果发现SLE患者CD4+ T细胞CREMα启动子区JMJD3显著增加,且JMJD3与H3K27me3水平呈负相关,而与CREMα mRNA水平呈正相关。然而,SLE患者和正常对照CD4+ T细胞CREMα启动子区的UTX及EZH2水平无明显差异。
综合上述结果,我们的研究提示SLE患者CD4+ T细胞CREMα启动子区JMJD3增加,这可导致此区域H3K27me3水平下降,从而促使CREMα增多,这一改变可能是引起SLE发病的重要机制之一。本研究为SLE的发病机理提供了新的理论依据,并为SLE的治疗提供了潜在的治疗靶点。
前文已述,H3K27me3能阻碍正性活化标志H3K4的甲基化。有趣的是,我们团队已证实了SLE患者CD4+ T细胞CREMα启动子区H3K4me3的水平明显高于正常对照[30]。而本次试验中,我们又发现SLE CD4+ T细胞CREMα启动子区H3K27me3水平显著降低,这就使得我们联想到这二者之间是否有因果关系,还是相互独立的事件?还有待进一步的研究。
| [1] | Moulton VR, Holcomb DR, Zajdel MC, et al. Estrogen upregulates cyclic AMP response element modulator alpha expression and downregulates interleukin-2 production by human T lymphocytes[J]. Mol Med, 2012, 18: 370-8. |
| [2] | Tenbrock K, Kyttaris VC, Ahlmann M, et al. The cyclic AMP response element modulator regulates transcription of the TCR zetachain[J]. J Immunol, 2005, 175(9): 5975-80. DOI: 10.4049/jimmunol.175.9.5975. |
| [3] | Hewagama A, Richardson B. The genetics and epigenetics of autoimmune diseases[J]. J Autoimmun, 2009, 33(1): 3-11. DOI: 10.1016/j.jaut.2009.03.007. |
| [4] | Pan Y, Sawalha AH. Epigenetic regulation and the pathogenesis of systemic lupus erythematosus[J]. Transl Res, 2009, 153(1): 4-10. DOI: 10.1016/j.trsl.2008.10.007. |
| [5] | Brooks WH, Le Dantec C, Pers JO, et al. Epigenetics and autoimmunity[J]. J Autoimmun, 2010, 34(3): J207-19. DOI: 10.1016/j.jaut.2009.12.006. |
| [6] | Zhang Q, Long H, Liao J, et al. Inhibited expression of hematopoietic progenitor kinase 1 associated with loss of jumonji domain containing 3 promoter binding contributes to autoimmunity in systemic lupus erythematosus[J]. J Autoimmun, 2011, 37(3): 180-9. DOI: 10.1016/j.jaut.2011.09.006. |
| [7] | Zhang P, Su Y, Lu Q. Epigenetics and psoriasis[J]. J Eur Acad Dermatol Venereol, 2012, 26(4): 399-403. DOI: 10.1111/jdv.2012.26.issue-4. |
| [8] | Hong S, Cho YW, Yu LR, et al. Identification of JmjC domaincontaining UTX and JMJD3 as histone H3 lysine 27 demethylases[J]. Proc Natl Acad Sci USA, 2007, 104(47): 18439-44. DOI: 10.1073/pnas.0707292104. |
| [9] | De Santa F, Totaro MG, Prosperini E, et al. The histone H3 lysine-27 demethylase Jmjd3 links inflammation to inhibition of polycomb-mediated gene silencing[J]. Cell, 2007, 130(6): 1083-94. DOI: 10.1016/j.cell.2007.08.019. |
| [10] | Banka S, Lederer D, Benoit V, et al. Novel KDM6A (UTX) mutations and a clinical and molecular review of the X-linked Kabuki syndrome (KS2)[J]. Clin Genet, 2015, 87(3): 252-8. DOI: 10.1111/cge.2015.87.issue-3. |
| [11] | Choi HJ, Park JH, Park M, et al. UTX inhibits EMT-induced breast CSC properties by epigenetic repression of EMT genes in cooperation with LSD1 and HDAC1[J]. EMBO Rep, 2015, 16(10): 1288-98. DOI: 10.15252/embr.201540244. |
| [12] | Fujii S, Ito K, Ito Y, et al. Enhancer of zeste homologue 2 (EZH2) down-regulates RUNX3 by increasing histone H3 methylation[J]. J Biol Chem, 2008, 283(25): 17324-32. DOI: 10.1074/jbc.M800224200. |
| [13] | Hedrich CM, Rauen T, Kis-Toth K, et al. cAMP-responsive element modulator alpha (CREMalpha) suppresses IL-17F protein expression in T lymphocytes from patients with systemic lupus erythematosus (SLE)[J]. J Biol Chem, 2012, 287(7): 4715-25. DOI: 10.1074/jbc.M111.323261. |
| [14] | Hedrich CM, Crispin JC, Rauen T, et al. cAMP response element modulator alpha controls IL2 and IL17A expression during CD4 lineage commitment and subset distribution in lupus[J]. Proc Natl Acad Sci USA, 2012, 109(41): 16606-11. DOI: 10.1073/pnas.1210129109. |
| [15] | Rauen T, Hedrich CM, Juang YT, et al. cAMP-responsive element modulator (CREM) alpha protein induces interleukin 17A expression and mediates epigenetic alterations at the interleukin-17A gene locus in patients with systemic lupus erythematosus[J]. J Biol Chem, 2011, 286(50): 43437-46. DOI: 10.1074/jbc.M111.299313. |
| [16] | Ohl K, Wiener A, Schippers A, et al. Interleukin-2 treatment reverses effects of cAMP-responsive element modulator alpha-overexpressing T cells in autoimmune-prone mice[J]. Clin Exp Immunol, 2015, 181(1): 76-86. DOI: 10.1111/cei.12629. |
| [17] | Gomez-Martin D, Diaz-Zamudio M, Crispin JC, et al. Interleukin 2 and systemic lupus erythematosus: beyond the transcriptional regulatory net abnormalities[J]. Autoimmun Rev, 2009, 9(1): 34-9. DOI: 10.1016/j.autrev.2009.02.035. |
| [18] | Koga T, Hedrich CM, Mizui M, et al. CaMK4-dependent activation of AKT/mTOR and CREM-alpha underlies autoimmunityassociated Th17 imbalance[J]. J Clin Invest, 2014, 124(5): 2234-45. DOI: 10.1172/JCI73411. |
| [19] | Crispin JC, Tsokos GC. IL-17 in systemic lupus erythematosus[J]. J Biomed Biotechnol, 2010, 2010: 943254. |
| [20] | Nalbandian A, Crispin JC, Tsokos GC. Interleukin-17 and systemic lupus erythematosus: current concepts[J]. Clin Exp Immunol, 2009, 157(2): 209-15. DOI: 10.1111/cei.2009.157.issue-2. |
| [21] | Tenbrock K, Juang YT, Leukert N, et al. The transcriptional repressor cAMP response element modulator alpha interacts with histone deacetylase 1 to repress promoter activity[J]. J Immunol, 2006, 177(9): 6159-64. DOI: 10.4049/jimmunol.177.9.6159. |
| [22] | Rauen T, Grammatikos AP, Hedrich CM, et al. cAMP-responsive element modulator alpha (CREMalpha) contributes to decreased Notch-1 expression in T cells from patients with active systemic lupus erythematosus (SLE)[J]. J Biol Chem, 2012, 287(51): 42525-32. DOI: 10.1074/jbc.M112.425371. |
| [23] | Verjans E, Ohl K, Yu Y, et al. Overexpression of CREMalpha in T cells aggravates lipopolysaccharide-induced acute lung injury[J]. J Immunol, 2013, 191(3): 1316-23. DOI: 10.4049/jimmunol.1203147. |
| [24] | Lippe R, Ohl K, Varga G, et al. CREMalpha overexpression decreases IL-2 production, induces a T(H)17 phenotype and accelerates autoimmunity[J]. J Mol Cell Biol, 2012, 4(2): 121-3. DOI: 10.1093/jmcb/mjs004. |
| [25] | Hochberg MC. Updating the American College of Rheumatology revised criteria for the classification of systemic lupus erythematosus[J]. Arthritis Rheum, 1997, 40(9): 1725. |
| [26] | Zhao M, Sun Y, Gao F, et al. Epigenetics and SLE: RFX1 downregulation causes CD11a and CD70 overexpression by altering epigenetic modifications in lupus CD4+ T cells[J]. J Autoimmun, 2010, 35(1): 58-69. DOI: 10.1016/j.jaut.2010.02.002. |
| [27] | Lu Q, Wu A, Tesmer L, et al. Demethylation of CD40LG on the inactive X in T cells from women with lupus[J]. J Immunol, 2007, 179(9): 6352-8. DOI: 10.4049/jimmunol.179.9.6352. |
| [28] | Lund AH, van Lohuizen M. Polycomb complexes and silencing mechanisms[J]. Curr Opin Cell Biol, 2004, 16(3): 239-46. DOI: 10.1016/j.ceb.2004.03.010. |
| [29] | Kondo Y, Shen L, Cheng AS, et al. Gene silencing in cancer by histone H3 lysine 27 trimethylation independent of promoter DNA methylation[J]. Nat Genet, 2008, 40(6): 741-50. DOI: 10.1038/ng.159. |
| [30] | Zhang Q, Ding S, Zhang H, et al. Increased Set1 binding at the promoter induces aberrant epigenetic alterations and up-regulates cyclic adenosine 5'-monophosphate response element modulator alpha in systemic lupus erythematosus[J]. Clin Epigenetics, 2016, 8: 126. DOI: 10.1186/s13148-016-0294-2. |
 2017, Vol. 37
2017, Vol. 37

