2. 广州医学院附属武警医院泌尿外科,广东 广州 510000
2. Department of Urology, Armed Police Hospital of Guangdong Province, Guangzhou 510507, China
肾癌(RCC)占成人恶性肿瘤的2%~3%,中位发病年龄65岁。在所有肾肿瘤中肾癌约占85%~90%,其中70%为肾透明细胞癌[1]。肾癌缺乏早期特异性诊断标志物,进展期肾癌或转移性肾癌5年生存率很低[2]。因此寻找早期诊断标志物和筛选有效的治疗靶点对提高肾癌生存率非常重要。
近年来,蛋白质组学技术已经成为研究临床肿瘤疾病普查和早期诊断蛋白质标记物的有效工具,同时为研究肿瘤产生和发展机制提供了技术平台[3-4]。国内外已有一些肾癌蛋白质组学研究[5-17],不少报道均采用普通双向凝胶电泳(2-DE)筛选肾癌相关蛋白。但普通2-DE技术存在敏感性、特异性、重复性较差,对疏水蛋白、小分子蛋白、膜蛋白分离有限等不足。国内未见采用荧光差异双向凝胶电泳(2-D DIGE)技术筛选肾癌差异表达蛋白的定量比较蛋白质组学研究报道。本课题组首先采用2-D DIGE联合质谱技术筛选并鉴定了中国人肾癌及其癌旁正常肾组织差异表达蛋白质,其中一些蛋白为首次筛选出的肾癌差异表达蛋白。
1 资料和方法 1.1 标本采集与分组随机选取标本库中肾癌患者15例,肾癌组织及配对癌旁正常肾组织均来自南方医科大学附属南方医院泌尿外科肾癌根治性切除患者,肿瘤标本均经病理及免疫组化证实为肾透明细胞癌,癌旁组织为癌灶周围5 cm以外正常肾组织,均经病理证实无癌细胞浸润。留取手术新鲜标本时用生理盐水冲洗3次,滤纸轻轻吸干标本中的生理盐水,并尽量切除结缔组织及坏死组织,所有标本装入已标记好的2 mL冷冻管内,在离体后20 min内放入液氮罐,实验前转存-80 ℃低温冰箱备用。
将15对标本随机分为3组,每组5对标本,分别取等量标本混合后得到相应的3组肿瘤组织和癌旁正常肾组织,即共6份样品。
1.2 实验试剂及仪器 1.2.1 主要试剂固相pH梯度干胶条(IPG dry strip pH 4-7NL,24 cm)、IPG缓冲液Bio-Lytes+4/7、荧光染料Cy2、Cy3和Cy5均为美国通用电气医疗集团(GE Healthcare, USA)公司产品;尿素、二硫苏糖醇、3-[(3-胆酰胺丙基)-二乙铵]-丙磺酸、十二烷基磺酸钠、三羧基氨基甲烷、丙烯酰胺、甲叉双丙烯酰胺、甘氨酸、过硫酸铵、碘代乙酰胺、矿物油、甘油、胰蛋白酶、碳酸氢铵、三氟乙酸、乙腈、α-氰基-4-羟基肉桂酸均为美国Sigma产品;硫脲为Fluka产品;硫代硫酸钠、无水碳酸钠、甲醛、硝酸银、乙醇、冰醋酸、醋酸钠、甘氨酸均为国产分析纯。所有缓冲液均用Milli-Q水配制。
1.2.2 仪器及软件等电聚焦仪(IPG-PhorⅡ)、垂直电泳系统[GE Ettan DALT six(26 cm×20 cm)]均购自GE Healthcare,LKB-MultiTempⅡ低温循环水浴箱购自Pharmacia,Milli-Q超纯水仪购自Millipore,低温高速离心机为Beckman产品。SpectraMax M5多功能酶标仪购自美国分子仪器公司。脱色摇床、漩涡振荡器均为国产。DIGE扫描仪为Typhoon 9410购自美国GE Healthcare。SpeedVac真空干燥仪(美国Thermo Savant),VoyagerDE STR质谱仪(美国Applied Biosystems)。凝胶扫描软件为Image Quant TL(Amersham Biosciences);凝胶分析软件为Decyder软件(Amersham Biosciences)。
1.3 Cy-染料标记和2-D DIGE采用美国GE Healthcare提供的实验指南进行实验设计。共6份样品,每份样品各50 μg,平均用CyDyeDIGE染料Cy3和Cy5分别随机交叉标记(400 pmoL染料:50 μg蛋白裂解液1 μL,pH值8.0~9.0之间),内标用等量肾癌及癌旁组织(共6份样品)裂解液混合,并用Cy2标记。4 ℃下标记反应30 min后用IPG胶条(pH 4~7, 24 cm)进行第1向等电聚焦电泳(IEF),溶胀液(DeStreak Reagent)由美国GE Healthcare生产。然后用Ettan DALT系统进行第二向十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE)。
1.4 图像采集和分析用Typhoon 9410荧光扫描仪对凝胶进行扫描,并用DeCyder软件进行分析。共3张胶,每张胶扫描后得到3张扫描胶图(共9张图),对每一张胶图上的蛋白点扫描进行胶内分析,然后对不同胶上的同一个蛋白点与内标匹配,进行胶间分析,对匹配后的蛋白点的相对量比较(Cy3/Cy2或Cy5/Cy2)后确定差异蛋白。
1.5 质谱鉴定和数据库检索用1 mg样品行双向凝胶电泳,建立制备胶,第2向凝胶电泳结束后进行考马斯蓝染色,与DIGE胶图匹配后手动切胶切取相匹配的蛋白质点。胶内胰蛋白酶消化并提取肽混合物,对其用基质辅助激光解吸电离飞行时间质谱(MALDI-TOF MS)和MALDI-TOF/TOF MS对差异蛋白点进行鉴定,获取肽质量指纹图谱。对蛋白质的质谱分析结果数据使用国际互联网上的蛋白质数据库MSDB提供的Masort检索程序进行检索(http://www.matrixscience.com),数据库选择Swiss-Prot。
1.6 统计学方法由DeCyder软件进行统计学分析,肾癌组及癌旁组之间差异蛋白采用t检验进行定量比较,两组蛋白含量比值在1.5倍及以上,P<0.05,表示差异有统计学意义,被认为是差异表达蛋白点。
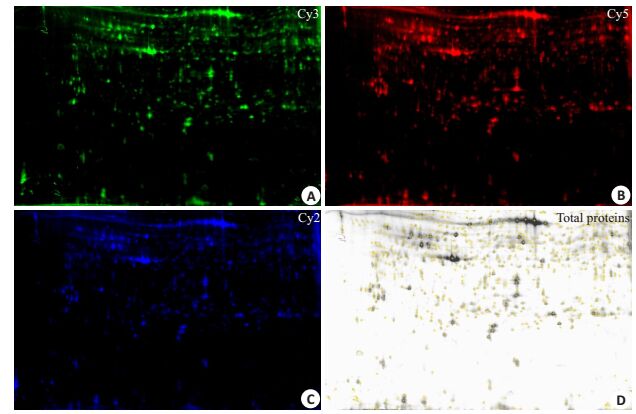
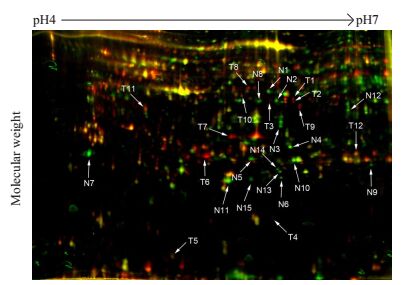
2 结果 2.1 DIGE凝胶扫描及Decyder软件分析结果经双向电泳获得3张胶,分别用3种不同波长488 nm(Cy2),532 nm(Cy3),633 nm(Cy5)的激光扫描,每张胶得3个扫描图谱,即共得到9个图谱。图 1是一张胶中3个激光扫描图及总蛋白点检测结果图。采用Decyder软件分析,从中筛选出35个差异表达蛋白质点(Average ratio≥1.5,P<0.05)。经考马斯蓝染色建立制备胶后在上述差异表达蛋白质点中共找到27个差异蛋白点。图 2为Cy3和Cy5的软件叠加图,图中箭头指示为27个差异蛋白质点作为下一步质谱鉴定之用。
|
图 1 一张胶上不同波长扫描得到的荧光图谱 Figure 1 2-D DIGE images of gel. A: Cy3-labeled adjacent normal renal tissue; B: Cy5-labeled renal cell carcinoma; C: Cy2-labeled the mixed sample internal standard; D: The total protein spots map. |
|
图 2 筛选出用于质谱鉴定的27个差异蛋白质点标记图 Figure 2 Identification of 27 differentially expessed proteins by 2D-DIGE for mass spectral analysis; N: Protein spots down regulated in tumor, T: Protein spots up regulated in tumor. |
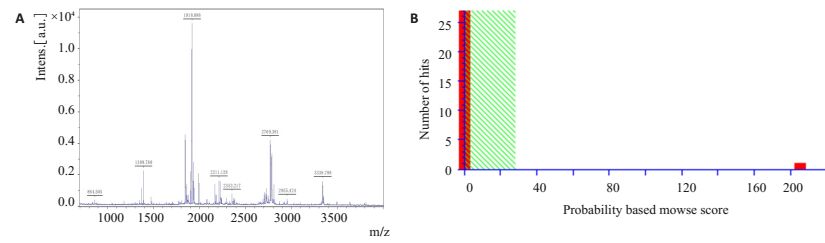
27个差异蛋白质点均得到成功鉴定,共鉴定出26种蛋白质,图 3所示为蛋白质点T1的肽质量指纹图谱及数据库匹配结果。其中11种蛋白质在肾癌组织中上调,分别为加帽蛋白G(CAPG)、纤维蛋白原-β(FIBB)、核苷二磷酸激酶A(NDPK-A)、β-防御素107(D107A)、尼克酰胺-N-甲基转移酶(NNMT)、膜联蛋白A4(ANXA4)、磷酸甘油酸激酶1(PGK1)、α-烯醇化酶(ENOA)、血清白蛋白(ALBU)、微纤维相关糖蛋白4(MFAP4)、磷酸甘油酸变位酶1(PGAM1)(表 1)。
|
图 3 CAPG蛋白(蛋白质点T1)肽指纹图(A)及匹配图(B) Figure 3 Peptide mass fingerprint (A) and probability based mowse score (B) of Macrophage-capping protein (protein spot T1). |
| 表 1 质谱鉴定在肾癌组织和癌旁正常肾组织差异表达蛋白(12个上调的差异蛋白质点) Table 1 Mass spectrometry identification of differentially expressed proteins between tumor and normal tissue (12 protein spots up regulated) |
15个在肾癌组织中表达下调的蛋白质分别是:线粒体琥珀酰辅酶A连接酶-β(SUCB1)、甜菜碱-高半胱氨酸S-甲基转移酶2(BHMT2)、甘油-3-磷酸脱氢酶辅酶Ⅰ(GPDA)、酮己糖激酶(KHK)、过氧化物酶-4(PRDX4)、含自水解酶结构域蛋白14B(ABHEB)、钙结合蛋白(CALB1)、线粒体短/支链特异性酰基辅酶A脱氢酶(ACDSB),细胞色素b-cl复合亚基Rieske样蛋白1(UCRIL)、线粒体烯酰辅酶A水合酶(ECHM)、谷胱甘肽S -转移酶P(GSTP1)、醛糖1-表异构酶(GALM)、线粒体NADH脱氢酶黄素蛋白2(NDUV2)、Ribosyldihydronicotinami脱氢酶(NQO2)、谷胱甘肽过氧化物酶3(GPX3)(表 2)。
| 表 2 质谱鉴定在肾癌组织和癌旁正常肾组织差异表达蛋白(15个下调的差异蛋白质) Table 2 Mass spectrometry identification of differentially expressed proteins between tumor and normal tissue (15 proteins down regulated) |
检索国外文献已有不少报道采用普通银染2-DE技术筛选肾癌组织差异表达蛋白的研究[10-15]。但国内对肾癌及配对癌旁组织的蛋白质组学研究报道较少,仅有靖永胜等[17]对5例肾癌患者组织标本采用普通2-DE筛选并质谱鉴定了7个差异表达蛋白。据我们所知国外采用2-D DIGE筛选肾癌组织差异表达蛋白的报道也较少[18-20]。肾癌是一种具有明显肿瘤异质性的恶性肿瘤,不同的肾癌患者其肿瘤行为、临床转归和分子生物学特点往往明显不同[21]。再者,因为不同的实验研究所采取的实验技术往往有一定的差异,所以筛选出来的差异蛋白的数量和种类也有一定的不同,很多有意义的差异蛋白不可能被一次性完全筛选鉴定出来。所以本研究以中国人肾癌患者组织标本为研究对象,依托我们的蛋白组学研究平台筛选可能有意义的差异表达蛋白质。目前国内尚无采用2-D DIGE筛选肾癌组织和配对癌旁正常肾组织中差异表达蛋白的研究报道。
2-D DIGE是近年来蛋白质组学研究的一种新技术,其在传统2-DE技术基础上,结合了多重荧光分析的方法,在同一块凝胶上共同分离多个分别由不同荧光染料标记的样品,并第一次引入了内标的概念,这样理论上使的每个蛋白点都有自己的内标,并且应用软件全自动地根据每个蛋白点的内标对其表达量进行校准,极大地提高了结果的准确性,可靠性和重复性[22-24]。降低了普通双向凝胶电泳中不同凝胶间的误差,减少了实验重复次数。本研究对15例肾透明细胞癌患者标本分为3组采用标本混合法,通过2-D DIGE技术,获得了较好的荧光标记双向凝胶电泳图,软件分析及考马斯亮蓝染色筛选并切取了27个差异表达蛋白点,用于后续质谱鉴定。显示DIGE技术具有较好的敏感性和重复性。
对本研究质谱鉴定出的26种差异表达蛋白质分析显示,肾癌发生、发展过程中蛋白质水平的改变非常复杂。根据蛋白质特点及功能的不同,这些差异表达蛋白质主要可以分为以下几类:细胞外基质相关蛋白类、肌动蛋白细胞骨架相关蛋白类、钙依赖性磷脂结合蛋白超家族类、内源性抗菌肽家族、抗氧化酶蛋白类、脂质代谢相关蛋白类、代谢酶类等。其中线粒体短/支链特异性酰基辅酶A脱氢酶、醛糖1-表异构酶、PRDX4、CAPG、微纤丝相关糖蛋白4、β-防御素107等6种蛋白是我们首次采用蛋白质组学技术成功筛选的肾癌差异表达蛋白质。线粒体短/支链特异性酰基辅酶A脱氢酶和醛糖1-表异构酶为代谢酶类,本研究发现其在肾癌组织中表达均显著下调;研究显示过氧化物酶家族与肿瘤有关,在不同类型肿瘤中过氧化物酶的不同成员呈现不同表达模式,起抑癌基因或癌基因的作用[25-26]。PRDX4作为过氧化物酶家族成员在肿瘤中的研究结果报道不尽相同,具有一定争议,在肾癌中的作用需进一步研究;CAPG作为肌动蛋白结合蛋白的一种,文献报道CAPG蛋白在肿瘤细胞浸润和转移过程中具有重要的作用[27-28],其在肾癌中的作用尚不清楚,值得后续研究;微纤丝相关糖蛋白4和β-防御素107为本研究首次筛选出来的肾癌差异蛋白,在肾癌组织中表达均显著上调,检索相关文献,均未发现在肾癌中的相关研究报道。此外,我们还筛选到了一个未知预测蛋白(UCRIL),其在肾癌中的作用更有待我们进一步的研究。
总之,我们采用2-D DIGE技术筛选的肾癌及癌旁组织差异表达蛋白质参与了肾癌发生、发展过程中的多个环节和步骤。后期研究工作中,本课题组将对感兴趣差异蛋白进行深入研究。相信对进一步揭示肾癌发生、发展及转移机制有一定价值,一些差异表达蛋白有可能成为肾癌的特异性肿瘤标志物和新的潜在治疗靶点。
| [1] | Rini B, Campbell SC, Escudier B. Renal cell carcinoma[J]. Lancet, 2009, 373(9669): 1119-32. DOI: 10.1016/S0140-6736(09)60229-4. |
| [2] | Lin-Tsai O, Kaffenberger S, Stratton K, et al. Statin use is associated with improved survival in patients undergoing surgery for renal cell carcinoma[J]. Urol Oncol, 2013, 189(4, S): E676-7. |
| [3] | Chinello C, L'imperio V, Stella M, et al. The proteomic landscape of renal tumors[J]. Expert Rev Proteomics, 2016, 13(12): 1103-20. DOI: 10.1080/14789450.2016.1248415. |
| [4] | Sandim V, Pereira DA, Ornellas AA, et al. Renal cell carcinoma and proteomics[J]. Urol Int, 2010, 84(4): 373-7. DOI: 10.1159/000296283. |
| [5] | Zhao ZH, Wu F, Ding ST, et al. Label-free quantitative proteomic analysis reveals potential biomarkers and pathways in renal cell carcinoma[J]. Tumour Biol, 2015, 36(2): 939-51. DOI: 10.1007/s13277-014-2694-2. |
| [6] | Neely BA, Wilkins CE, Marlow LA, et al. Proteotranscriptomic analysis reveals stage specific changes in the molecular landscape of clear-cell renal cell carcinoma[J]. PLoS One, 2016, 11(4): e0154074. DOI: 10.1371/journal.pone.0154074. |
| [7] | Na CH, Hong JH, Kim WS, et al. Identification of protein markers specific for papillary renal cell carcinoma using imaging mass spectrometry[J]. Mol Cells, 2015, 38(7): 624-9. DOI: 10.14348/molcells.2015.0013. |
| [8] | Lebdai S, Verhoest G, Parikh H, et al. Identification and validation of TGFBI as a promising prognosis marker of clear cell renal cell carcinoma[J]. Urol Oncol, 2015, 33(2): e11-8. |
| [9] | Weisser J, Lai ZW, Bronsert P, et al. Quantitative proteomic analysis of formalin-fixed, paraffin-embedded clear cell renal cell carcinoma tissue using stable isotopic dimethylation of primary amines[J]. BMC Genomics, 2015, 16(4): 559. |
| [10] | Zhuang ZP, Huang S, Kowalak JA, et al. From tissue phenotype to proteotype: Sensitive protein identification in microdissected tumor tissue[J]. Int J Oncol, 2006, 28(1): 103-10. |
| [11] | Perroud B, Lee J, Valkova N, et al. Pathway analysis of kidney cancer using proteomics and metabolic profiling[J]. Mol Cancer, 2006, 5(3): 64. |
| [12] | Seliger B, Fedorushchenko A, Brenner WA, et al. Ubiquitin COOHterminal hydrolase 1: A biomarker of renal cell carcinoma associated with enhanced tumor cell proliferation and migration[J]. Clin Cancer Res, 2007, 13(1): 27-37. DOI: 10.1158/1078-0432.CCR-06-0824. |
| [13] | Lichtenfels R, Dressler SP, Zobawa M, et al. Systematic comparative protein expression profiling of clear cell renal cell carcinoma: a pilot study based on the separation of tissue specimens by twodimensional gel electrophoresis[J]. Mol Cell Proteomics, 2009, 8(12): 2827-42. DOI: 10.1074/mcp.M900168-MCP200. |
| [14] | Vieira DA, Sandim V, Ornellas AA, et al. Differencial proteome of clear-cell renal cell carcinoma (ccRCC) tissues[J]. Int Braz J Urol, 2013, 39(1): 83-94. DOI: 10.1590/S1677-5538.IBJU.2013.01.11. |
| [15] | Lu ZJ, Yao YQ, Song Q, et al. Metabolism-related enzyme alterations identified by proteomic analysis in human renal cell carcinoma[J]. Onco Targets Ther, 2016, 9(4): 1327-37. |
| [16] | Wang J, Zhao XL, Qi J, et al. Eight proteins play critical roles in RCC with bone metastasis via mitochondrial dysfunction[J]. Clin Exp Metastasis, 2015, 32(6): 605-22. DOI: 10.1007/s10585-015-9731-4. |
| [17] | 靖永胜, 刘师莲, 张旭华, 等. 肾癌及癌旁组织蛋白质组学的比较研究[J]. 山东大学学报:医学版, 2007, 45(8): 781-4. |
| [18] | Junker H, Venz S, Zimmermann U, et al. Stage-Related alterations in renal cell carcinoma -comprehensive quantitative analysis by 2D-DIGE and protein network analysis[J]. PLoS One, 2011, 6(7): e21867. DOI: 10.1371/journal.pone.0021867. |
| [19] | Raimondo F, Salemi C, Chinello CA, et al. Proteomic analysis in clear cell renal cell carcinoma: identification of differentially expressed protein by 2-D DIGE[J]. Mol Biosyst, 2012, 8(4): 1040-51. DOI: 10.1039/c2mb05390j. |
| [20] | Hosoya N, Sakumoto M, Nakamura Y, et al. Proteomics identified nuclear N-myc downstream-regulated gene 1 as a prognostic tissue biomarker candidate in renal cell carcinoma[J]. Biochim Biophys Acta, 2013, 1834(12): 2630-9. DOI: 10.1016/j.bbapap.2013.08.009. |
| [21] | Nelson EC, Evans CP, Lara J. Renal cell carcinoma: Current status and emerging therapies[J]. Cancer Treat Rev, 2007, 33(3): 299-313. DOI: 10.1016/j.ctrv.2006.12.005. |
| [22] | Gharbi S, Gaffney P, Yang A, et al. Evaluation of two-dimensional differential gel electrophoresis for proteomic expression analysis of a model breast cancer cell system[J]. Mol Cell Proteomics, 2002, 1(2): 91-8. DOI: 10.1074/mcp.T100007-MCP200. |
| [23] | Zhou G, Li H, Decamp D, et al. 2D differential in-gel electrophoresis for the identification of esophageal scans cell cancer-specific protein markers[J]. Mol Cell Proteomics, 2002, 1(2): 117-24. DOI: 10.1074/mcp.M100015-MCP200. |
| [24] | Greengauz-Roberts O, Stoppler H, Nomura S, et al. Saturation labeling with cysteine-reactive cyanine fluorescent dyes provides increased sensitivity for protein expression profiling of lasermicrodissected clinical specimens[J]. Proteomics, 2005, 5(7): 1746-57. DOI: 10.1002/(ISSN)1615-9861. |
| [25] | Nicolussi A, D'inzeo S, Capalbo C, et al. The role of peroxiredoxins in cancer[J]. Mol Clin Oncol, 2017, 6(2): 139-53. DOI: 10.3892/mco.2017.1129. |
| [26] | Park MH, Jo M, Kim YR, et al. Roles of peroxiredoxins in Cancer, neurodegenerative diseases and inflammatory diseases[J]. Pharmacol Ther, 2016, 163(3): 1-23. |
| [27] | Kimura K, Ojima H, Kubota D, et al. Proteomic identification of the macrophage-capping protein as a protein contributing to the malignant features of hepatocellular carcinoma[J]. J Proteomics, 2013, 78(2): 362-73. |
| [28] | Westbrook JA, Cairns DA, Peng JH, et al. CAPG and GIPC1: breast cancer biomarkers for bone metastasis development and treatment[J]. J Natl Cancer Inst, 2016, 108(4): 782. |
 2017, Vol. 37
2017, Vol. 37

