糖尿病足骨髓炎(DFO)是由糖尿病足软组织感染蔓延至深部骨及骨髓组织的糖尿病(DM)严重并发症[1]。有研究显示,约20%的糖尿病足溃疡患者合并有骨髓炎[2],且DFO患者的截趾/肢率高达59.4%[3]。近年研究表明,内科抗感染治疗可降低骨髓炎患者的截趾/肢率和截肢风险[4-5],而感染骨组织病原菌及抗生素敏感性的鉴定是其关键环节。据估计,生物界99%以上的细菌和真菌目前无法进行人工培养[6-7],因而临床上基于培养法的微生物鉴别方式在鉴定某些新物种及难以培养微生物时存在一定的困难[8],无法有效地指导感染的控制治疗,使半数以上的骨髓炎患者仍难以摆脱截肢的厄运。16s rRNA基因普遍存在于原核微生物中,是研究微生物多样性应用最广泛的分子标记[9],可运用于微生物菌落及细菌分型。高通量测序较第1代测序通量高、速度快、读长长、准确度高,早在2010年已超过1200种细菌的全基因组被完整测序[10]。此前已有研究将16s rRNA测序技术运用于DFO病原微生物的分析,但其标本收集方法不统一,未阐明DFO病原微生物的分布特点,且对优势菌属的分析不够全面[11]。本研究统一采用临床上常用的清创术收集标本,并全面分析DFO病原微生物多样性及分布特点,深度探索16s rRNA测序在菌群鉴定方面的优势。
1 资料和方法 1.1 研究对象2016年9月~2017年4月于本院内分泌科住院治疗的16例确诊为DFO患者,男性9例,女性7例,年龄60.69±9.69岁,DM病程8.74±5.95年,糖尿病足病程17.50(10.00~52.50)d。患者DM的诊断依据于1999年世界卫生组织(WHO)的诊断标准。
1.2 诊断标准 1.2.1 足部骨髓炎诊断依据[1]足部红肿、肿胀;感染指标[白细胞计数(WBC)、中性粒细胞计数(n)、降钙素原(PCT)、C反应蛋白(CRP)、红细胞沉降率(ESR)等]升高;骨探针试验阳性或骨外露;X线显示骨质破坏;MRI显示骨髓炎性改变;骨微生物培养阳性或骨病理检查阳性。
1.2.2 糖尿病周围神经病变(DPN)诊断标准[12]明确的糖尿病病史或至少糖代谢异常证据;在诊断糖尿病时或之后出现的神经病变;临床症状和体征与DPN的表现相符;有临床症状(疼痛、麻木、感觉异常等)者,存在5项检查异常(温度觉异常、尼龙丝检查足部感觉减退或消失、震动觉异常、踝反射消失、神经传导速度有2项或2项以上减慢)中任1项及1项以上;或无临床症状者,存在5项检查异常中任2项及2项以上异常;排除其他病变如颈椎病变、脑梗塞等及药物引起的神经损害。
1.2.3 糖尿病周围血管病变(PAD)诊断标准[13]有间歇性跛行、静息痛、坏疽等下肢血管病变表现;局部动脉搏动减弱或消失,皮温降低;踝肱指数(ABI)≤0.9;血管超声、CTA、MRA或动脉造影等检查示血管硬化、阻塞或闭塞;排除血管闭塞性脉管炎等其它动脉性疾病。
1.2.4 糖尿病视网膜病变(DR)诊断标准[12]眼底照相或眼底检查出现硬性渗出、棉绒斑、视网膜内微血管异常、微血管瘤、静脉串珠、新生血管、视网膜内出血、玻璃体积血、视网膜前出血、纤维增生等任一异常改变。
1.2.5 Wagner分级法[14]0级,目前尚无溃疡,但存在发生足溃疡的危险因素;1级,足部皮肤表面溃疡,临床上无感染;2级,较深的溃疡,常合并软组织感染,可累及肌肉、筋膜或关节,但并无脓肿或骨的感染;3级,深部溃疡,伴有深部脓肿或骨髓炎;4级,局限性坏疽;5级,全足坏疽。
1.3 纳入及排除标准 1.3.1 纳入标准年龄≥18岁;符合糖尿病足骨髓炎的诊断;存在感染骨暴露;患者全身状况较好;患者及家属均同意参与研究。
1.3.2 排除标准创面周围存在严重的皮肤病;入院前有使用较长时间的免疫抑制剂治疗;存在影响骨髓炎创面的肿瘤;患者不配合;根据医师判断不适合入组。
1.4 方法 1.4.1 主要试剂动物组织/细胞DNA磁珠提取试剂盒(深圳易瑞)、TransStart Fastpfu DNA Polymerase PCR试剂盒(北京全式金)、蛋白酶K(Sigma)。
1.4.2 基本病例资料收集整理患者的病史、体格检查、物理检查、实验室检查、影像学检查等数据资料,包括性别、年龄、DM病程、糖尿病足病程、Wagner分级、ABI、糖化血红蛋白(HbAlc)、血常规、血生化、感染2项、肾功能、慢性并发症筛查等。
1.4.3 标本采集用大量生理盐水及过氧化氢溶液冲洗、消毒创面,清除创面周围坏死组织后,用无菌剪刀或咬骨钳取少量感染骨组织均分为3份,整个操作过程由专业清创师完成。其中两份分别立即送检验科进行培养鉴定和病理科进行病理组织学检查,另一份保存于-80 ℃中以进行16s rRNA测序。患者标本编号为Dd1、Dd2·····Dd16。
1.4.4 微生物培养标本的接种、分离等过程由专业的检验科人员参照全国临床检验操作规程进行。仅做一般细菌及真菌的培养及鉴定(不包括厌氧菌等特殊细菌培养),微生物及药敏鉴定采用BD phonex血培养分析仪和BD100全自动细菌鉴定药敏系统。
1.4.5 16s rRNA测序按照说明书用动物组织DNA提取试剂盒提取细菌基因组DNA,1%琼脂糖凝胶电泳检测后,用PCR试剂盒根据说明书使用特异性引物(341F:5'-CCTAYGGGRBGCASCAG-3'和806R:5'-GGACTACNNGGGTATCTAAT-3)'对16s rRNA V3-V4区基因片段运用ABI GeneAmp 9700 PCR仪进行扩增。将获得的扩增产物再次进行1%琼脂糖凝胶电泳检测,并混样、纯化,构建DNA文库,最后运用Illumina Hiseq2500测序平台检测(赛哲生物,广州)。
1.5 数据的整理与分析数据统计分析采用SPSS 22.0软件包,计量资料用均数±标准差(正态分布)或中位数(非正态分布)表示,采用t检验或非参数检验;计数资料用百分比表示,采用χ2检验。P < 0.05为差异存在统计学意义。
16s rRNA测序数据分析:根据Barcode序列和PCR扩增引物序列从下机数据中拆分出各样品数据,截去Barcode序列和引物序列,对样品的reads进行拼接,得到的拼接序列为原始Tags数据,Raw Tags经过过滤处低质量,和不符合长度的tags,以及过滤嵌合体和宿主序列,得到高质量的Tags数据(Clean Tags)。得到Clean Tags后,基于Clean Tags进行OTUs聚类分析。根据OTUs聚类结果,一方面对每个OTU的代表序列做物种注释,得到对应的物种注释信息和基于物种的丰度分布情况。同时,对OTUs进行丰度(统计,可视化)、Alpha多样性分析等,以得到样品内物种丰富度和均匀度信息、不同样品间的共有和特有OTUs信息等。另一方面,对OTUs进行多序列比对并构建系统发生树,并基于进化树信息和丰度信息进一步进行Beta多样性分析等,获得不同样品和分组的群落结构差异信息,通过PCoA和PCA、NMDS和距离矩阵热图等方式进行展示。
2 结果 2.1 一般资料(表 1)| 表 1 患者的一般资料 Table 1 Demographic and clinical data of the patients (n=16) |
Wagner分级为3~4级,3级与4级患者各8例;68.75%的患者入院前有抗生素治疗史;合并DPN、PAD和DR的患者分别占87.50%、68.75%和62.50%。
2.2 16s rRNA高通量测序结果 2.2.1 序列聚类分析(OTU分析)16例患者骨组织细菌16s rRNA V3-V4区基因片段扩增后,经1%琼脂糖凝胶电泳检测大小为500~600 bp。将原始序列过滤后所得的序列在相似度为97%的水平上进行聚类分析,共得到2488个操作分类单元(OTUs)和499896个优质序列。
2.2.2 Alpha多样性分析(图 1、2)
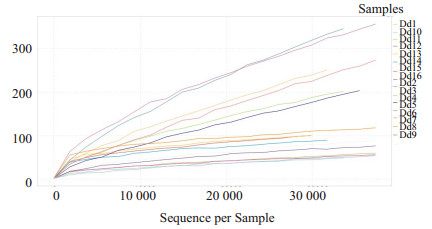
|
图 1 OUT稀释曲线 Figure 1 Rarefaction curve of OTU. |
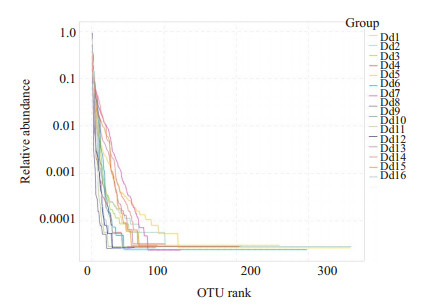
|
图 2 Rank abundance曲线 Figure 2 Rank abundance curve of OTU. |
样本细菌OTU稀释曲线趋于平缓,表明本次测序已基本覆盖样本的所有物种,能较好的展现样本细菌的多样性和分布。同时,样本Shannon曲线已到达平台期,表明测序量趋于饱和,本次测序深度足够体现样本的多样性,即使增加测序深度也不会影响物种多样性。Rank Abundance曲线在水平方向上跨度较大,在垂直方向上较为平缓,表明样本细菌OTU分类丰度较高,且分布较为均匀。
2.2.3 物种注释利用RDP比对注释软件根据16s rRNA基因数据库将OTU聚类序列相对丰度分别在界、门、纲、目、科、属、种水平上进行注释,以相对丰度大于或等于1%的物种为优势菌。16例样本中共得到20种优势微生物菌属,占所有菌属的87.00%。
在按照各样本优势菌属的相对丰度制成的堆叠图中(图 3),样本Dd1~Dd16的最主要优势菌属分别为Anaerococcus(37.51%)、Pseudomonas(46.20%)、Prevotella(59.27%)、Finegoldia(48.04%)、Prevotella(41.80%)、Streptococcus(39.14%)、Prevotella(28.39%)、Staphylococcus(94.82%)、Prevotella(46.72%)、Prevotella(61.03%)、Citrobacter(52.31%)、Fusobacterium(44.57%)、Porphyromonas(21.06%)、Prevotella(37.35%)、Streptococcus(48.46%)、Enterococcus(70.12%)。
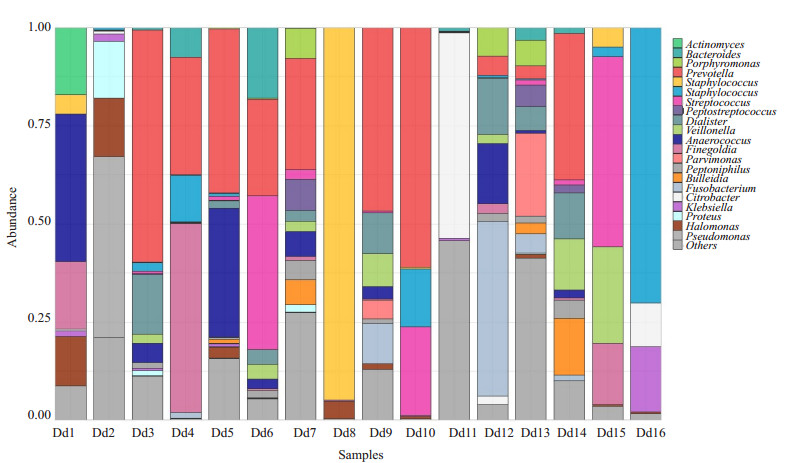
|
图 3 菌属相对丰度堆叠图 Figure 3 Stack of relative abundance dominant genera. |
在根据计算各水平物种间的距离进行各水平物种聚类分析,并进行colunm标准化所得的热图中(图 4),在属水平上,各样本的优势菌属整体上分布较为离散,但Anaerococcus、Veillonella、Dialister、Peptoniphilus、Streptococcus和Enterococcus等菌属在样本中的分布较为集中。
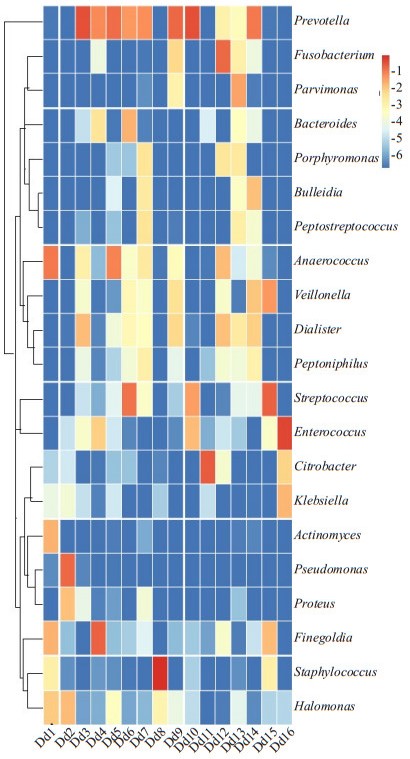
|
图 4 菌属相对丰度聚类热图 Figure 4 Heatmap of relative abundance dominant genera. |
根据所有样本优势菌属相对丰度制成的箱形图(图 5)可发现,优势菌属在各样本中的分布不均匀,呈偏态分布。Prevotella具有显著较高的相对丰度,占所有菌属的21.16%。
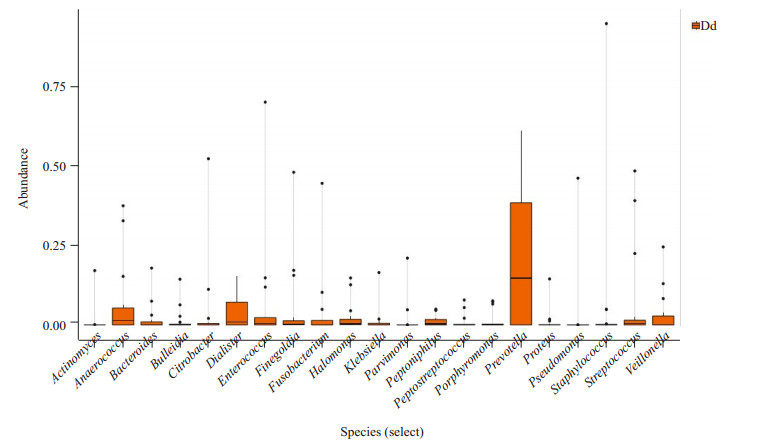
|
图 5 菌属相对丰度箱式图 Figure 5 Boxplot of relative abundance dominant genera. |
| 表 2 培养法与16s rRNA测序结果中DFO病原菌分布的特点 Table 2 Comparison of pathogen distribution in DFO bone tissues detected by routine culture and 16S rRNA sequencing |
16例标本中,培养法的阳性率仅为88.24%(14/ 16),而16s rRNA测序法的阳性率高达100%(16/16)。培养法和16s rRNA测序法在优势菌属种类数上的区别相差不大,分别为16种和20种,但后者平均每个样本优势菌属的种类数是前者的8倍以上(12.56种vs 1.50种)。两种微生物鉴定方法结果均表明革兰氏阴性菌所占比例高于革兰氏阳性菌,且16s rRNA测序结果中革兰氏阴性菌所占比例高于培养法(67.16% vs 50.00%),但差异无统计学意义(P > 0.05)。
培养结果中Staphylococcus aureus(12.50%)为最主要病原菌,其次为Enterococcus faecalis、Streptococcus acidominimus、Escherichia coli、Proteus mirabilis和Serratia marcescens(各8.33%);16s rRNA测序结果中Halomonas(7.96%)为最主要病原菌属,其次为Enterococcus、Finegoldia 和Prevotella(各6.97%)。两种微生物鉴定法中共同出现的病原菌属为Staphylococcus、Enterococcus、Streptococcus、Proteus、Pseudomonas、Klebsiella、Citrobacter,培养法结果中仅有Escherichia coli、Serratia marcescens及Enterobacter cloaca 3种病原菌未通过16s rRNA测序法检测出来,而高达13种病原菌仅出现在16s rRNA测序法结果中,且其中的Anaerococcus、Veillonella、Bacteroides、Fusobacterium、Porphyromonas、Finegoldia、Prevotella、Peptostreptococcus、Parvimonas、Peptoniphilus和Bulleidia均为专性厌氧菌或严格厌氧菌。仅培养法检测出真菌(占4.17%),为Candida albicans。培养法中多重耐药菌(MDR)占58.33%。
3 讨论糖尿病足骨髓炎是糖尿病致残率及致死率均较高的并发症,近年研究表明有效的抗生素治疗能提高骨髓炎的治愈率、降低外科治疗风险,是降低截趾/肢率和死亡率的重要治疗手段[4-5, 15]。了解临床上DFO创面病原生物菌群的多样性及分布特点,对于指导抗生素的使用有着重要的意义。本研究显示16s rRNA高通量测序可展示DFO骨组织中病原微生物的多样性及分布特点。Alpha多样性分析表明本次测序的深度和广度基本覆盖样本的所有物种,各样本细菌OTU分类丰度较高,且分布较为均匀。但优势菌属在各样本中的分布并不均匀,呈偏态分布,各样本间的优势菌属种类及数量差异均较大。
本研究无论是在16s rRNA测序还是培养法结果中,DFO感染骨组织病原菌中革兰氏阴性菌所占比例均高于革兰氏阳性菌。在一些欠发达且气候显著温暖地区的研究中,DFO病原菌中革兰氏阴性菌的比例较高[16]。此前也有研究揭示,糖尿病足创面主要病原菌会因Wagner级别的不同而有所差异:随着Wagner级别的升高,G-菌所占的比例逐渐增大[17-18]。黄莺等[19]进一步回顾性研究发现,糖尿病足感染一旦合并骨髓炎,其病原菌将从以革兰氏阳性菌为主转变为以革兰氏阴性菌为主。有趣的是,本研究中16s rRNA测序G-菌属的比例高于培养法,且Anaerococcus、Veillonella、Bacteroides、Fusobacterium、Porphyromonas、Finegoldia、Prevotella、Peptostreptococcus、Parvimonas、Peptoniphilus和Bulleidia等专性厌氧菌或严格厌氧菌仅出现于16s rRNA测序结果中,而培养结果中并无厌氧菌的出现,表明16s rRNA测序对于G-菌属及厌氧菌属的鉴定较培养法有显著的优势。
研究发现16s rRNA测序对细菌鉴定的敏感性是微生物培养的2倍,而且可分离出某些少见或含量较少、难以或不能培养的菌种[20]。本研究中,16s rRNA测序结果平均每个样本病原菌种类数是培养法的8倍以上,除Escherichia coli、Serratia marcescens及Enterobacter cloacae外,16s rRNA测序结果覆盖了培养结果的所有病原菌,且Halomonas、Peptostreptococcus、Parvimonas、Finegoldia、Prevotella、Anaerococcus、Dialister、Veillonella、Bacteroides、Fusobacterium、Porphyromonas、Peptoniphilus和Bulleidia等菌属仅出现在16s rRNA测序结果中,进一步证实了16s rRNA测序在病原微生物鉴定灵敏度方面的优越性,与Choi等[20]的结果一致。此外,16s rRNA测序的阳性率高于培养法(100% vs 88.24%)。需要指出的是,16s rRNA测序目前并不能及时鉴定病原菌的抗生素敏感性,而培养法结果显示58.33%的病原菌为多重耐药菌。有研究证实,长期抗生素使用史是多重耐药菌出现的危险因素[21],本研究中约高达68.75%的患者入院前有抗感染治疗史,而有研究认为多重耐药菌的出现是导致抗感染治疗失败的重要因素[22]。
因此,对于DFO病原菌的鉴定,应常规结合一般细菌及厌氧菌培养,甚至其他它更为复杂的培养方法,因而取样要求严格,鉴定程序繁琐;而16s rRNA测序仅通过一次测序即可完成鉴定,方法简单,不易漏诊,在对革兰氏阴性菌及厌氧菌的鉴定方面较培养法有显著的优势。由于16s rRNA测序在对真菌及病原菌药物敏感性的鉴定方面仍存在不足,因而应结合药敏试验才能更准确地指导临床抗生素的选择。临床上DFO感染的控制应该在病原微生物及其药物敏感性的准确鉴定结果指导下进行,必要时首选以抗革兰氏阴性菌为主的抗生素作为经验性用药,并联合抗厌氧菌药物。
本研究并未进行抗生素洗脱,与临床活检条件完全一致,且16s rRNA测序灵敏度高,不会因为抗生素的使用而出现假阴性结果,因而本研究的微生物分析结果更能反映临床DFO创面病原微生物的特征。16s rRNA测序结果发现,DFO病原微生物的多样性较大,各样本间物种的种类及分布差异较大,Prevotella、Streptococcus、Staphylococcus、Enterococcus、Anaerococcus等菌属成为丰度最大的菌属,占所有菌属的49.09%。与van Asten等[11]的结果不同的是,本研究16s rRNA测序中Staphylococcus并非最优势菌属,但Dowd等[23]认为导致DFO发生的可能不是某一个细菌,而是某一群细菌的共同作用。越来越多的研究认为,由多种细菌共同形成的生物膜及其代谢产物是导致骨髓炎发生的重要原因[24-25]。本研究中68.75%的患者入院前有长期抗生素使用史,因而可能是由于抗生素的“挑选作用”使某些耐药的条件致病菌成为优势菌,但并不一定是致病的关键菌。此外,糖尿病患者本身的免疫力下降,组织微环境也发生了改变[26-27],因而更有利于其他条件致病菌的定植。
尽管16s rRNA测序法较传统培养法有显著优势,但仍然存在一系列亟待解决的问题。目前16s rRNA测序法对病原菌的鉴定只能精确到属水平,对种水平的鉴定仍然不够完善;此外,尽管16s rRNA高通量测序速度极快,但对产生的大量数据的分析和充分利用需要专业的数据分析团队。但是,16s rRNA高通量测序技术仍然是一项有前景的微生物分析方法,不仅可指导抗感染治疗,还可进一步研究微生物物种间的相互关系和致病作用机制,开创新的控制感染方法[28]。
本研究中的病原微生物大多是条件致病菌,这些条件致病菌在骨髓炎的发生及发展过程中究竟扮演什么样的角色?此外,检测结果中某些丰度较大的微生物是否是真正导致骨髓炎难以清除的罪魁祸首?这些问题需待后期进一步实验进行探讨。
| [1] | Lipsky BA, Aragón-Sánchez J, Diggle M, et al. IWGDF guidance on the diagnosis and management of foot infections in persons with diabetes[J]. Diabetes Metab Res Rev, 2016, 32(Suppl 1): 45-74. |
| [2] | Lavery LA, Peters EJ, Armstrong DG, et al. Risk factors for developing osteomyelitis in patients with diabetic foot wounds[J]. Diabetes Res Clin Pract, 2009, 83(3): 347-52. DOI: 10.1016/j.diabres.2008.11.030. |
| [3] | Mutluoglu M, Sivrioglu AK, Eroglu M, et al. The implications of the presence of osteomyelitis on outcomes of infected diabetic foot wounds[J]. Scand J Infect Dis, 2013, 45(7): 497-503. DOI: 10.3109/00365548.2013.765589. |
| [4] | Lazaro-Martinez JL, Aragon-Sanchez J. Garcia-Morales E.antibiotics versus conservative surgery for treating diabetic foot osteomyelitis: a randomized comparative trial[J]. Diabetes Care, 2014, 37(3): 789-95. DOI: 10.2337/dc13-1526. |
| [5] | Jordano-Montanez Q, Muniz-Tatay M, Viade-Julia J, et al. Diabetic foot osteomyelitis: Is conservative treatment possible?[J]. Enferm Infecc Microbiol Clin, 2014, 32(9): 555-9. DOI: 10.1016/j.eimc.2014.03.012. |
| [6] | Streit WR, Schmitz RA. Metagenomics--the key to the uncultured microbes[J]. Curr Opin Microbiol, 2004, 7(5): 492-8. DOI: 10.1016/j.mib.2004.08.002. |
| [7] | Amann RI, Ludwig W, Schleifer KH. Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J]. Microbiol Rev, 1995, 59(1): 143-69. |
| [8] | Clarridge JE. Impact of 16S rRNA gene sequence analysis for identification of bacteria on clinical microbiology and infectious diseases[J]. Clin Microbiol Rev, 2004, 17(4): 840. DOI: 10.1128/CMR.17.4.840-862.2004. |
| [9] | 张军毅, 朱冰川, 徐超, 等. 基于分子标记的宏基因组16S rRNA基因高变区选择策略[J]. 应用生态学报, 2015, 26(11): 3545-53. |
| [10] | Liolios K, Chen I-, Mavromatis K, et al. The genomes on line database (Gold) in 2009: status of genomic and metagenomic projects and their associated metadata[J]. Nucleic Acids Res, 2010, 38(1): D346-54. |
| [11] | van Asten SA, La Fontaine J, Peters EJ, et al. The microbiome of diabetic foot osteomyelitis[J]. Eur J Clin Microbiol Infect Dis, 2016, 35(2): 293-8. DOI: 10.1007/s10096-015-2544-1. |
| [12] | 中华医学会糖尿病学分会. 中国2型糖尿病防治指南(2013年版)[J]. 中国医学前沿杂志:电子版, 2015, 30(3): 26-89. |
| [13] | Conte MS, Pomposelli FB, Clair DG, et al. Society for vascular surgery practice guidelines for atherosclerotic occlusive disease of the lower extremities: management of asymptomatic disease and claudication[J]. J Vasc Surg, 2015, 61(3, S): 2S-41S. DOI: 10.1016/j.jvs.2014.12.009. |
| [14] | Wagner FW. The dysvascular foot: a system for diagnosis and treatment[J]. Foot Ankle, 1981, 2(2): 64-122. DOI: 10.1177/107110078100200202. |
| [15] | Acharya S, Soliman M, Egun A, et al. Conservative management of diabetic foot osteomyelitis[J]. Diabetes Res Clin Pract, 2013, 101(3): e18-20. DOI: 10.1016/j.diabres.2013.06.010. |
| [16] | Aragon-Sanchez J, Lipsky BA, Lazaro-Martinez JL. Gram-Negative diabetic foot osteomyelitis: risk factors and clinical presentation[J]. Int J Low Extrem Wounds, 2013, 12(1): 63-8. DOI: 10.1177/1534734613477423. |
| [17] | 李永恒, 何利平, 王椿, 等. 糖尿病足合并感染患者532株病原菌分布及耐药性分析[J]. 中华糖尿病杂志, 2011, 3(4): 296-300. |
| [18] | 黄莺, 曹瑛, 邹梦晨, 等. 糖尿病足感染病原菌特点及耐药性15年变迁[J]. 中华糖尿病杂志, 2016(2): 87-92. |
| [19] | Huang Y, Cao Y, Zou M, et al. Distribution of pathogens in diabetic foot osteomyelitis and risk factors of osteomyelitis[J]. Nan Fang Yi Ke Da Xue Xue Bao, 2015, 35(12): 1782-6. |
| [20] | Choi SH, Sung H, Kim SH, et al. Usefulness of a direct 16S rRNA gene PCR assay of percutaneous biopsies or aspirates for etiological diagnosis of vertebral osteomyelitis[J]. Diagn Microbiol Infect Dis, 2014, 78(1): 75-8. DOI: 10.1016/j.diagmicrobio.2013.10.007. |
| [21] | Ertugrul BM, Oncul O, Tulek N, et al. A prospective, multi-center study: factors related to the management of diabetic foot infections[J]. Eur J Clin Microbiol Infect Dis, 2012, 31(9): 2345-52. DOI: 10.1007/s10096-012-1574-1. |
| [22] | Vardakas KZ, Horianopoulou M, Falagas ME. Factors associated with treatment failure in patients with diabetic foot infections:An analysis of data from randomized controlled trials[J]. Diabetes Res Clin Pract, 2008, 80(3): 344-51. DOI: 10.1016/j.diabres.2008.01.009. |
| [23] | Dowd SE, Wolcott RD, Sun Y, et al. Polymicrobial Nature of chronic diabetic foot ulcer biofilm infections determined using bacterial tag encoded FLX amplicon pyrosequencing (bTEFAP)[J]. PLoS One, 2008, 3(10): e3326. DOI: 10.1371/journal.pone.0003326. |
| [24] | Junka A, Szymczyk P, Ziółkowski G, et al. Bad to the bone: on in vitro and Ex vivo microbial biofilm ability to directly destroy colonized bone surfaces without participation of host immunity or osteoclastogenesis[J]. PLoS One, 2017, 12(1): e169565. |
| [25] | Suleman L. Extracellular bacterial proteases in chronic wounds: A potential therapeutic target[J]. Adv Wound Care (New Rochelle), 2016, 5(10): 455-63. DOI: 10.1089/wound.2015.0673. |
| [26] | Lazaro JL, Izzo V, Meaume S, et al. Elevated levels of matrix metalloproteinases and chronic wound healing: an updated review of clinical evidence[J]. J Wound Care, 2016, 25(5): 277-87. DOI: 10.12968/jowc.2016.25.5.277. |
| [27] | Nielsen TB, Pantapalangkoor P, Yan J, et al. Diabetes exacerbates infection via hyperinflammation by signaling through TLR4 and RAGE[J]. MBio, 2017, 8(4): 45. |
| [28] | Franz P, Betat H, Morl M. Genotyping bacterial and fungal pathogens using sequence variation in the gene for the CCA-adding enzyme[J]. BMC Microbiol, 2016, 25(4): 47. |
 2017, Vol. 37
2017, Vol. 37

