2. 南方医科大学基础医学院医学遗传学教研室,广东 广州 510515
2. Department of Medical Genetics, School of Basic Medical Sciences, Southern Medical University, Guangzhou 510515, China
β-地中海贫血(简称“β-地贫”)是我国南方省区高发的严重致死致残单基因遗传病之一,据其临床表型可以分为重型、轻型和中间型3种主要类型[1-3]。其中中间型β-地贫的表型多样,轻者只有轻微地贫表征,可无明显临床症状;而重者需要不定时输血,出现肝脾肿大等明显症状。中间型β-地贫的分子基础比较复杂,其病理学机制是源于α-和β-类珠蛋白不平衡的程度。β-地贫杂合子合并α珠蛋白基因三联体或四联体,可加剧珠蛋白比例失衡,使患者由无临床症状转变为中间型地贫表型[4-6]。αααanti-3.7为α珠蛋白基因三联体,在人群中有一定的基因频率[7-9]。αααanti-3.7的检测,在中间型β-地贫的分子机制研究、临床诊断、治疗方案及预后中,具有重要的现实意义,但目前尚无稳定有效而操作简单的检测方法。本研究即采用微滴式数字PCR(ddPCR)技术,建立了一种快速检测α珠蛋白基因αααanti-3.7三联体的方法,报道如下。
1 材料和方法 1.1 研究对象 1.1.1 已知基因型标本从本实验室样本资源库中,选取基因型为αα/αα(正常基因型)gDNA标本20例,αα/ αααanti-3(.7 α珠蛋白基因三联体)gDNA标本8例。所有标本来自2015年~2016年在本实验室进行基因诊断的患者或孕妇,均已签署知情同意书,基因型均已采用Wen等报道的PCR方法[10-11]和α珠蛋白基因多重连接探针扩增技术(MLPA)[12-13]予以验证。
1.1.2 临床标本收集2016年7月~12月在韶关市妇幼保健院产前诊断中心进行地贫基因检测的外周血标本60例,乙二胺四乙酸(EDTA)抗凝,男、女各30例,年龄3~40岁,其中确诊为中间型β-地贫标本30例,未检出地贫基因携带的正常标本30例。所有受检者均已签署知情同意书。
1.1.3 仪器与试剂Bio-rad QX200微滴式数字PCR仪及配套Digital PCR Supermixes、Droplet DigitalTM PCR Oils等试剂耗材购自美国Bio-rad公司;引物及探针由上海英骏(Invitrogen)生物技术有限公司合成。
1.2 方法 1.2.1 基因组DNA(gDNA)提取及研究样本准备将临床收集的60例外周血标本用血液基因组DNA提取试剂盒(天根生化科技北京有限公司,货号DP348),按操作说明提取gDNA。将临床标本和已知基因型标本gDNA,用灭菌双蒸水,调其浓度至5~10 ng/µL备用。
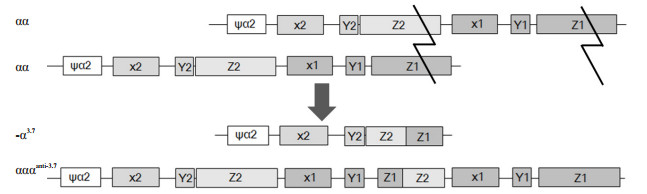
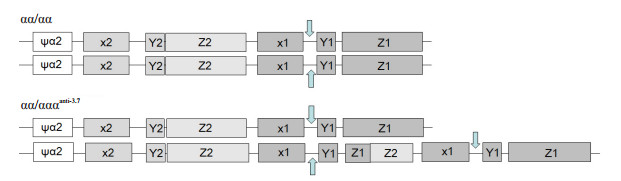
1.2.2 引物及探针设计α珠蛋白基因簇定位于16号染色体16p13.3,包含两个高度同源的α2和α1基因,α2基因X2、Y2、Z2盒分别与α1基因X1、Y1、Z1盒的DNA序列相同。如图 1所示αααanti-3.7形成和结构示意图,其分子机制是源于非等位交换,进而分别形成-α3.7和αααanti-3.7携带单倍型[14]。据图 2所示的基因拷贝数示意图,如果以α 1基因X1与Y1之间的差异序列(箭头所示位置)为定量靶标,基因型为αα/αα的正常个体为2拷贝,而基因型为αα/αααanti-3.7的个体为3拷贝。据此,本研究即以X1与Y1之间的单一特异性序列为定量检测靶序列,以7号染色体上的看家基因β-actin为参比序列,分别设计特异性PCR引物和TaqMan探针(表 1)。
|
图 1 -α3.7和αααanti-3.7形成及结构示意图 Figure 1 Diagram showing the formation and structure of -α3.7 and αααanti-3.7. |
|
图 2 αα/αα和αα/αααanti-3.7基因拷贝数示意图 Figure 2 Diagram showing -α3.7 and αααanti-3.7 gene copies. |
| 表 1 引物和探针序列 Table 1 Sequences of primers and probes |
以Bio-rad ddPCRTM Supermix for Probes(No dUTP)为基本体系,于同一反应管中加入对应目的基因(α1)和参比基因(β-actin)扩增引物及TaqMan探针、gDNA模板,于Bio-rad QX200 ddPCR系统,按操作说明进行扩增和检测分析,根据检测结果对体系组份进行优化调整,建立稳定有效的α1基因拷贝数ddPCR检测体系。
1.2.4 检测方法的灵敏度和准确性验证与评价应用本研究建立的方法,检测已备的28例已知基因型标本(αα/ αα标本20例,αα/αααanti-3.7标本8例)以评价此方法的准确性与灵敏度;检测已收集的60例临床样本,并将所有检出的阳性样本及10例随机阴性样本采用MLPA方法(SALSA MLPA probemix P140-B4 HBA试剂盒,荷兰MRC-Holland公司产品)进行验证,以评价此方法的应用性。
然后将所有88例gDNA样本(28例已知基因型样本和60例已检测验证临床样本),进行盲法编号后用本方法检测,以全面验证准确性与灵敏度。
2 结果 2.1 α1基因拷贝数微滴式数字PCR(ddPCR)检测体系建立的检测体系总体积20.0 μL,包含2×ddPCRTM Supermix for Probes(No dUTP)10.0 μL、10 pmol/μL α1和β-actin正反向引物及TaqMan探针各0.5 μL、5~ 10 ng/μL gDNA 1.0 μL、灭菌双蒸水4.0 μL。配制好的PCR扩增体系,经混匀,瞬时离心后,于Bio-rad ddPCR系统配套QX200 Droplet Generator,按操作说明制备微滴;然后转移微滴至96孔PCR板,封膜后于Bio-rad C1000 Touch PCR仪进行扩增(PCR反应条件为95 ℃预变性6 min,94 ℃ 30 sec+60 ℃ 30sec 30个循环)。扩增完成后,将PCR反应板转移至Bio-rad ddPCR系统配套QX200 Droplet Reader,按仪器和软件操作说明,进行微滴计数及荧光信号检测。
2.2 检测数据及结果分析仪器配套软件分别显示具有FAM和HEX荧光信号微滴数(即目的序列拷贝数)的绝对值及分布直方图,FAM标记的为2拷贝的参比基因,HEX记的为待检的目的基因,如果HEX通道的荧光微滴数与FAM通道的荧光微滴数之比≈1,待检目的基因的拷贝数为2;如果≈ 1.5,则待检目的基因的拷贝数为3。
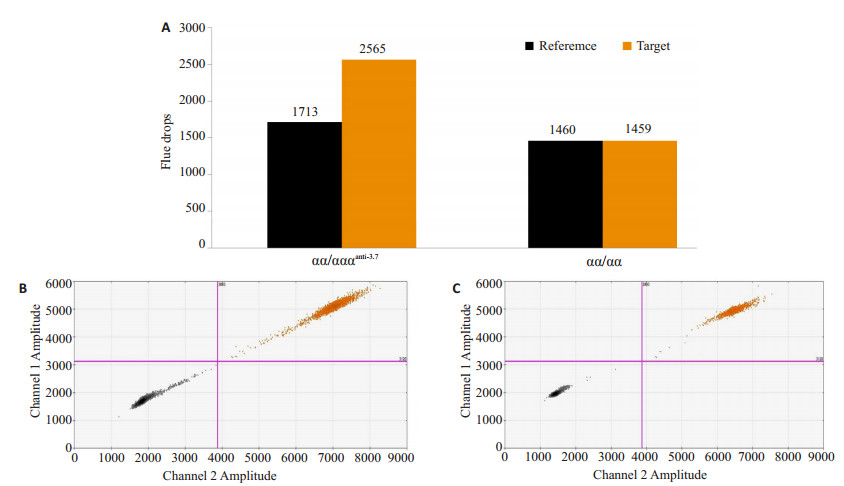
2.3 28例已知基因型样本检测结果经检测分析,20例为αα/αα的正常基因型,8例为αα/ αααanti-3.7的α珠蛋白基因三联体,各样本的基因型检测结果与已知基因型结果相符。基因型分别为αα/αα和αα/αααanti-3.7的ddPCR检测结果如图 3所示:已知参比基因相对拷贝数为2,图中右侧所检样本检测结果显示,参比基因目的序列拷贝数为1460,α1基因目的序列拷贝数为1459,比例≈1,即说明α1基因的相对拷贝数也为2,此样本的基因型为αα/αα;而图中左侧所检样本的参比基因与α1基因目的序列拷贝数为1713: 2565≈1.5,即说明α1基因的相对拷贝数为3,此样本的基因型为αα/αααanti-3.7。
|
图 3 αα/αααanti-3.7和αα/αα ddPCR检测结果图 Figure 3 Results of αα/αααanti-3.7 and αα/αα detection using the ddPCR-based method. A: Comparison between the fluorescence drops of the reference gene and the target gene; B and C: Fluorescence drop distribution histograms of the two genotype samples, respectively. |
将收集的60例临床样本,采用本研究建立的方法进行检测,30例中间型β-地贫标本中,检出3例为αα/ αααanti-3.7;30例正常样本中,检出2例αα/αααanti-3.7。5例阳性样本及随机选取的10例阴性样本,经MLPA方法验证,两种方法的检测结果相符。
2.5 盲法编号样本检测灵敏度与准确性评价采用本方法检测88例盲法编号样本的基因型结果均与靶基因型相符,准确性与灵敏度均达100%(表 2)。
| 表 2 88例盲法样本的检测结果统计分析表 Table 2 Detection results of 88 blind samples using the ddPCR-based method |
地中海贫血的病理学机制是α-和β-类珠蛋白不平衡,其临床症状的轻重与珠蛋白比例失衡程度直接相关[15-18]。研究已经表明,β-地贫杂合子和并α珠蛋白基因多拷贝是中间型β-地贫的重要分子基础之一[9]。分子流行病学研究报道-α3.7缺失突变在广东和广西人群中的携带率分别高达3.10%和4.78%,仅次于--SEA突变类型[19-21]。如图 1所示配子生成过程中α珠蛋白基因的非等位交叉产生了-α3.7缺失和αααanti-3.7三拷贝两种等位基因,由此可推断与-α3.7缺失对应的αααanti-3.7在我国南方人群中有一定的携带率,只是由于检测方法等多方面的局限,目前尚未获得相应的数据资料。
αααanti-3.7的检测方法,最早是采用PCR扩增给合酶切电泳[22]。2003年所报道的方法,则根据非等位交叉而形成融合基因的序列特征(于图 1所示形成的Z1/Z2融合基因),于两端设计特异性引物进行PCR扩增、凝胶电泳目的产物分析进行检测[10-11],此方法在一定程度上简化了操作,但由于α珠蛋白基因DNA序列高GC、高同源,特异性引物只能选择Y1/Z1和Z2/X1之间的差异序列(图 1),所以此检测体系为高GC目的序列长片段扩增,扩增时间长且易出现扩增失败导致假阴性结果。随着MLPA技术的应用,近年来多采用此方法进行α珠蛋白基因多拷贝检测,此方法结果准确,但需经DNA杂交过夜、PCR扩增、毛细管电泳等步骤,操作繁琐、设备要求高,不适合大规模常规检测[23-24]。
据图 1,2所示的αααanti-3.7结构基础,采用合适的技术策略对α1基因拷贝数进行准确定量,即可实现αααanti-3.7的有效检测。目前普遍采用的Real-Time定量PCR技术,能准确分析的拷贝数比例为1:2,而不适应于拷贝数比例为2:3的αα/αααanti-3.7检测[25]。微滴式数字PCR(ddPCR)是Bio-rad公司近年推出的专利技术,在传统的PCR扩增前对样品进行微滴化处理,即将含有核酸分子的反应体系分成成千上万个纳升级的微滴,其中每个微滴不含或含一至数个待检靶分子,扩增后对微滴进行逐个检测,根据泊松分布原理及荧光信号的有无,即可得出靶分子的初始拷贝数或浓度,实现原始模板的绝对定量[26-27]。本方法的技术原理就是在α1基因的特异性单一序列区域,选取非高GC短片段( < 90 bp)扩增子,采用ddPCR技术对α1基因进行拷贝数定量。实验结果显示,本研究建立的方法,能对靶基因DNA目的序列进行绝对定量,参比基因与目的基因比例直接反映α1基因拷贝数。应用此方法对28例已知基因型样本的检测结果均与靶基因型结果相符,60例临床样本的检测结果也与验证结果一致,灵敏度与准确性均达100%。与目前采用的MLPA技术相比较,本方法在PCR扩增完成后直接将反应板置于QX200 Droplet Reader仪器平台进行自动化检测与分析,2~3 h即可获得检测结果,无其它中间环节而操作简单,且结果准确、检测成本低,适合大规模常规检测。
总言之,本研究建立的方法,为当前地贫的临床诊断及相关基础研究提供了一种α珠蛋白基因αααanti-3.7快速检测新体系,是数字PCR技术在此领域的应用,也为诸如αααanti-4.2、SMN等基因拷贝数相关单基因遗传病的ddPCR检测方法学研究提供参考和借鉴。
| [1] | Barrett AN, Saminathan R, Choolani M. Thalassaemia screening and confirmation of carriers in parents[J]. Best Pract Res Clin Obstet Gynaecol, 2017, 39: 27-40. DOI: 10.1016/j.bpobgyn.2016.10.015. |
| [2] | Rund D. Thalassemia 2016: modern medicine battles an ancient disease[J]. Am J Hematol, 2016, 91(1): 15-21. DOI: 10.1002/ajh.24231. |
| [3] | Bonifazi F, Conte R, Baiardi P, et al. Pattern of complications and burden of disease in patients affected by beta thalassemia major[J]. Curr Med Res Opin, 2017, 7: 1-9. |
| [4] | Brancaleoni V, Di Pierro E, Motta I, et al. Laboratory diagnosis of thalassemia[J]. Int J Lab Hematol, 2016, 38(Suppl 1): 32-40. |
| [5] | YusCebrian F, Recasens Flores Mdel V, Izquierdoálvarez S, et al. Combination of a triple alpha-globin gene with beta-thalassemia in a gypsy family: importance of the genetic testing in the diagnosis and search for a donor for bone marrow transplantation for one of their children[J]. BMC Res Notes, 2016, 9: 220. DOI: 10.1186/s13104-016-2027-1. |
| [6] | Ben Salah N, Bou-Fakhredin R, Mellouli F, et al. Revisiting beta thalassemia intermedia: past, present, and future prospects[J]. Hematology, 2017, 7: 1-10. |
| [7] | Xie XM, Wu MY, Li DZ. Evidence of selection for the α-Globin gene deletions and triplications in a southern Chinese population[J]. Hemoglobin, 2015, 39(6): 442-4. DOI: 10.3109/03630269.2015.1072551. |
| [8] | Farashi S, Bayat N, FaramarziGarous N, et al. Interaction of an α-Globin gene triplication with β-Globin gene mutations in iranian patients with β-Thalassemia intermedia[J]. Hemoglobin, 2015, 39(3): 201-6. DOI: 10.3109/03630269.2015.1027914. |
| [9] | Chen W, Zhang X, Shang X, et al. The molecular basis of beta-thalassemia intermedia in southern China: genotypic heterogeneity and phenotypic diversity[J]. BMC Med Genet, 2010, 11: 31. |
| [10] | Kimura EM, Grignoli CR, Pinheiro VR, et al. Thalassemia intermedia as a resultof heterozygosis for β0-thalassemiaand αααanti-3.7/ ααgenotype in aBrazilian patient[J]. Braz J Med Biol Res, 2003, 36(6): 699-701. DOI: 10.1590/S0100-879X2003000600003. |
| [11] | Wang W, Ma ES, Chan AY, et al. Single-tube multiplex-PCR screen for anti-3.7 and anti-4.2 alpha-globin gene triplications[J]. ClinChem, 2003, 49(10): 1679-82. |
| [12] | 郝颖, 徐晓昕, 徐志勇, 等. 多重连接依赖性探针扩增技术在α地中海贫血产前诊断中的应用[J]. 中华医学遗传学杂志, 2015, 5: 683-6. DOI: 10.3760/cma.j.issn.1003-9406.2015.05.016. |
| [13] | 陈亚军, 杨学煌, 曾宪琪, 等. 多重连接依赖式探针扩增技术在α地中海贫血基因诊断与产前诊断中的应用[J]. 中华血液学杂志, 2013, 34(7): 591-4. DOI: 10.3760/cma.j.issn.0253-2727.2013.07.007. |
| [14] | De Mare A, Groeneger AH, Schuurman S, et al. A rapid single-tube multiplex polymerase chain reaction assay for the seven most prevalent alpha-thalassemia deletions and alphaalphaalpha(anti 3.7) alpha-globin gene triplication[J]. Hemoglobin, 2010, 34(2): 184-90. DOI: 10.3109/03630261003670259. |
| [15] | Giordano PC, Bakker-Verwij M. Frequency ofalpha-globingenetriplicationsandtheirinteraction with beta-thalassemia mutations[J]. Hemoglobin, 2009, 33(2): 124-31. DOI: 10.1080/03630260902827684. |
| [16] | Helmi N, Bashir M, Shireen A, et al. Thalassemia review: features, dental considerations and management[J]. Electron Physician, 2017, 9(3): 4003-8. DOI: 10.19082/4003. |
| [17] | Origa R. β-Thalassemia[J]. Genet Med, 2017, 19(6): 609-19. DOI: 10.1038/gim.2016.173. |
| [18] | Constan?o MC, Rocha P, Bento C, et al. Heterozygous β thalassemia with triplication of the α globin gene[J]. Acta Med Port, 2012, 24(4): 633-6. |
| [19] | Xu XM, Zhou YQ, Luo GX, et al. The prevalence and spectrum of alpha and beta thalassaemia in Guangdong Province: implications for the future health burden and population screening[J]. J ClinPathol, 2004, 57(5): 517-22. |
| [20] | Xiong F, Sun M, Zhang X, et al. Molecular epidemiological survey of haemoglobinopathies in the Guangxi Zhuang Autonomous Region of southern China[J]. Clin Genet, 2010, 78(2): 139-48. DOI: 10.1111/cge.2010.78.issue-2. |
| [21] | Wu MY, Li J, Li SC, et al. Frequencies of HKαα and anti-HKαα alleles in Chinese carriers of silent deletionalα-Thalassemia[J]. Hemoglobin, 2015, 39(6): 407-11. DOI: 10.3109/03630269.2015.1071268. |
| [22] | Dodé C, Krishnamoorthy R, Lamb J, et al. Rapid analysis of alpha3.7thalassaemiaandalpha-alpha-alpha-anti3.7triplicationbyenxy maticamplificationanalysis[J]. Br J Haematol, 1993, 83(1): 105-11. DOI: 10.1111/j.1365-2141.1993.tb04639.x. |
| [23] | Yuregir OO, Ayaz A, Yalcintepe S, et al. Detection of α-Thalassemia by using multiplex Ligation-Dependent probe amplification as an additional method for rare mutations in southern Turkey[J]. Indian J Hematol Blood Transfus, 2016, 32(4): 454-9. DOI: 10.1007/s12288-015-0617-z. |
| [24] | Belvini D, Salviato R, Radossi P, et al. Multiplex ligation-dependent probe amplification as first mutation screening for large deletions and duplications in haemophilia[J]. Haemophilia, 2017, 23(2): e124-32. DOI: 10.1111/hae.13143. |
| [25] | Zhou W, Wang G, Zhao X, et al. A multiplex qPCR gene dosage assay for rapid genotyping and large-scale population screening for deletionalα-thalassemia[J]. J Mol Diagn, 2013, 15(5): 642-51. DOI: 10.1016/j.jmoldx.2013.05.007. |
| [26] | Taly V, Huggett J. Digital PCR, a technique for the future[J]. Biomol Detect Quantif, 2016, 10: 1. DOI: 10.1016/j.bdq.2016.10.003. |
| [27] | Perkins G, Lu H, Garlan F, et al. Droplet-Based digital PCR: application in cancer research[J]. Adv Clin Chem, 2017, 79: 43-91. DOI: 10.1016/bs.acc.2016.10.001. |
 2017, Vol. 9
2017, Vol. 9

