2. 南方医科大学 放疗科, 广东 广州 510515;
3. 南方医科大学 基础医学院病理学系, 广东 广州 510515
2. Department of Radiotherapy, Nanfang Hospital, Southern Medical University, Guangzhou 510515, China;
3. Department of Pathology, School of Basic Medical Sciences, Southern Medical University, Guangzhou 510515, China
结直肠癌目前是全世界癌症发病率居第3位的恶性肿瘤,在我国其发病率列恶性肿瘤的第2位,而且近年来其发病率呈逐年上升及年轻化趋势[1]。由于疾病的早期诊断及治疗方案的不断改进,结直肠癌患者的病死率在逐年下降,而发病率仍然居高不下。目前针对于人表皮细胞生长因子受体(EGFR)及血管内皮细胞生长因子受体(VEGFR)的靶向药物已在临床应用中取得了可喜的成绩,然而其疗效仍具有局限性,而且约40%结直肠癌患者表现为K-RAS突变型,这部分患者对EGFR抑制剂具有天然耐药性[2]。据文献报导,有20%~30%的结直肠癌患者存在基因遗传突变及表观遗传修饰,较常见的有P53突变、APC高甲基化、DNA错配修复基因突变等[3]。因此阐明结直肠癌发生发展的相关分子机制及新的治疗靶点是当前结直肠癌研究的重要内容。最近美国肿瘤基因组图谱研究网络计划的研究人员通过大规模基因组测序研究方法分析结直肠癌的分子遗传学特征,获得一系列结直肠癌相关基因,除了人们所熟知的APC、BRAF及K-ras等结直肠癌相关基因外,还发现一些功能未知的结直肠癌相关基因,其中MIER3以其在结直肠癌中异常的高突变率引起我们的关注[4]。
MIER3基因位于5号染色体(5q11.2),编码的蛋白质相对分子质量为61 000,属于MIER基因家族。该家族目前被人们所知的只有三个成员(MIER1、MIER2及MIER3),在进化上相对保守,普遍存在于从果蝇到人类的各种生物体中,目前对于这一家族成员的相关研究尚不多见,具体功能尚不清楚。值得注意的是Pitule课题小组最近利用real-time PCR方法研究发现,与正常结直肠粘膜组织相比,MIER3在结直肠癌组织中mRNA的表达显著下调,但对其具体功能未进一步探讨[5]。因此,MIER3在结直肠癌中的异常高突变率以及表达失调,均提示MIER3与结直肠癌密切相关,但目前尚缺乏对MIER3的进一步研究,其在结直肠癌发生发展中的作用及相关机制尚有待进一步阐明。在本研究中我们初步检测比较MIER3蛋白在结直肠癌及正常肠粘膜组织中的表达,验证MIER3与结直肠癌的相关性,通过克隆构建MIER3基因真核表达载体,检测过表达MIER3对结直肠癌细胞株生物学特性的影响,并应用生物信息学方法对MIER3及其相互作用蛋白进行了初步预测分析,为深入研究MIER3在结直肠癌发生发展中的作用及其相关分子机制奠定基础。
1 材料和方法 1.1 材料胎牛血清和RMPI 1640培养基(Life, 美国);Trizol试剂、RT-PCR试剂盒、PrimeSTAR DNA高保真酶、限制性内切酶、标准相对分子质量DNA Marker、T4连接酶、质粒小量提取试剂盒、DNA凝胶回收试剂盒、DNA片段纯化试剂盒(Takara, 日本);CCK8试剂(东仁化学科技(上海)有限公司)、Transwell小室(BD公司);过表达MIER3转染质粒(上海吉凯公司);SW620、DLD1人大肠癌细胞和DH5α菌株为本实验室保存。
1.2 方法 1.2.1MIER3在结肠癌及相应癌旁正常组织中mRNA和蛋白表达水平的表达。
1.2.1.1 组织RNA及总蛋白提取收集手术切除的结肠癌及相应癌旁粘膜组织标本,在液氮冷冻下充分研磨,加入Trizol试剂或蛋白裂解液,按常规方法提取组织标本RNA及总蛋白,并进行相应浓度测定。
1.2.1.2 应用Real-time PCR方法检测MIER3 mRNA在结肠癌组织中表达应用Mx3000P定量PCR仪(Strata-gene)进行Real-time PCR检测,反应条件为:95 ℃ 20 s,57 ℃ 30 s,72 ℃ 34 s,共40个循环。MIER3扩增引物为5'-ACACACACAAGGGACAATGAACA GG-3'和5'-TTCTTCCGTCCATGCAGTCATTCCT-3';对照β-actin扩增引物为:5'-ACTCGTCATACTCCTG CT-3'和5'-GAAACTACCTTCAACTCC-3'。MIER3相对表达量为2-△△CT,ΔΔCt=C(t MIER3)-Ct(β-actin);将正常组织(N)与癌组织(T)中的相对表达量相比较(N/T),以评价MIER3基因在癌组织中表达下调倍数。实验重复3次,取平均值。
1.2.1.3 应用Western blot方法检测MIER3蛋白在结肠癌组织中的表达30 μg蛋白8% SDS-PAGE凝胶电泳分离,湿转法将蛋白转印至PVDF膜后,5%脱脂奶粉封闭1 h,鼠抗人MIER3多克隆抗体4 ℃孵育过夜;1× TBS-T漂洗15 min×3次,辣根过氧化物酶标记的二抗室温孵育1 h,1×TBS-T漂洗3次,ECL发光液显色,X胶片曝光,胶片常规显影、定影。
1.2.2 真核载体的构建 1.2.2.1 引物设计与合成根据GenBank上MIER3基因序列(GeneBank Accession Number: NM_001297598),依据PCR引物设计原则,利用primer5.0软件设计可以特异性扩增MIER3-cDNA全长的引物序列,同时分别在其上游引物和下游引物的5'端引入Kpn1和EcoR1两酶切位点,引物由英潍捷基(上海)贸易有限公司合成,序列如下:
F:5'GGTACCATGGCGGAGGCTTCTTTTGG3'。
R:5'GAATTCTCACTCAGAGTGTAGGGCCG3'。
1.2.2.2 RT-PCR选取适量生长状态良好的培养大肠癌细胞SW620,加入1 mL Trizol试剂提取总RNA,以3 μg总RNA为模板,按照逆转录试剂盒说明书进行逆转得到cDNA,以cDNA产物为模板,加入上下游引物,PCR扩增约1662 bp的片段,总反应体系为50 μL,循环参数为:94 ℃ 2 min;94 ℃ 20 s,56 ℃ 20 s,72 ℃ 2 min,30个循环,72 ℃延伸5 min。取5 μL PCR产物用于1%琼脂糖凝胶电泳分析。
1.2.2.3 酶切取1 μg pcDNA3质粒和0.2 μg PCR产物,加入1.5 μL Kpn Ⅰ和1.5 μL EcoR Ⅰ双酶切,37 ℃ 50 min,1%琼脂糖电泳,胶回收载体大片段和目的片段。
1.2.2.4 连接在T4连接酶的作用下将PCR产物及质粒以10:1的比例连接,16 ℃过夜。
1.2.2.5 转化无菌状态下取100 μL DH5α感受态细胞至1.5 mL离心管中,加入上述20 μL连接体系,轻轻混匀放置在冰上30 min,42 ℃热休克45 s,快速置于冰上冷却2~3 min,每管加入500 μL无抗性LB,置于细菌摇床中37 ℃ 160 r/min培养60 min,然后瞬时离心,去上清。取100 μL的菌液均匀涂布于含氨苄青霉素抗性的琼脂板上,晾干后倒置,于恒温箱中37 ℃培养14 h。
1.2.2.6 阳性克隆挑取及鉴定用含Amp+的LB平板筛选阳性克隆,小量抽提质粒,经Kpn Ⅰ和EcoR Ⅰ双酶切鉴定并进行测序分析。
1.2.3 MIER3对结肠癌细胞增殖及迁移能力的影响 1.2.3.1 瞬时转染转染前1 d将细胞以2×106孔接种到六孔板上,转染当天当细胞的融合度达80%时吸去旧培养基,PBS洗两次,每孔加入2 mL新鲜培基。配置AB混合液:A液为4 μg质粒+250 μL opti-MEM,轻柔混匀,B液为10 μL lip2000+250 μL opti-MEM,轻柔混匀后静置5 min,将AB两液轻柔混合后静置20 min,然后将AB混合液加入到六孔板中,置入37 ℃,5% CO2培养箱中培养,6 h后更换新鲜培养基。
1.2.3.2 CCK8细胞增殖实验(1)将转染过表达载体、转染NC及野生型的DLD1细胞分别接种于96孔板,每孔加入细胞悬被100 μL,细胞数为5000/孔,每组设6个复孔。置37 ℃,5% CO2孵箱培养24 h;(2)分别在细胞贴壁后的0、24、48、72、96及120 h,取出细胞培养板,加入CCK8溶液10 μL/孔,置37 ℃,5% CO2孵箱继续培养2 h;(3)取出培养板,于Quant分光光度计中测定波长为450 nm的各孔吸光度值(A450)。实验进行3次独立重复实验。
1.2.3.3 细胞侵袭实验转染24 h后,用无血清的培养基饥饿细胞6~8 h,消化的细胞用无血清培养基洗2遍,以每孔5万个细胞量接种于Costar24孔Transwell上室,在下室中加入500 μL含10%胎牛血清的培养基,37 ℃,5% CO2,培养24 h。24 h后,用4%多聚甲醛固定transwell小室5 min,用0.1%的结晶紫染色5 min,用棉棒擦去小室内底部表面的细胞。每个培养孔随机选择5个视野用正置显微镜(×200倍)拍照,计数,计算每组小室细胞的平均数。实验进行3次独立重复实验。
1.2.4 MIER3及其相互作用蛋白的生物信息学分析(1)运用NCBI在线数据库,对MIER3进行Blast分析,搜索其直系和旁系同源蛋白,对不同种属包括人的MIER1家族成员进行进化树分析。
(2)应用模式生物果蝇相互作用蛋白数据库DIP (http://dip.doe-mbi.ucla.edu/),分析果蝇MIER相互作用网络,寻找果蝇中MIER相互作用蛋白。
(3)应用在线数据库寻找果蝇中MIER相互作用蛋白的人类的同源蛋白(http://www.ensembl.org/index.html),对人MIER3的相互作用蛋白进行初步分析预测。
(4)通过NCBI的UniGene数据库,对MIER3及预测的相互作用蛋白在各个组织进行表达分析。
1.3 统计学分析所有数据采用SPSS19.0统计软件包进行测算统计处理,RT-PCR实验数据以均数±标准差表示,两样本均数的比较采用采用单因素方差分析(one-way ANOVA)或两独立样本的t检验(Independent-Sample T Test),取P < 0.05为差异具有统计学意义。
2 结果 2.1 Real-time PCR检测MIER3在结肠癌组织中mRNA的表达提取8对结肠癌及相应正常粘膜组织总RNA,逆转录为cDNA后进行相应Real-time PCR检测,结果显示MIER3在8例结肠癌组织中的相对表达量明显低于癌旁组织的2倍以上(图 1A)。
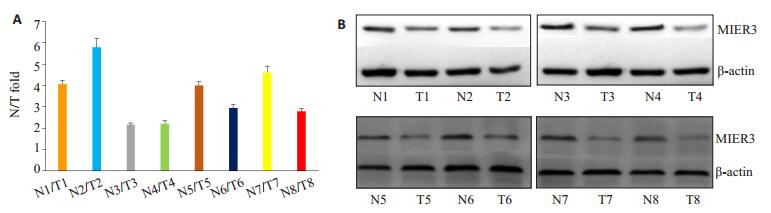
|
图 1 Real-time PCR检测MIER3在结肠癌及癌旁组织中mRNA的表达及Western blot检测MIER3蛋白在结肠癌组织中的表达 Figure 1 Expression of MIER3 in colorectal carcinoma detected by real-time PCR (A) and Western blotting (B). T: Tumor; N: Normal. |
提取8例结肠癌及相应癌旁正常粘膜组织蛋白,Western blot检测MIER3蛋白表达,结果显示MIER3在8例结肠癌组织中的蛋白表达明显低于相应正常癌旁组织(图 1B)。
2.3 MIER3基因cDNA的克隆及真核表达载体构建PCR成功扩增出人MIER3 cDNA全长,PCR产物电泳结果可见大小为1662 bp左右的特异性条带(图 2A),将电泳回收片段插入pcDNA3真核表达载体中,形成pcDNA3-MIER3真核表达载体。Kpn Ⅰ和EcoR Ⅰ双酶切鉴定得到约1662 bp左右的片段(图 2B),测序结果与GeneBank里的MIER3基因序列相一致。

|
图 2 pcDNA3-MIER3的真核表达载体 Figure 2 Full-length human MIER3 cDNA (A) and restriction endonuclease analysis of the recombinant plasmid pcDNA3-MIER3 (B). |
MIER3在DLD1稳定转染MIER3过表达细胞株中高表达(图 3A,P < 0.05)。利用CCK8实验检测发现MIER3过表达的DLD1细胞株增殖能力明显下降(图 3B,P < 0.05)。同时利用Transwell实验发现在MIER3过表达的DLD1细胞株侵袭能力明显降低(图 3C,P < 0.05)。
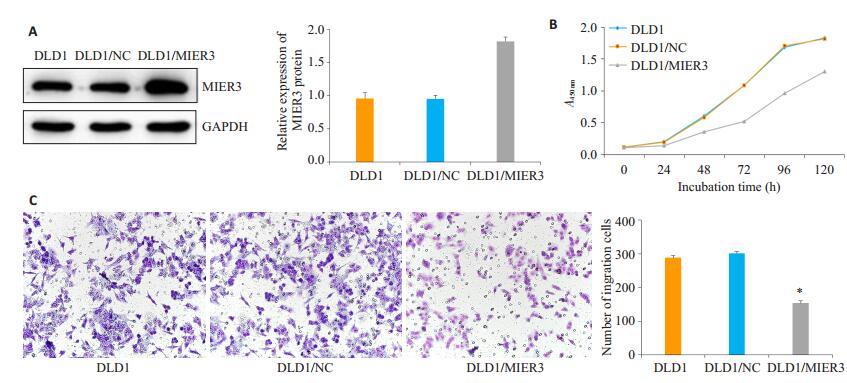
|
图 3 MIER3可抑制结肠癌细胞株的增殖和迁移能力 Figure 3 MIER3 over-expression inhibits colorectal carcinoma cell migration and proliferation in vitro. A: Transfection efficiency of MIER3 vector in DLD1 cells; B: CCK8 assay showing lowered proliferation ability of MIER3-overexpressing cells; C: Transwell assay showing lowered migration ability of MIER3-overexpressing cells. *P < 0.05 vs DLD1. |
利用NCBI在线数据库我们寻找到包括小鼠、大鼠、黑猩猩、果蝇、非洲爪蟾、酵母、斑马鱼、线虫以及人等不同种属的MIER蛋白的直系及旁系同源蛋白,对不同种属的MIER家族成员进行进化树分析,结果显示人MIER3在果蝇中的同源蛋白为CG1620(图 4A),实际上在果蝇中MIER只有一个成员即CG1620,这提示MIER3在进化上的相对保守性。
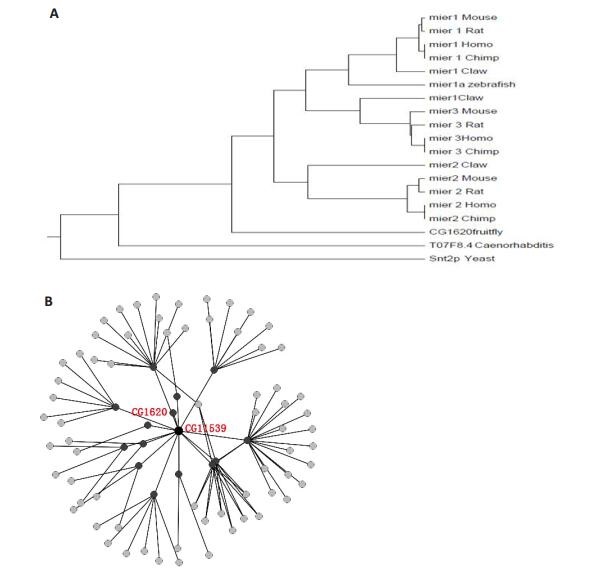
|
图 4 不同种属MIER1家族进化树分析及果蝇CG1620相互作用蛋白网络 Figure 4 Phylogenetic analysis of members in MIER1 family (A) and the CG1620-interacting protein network in Drosophila melanogaster (B). |
CG1620相互作用蛋白的确定应用在线的模式生物果蝇相互作用蛋白数据库DIP,分析果蝇CG1620相互作用蛋白的网络,发现CG1620的相互作用蛋白为CG11539,而CG11539是连接CBP、msl-1、CG4463、CG13277等多个蛋白的重要桥梁(图 4B),CG1620与CG11539的相关性在功能上提示其可能承担着重要角色。
2.5.3 人类MIER3相互作用蛋白的预测根据确定的模式生物果蝇中CG1620的相互作用蛋白CG11539的序列,利用在线数据库,寻找到其在人类的同源蛋白为NAT9(N-acetyltransferase 9),同源蛋白之间具有较高的同源性,而且具有较为一致的作用域。
2.5.4 数字Northern杂交分析MIER3及预测的相互作用蛋白的表达通过查询NCBI的UniGene数据库,我们对MIER3及预测的相关相互作用蛋白在正常及相应肿瘤组织中的表达进行数字Northern杂交分析(表 1),结果显示NAT9与MIER3及MIER1、MIER2的在正常组织中的基因表达谱非常接近,而在正常与相应肿瘤组织中的表达比较显示,MIER3在肺、胰腺及睾丸等肿瘤中的表达显著上调;而在结直肠及卵巢等肿瘤中的表达明显下调,显示出一定的组织特异性(表 2)。
| 表 1 MIER3相关蛋白在正常组织中虚拟Northern杂交表达情况 Table 1 Expression tendency of proteins related with MIER3 in normal tissues by virtual Northern hybridization |
| 表 2 MIER3在部分正常组织及肿瘤中虚拟Northern杂交表达情况 Table 2 Expression tendency of MIER3 in normal tissues and carcinoma tissues by virtual Northern hybridization |
MIER3是MIER家族的第3个成员,目前对于这一家族,人们已对MIER1有初步认识。研究显示MIER1蛋白在结构上因其N端所含有的酸性结构域以及C端的ELM2、SANT等结构域,可通过募集并活化组蛋白去乙酰化酶[7-8],或与转录激活因子Sp1相互作用,干扰Sp1与下游位点的结合以及结合组蛋白乙酰化酶及CBP等多种途径在相关基因的转录调控以及肿瘤的发生发展中承担着重要角色[9-11]。MIER3与MIER1具有明显的同源性,两者有54%相同氨基酸序列,在结构上MIER3蛋白C端同样具有ELM2、SANT两个重要的功能结构域。这一进化上的高度保守提示MIER3在功能上可能类同于MIER1,也可能在组蛋白乙酰化修饰、基因转录调控中发挥重要作用[12-15],其在肿瘤中可能承担的重要作用也得到人们的日益关注。在乳腺癌中的研究发现,MIER3是乳腺癌的易感基因,其在乳腺癌中表达明显高于正常乳腺组织,提示MIER3与乳腺癌的发生发展密切相关[5];而在结直肠癌中的初步研究显示,MIER3在结直肠癌细胞中明显低表达,可能与肿瘤的分化程度及预后密切相关[16]。这些不同研究结果提示MIER3在不同肿瘤中功能及相关作用机制具有的特异性与复杂性,MIER3在结直肠癌等肿瘤中的确切功能还需要更多实验去证实。
我们通过对MIER3在结直肠癌组织中的表达进行初步检测,证实MIER3在结直肠癌组织中出现异常的表达下调,提示MIER3与结直肠癌发生发展密切相关。同时,我们通过克隆构建真核表达载体,利用体外过表达功能实验检测发现MIER3表达上调后可明显抑制结直癌细胞株的增殖及侵袭能力。这些发现提示MIER3在结直肠癌中可能具有抑癌基因的特点,MIER3的表达失调可能在结直肠癌发生演进中承担着重要生物学功能。进一步,我们应用基于保守的蛋白质相互作用的生物信息学预测方法,通过查询模式生物黑腹果蝇的蛋白数据库[17],初步确定NAT9为MIER3的候选相互作用蛋白。NAT9是N-乙酰基转移酶超家族(NAT)成员之一,NAT家族是人体内重要的Ⅱ相代谢酶,在人体对芳香胺类致癌物质的活化及灭活以及某些药物的代谢过程中起着重要作用,其活性存在遗传多态性[18-19],并与肿瘤的易感性密切相关。如研究显示NAT家族成员NAT2快速乙酰化表型是结直肠癌患病的高发因素[21],而其慢速乙酰化表型则可能是膀胱癌和结直肠癌的易感因素[21-22]。而另一家族成员NAT10被证实可通过EMT相关信号通路促进肝癌细胞的侵袭转移,而其在结肠癌细胞中出现的亚细胞重定位,常提示不良预后[23-24]。目前关于NAT9的相关研究尚不多见,初步的研究显示NAT9在乳腺癌、卵巢癌、子宫内膜癌等多种肿瘤中出现异常高表达[25],但具体功能尚不明确。NAT9在结直肠癌中的功能,与MIER3可能的直接相互作用尚有待于进一步实验证实。
综合上述实验结果与分析,我们推测MIER3与结直肠癌发生发展密切相关,MIER3的表达失调和功能异常在结直肠癌的发生演进中可能承担着重要角色,MIER3在结直肠癌中的具体功能及相关机制值得进一步深入研究。
| [1] | Chen W, Zheng R, Baade PD, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2): 115-32. DOI: 10.3322/caac.21338. |
| [2] | Gong J, Cho M, Fakih M. RAS and BRAF in metastatic colorectal cancer management[J]. J Gastrointest Oncol, 2016, 7(5): 687-704. DOI: 10.21037/jgo. |
| [3] | Hahn MM, De Voer RM, Hoogerbrugge N, et al. The genetic heterogeneity of colorectal cancer predisposition-guidelines for gene discovery[J]. Cell Oncol, 2016, 39(6): 491-510. DOI: 10.1007/s13402-016-0284-6. |
| [4] | Muzny DM, Bainbridge MN, Chang K, et al. Comprehensive molecular characterization of human colon and rectal cancer[J]. Nature, 2012, 487(747): 330-7. |
| [5] | Pitule P, Vycital O, Bruha J, et al. Differential expression and prognostic role of selected genes in colorectal cancer patients[J]. Anticancer Res, 2013, 33(11): 4855-65. |
| [6] | Roos DS. Computational biology. Bioinformatics--trying to swim in a sea of data[J]. Science,, 2001, 291(557): 1260-1. |
| [7] | Matthews LR, Vaglio P, Reboul J, et al. Identification of potential interaction networks using sequence-based searches for conserved protein-protein interactions or "interologs"[J]. Genome Res, 2001, 11(12): 2120-6. DOI: 10.1101/gr.205301. |
| [8] | Ding ZH, Gillespie LL, Paterno GD. Human MI-ER1 alpha and beta function as transcriptional repressors by recruitment of histone deacetylase 1 to their conserved ELM2 domain[J]. Mol Cell Biol, 2003, 23(1): 250-8. DOI: 10.1128/MCB.23.1.250-258.2003. |
| [9] | Millard CJ, Fairall L, Schwabe JW. Towards an understanding of the structure and function of MTA1[J]. Cancer Metastasis Rev, 2014, 33. |
| [10] | Ding Z, Gillespie LL, Mercer FC, et al. The SANT domain of human MI-ER1 interacts with Sp1 to interfere with GC box recognition and repress transcription from its own promoter[J]. JBiol Chem, 2004, 279(27): 28009-16. DOI: 10.1074/jbc.M403793200. |
| [11] | Lakisic G, Lebreton A, Pourpre R, et al. Role of the BAHD1 Chromatin-Repressive complex in placental development and regulation of steroid metabolism[J]. PLoS Genet, 2016, 12(3): e1005898. DOI: 10.1371/journal.pgen.1005898. |
| [12] | Li S, Paterno GD, Gillespie LL. Insulin and IGF-1, but not 17beta-estradiol, alter the subcellular localization of MIER1alpha in MCF7 breast carcinoma cells[J]. BMC Res Notes, 2015, 8: 356. DOI: 10.1186/s13104-015-1336-0. |
| [13] | Mccarthy PL, Mercer FC, Savicky MW, et al. Changes in subcellular localisation of MI-ER1 alpha, a novel oestrogen receptor-alpha interacting protein, is associated with breast cancer progression[J]. Br J Cancer, 2008, 99(4): 639-46. DOI: 10.1038/sj.bjc.6604518. |
| [14] | Derwish R, Paterno GD, Gillespie LL. Differential HDAC1 and 2 recruitment by members of the MIER family[J]. PLoS One, 2017, 12(1): e169338. |
| [15] | Dendekker AD, Xu X, Vaughn MD, et al. Rat Mcs1b is concordant to the Genome-Wide Association-Identified breast cancer risk locus at human 5q11.2 and MIER3 is a candidate cancer susceptibility gene[J]. Cancer Res, 2012, 72(22): 6002-12. DOI: 10.1158/0008-5472.CAN-12-0748. |
| [16] | Giot L, Bader JS, Brouwer C, et al. A protein interaction map of Drosophila melanogaster[J]. Science, 2003, 302(5651): 1727-36. DOI: 10.1126/science.1090289. |
| [17] | Jiugang Z, Jing L, Characterization YL, et al. Polymorphism and association of porcine NAT9 gene[J]. Mol Biol Rep, 2012, 39(3): 3137-42. DOI: 10.1007/s11033-011-1079-5. |
| [18] | Kubiak X, Duval R, Pluvinage B, et al. Xenobiotic-metabolizing enzymes in Bacillus anthracis: molecular and functional analysis of a truncated arylamine N-acetyltransferase isozyme[J]. Br J Pharmacol, 2017, 174(14): 2174-82. DOI: 10.1111/bph.v174.14. |
| [19] | Hein DW. Molecular genetics and function of NAT1 and NAT2: role in aromatic amine metabolism and carcinogenesis[J]. Mutat Res, 2002, 506(SI): 65-77. |
| [20] | Xu WJ, Wen LP, Jiang XX, et al. Association between Nacetyltransferase 2 polymorphism and bladder cancer risk: a meta-analysis in a single ethnic group[J]. Clin Lab, 2017, 63(2): 287-93. |
| [21] | Procopciuc LM, Osian G, Iancu M. N-acetyl transferase 2/ environmental factors and their association as a modulating risk factor for sporadic colon and rectal cancer[J]. 2016 Nov 24. doi: 10.1002/jcla.22098.[Epub ahead of print]. https://www.ncbi.nlm.nih.gov/pubmed/?term=Iancu%20M%5Bauth%5D |
| [22] | Marzuillo P, Di Sessa A, Umano GR, et al. Novel association between the nonsynonymous A803G polymorphism of the Nacetyltransferase 2 gene and impaired glucose homeostasis in obese children and adolescents[J]. Pediatr Diabetes, 2016 Aug 2. doi: 10.1111/pedi.12417.[Epub ahead of print]. |
| [23] | Ma R, Chen J, Jiang SJ, et al. Up regulation of NAT10 promotes metastasis of hepatocellular carcinoma cells through epithelial-tomesenchymal transition[J]. Am J Transl Res, 2016, 8(10): 4215-23. |
| [24] | Zhang H, Hou W, Wang HL, et al. GSK-3 beta-regulated Nacetyltransferase 10 is involved in colorectal cancer invasion[J]. Clin Cancer Res, 2014, 20(17): 4717-29. DOI: 10.1158/1078-0432.CCR-13-3477. |
| [25] | Helms C, Cao L, Krueger JG, et al. A putative RUNX1 binding site variant between SLC9A3R1 and NAT9 is associated with susceptibility to psoriasis[J]. Nat Genet, 2003, 35(4): 349-56. DOI: 10.1038/ng1268. |
 2017, Vol. 37
2017, Vol. 37



