2. 军事医学科学院基础医学研究所,北京 100850;
3. 解放军总后勤部第一门诊部口腔中心,北京 100842
2. Institute of Basic Medical Sciences, Academy of Military Medical Sciences, Beijing 100850, China;
3. Center of Stomatology, First Out-patient Clinics, Department of General Logistics of PLA, Beijing 100842, China
变异链球菌(SM)是人类口腔中的首要致龋菌,可承受持续的宿主防御反应并适应口腔内环境的变化。变异链球菌在动态的口腔唾液环境中可获得最优的生长,分解代谢多种糖类,尤其是适应酸环境的能力较强[1-3]。针对变异链球菌中不同致龋性菌株间致龋能力的分子差异的研究及在耐酸适应性反应中的表达调控是研究变异链球菌致龋机制的一个热点内容,包括蛋白质及调控RNA尤其是小分子RNA等的研究[4-8]。
本课题组通过前期实验已经分离、鉴定出高致龋性变异链球菌临床分离株17和低致龋性变异链球菌临床分离株5 [9],并通过SELEX技术成功获得特异识别高致龋性变异链球菌临床分离株的ssDNA适配体H19 [10],采用pull-down技术钓取的差异蛋白SDS-PAGE胶银染后经质谱测序的结果表明,GroEL是该适配体的候选靶标,提示了GroEL在致龋方面的关键作用。文献报道[11-14]酸性环境下变异链球菌通过上调分子伴侣GroEL的表达来完成其适应机制。但该蛋白参与致龋过程的分子机制及其调控机制并未得到清晰的诠释。
细菌的非编码小RNA通常被称为sRNA是一类长度在50~500个核苷酸,不能编码蛋白质的RNA,能通过与靶基因互补配对,调节mRNA的翻译和转录等途径调控靶基因的表达及蛋白质的稳定性15]。我们课题组通过提取细菌总RNA及高通量测序的方法分析获得一组在高低致龋变异链球菌中具有表达差异的小分子RNA(未发表数据)。在分析已经标注过的差异表达sRNAs过程中,生物信息学分析发现sRNA SpR19可能靶向GroEL的mRNA及上下游调控区。因此,对该SpR19及GroEL的表达水平的详尽研究具有重要意义,不仅可以探讨这一对分子联合应用作为高低致龋变异链球菌的标志物,还可以揭示GroEL在酸性环境下的sRNA调控机制。
1 材料和方法 1.1 实验菌株和实验动物已获得的高致龋性变异链球菌临床分离株17,低致龋性临床分离株5 [9]50%甘油条件,-70 ℃冰箱,军事医学科院保存;新西兰大白兔,2.5~3 kg雌性。同时选用两只进行免疫。动物来自军事医学科学院实验动物中心。
1.2 细菌纯化培养将冻存的变异链球菌的高致龋性临床分离株17和低致龋性临床分离株5在添加了1%杆菌肽和亚碲酸钾的含有20%无菌蔗糖溶液的MSB轻唾液链球菌培养基中,37 ℃厌氧环境(80% N2、10% H2、10% CO2),培养24 h。挑取单克隆菌株,分别接种于以HCl调节的pH5.5及pH7的5 mL BHI培养液中,扩大培养,依据变异链球菌生长规律,于37 ℃厌氧袋(三菱,MGC)内培养至对数生长期,紫外分光光度计下计量A600浓度[9]。
1.3 生物信息学分析应用NCBI中的BLAST软件(http://blast.ncbi.nlm.nih.gov/Blast.cgi)分别将GroEL位于1 833 204~1 834 832的全部1629 bp序列并取其上游下游各1000 bp序列(含GroEL的共伴侣分子GroES区段)与sRNA SpR19序列使用GUUGle软件进行序列比对,分别得到连续匹配的种子序列数据,取其中位点匹配范围从11~13 bp的具有高匹配率的数据[16]。
1.4 细菌RNA的提取分别取培养的变异链球菌菌液1 mL,离心收集细菌,用0.1% DEPC水洗涤1次。-70 ℃冰箱中反复冻融3次,50 µL DEPC H2O重悬,加50 µL溶菌酶液50 g/L溶菌酶的TE缓冲液(DEPC水配制,pH=8.0),37 ℃水浴30 min,加20% SDS沸水浴1 min,于冰上冷却并加入1 mL Trizol(Sigma)依产品RNA提取说明书操作,乙醇洗涤、干燥后用42 µL DEPC H2O溶解,分光光度计测定A260/280计算RNA浓度及纯度。RNase-Free DNase(Progema公司的M6101)依产品说明处理上述RNA样品。处理后进行酚氯仿抽提、乙醇沉淀,DEPC水溶解RNA,A260/280定量,-70 ℃保存。
1.5 qRT-PCR检测取1 µg所提总RNA进行一步法加尾及反转录反应:M-MLV逆转录酶0.5 µL,100 mmol/L ATP 2.5 µL,Poly(A)聚合酶,10×PAP buffer 2.5 µL,MnCl2 2.5 µL,RNase抑制剂0.5 µL,加尾法RNA通用反转引物RTPrimer(参表 1,500 ng/µL)1 µL,DEPC水补至21 µL,37 ℃水浴90 min。95 ℃处理5 min灭活酶,放-20 ℃保存。反转录产物进行荧光定量PCR:95 ℃ 10 min;95 ℃20 s,56 ℃ 30 s,72 ℃ 32 s,扩增40个循环,95 ℃ 20 s,56 ℃ 1 min,95 ℃ 32 s,采集熔解曲线。选用16sRNA为内参,sRNA SpR19引物见表 1。GroEL mRNA水平的荧光定量PCR检测基本同上,提取细菌总RNA后随机引物反转录,荧光定量PCR反应条件如下:95 ℃ 3 min;95 ℃ 15 s,57 ℃ 20 s,72 ℃ 30 s,扩增45个循环;95 ℃ 15 s,57 ℃ 1 min,95 ℃ 30 s,采集熔解曲线。GroEL引物序列见表 1,内参引物仍为16sRNA。将4次实验结果采用two way-ANOVA对数据标准差和均值进行统计学分析,计算P值。
| 表 1 引物序列 Table 1 Primer sequences |
根据GroEL蛋白序列设计两段抗原多肽:NVTAGANPIGIR;N端肽:MAKDIKFSADAR,合成多肽,以合成多肽为抗原,免疫兔子,免疫前采血,免疫前每只家兔耳缘静脉采集1 mL正常全血。以背部皮下多点注射(10点)的方式完成第1次免疫,抗原的免疫剂量为400 g/只,注射体积为2 mL/只(即每点200 µL)间隔两周同样的方式完成第2次免疫。采用ELISA间接法检测1周后血清效价并完成第3次免疫,大量采集血清(第3次免疫3~5 d后,效价大于50 000),完成血清的后处理和保存、效价检测,纯化后分装,获得抗体[17-18]。
1.7 细菌蛋白的提取及Western bolt实验验证GroEL表达水平HCl调节配制pH5.5及pH7的BHI培养基,以1:50分别将低致龋性临床分离株4,高致龋性临床分离株17接种于10 mL BHI中,37 ℃厌氧袋(三菱,MGC)中培养至A600=0.7,5000 r/min离心5 min,弃培养液,0.5×PBS漂洗1次,得白色菌体沉淀,加入350 µL RIPA裂解液(普利莱,北京)及0.35 µL 10 mg/mL溶菌酶溶液(TIANGEN)及0.35 µL蛋白酶抑制剂(Progema),低速下摇床冰浴30 min,超声裂解10次,超5 s,停10 s。4 ℃ 12 000 r/min离心15 min。取上清并进行蛋白定量。等量匀浆上样,进行0.75 mm厚度的12% SDS PAGE。电泳结束后,余胶作考马斯亮蓝R250染色,鉴定电泳效果。电转仪恒流200 mA将蛋白转移到硝酸纤维素膜(NC膜)1 h。丽春红染色,NC膜经5%牛奶封闭液封闭,漂洗后,1:5000孵育一抗(兔源多克隆GroEl抗体),洗膜3次后1:8000孵育羊抗兔Igg(CWBio)。应用化学发光成像仪(Clinx Scirnce Instruments Co., Ltd)显示。
2 结果 2.1 sRNA SpR19潜在靶标GroEL mRNA序列分析groEL的全长1629 bp,取其上下游各1000 bp调控序列区段(含GroES区段),起始密码子为AUG,终止密码子UAA。取高致龋菌株17及低致龋菌株5经过高通量测序得到的差异sRNA SpR19序列,经过生物信息学分析,表 2罗列出了预测得到的可能性最大的7条SpR19与groEL靶向的结合位点。
| 表 2 sRNA SpR19潜在靶向groEL的种子区序列与靶向位点的生物信息学预测结果 Table 2 Seed sequences of sRNA SpR19 and their corresponding potential target sites in groEL mRNA and inter-genic regions predicted by bioinformatics analysis |
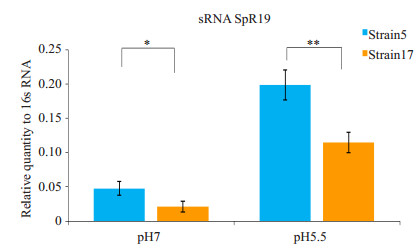
分别比较高致龋性变异链球菌临床分离株17和低致龋性变异链球菌临床分离株5在pH5.5和pH7情况下sRNA SpR19的表达差异,发现无论是正常培养条件,还是模拟致龋关键因素的酸性培养条件,相较于低致龋菌,高致龋菌中sRNA SpR19表达下降(P < 0.05,图 1)。
|
图 1 不同pH培养条件下高低致龋性变异链球菌中sRNA SpR19表达水平 Figure 1 SpR19 expression level in Streptococcus mutans strains with different cariogenicities under different pH condition. *P < 0.05; **P < 0.05. |
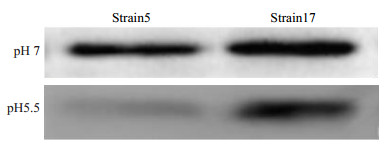
因为没有商品化变异链球菌内参蛋白抗体,准确定量后,取相同量蛋白电泳并转膜,丽春红染色后进行抗体免疫印记。结果显示,GroEL在不同致龋能力的临床分离株中存在明显的表达差异,且在与致龋密切相关的酸性培养条件(pH5.5)下高致龋性变异链球菌中GroEL表达呈明显优势(P < 0.05,图 2)。
|
图 2 不同pH培养条件下高低致龋变异链球菌中GroEL蛋白表达水平 Figure 2 GroEL protein expression level of different cariogenic Streptococcus mutans strains under different pH culture condition. |
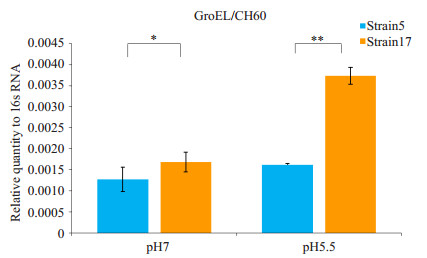
提取不同培养条件下的高、低致龋变异链球菌的细菌总RNA,随机引物反转录后荧光定量PCR检测目的基因的mRNA表达水平变化。结果显示,无论是正常(pH7)培养条件下还是在与致龋密切相关的酸性培养条件(pH5.5)下,高致龋性变异链球菌中GroEL mRNA均高表达(P < 0.05),与蛋白表达趋势一致(图 3)。
|
图 3 不同pH培养条件下高低致龋性变异链球菌中GroEL mRNA表达水平 Figure 3 GroEL mRNA expression in Streptococcus mutans strains with different cariogenicities under different pH condition detected by qRT-PCR. *P < 0.05, **P < 0.05 |
酸性环境可以直接导致牙釉质脱钙并诱发龋齿。变异链球菌的致龋性主要取决于其耐酸性。目前大部分研究关注在酸性环境下变异链球菌的生存机理。研究已证实多个与耐酸性相关的热休克蛋白家族基因htrA,dnaK,groEL,起到关键作用[6-7]。这些耐酸基因的调控机制,尤其是蛋白-蛋白以及sRNAs在转录后的基因表达过程中起到的调控作用也逐步得以解析[8, 19]。Liu等[8]首次通过构建酸性条件下变异链球菌(18~50 nt)sRNAs文库、高通量测序分析及生物信息学分析得到srn884837和srn133480及其靶向的5种匹配的基因,证实其对靶基因的调控影响变异链球菌的耐酸特性。但对于变异链球菌致病相关的关键蛋白及其酸性环境中的调控机制尚需要更多的数据。本研究从致龋力不同的变异链球菌的sRNA高通量测序比较分析数据入手,针对变异链球菌耐酸相关的蛋白GroEL,利用生物信息学手段,筛选出一条可能靶向GroEL的sRNA SpR19。本研究进一步检测了上述分子的表达水平及其在酸性条件下的表达变化,为获得区分变异链球菌致龋能力的分子标志物及致龋相关分子的sRNA调控机制提供数据支持。
高龋患者与无龋健康变异链球菌临床分离株蛋白表达谱提示二者在60 000左右蛋白表达量上存在差异[20];GroEL又称CH60或60 000伴侣分子,由含有高度保守性的groES和groEL两个相距111 bp的基因构成,由groE操纵子表达,是属于热休克蛋白(HSP)60家族的一种可溶性蛋白,相对分子质量为60 000左右,它可捕捉并重折叠50 000~60 000非自身底物蛋白,防止其与其它非自身蛋白相互聚集、辅助新合成的及变性的蛋白折叠、组装、转运及降解,从而增强其酸适应性[7, 21]。本研究通过制备GroEL的抗体和免疫印迹,进一步验证了GroEL在高致龋性变异链球菌中的高表达,与上述报道一致。另外,高致龋变异链球菌临床分离株中GroEL蛋白在酸性培养条件下无明显下降,而在低致龋变异链球菌中降低明显,也支持了GroEL蛋白在耐酸与致龋方面的重要作用。
但GroEL蛋白的调控机制,尤其是与致龋相关的sRNA调控机制并未见报道。我们进一步通过高、低致龋菌株的差异sRNA高通量测序数据,结合生物信息学手段,筛选出一条可能靶向GroEL的sRNA SpR19。细菌sRNA根据作用机制的不同主要可以分为3类:第1类sRNA称为核糖体开关,常位于mRNA前导序列5'-UTR区,随着结构改变调节下游基因表达[22];第2类sRNA主要通过与靶核苷酸碱基特异性配对或与伴侣蛋白结合来发挥调节作用,如大肠杆菌中发现的大部分sRNA都与伴侣分子Hfq结合[23];第3类为成簇规律性间隔的重复短回文序列CRISPRs,是基因组上的短回文序列串联排列重复规律间隔,目前认为可以在DNA的复制环节干扰噬菌体或质粒[24]。
细菌中最为普遍存在的是第2类sRNA,也是研究最广泛的一类。在细菌中以trans-sRNA作用为主[23],通常由基因间隔区转录产生,在基因组中存在多个结合靶点。处于基因间隔区的sRNA SpR19可能是上述第2类调控机制的sRNA。生物信息学分析发现在GroEL的基因编码区及上下游调控区(基因间区)均存在多组高度匹配的SpR19的种子区。随后的数据证实,二者在酸性环境下的表达在高低致龋菌中存在负相关。由于缺乏针对变异链球菌有效的转染技术手段,本研究没有进一步的SpR19直接靶向GroEL的实验。下一步,我们将利用体外实验相关技术手段,包括构建荧光报告系统,提供SpR19直接靶向GroEL的实验数据支持。另外,我们的结果证实,在模拟致龋的耐酸环境下,GroEL与sRNA SpR19在高低致龋变异链球菌临床分离株中的明显表达差异,提示这一对分子可以作为分子标志物用于区分变异链球菌致龋能力。本研究目前正在搜集临床变异链球菌分离株,生化分析其高低致龋性,并与该一对潜在靶向的分子标志物鉴定结果比较,从而为临床变异链球菌致龋能力的鉴定提供更加便捷、细致的分子诊断手段。综上,本文从致龋高低不同菌株入手,结合高通量测序与耐酸关键蛋白,筛选到了与致龋、耐酸相关的一对具有潜在调控作用的分子标志物。
| [1] | Smith EG, Spatafora GA. Gene regulation in S. mutans: complex control in a complex environment[J]. J Dent Res, 2012, 91(2): 133-41. DOI: 10.1177/0022034511415415. |
| [2] | Liu CC, Niu YL, Zhou XE, et al. Streptococcus mutans copes with heat stress by multiple transcriptional regulons modulating virulence and energy metabolism[J]. Sci Rep, 2015, 5: 12929. DOI: 10.1038/srep12929. |
| [3] | Matsui R, Cvitkovitch D. Acid tolerance mechanisms utilized by Streptococcus mutans[J]. Future Microbiol, 2010, 5(3): 403-17. DOI: 10.2217/fmb.09.129. |
| [4] | Jiang S, Chen S, Zhang C, et al. Effect of the biofilm age and starvation on acid tolerance of biofilm formed by Streptococcus mutans isolated from Caries-active and caries-free adults[J]. Int J Mol Sci, 2017, 18(4): pii: E713. DOI: 10.3390/ijms18040713. |
| [5] | Xia L, Xia W, Li SH, et al. Identification and expression of small Non-coding RNA, L10-Leader, in different growth phases of Streptococcus mutans[J]. Nucleic Acid Ther, 2012, 22(3): 177-86. |
| [6] | Moye ZD, Zeng L, Burne RA. Fueling the caries process: carbohydrate metabolism and gene regulation by Streptococcus mutans[J]. J Oral Microbiol, 2014, Sep 5: 6. DOI: 10.3402/jom.v6.24878.eCollection2014. |
| [7] | 王一舟, 张雅琪, 牛雪微, 等. 变异链球菌groE操纵子及其表达与调控[J]. 国际口腔医学杂志, 2016, 43(3): 348-51. |
| [8] | Liu S, Tao Y, Yu L, et al. Analysis of small RNAs in Streptococcus mutans under acid Stress-A new insight for caries research[J]. Int J Mol Sci, 2016, 17: 1529. DOI: 10.3390/ijms17091529. |
| [9] | 王成龙, 刘佼佼, 苏东华, 等. 高致龋性变异链球菌临床分离株的初步筛选[J]. 华西口腔医学杂志, 2013, 31(2): 136-40. DOI: 10.7518/hxkq.2013.02.006. |
| [10] | 王成龙, 胡丹阳, 刘佼佼, 等. 不同致龋性变形链球菌临床分离株差异ssDNA配基的筛选和鉴定[J]. 南方医科大学学报, 2013, 33(5): 738-41. |
| [11] | Wilkins JC, Homer KA, Beighton D. Analysis of Streptococcus mutans proteins modulated by culture under acidic conditions[J]. Appl Environ Microbiol, 2002, 68(5): 2382-90. DOI: 10.1128/AEM.68.5.2382-2390.2002. |
| [12] | Klein MI, Jin X, Lu B, et al. Streptococcus mutans Protein synthesis during mixed-species biofilm development by high-throughput quantitative proteomics[J]. PLoS One, 2012, 7(9): e45795. DOI: 10.1371/journal.pone.0045795. |
| [13] | Bolean M, Paulino TD, Thedei J. Photodynamic therapy with rose Bengal induces GroEL expression in Streptococcus mutans[J]. Photomed Laser Surg, 2010, 28(1): S79-84. |
| [14] | Klein MI, Xiao J, Lu BW, et al. Streptococcus mutans protein synthesis during mixed-species biofilm development by highthroughput quantitative proteomics[J]. PLoS One, 2012, 7(9): 348-51. |
| [15] | 夏丽, 王成龙, 储冰峰. 生物信息学在预测和鉴定变形链球菌非编码RNA中的应用[J]. 中华老年口腔医学杂志, 2015, 13(4): 193-6. |
| [16] | 王立贵, 赵雅琳, 李伍举. 细菌sRNA基因及其靶标预测研究进展[J]. 微生物学报, 2009, 49(1): 1-5. |
| [17] | 肖斌, 陈瑜, 李林海, 等. 兔抗弓形虫CorA家族Mg-(2+)转运蛋白多肽抗体的制备及鉴定[J]. 生物技术通讯, 2016, 27(6): 778-82. |
| [18] | 何胜平, 陈雅华, 赵瑛瑛, 等. 肝螺杆菌甲基基团趋化信号转导蛋白多克隆抗体的制备及鉴定[J]. 南方医科大学学报, 2013(9): 1295-8. |
| [19] | Lalaouna D, Simoneau-Roy M, Lafontaine DA. Regulatory RNAS and target mRNA decay in prokaryotes[J]. Biochim Biophys Acta, 2013, 1829(6/7, SI): 742-7. |
| [20] | 赵兴福, 黄晓晶, 蔡志宇, 等. 高龋患者与无龋健康人口内变形链球菌蛋白表达差异的初步分析[J]. 口腔医学研究, 2008, 24(2): 127-30. |
| [21] | 闫啸. 分子伴侣GroEL/GroES介导重组蛋白可溶表达及折叠与组装初步研究[D]. 杭州: 浙江大学, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10335-1013303279.htm |
| [22] | Liu HH, Yan F, Liu ZR. Oscillatory dynamics in a gene regulatory network mediated by small RNA with time delay[J]. Nonlinear Dyn, 2014, 76(1): 147-59. DOI: 10.1007/s11071-013-1117-z. |
| [23] | Zhang AX, Schu DJ, Tjaden BC, et al. Mutations in interaction surfaces differentially impact E. coli Hfq association with small RNAs and their mRNA targets[J]. J Mol Biol, 2013, 425(19): 3678-97. DOI: 10.1016/j.jmb.2013.01.006. |
| [24] | Wagner R, Ümit P. CRISPR: A bacterial immunity system based on small RNAs[M]. Berlin Heidelberg: Springer, 2012: 121-43. |
 2017, Vol. 37
2017, Vol. 37

