2. 南方医科大学 口腔医学院,广东 广州 510515;
3. 南方医科大学附属广东省口腔医院,广东 广州 510282;
4. 深圳市龙华新区中心医院口腔科,广东 深圳 518110
2. 2 College of Stomatology, Southern Medical University, Guangzhou 510515, China;
3. Guangdong Provincial Stomatological Hospital, Southern Medical University, Guangzhou 510282, China;
4. Department of Stomatology, Longhua New District Central Hospital, Shenzhen 518110, China
龋病是常见的口腔疾病,变异链球菌(S. mutans)作为最主要的致龋菌之一,在龋病的发生发展中起着重要的作用。S. mutans可通过酵解食物中的多种碳水化合物产生有机酸,使菌斑pH下降,导致牙面硬组织脱矿与再矿化的失衡,形成龋齿。同时S. mutans是产酸耐酸菌,其能在亚致死性酸环境中生长,并通过引起自身基因表达的变化提高在致死酸性环境中的生存能力,即酸耐受反应(ATR),从而较其他不耐酸菌更具有生存优势[1]。
SMU.2055蛋白被认为是假想的乙酰基转移酶,通过同源性序列比对属于GNAT家族,这类酶的主要作用是将乙酰辅酶A(AcCoA)上的乙酰基转移给底物上的氨基,选择性乙酰化4~5个氨基之一。而乙酰化修饰与细菌的形态、生长代谢、应激反应等方面有着密切的关系[2-3]。第一个确定的GNAT家族蛋白是细菌的乙酰基转移酶[4]。GNAT家族是蛋白乙酰基转移酶中极为重要的一个家族,该家族的乙酰基转移酶参与了细胞生长调控、基因转录激活和DNA损伤修复[5]。因此,我们推测SMU.2055蛋白与变异链球菌的生长、应激等有关。本课题组前期已通过结构生物学方法对SMU.2055蛋白进行研究,培养蛋白晶体并成功解析其三维晶体结构,晶体衍射率为2.3 Ǻ,属于C2221空间群,晶胞参数为a= 92.0 Ǻ,b=95.0 Ǻ,c=192.2 Ǻ[6]。对该蛋白进行结构分析,确定底物的结合位点,筛选出一部分与靶标蛋白具有较好结合活性的小分子抑制剂,旨在通过小分子抑制剂改变S. mutans的生物表型和生物学行为,进而改变其致龋性,达到防龋的效果[7]。目前,国内外对SMU.2055基因的功能尚缺乏研究。因此,本研究拟构建SMU.2055基因缺陷菌株,观察其形态、生长能力、产酸能力及耐酸能力的变化,探讨SMU.2055基因在S. mutans的形态及与致龋性相关的生物学特性中发挥的功能作用。
1 材料和方法 1.1 菌株及培养条件本实验所用菌株S.mutans UA159(北京大学口腔医学院中心实验室)采用脑心浸液(BHI)培养基(青岛海博生物技术有限公司)培养。基因缺陷菌株采用含1 mg/mL壮观霉素(spectinomycin, spe)(Sigma公司,美国)选择性BHI培养基培养。S. mutans的培养条件为37 ℃、静置厌氧培养(10% CO2、10% H2、80% N2)。大肠杆菌(E.coli)DH5α[天根生化科技(北京)有限公司]采用Luria-Bertani(LB)培养基(青岛海博生物技术有限公司)培养,选择性LB培养基采用含0.1 mg/mLspe抗性培养基。E. coli的培养条件为37 ℃、200 r/min振荡需氧培养。
1.2 SMU.2055基因缺陷菌株的构建根据NCBI提供的S.mutans UA159全基因组序列,利用Primer Premier 5.0软件设计SMU.2055基因上下游引物,具体见表 1。
| 表 1 SMU.2055基因缺陷菌株构建所用引物序列 Table 1 Sequences of primers of constructing SMU.2055-deficient mutant |
采用PCR扩增SMU.2055基因上下游片段,并对该片段进行双酶切。经纯化后,在T4连接酶作用下,分别将SMU.2055基因上下游片段与pFW5质粒(第四军医大学口腔医学院余擎教授惠赠)进行连接,并使用E.coli DH5α感受态细胞进行转化。采用选择性LB培养基过夜培养,筛选阳性克隆,并提取阳性克隆的质粒进行测序鉴定。使用1 μg/mL的感受态刺激肽对野生型S. mutans UA159进行刺激,并将构建成功的SMU.2055基因缺陷质粒与其混合进行转化。采用选择性BHI培养基过夜培养,筛选阳性克隆,并提取阳性克隆的DNA进行PCR及测序鉴定[8]。
1.3 SMU.2055基因缺陷菌株生长能力检测将野生型S. mutans UA159及SMU.2055基因缺陷菌株复苏后,调节菌液A600≈0.8,按1:100比例分别加入BHI液体培养基中,并在SMU.2055基因缺陷菌株菌液中加入终浓度为1 mg/mL的spe,37 ℃厌氧培养16 h,每隔1 h测量菌液A600值,重复3次,绘制S. mutans UA159及SMU.2055基因缺陷菌株的生长曲线[7]。
1.4 SMU.2055基因缺陷菌株的形态结构观察 1.4.1 扫描电镜观察将野生型S. mutans UA159及SMU.2055基因缺陷菌株复苏后,调节菌液A600≈0.8,在6孔板中放入2 cm×2 cm大小的盖玻片,每孔加入菌液40 μL及BHI液体培养基4 mL。SMU.2055基因缺陷菌株菌液中另外加入终浓度为1 mg/mL的spe。在37 ℃厌氧环境中培养16 h后,小心弃去菌液,取出盖玻片,2%戊二醛固定1 h,乙醇梯度脱水30 min,干燥,喷金,扫描电子显微镜下观察细菌形态。
1.4.2 透射电镜观察将野生型S. mutans UA159及SMU.2055基因缺陷菌株复苏后,调节菌液A600≈0.8,按1:100加入液体培养基中,37 ℃厌氧培养至对数生长期,3000 r/min离心,去上清液后加入0.3%戊二醛预固定15~30 mim,10 000 r/min离心15 min,采用2%戊二醛固定,1%四氧化锇再固定,丙酮梯度脱水,包埋,切片,醋酸铀及枸橼酸铅双重染色,透射电子显微镜观察。
1.5 SMU.2055基因缺陷菌株产酸能力检测将野生型S. mutans UA159及SMU.2055基因缺陷菌株复苏后,使用无菌PBS溶液洗涤菌体3次,重悬,调节菌液A600≈0.8,按1:100加入BHI液体培养基中,37 ℃厌氧培养16 h。测量培养前菌液的pH值,并从第4 h起,每隔2 h测量1次菌液pH值,计算每个测量时间点菌液与培养前菌液的△pH值,实验重复3次。使用SPSS 20.0软件进行两独立样本t检验,检验水平α=0.05。
1.6 SMU.2055基因缺陷菌株耐酸能力检测将野生型S. mutans UA159及SMU.2055基因缺陷菌株置于BHI液体培养基中(pH7.0),培养至A600≈0.2。将菌液于4000 r/min、4 ℃下离心10 min,弃上清液。加入等体积的BHI液体培养基(pH5.0)预酸化处理,培养2 h,调整至A600≈0.3。离心,弃上清液,漂洗,置于甘氨酸缓冲液(1 mol/L,pH2.8)(上海生工生物工程有限公司)中酸化0、15、30、45 min后进行梯度稀释,并涂板。培养24 h后进行菌落计数,计算生存率。实验重复3次。使用SPSS 20.0软件进行两独立样本t检验,检验水准α=0.05。
2 结果 2.1 SMU.2055基因缺陷菌株的鉴定SMU.2055基因缺陷菌株PCR鉴定结果见图 1。如图 1所示,SMU.2055基因缺陷菌株的PCR扩增片段大小与以SMU.2055重组质粒为模板所扩增的片段大小一致,且与预计相符。对片段进行测序,并将结果在NCBI上进行BLAST比对,结果显示99%相符。SMU.2055基因缺陷菌株构建成功。
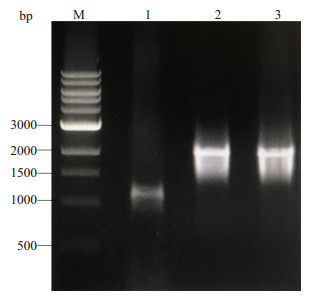
|
图 1 SMU.2055基因缺陷菌株的PCR鉴定 Figure 1 Identification of SMU.2055-deficient mutant by PCR. M: 1kb ladder; Lane 1: Wild-type S. mutans UA159; Lane 2: SMU.2055 recombinant plasmid; 3: SMU.2055-deletion mutants. All amplifications used the primers SMU.2055-UP-F and SMU.2055-DOWN-R. |
SMU.2055基因缺陷菌株在BHI培养基中,与野生菌株相比,该基因缺陷菌株的生长受到明显的影响。如图 2所示,野生菌株在约第4 h进入对数生长期,约第8 h达到平台期,而缺陷菌株的迟缓期明显延长,在约第7 h开始进入对数生长期,约第13 h达到平台期。
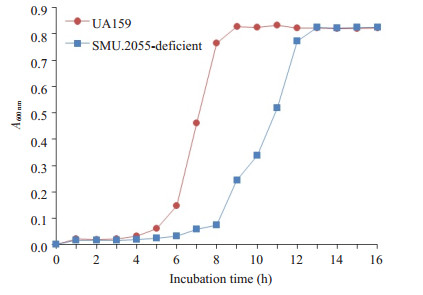
|
图 2 野生型S. mutans UA159及SMU.2055基因缺陷菌株的生长曲线 Figure 2 Growth curve of wild-type S. mutans UA159 and SMU.2055-deficient mutant. |
两种菌株经过16 h培养后,在SEM下随机选取一个视野,观察其表面形态结果,如图 3所示,SMU.2055基因缺陷株形态及菌体间物质出现明显的改变。野生型S. mutans UA159细菌形态规则,包膜光滑完整,呈球杆状,菌体间存在不规则物质。而SMU.2055基因缺陷菌株菌体较野生型的长,部分细菌呈团块状分布,菌体间不规则物质较少。

|
图 3 野生型S. mutans UA159及SMU.2055基因缺陷菌株细菌SEM观察 Figure 3 Observation of wild-type S. mutans UA159 and SMU.2055-deficient mutant by scanning electron microscopy. A: Wild-type S. mutans UA159 (Original magnification: × 12 500); B: SMU.2055-deletion mutants (× 12 500); C: Wild-type S. mutans UA159 (× 25 000); D: SMU.2055-deletion mutants (×25 000). |
将制备好的切片在TEM下观察,随机选取一个视野,结果如图 4所示,野生型S. mutans UA159形态正常,细胞膜结构完整,分裂状态下细菌两级对称,分裂后细菌形态一致。SMU.2055基因缺陷菌株细胞膜较模糊,完整性被破坏。
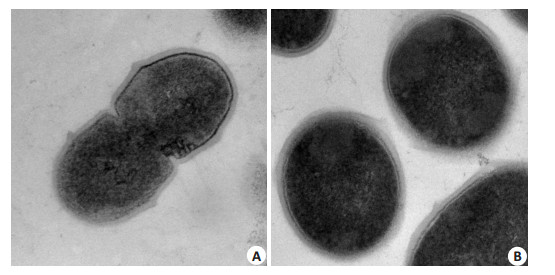
|
图 4 野生型S. mutans UA159及SMU.2055基因缺陷菌株细菌TEM观察 Figure 4 Morphological observation of wild-type S. mutans UA159 (A) and SMU.2055-deficient mutant (B) by transmission electron microscopy (×40 000). |
如图 5所示,两种菌株的菌液pH值均下降,但野生株ΔpH值更大,即产酸能力更强。野生菌株在前8 h菌液pH值下降明显,到达第8 h后,下降幅度减小,而SMU.2055基因缺陷菌株在第6~12 h pH值下降明显,随后下降幅度逐渐减小。在第6、8、10 h时,两种菌株的ΔpH值间的差异具有统计学意义(P < 0.05)。
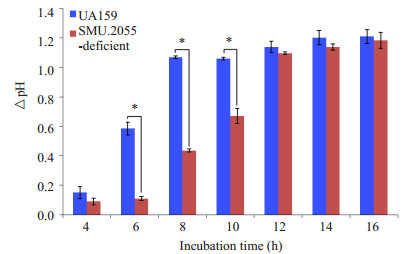
|
图 5 野生型S. mutans UA159及SMU.2055基因缺陷菌株的产酸能力 Figure 5 Acid production capacity of wild-type S. mutans UA159 and SMU.2055-deficient mutant (*P < 0.05). |
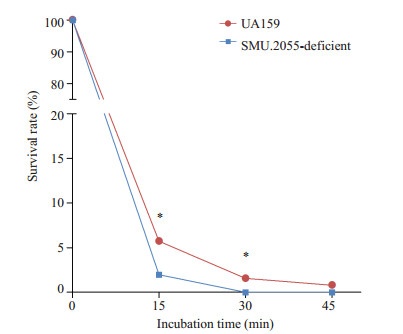
在对两种菌株进行预酸化处理后,进行酸冲击,结果如图 6所示。可见随着酸冲击时间的延长,两种菌株生存率均明显下降,缺陷菌株下降幅度较大。在作用时间为15及30 min时,两种菌株的细菌生存率间的差异具有统计学意义(P < 0.05),且当酸作用时间为30及45 min时,SMU.2055基因缺陷菌株未见生长。
|
图 6 酸性条件下野生型S. mutans UA159及SMU.2055基因缺陷菌株生存率 Figure 6 Survival rate of wild-type S. mutans UA159 and SMU.2055-deficient mutant in acidic condition (*P < 0.05). |
变异链球菌SMU.2055蛋白被认为是假定的乙酰基转移酶,属于GNAT超家族。乙酰化过程广泛存在于真核生物及原核生物中,并参与蛋白质合成等多个过程[10-11]。Zhang等[12]在大肠杆菌中发现91个乙酰化修饰的蛋白,其中超过70%的蛋白质是与代谢相关的酶类和翻译调节蛋白,提示乙酰化修饰与细菌代谢密切相关。除此之外,乙酰基转移酶在多种生命活动过程中如生长、氧化应激耐受、蛋白质活性调控等均具有重要作用[13-17]。因此,我们推测SMU.2055基因在变异链球菌的生长及其生物学特性中发挥一定的作用。本课题组前期通过靶基因筛选、高通量自动化基因克隆和表达、蛋白纯化、晶体筛选、晶体衍射数据收集和晶体结构解析等方法,成功解析了变异链球菌SMU.2055蛋白的三维晶体结构[6],且该结构已被蛋白质数据库接受,编号为3LD2。并基于该结构设计与筛选出一部分与该蛋白结构相匹配的小分子化合物抑制剂,建立S.mutans UA159蛋白抑制剂虚拟筛选模型[7],因此,有必要研究SMU.2055蛋白在S. mutans的生长及致龋中发挥的作用。
本研究采用同源重组的方法,将目的基因上下游两片段分别插入拥有壮观霉素抗性基因aad9的Pfw5载体的两个多克隆位点中,使目的基因被抗性基因替换。经过PCR及测序鉴定,成功构建带有壮观霉素抗性的SMU.2055基因缺陷菌株。
在SMU.2055基因缺陷菌株生长能力研究中,从生长曲线可以看出,缺陷菌株的迟缓期明显延长,在观察的前8 h内未见明显生长,表明SMU.2055基因在变异链球菌的生长过程中起着一定的作用,这可能是因为乙酰化修饰参与了细菌糖酵解、三羧酸循环等生长代谢过程[18],当乙酰化修饰受到影响时,细菌的生长代谢受到影响。但在8 h后,缺陷菌株开始生长,且终末菌量与野生菌株基本相同,推测当SMU.2055基因缺陷导致生长缓慢时,存在代偿机制对其生长进行补偿以维持生命。
扫描电镜及透射电镜下观察野生型S. mutans UA159及SMU.2055基因缺陷菌株形态结构,结果显示,SMU.2055基因缺陷菌株结构与野生菌株相比,细菌细胞膜完整性及不规则物质的形成均受到影响。这可能与乙酰化修饰可改变细菌细胞壁结构和通透性有关[19]。S. mutans菌体间的不规则物质主要由胞外多糖、蛋白质等组成[20],本研究中,SMU.2055基因缺陷菌株菌体间不规则物质减少,推测SMU.2055基因可能通过影响变异链球菌细胞膜结构完整性及通透性,进一步影响其合成胞外多糖、蛋白质等能力。
S. mutans的致龋性与其产酸特性密切相关。S. mutans可依赖糖酵解途径,在代谢过程中产生大量有机酸,导致牙体硬组织脱矿,形成龋齿[21]。由生长曲线可知,在第12~16小时,SMU.2055基因缺陷菌株与野生型UA159菌株均处于平台期,菌量相同,而两种菌株的菌液△pH值未见明显的差异,推测SMU.2055基因对S. mutans的产酸能力不造成明显影响。在第6、8、10小时,缺陷菌株与野生菌株的△pH值间的差异具有统计学意义,这可能与缺陷菌株的生长受到影响有关。
细菌在亚致死性的低pH值环境中生长一定时间,可产生酸适应,耐酸能力会有所提高[22]。当对野生型S. mutans UA159及SMU.2055基因缺陷菌株进行酸适应后,加入酸冲击,结果显示缺陷菌株较野生菌株对酸环境更敏感,在酸性条件下的生长受到明显的抑制。有研究表明,变异链球菌为应对酸性环境,会降低自身细菌细胞膜的质子通透性[23],从而能在较低pH的环境下生存。在本研究中,透射电镜及扫描电镜结果显示,SMU.2055基因缺陷菌株细菌细胞膜结构发生改变。我们推测SMU.2055基因可能通过影响细菌细胞膜结构,进一步影响变异链球菌质子通透性和质子移位膜腺苷三磷酸酶的活性[24],从而使变异链球菌SMU.2055基因缺陷菌株在应对酸环境时,降低自身质子通透性能力下降,进而对酸的耐受力下降。
本研究成功构建了变异链球菌SMU.2055基因缺陷菌株,并初步探讨了该基因对S. mutans的形态及与致龋性相关的生物学特性的影响。SMU.2055基因影响S. mutans的形态、生长能力及耐酸能力,但其具体调控机制仍有待进一步研究。
| [1] | Matsui R, Cvitkovitch D. Acid tolerance mechanisms utilized by Streptococcus mutans[J]. Future Microbiol, 2010, 5(3): 403-17. DOI: 10.2217/fmb.09.129. |
| [2] | Sang Y, Ren J, Ni J, et al. Protein acetylation is involved in Salmonella enterica serovar typhimurium virulence[J]. J Infect Dis, 2016, 213(11): 1836-45. DOI: 10.1093/infdis/jiw028. |
| [3] | Pan J, Ye Z, Cheng Z, et al. Systematic analysis of the lysine acetylome in Vibrio parahemolyticus[J]. J Proteome Res, 2014, 13(7): 3294-02. DOI: 10.1021/pr500133t. |
| [4] | Vetting MW, S DCL, Yu M, et al. Structure and functions of the GNAT superfamily of acetyltransferases[J]. Arch Biochem Biophys, 2005, 433(1): 212-26. DOI: 10.1016/j.abb.2004.09.003. |
| [5] | Carrozza MJ, Utley RT, Workman JL, et al. The diverse functions of histone acetyltransferase complexes[J]. Trends Genet, 2003, 19(6): 321-9. DOI: 10.1016/S0168-9525(03)00115-X. |
| [6] | Zhao WH, Zhan XR, Gao XZ, et al. Preliminary X-ray crystallographic analysis of SMU.2055 protein from the caries pathogen Streptococcus mutans[J]. Acta Crystallogr Sect F Struct Biol Cryst Commun, 2010, 66(Pt 5): 530-3. |
| [7] | 陈晓丹, 展秀荣, 吴昕彧, 等. 变异链球菌SMU.2055蛋白晶体结构及其小分子抑制剂设计与筛选[J]. 西口腔医学杂志, 2015(2): 182-6. DOI: 10.7518/hxkq.2015.02.016. |
| [8] | 吴昕彧, 陈晓丹, 赵望泓, 等. 变异链球菌磷酸转移酶系统ptxA、ptxB基因对细菌生长能力的影响[J]. 华西口腔医学杂志, 2016(3): 302-6. DOI: 10.7518/hxkq.2016.03.017. |
| [9] | Wu X, Hou J, Chen X, et al. Identification and functional analysis of the L-ascorbate-specific enzyme Ⅱ complex of the phosphotransferase system in Streptococcus mutans[J]. BMC Microbiol, 2016, 16: 51. DOI: 10.1186/s12866-016-0668-9. |
| [10] | Henriksen P, Wagner SA, Weinert BT, et al. Proteome-wide analysis of lysine acetylation suggests its broad regulatory scope in Saccharomyces cerevisiae[J]. Mol Cell Proteomics, 2012, 11(11): 1510-22. DOI: 10.1074/mcp.M112.017251. |
| [11] | Zhang K, Zheng S, Yang JS, et al. Comprehensive profiling of protein lysine acetylation in Escherichia coli[J]. J Proteome Res, 2013, 12(2): 844-51. DOI: 10.1021/pr300912q. |
| [12] | Zhang J, Sprung R, Pei J, et al. Lysine acetylation is a highly abundant and evolutionarily conserved modification in Escherichia coli[J]. Mol Cell Proteomics, 2009, 8(2): 215-25. DOI: 10.1074/mcp.M800187-MCP200. |
| [13] | Nasuno R, Hirano Y, Itoh T, et al. Structural and functional analysis of the yeast N-acetyltransferase Mpr1 involved in oxidative stress tolerance via proline metabolism[J]. Proc Natl Acad Sci USA, 2013, 110(29): 11821-6. DOI: 10.1073/pnas.1300558110. |
| [14] | Kosono S, Tamura M, Suzuki S, et al. Changes in the acetylome and succinylome of Bacillus subtilis in response to carbon source[J]. PLoS One, 2015, 10(6): e131169. |
| [15] | Liao G, Xie L, Li X, et al. Unexpected extensive lysine acetylation in the trump-card antibiotic producer Streptomyces roseosporus revealed by proteome-wide profiling[J]. J Proteomics, 2014, 106: 260-9. DOI: 10.1016/j.jprot.2014.04.017. |
| [16] | Xie L, Wang X, Zeng J, et al. Proteome-wide lysine acetylation profiling of the human pathogen Mycobacterium tuberculosis[J]. Int J Biochem Cell Biol, 2015, 59: 193-202. DOI: 10.1016/j.biocel.2014.11.010. |
| [17] | Ren J, Sang Y, Tan Y, et al. Acetylation of lysine 201 inhibits the DNA-binding ability of PhoP to regulate salmonella virulence[J]. PLoS Pathog, 2016, 12(3): e1005458. DOI: 10.1371/journal.ppat.1005458. |
| [18] | Song L, Wang G, Malhotra A, et al. Reversible acetylation on Lys501 regulates the activity of RNase Ⅱ[J]. Nucleic Acids Res, 2016, 44(5): 1979-88. DOI: 10.1093/nar/gkw053. |
| [19] | Lee DW, Kim D, Lee YJ, et al. Proteomic analysis of acetylation in thermophilic Geobacillus kaustophilus[J]. Proteomics, 2013, 13(15): 2278-82. DOI: 10.1002/pmic.v13.15. |
| [20] | 刘凤英. 可逆的赖氨酸乙酰化修饰对细菌生理功能的影响[D]. 中国科学院大学, 2014. |
| [21] | Hasan S, Singh K, Danisuddin M, et al. Inhibition of major virulence pathways of Streptococcus mutans by quercitrin and deoxynojirimycin: a synergistic approach of infection control[J]. PLoS One, 2014, 9(3): e91736. DOI: 10.1371/journal.pone.0091736. |
| [22] | Sawada D, Ogawa T, Miyake M, et al. Potent inhibitory effects of D-tagatose on the acid production and water-insoluble glucan synthesis of Streptococcus mutans GS5 in the presence of sucrose[J]. Acta Med Okayama, 2015, 69(2): 105-11. |
| [23] | Guo L, Mclean JS, Lux R, et al. The well-coordinated linkage between acidogenicity and aciduricity via insoluble glucans on the surface of Streptococcus mutans[J]. Sci Rep, 2015, 5: 18015. |
| [24] | Fozo EM, Quivey RJ. Shifts in the membrane fatty acid profile of Streptococcus mutans enhance survival in acidic environments[J]. Appl Environ Microbiol, 2004, 70(2): 929-36. DOI: 10.1128/AEM.70.2.929-936.2004. |
| [25] | Lemos JA, Abranches J, Burne RA. Responses of cariogenic streptococci to environmental stresses[J]. Curr Issues Mol Biol, 2005, 7(1): 95-107. |
 2017, Vol. 37
2017, Vol. 37

