2. 南方医科大学 基础医学院神经生物学教研室,广东 广州 510515
2. Department of Neurobiology, College of Basic Medical Science, Southern Medical University, Guangzhou 510515, China
环二鸟苷酸(c-di-GMP)是一种由两分子GTP缩合形成的环二核苷酸,为细菌中广泛存在的第二信使,参与调控多项细菌生理功能,并在人体固有的免疫应答中起免疫调节剂的作用[1-2]。c-di-GMP信号通路的研究早期主要集中在革兰氏阴性菌中[3],近年来,在许多革兰氏阳性菌中也证明了其信号通路的存在,这些革兰氏阳性菌主要包括耻垢分枝杆菌(Mycobacterium smegmatis)[4]、艰难梭菌(Clostridium difficile)[5]、枯草芽孢杆菌(Bacillus subtilis)[6]和单核增生李斯特菌(Listeria monocytogenes)[7]等,而在乳杆菌属中尚未见报道。嗜酸乳杆菌是革兰氏阳性菌,能发酵多种糖,产酸能力强,可使菌斑pH下降,同时具有很强的耐酸能力,是公认的龋病相关主要细菌之一,与变异链球菌等一起被列为“龋标志菌”[8-9]。
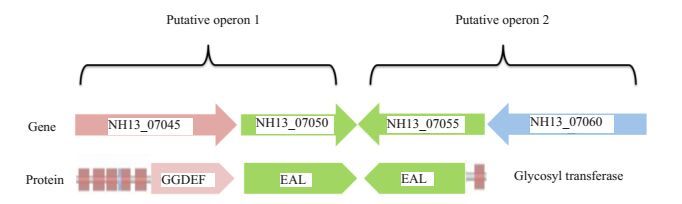
前期通过生物信息学分析发现:在嗜酸乳杆菌的基因组中存在一个c-di-GMP合成酶(GGDEF)的编码基因(NH13_07045)和其两个分解酶(EAL)的编码基因(NH13_07050和NH13_07055)。
为验证嗜酸乳杆菌是否存在c-di-GMP的体内代谢途径,并为后续进一步探究c-di-GMP在嗜酸乳杆菌中的调控机制,基于上述分析及前期预试验的结果,本研究拟对NH13_07045-GGDEF结构域编码基因,及全长的NH13_07050和NH13_07055基因进行克隆、表达及纯化。以获得高纯度的目的蛋白并进行体外酶活性实验。
1 材料和方法 1.1 材料 1.1.1 主要材料及试剂pMAL-c2-His表达质粒[10]由本研究组提供。细菌基因组DNA抽提试剂盒,高保真DNA聚合酶及T4 DNA连接酶(TaKaRa,日本);DNA凝胶回收试剂盒和质粒小量抽提试剂盒(天根生物,北京);BCA蛋白定量试剂盒,引物合成,三磷酸鸟苷(GTP),二磷酸鸟苷(GDP)及DNA测序(生工生物,上海);限制性内切酶BamHⅠ、SalⅠ和PstⅠ(Promega,美国);直链淀粉树脂(NEB公司,美国);抗His标签小鼠多克隆抗体和辣根过氧化物酶标记的山羊抗小鼠IgG(博士徳公司,武汉);ECL化学发光底物(PerkinElmer,美国);c-di-GMP,线性二鸟苷酸(pGpG)(Biolog,德国)。
1.1.2 细菌和培养基嗜酸乳杆菌ATCC4356(广东省微生物菌种保藏中心),MRS培养基,LB培养基(青岛海博),大肠杆菌TOP10感受态细胞,BL21感受态细胞(本实验室提供),C43(DE3)感受态细胞(武汉淼灵生物)。
1.1.3 引物根据GenBank中嗜酸乳杆菌ATCC4356的NH13_07045-GGDEF、NH13_07050和NH13_07055片段序列设计合成特异性引物(表 1)。
| 表 1 基因片段特异性引物 Table 1 Gene specific primers |
对3个目的基因进行计算机分析。将它们相应目的蛋白的氨基酸序列输入NCBI网页的CDD[11]数据库中进行序列同源性分析,寻找保守结构域。再使用TMHMM 2.0[12]软件预测蛋白疏水跨膜区(图 1)。将冻干的嗜酸乳杆菌用MRS液体培养基37 ℃厌氧培养24 h,涂布于MRS固体培养基平板中,37 ℃厌氧培养48 h后常规接种,传代,涂片。取3 mL菌液离心获得菌沉淀后,用细菌基因组DNA提取试剂盒提取嗜酸乳杆菌基因组DNA,并以其作为模板进行PCR扩增,分别获得3个目的基因片段。通过1%琼脂糖凝胶电泳检测反应产物并从胶中分别回收,再用微量分光光度计NanoDrop2000定量浓度。
|
图 1 嗜酸乳杆菌中c-di-GMP信号通路相关基因及蛋白的生物信息学分析 Figure 1 In silico analysis of the genes and proteins involved in c-di-GMP signaling in Lactobacillus acidophilus. |
将pMAL-c2-His质粒及PCR产物分别进行双酶切,通过琼脂糖凝胶电泳分别回收酶切产物。微量分光光度计定量后,按照插入片段与线性化载体的摩尔比为5-10:1,用T4 DNA连接酶于16 ℃连接过夜。次日转化大肠杆菌TOP10感受态细胞,涂布于含氨苄(100 μg/mL)的LB培养基琼脂平板,37 ℃培养过夜。然后挑选阳性克隆进行增菌培养,并提取质粒进行双酶切和PCR鉴定。将重组正确的质粒送至生工生物公司(广州)进行测序。测序正确的上述重组质粒分别命名为pMAL-7045-GGDEF、pMAL-7050和pMAL-7055。
1.2.3 目的蛋白的表达将鉴定正确的重组质粒pMAL-7045-GGDEF,pMAL-7050转化到大肠杆菌BL21感受态细胞中,pMAL-7055转化到大肠杆菌C43(DE3)感受态细胞中,涂布于含有氨苄(100 μg/mL)的LB琼脂平板上,37 ℃孵育过夜。挑选阳性克隆分别接种至3 mL含氨苄(100 μg/mL)的LB液体培养基中,37 ℃、180 r/min震荡培养过夜。次日以1:100比例加入5 mL含氨苄(100 μg/mL)和0.2%葡萄糖的LB液体培养基中,37 ℃、180 r/min震荡3 h至A600 nm为0.6。此时,在pMAL-7045-GGDEF和pMAL-7050的转化菌培养基中加入终浓度为0.5 mmol的IPTG,在同样条件下诱导4 h;而在pMAL-7055的转化菌培养基中加入终浓度为0.25 mmol的IPTG,于25 ℃,120 r/min条件下诱导10 h。各诱导菌样品的制备和鉴定步骤为:取适量菌液在4 ℃、6000 r/min离心5 min,弃上清。沉淀菌以250 μL TBS重悬,吸取32 μL混悬液并加入8 μL 5×SDS上样缓冲液,混匀后沸水煮10 min,于12 000 g离心1 min,取20 μL上清进行上样,进行SDS-PAGE,一块胶用考马斯亮蓝(R-250)染色做蛋白条带分析,另一块胶用于表达蛋白的免疫印迹鉴定(详见1.2.4)。
1.2.4 Western blot实验上述蛋白样品经SDS-PAGE电泳后,将胶中蛋白电转移至PVDF膜上并在室温下用5%脱脂奶粉封闭液封闭1 h,TBST(20 mmol Tris-HCl、150 mmol NaCl、0.1% Tween-20、pH=7.4)洗涤2次,然后加入小鼠抗His-tag(一抗,1:300)于含5%脱脂奶粉的TBST缓冲液中4 ℃孵育过夜。接着用TBST洗涤3次,然后用羊抗鼠IgG-HRP(二抗,1:5000)于室温中再孵育1 h,TBST洗涤3次。最后将吸干水分的PVDF膜置于BioRad荧光成像仪的托板上,在膜上均匀加上ECL化学发光液并进行自动曝光拍照,选取曝光适当的照片进行分析。
1.2.5 目的蛋白的亲和纯化按上述表达菌的制备方法(1.2.3)制备200 mL的表达菌,离心后向沉淀菌中加入15 mL裂解液(10 mmol Tris-HCl、150 mmol NaCl、1 mmol EDTA、PMSF 17 ng/mL、0.2% Triton-X 100、Cocktail蛋白酶抑制剂、pH=7.4)重悬,于冰浴上进行超声裂解。而后16 000 g离心20 min取上清,缓缓加入并流经直链淀粉树脂柱(2 mL/min),再用Column buffer(10 mmol Tris-HCl、150 mmnol NaCl、1 mmol EDTA、pH=7.4)洗柱4次后,改用洗脱液(20 mmol Tris-HCl、150 mmol NaCl、1 mmol EDTA、0.2% Triton-X 100、10 mmol麦芽糖、pH=7.4)洗脱,收集洗脱液。取20 μL洗脱液行SDS-PAGE分析,剩余洗脱液加入甘油至终浓度为10%,储藏于-20 ℃备用。
1.2.6 目的蛋白的体外酶活性实验将纯化的蛋白加入离心浓缩管(10000 cut-off),分别加入DGC酶活性缓冲液(50 mmol Tris-HCl、50 mmol NaCl、10 mmol MgCl2、0.5 mmol EDTA、pH=7.4)或PDE酶活性缓冲液(50 mmol Tris-HCl、50 mmol NaCl、5 mmol MgCl2、0.5 mmol EDTA、pH=9.35),进行蛋白浓缩及缓冲液置换,调整蛋白浓度至5 μmol。分别加入GTP和c-diGMP作为反应底物至终浓度为100 μmol,在100 μL体积中37 ℃反应2 h。随后,将样品加热至100 ℃,3 min灭活,16 000 g离心后取上清,使用0.22 μm滤器过滤,用作高效液相色谱分析。
1.2.7 高效液相色谱分析高效液相色谱条件:安捷伦C18色谱柱(15×4.6 mm);检测波长254 nm;流速0.7mL/min;进样量15 μL;柱温25 ℃;流动相A:100 mmol磷酸二氢钾,4 mmol四丁基硫酸氢铵,pH=5.9,流动相B:75%流动相A+25%甲醇;梯度条件(前者为时间(min),后者为流动相B的百分率):0.0,0;2.5,0;5.0,30;10.0,60;14.0,100;21.0,100;22.0,50;23.0,0。通过将c-di-GMP、pGpG、GDP和GTP标准品的保留时间(RT)与样品进行比较后进行酶活性判定。
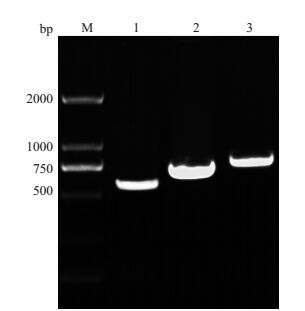
2 结果 2.1 目的基因的结构分析与扩增片段鉴定对嗜酸乳杆菌ATCC4356的基因组分析表明,3个目的基因分别是:一个含GGDEF结构的蛋白(NH13_07045)和两个含EAL结构的蛋白(NH13_07050和NH13_07055,图 1)。在此基础上,以ATCC4356基因组为模板,PCR扩增NH13_07045-GGDEF、NH13_07050和NH13_07055片段,1%琼脂糖凝胶电泳显示:特异性扩增产物与目的片段预期大小相符,分别为546、696和804 bp(图 2)。
|
图 2 目的基因扩增片段的1%琼脂糖电泳鉴定 Figure 2 Identification of 3 amplified target gene fragments in 1% agarose gel. M: DL2000 DNA marker; 1: amplified fragment of NH13_07045-GGDEF; 2: amplified fragment of NH13_07050; 3: amplified fragment of NH13_07055. |
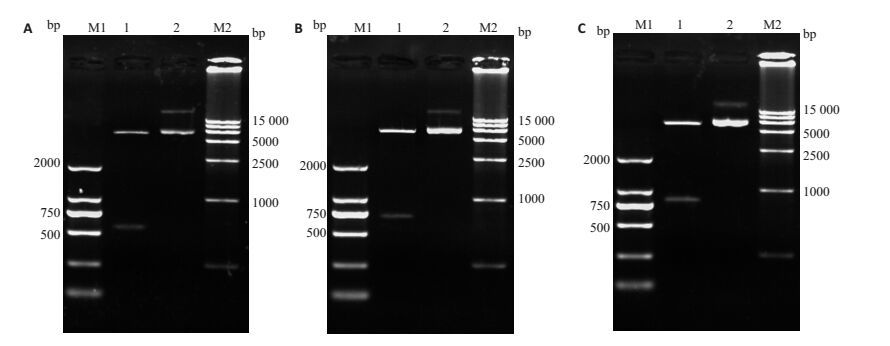
对3种重组质粒分别进行双酶切,酶切产物与自身未切割质粒进行1%琼脂糖凝胶电泳。结果表明,它们的酶切片段大小与预期相符。pMAL-7045-GGDEF为546 bp(图 3A);pMAL-7050为696 bp(图 3B)及pMAL-7055为804 bp(图 3C)。此后对重组片段的测序结果也与GenBank中嗜酸乳杆菌ATCC4356基因序列一致。表明重组表达质粒构建成功。
|
图 3 重组质粒双酶切产物的1%琼脂糖凝胶电泳 Figure 3 Identification of double enzymes digestion of the recombinant plasmids in 1% agarose electrophoresis. M1: DL2000DNA marker; M2: DL15000 DNA marker; 1: Double digested recombinant plasmid; 2: Undigested recombinant plasmid. |
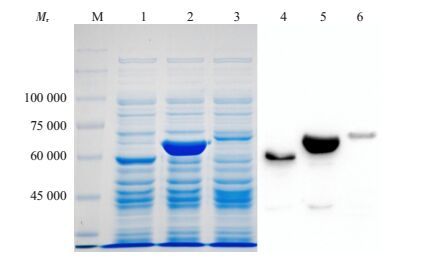
重组表达质粒pMAL-7045-GGDEF,pMAL-7050转化至大肠杆菌BL21中,pMAL-7055转化至大肠杆菌C43(DE3)中,经IPTG诱导表达后,产物经SDS-PAGE和Western blot分析(图 4),相对分子质量分别约为59 000、67 000和72 000,与预期的3个目的蛋白大小一致。用抗His-tag抗体的WB鉴定结果显示,3个特异性条带为含His标签的目的蛋白。
|
图 4 3种目的蛋白表达产物的SDS-PAGE和Western Blot分析 Figure 4 SDS-PAGE and Western blot analysis for the expression of the 3 target proteins. M: Protein ladder; 1: Lysate from pMAL-7045-GGDEF/BL21; 2: Lysate from pMAL-7050/BL21; 3:Lysate from pMAL-7055/C43(DE3); 4, 5 and 6: Western blotting results corresponding to 1, 2 and 3 with anti-His tag Ab, respectively |
分别将上述2.3中正确表达的菌体沉淀用超声裂解,其上清液经直链淀粉树脂亲和纯化后,用SDS-PAGE分析纯化前后样品的纯度及其相对分子质量。结果显示除pMAL-7055的纯化蛋白(72 000)中含少量降解蛋白外(图 5C),pMAL-7045-GGDEF(59 000,图 5A)和pMAL-7050(67 000,图 5B)均为较高纯度的目的蛋白。且3种纯化蛋白的相对分子质量与纯化前一致。
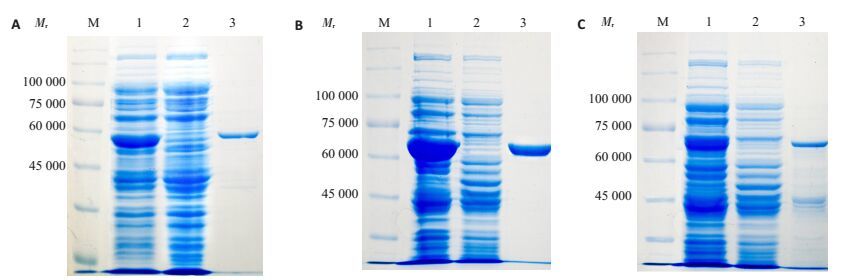
|
图 5 目的蛋白纯化的SDS-PAGE分析 Figure 5 Analysis of purified proteins in SDS-PAGE. M: Protein ladder; 1: Lysate from induced bacterial; 2: Lysate from non-induced bacterial; 3: Purified target protein. A: pMAL-7045-GGDEF/BL21; B: pMAL-7050/BL21; C: pMAL-7055/C43(DE3) |
高效液相色谱检测结果显示:以标准品的保留时间为参照(图 6A),NH13_07045的GGDEF结构域蛋白酶促反应产物与GDP和GTP标准品的出峰时间一致(图 6B);NH13_07050酶促反应产物与pGpG标准品的出峰时间一致(图 6C);NH13_07055酶促反应产物与c-di-GMP标准品的出峰时间一致(图 6D)。结果表明,NH13_07045-GGDEF结构域蛋白体外无DGC活性,仅表现出鸟苷三磷酸酶(GTPse)活性,把GTP转化成GDP;NH13_07050蛋白体外具有PDE活性,把c-di-GMP转化为链状二鸟苷酸(pGpG),而NH13_07055体外并无PDE活性。
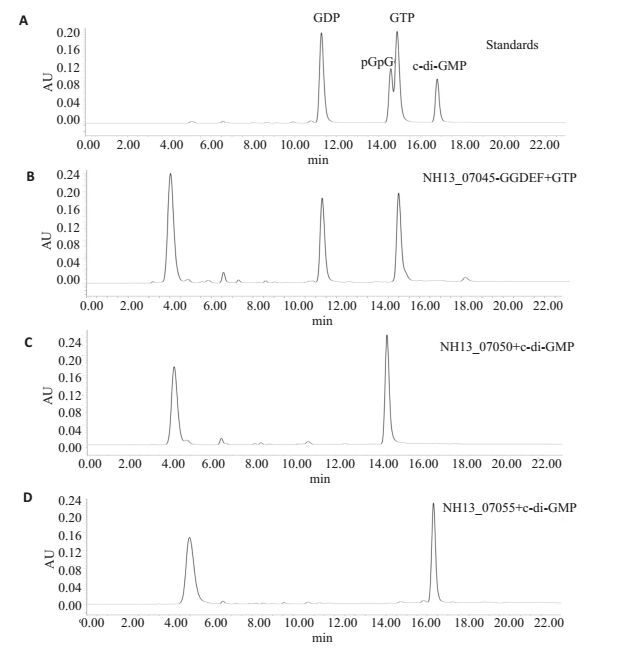
|
图 6 体外酶促反应产物的高效液相色谱分析 Figure 6 HPLC detection of in vitro enzymatic activity of the obtained proteins. A: Retention time of the peaks from standard samples; B: NH13_07045-GGDEF-encoded protein can hydrolyze GTP to GDP; C: NH13_07050-encoded protein hydrolyzes c-di-GMP to pGpG as PDE; D: NH13_07055-encoded protein has no PDE activity and can't hydrolyze c-di-GMP to pGpG. |
c-di-GMP作为一种细菌纤维素合成酶的别构激活剂于1987年被首次发现[13]。研究表明,c-di-GMP参与调节细菌的多项生理功能,包括细胞周期及分化、生物膜形成及分布、形态变化、致病因子和群感效应等[14]。参与调节c-di-GMP合成和降解的蛋白酶广泛存在于各种细菌中,因此c-di-GMP被认为是一种细菌内普遍存在的第二信使分子[3]。c-di-GMP的合成酶是含保守的GGDEF(Gly-Gly-Asp-Glu-Phe)氨基酸基序的二鸟苷酸环化酶(DGC),它可将GTP转化为c-di-GMP。而含有EAL或HD-GYP结构域的磷酸二酯酶(PDE)为c-di-GMP的降解酶[15]。这两种酶以底物循环方式维持菌体内c-di-GMP的平衡。
龋病是人类最常见的细菌感染性疾病之一,发病率高,发病涉及范围广。目前认为与龋病发生发展密切相关的菌种主要有变异链球菌、乳杆菌和放线菌等[9]。其中乳杆菌在牙本质龋中具有明显的促进作用[8]。对嗜酸乳杆菌ATCC4356的基因组分析表明,该菌株中含有一个编码具有GGDEF结构域的c-di-GMP合成酶基因及两个编码含EAL结构域的c-di-GMP水解酶基因。因此,嗜酸乳杆菌在引起龋病的进程中是否与c-di-GMP信号通路的调控相关,值得进一步探讨。
在预试验中我们发现,由于NH13_07045 N端含疏水性强的跨膜结构域,插入了NH13_07045全长基因的pMAL-c2-His质粒在IPTG诱导条件下,对大肠杆菌表达宿主BL21、TB1、ER2655和C43(DE3)均产生明显致死毒性,可能与其N端疏水的跨膜结构域有关。因而无法获得纯化蛋白用于后续实验。因此本课题组对NH13_07045的GGDEF结构域进行了单独表达纯化,用于酶活性实验。
ProOpBD[16]数据库中检索结果提示,NH13_07045与NH13_07050存在于同一操纵子。用NH13_07045的氨基酸序列在NCBI CDD[11]和TMHMM 2.0[12]数据库检索,结果显示N端序列未发现已知的保守结构域,C端结构域含有保守的GGDEF结构。对跨膜区的预测显示,NH13_07045蛋白的N端结构域包含多次的跨膜结构,而其C端结构域则位于胞质。这种结构提示,c-di-GMP的合成有可能受到细菌外部或细菌细胞璧上的信号调控。研究显示,单独GGDEF结构域蛋白往往表现出较低甚至无DGC活性,仅表现出GTPse活性,原因可能是缺少了信号感受域,无法形成具有酶活性的同源二聚体构象[3, 17]。据此,本课题组推测,NH13_07045-GGDEF结构域蛋白DGC活性的缺失,可能与缺少N端跨膜结构有关,相关内容将于下一步研究。
NH13_07050只含一个EAL结构域,其基因的5'端为一段121bp的非编码区,我们推测NH13_07050基因的表达调控与其密切相关。体外酶活性实验显示,NH13_07050具有高效的PDE活性,能使c-di-GMP水解成pGpG。
NH13_07055与NH13_07060存在于同一操纵子,NH13_07055的N端为单次跨膜结构域,C端为EAL结构域。NH13_07060为细菌表多糖聚-β-1,6-N-乙酰葡萄糖胺(PNAG)的合成酶,与大肠杆菌的PgaC,单核李斯特菌的PssE蛋白序列比较,一致性为30%,且这两种蛋白合成表多糖的过程均受c-di-GMP的调控[7, 18]。结合实验结果,我们认为,不具有PDE活性的NH13_07055可作为c-di-GMP的受体(本课题组实验结果表明其可与c-di-GMP特异性结合,数据另文发表),激活或者抑制嗜酸乳杆菌表多糖的合成通路。
含GGDEF结构域的膜蛋白功能分析[19-20]以及c-di-GMP调控细菌表多糖合成[21-22]的研究是近年来c-di-GMP信号通路的研究热点。因此本课题组在后续实验中将进行NH13_07045的体内酶活性实验,并对其跨膜结构进行功能分析。并且,对NH13_07055在c-di-GMP调控嗜酸乳杆菌表多糖合成中的作用也作进一步研究。
综上所述,本研究通过体外酶活性实验,检测到NH13_07050具有PDE活性,为后续阐明c-di-GMP在嗜酸乳杆菌中的调控机制提供了实验依据,从而为龋病的防治提供了新视野。
| [1] | Hengge R. Principles of c-di-GMP signalling in bacteria[J]. Nat Rev Microbiol, 2009, 7(4): 263-73. DOI: 10.1038/nrmicro2109. |
| [2] | Gray PM, Forrest G, Wisniewski T, et al. Evidence for cyclic diguanylate as a vaccine adjuvant with novel immunostimulatory activities[J]. Cell Immunol, 2012, 278(1/2): 113-9. |
| [3] | Römling U, Galperin MY, Gomelsky M. Cyclic di-GMP: the first 25 years of a Universal bacterial second messenger[J]. Microbiol Mol Biol Rev, 2013, 77(1): 1-52. DOI: 10.1128/MMBR.00043-12. |
| [4] | Kumar M, Chatterji D. Cyclic di-GMP: a second messenger required for long-term survival, but not for biofilm formation, in Mycobacterium smegmatis[J]. Microbiology, 2008, 154(Pt 10): 2942-55. |
| [5] | Bordeleau E, Fortier LC, Malouin F, et al. c-di-GMP turn-over in clostridium difficile is controlled by a plethora of diguanylate cyclases and phosphodiesterases[J]. PLoS Genet, 2011, 7(3): e1002039. DOI: 10.1371/journal.pgen.1002039. |
| [6] | Gao X, Mukherjee S, Matthews PM, et al. Functional characterization of core components of the Bacillus subtilis cyclic-di-GMP signaling pathway[J]. J Bacteriol, 2013, 195(21): 4782-92. DOI: 10.1128/JB.00373-13. |
| [7] | Chen LH, Köseoğlu VK, Güvener ZT, et al. Cyclic di-GMPdependent signaling pathways in the pathogenic Firmicute Listeria monocytogenes[J]. PLoS Pathog, 2014, 10(8). |
| [8] | Chhour KL, Nadkarni MA, Byun R, et al. Molecular analysis of microbial diversity in advanced caries[J]. J Clin Microbiol, 2005, 43(2): 843-9. DOI: 10.1128/JCM.43.2.843-849.2005. |
| [9] | Jenkinson HF, Lamont RJ. Oral microbial communities in sickness and in health[J]. Trends Microbiol, 2005, 13(12): 589-95. DOI: 10.1016/j.tim.2005.09.006. |
| [10] | Wang F, Robotham JM, Teuber SS, et al. Ana o 1, a cashew (Anacardium occidental) allergen of the vicilin seed storage protein family[J]. J Allergy Clin Immunol, 2002, 110(1): 160-6. DOI: 10.1067/mai.2002.125208. |
| [11] | Marchler-Bauer A, Derbyshire MK, Gonzales NR, et al. CDD: NCBI's conserved domain database[J]. Nucleic Acids Res, 2015, 43: D222-6. DOI: 10.1093/nar/gku1221. |
| [12] | Chen YJ, Yu P, Luo JC, et al. Secreted protein prediction system combining CJ-SPHMM, TMHMM, and PSORT[J]. Mammalian Genome, 2003, 14(12): 859-65. DOI: 10.1007/s00335-003-2296-6. |
| [13] | Ross P, Weinhouse H, Aloni Y, et al. Regulation of cellulose synthesis in Acetobacter xylinum by cyclic diguanylic acid[J]. Nature, 2008, 325(611): 279-81. |
| [14] | Sondermann H, Shikuma NJ, Yildiz FH. You've come a long way: c-di-GMP signaling[J]. Curr Opin Microbiol, 2012, 15(2): 140-6. DOI: 10.1016/j.mib.2011.12.008. |
| [15] | Dow JM, Fouhy Y, Lucey JF, et al. The HD-GYP domain, cyclic di-GMP signaling, and bacterial virulence to plants[J]. Mol Plant Microbe Interact, 2006, 19(12): 1378-84. DOI: 10.1094/MPMI-19-1378. |
| [16] | Taboada B, Ciria R, Martinez-Guerrero CE, et al. ProOpDB: prokaryotic operon DataBase[J]. Nucleic Acids Res, 2012, 40: D627-31. DOI: 10.1093/nar/gkr1020. |
| [17] | Zhu B, Liu C, Liu S, et al. Membrane association of SadC enhances its diguanylate cyclase activity to control exopolysaccharides synthesis and biofilm formation in Pseudomonas aeruginosa[J]. Environ Microbiol, 2016, 18(10): 3440-52. DOI: 10.1111/1462-2920.13263. |
| [18] | Steiner S, Lori C, Boehm A, et al. Allosteric activation of exopolysaccharide synthesis through cyclic di-GMP-stimulated protein-protein interaction[J]. EMBO J, 2013, 32(3): 354-68. |
| [19] | Lacey M, Agasing A, Lowry R, et al. Identification of the YfgF MASE1 domain as a modulator of bacterial responses to aspartate[J]. Open Biol, 2013, 3(6): 130046. DOI: 10.1098/rsob.130046. |
| [20] | Schirmer T. C-di-GMP synthesis: structural aspects of evolution, catalysis and regulation[J]. J Mol Biol, 2016, 428(19): 3683-701. DOI: 10.1016/j.jmb.2016.07.023. |
| [21] | Pérez-Mendoza D, Sanjuán J. Exploiting the Commons: cyclic diguanylate regulation of bacterial exopolysaccharide production[J]. Curr Opin Microbiol, 2016, 30: 36-43. DOI: 10.1016/j.mib.2015.12.004. |
| [22] | Köseoğlu VK, Heiss C, Azadi P, et al. Listeria monocytogenes exopolysaccharide: origin, structure, biosynthetic machinery and c-di-GMP-dependent regulation[J]. Mol Microbiol, 2015, 96(4): 728-43. DOI: 10.1111/mmi.12966. |
 2017, Vol. 37
2017, Vol. 37

