2. 公安部物证鉴定中心//现场物证溯源国家工程实验室//法医遗传学公安部重点实验室,北京 100038;
3. 广州市刑事科学技术研究所//广东省法医遗传学重点实验室,广东 广州 510030;
4. 天津医科大学,天津 300070
2. National Engineering Laboratory for Crime Scene Evidence Examination, Key Laboratory of Forensic Genetics of Ministry of Public Security, Institute of Forensic Science, Beijing 100038, China;
3. Guangzhou Institute of Criminal Science and Technology/Key Laboratory of Forensic Pathology of Ministry of Public Security, Guangzhou 510030, China;
4. Tianjin Medical University, Tianjin 300070, China
祖先信息位点(AIMs)指在不同人群之间等位基因频率差异非常大的多态性遗传位点,是在法医学中推断未比中来源人DNA样品种族来源的一类有效标记[1]。单核苷酸多态性(Single nucleotide polymorphisms, SNPs)位点因数量多、人群间基因频率差异显著,是目前筛选AIMs的一种重要遗传标记[2]。目前有研究应用基因芯片或二代测序技术检测上百个AIM-SNPs进行种族来源推断[3-4],但基因芯片、二代测序检测平台均未在目前的法医实验室普及,且技术也有待优化和证实。基于数据库的方法建立的复合扩增体系也见之于报道[5]。国外报道了基于毛细管电泳检测平台的AIM-SNPs复合扩增系统[6-9],例如Zeng等[7]建立23重祖先SNP复合扩增系统,用于区分美国主要人群。然而,目前尚无此类型的商品化试剂盒。因此我国法医遗传学公安部重点实验室近年来建立27重SNP系统,该系统可为目前普及的毛细管电泳平台检测。Wei等[10]首次报道了27重SNP系统,表明该系统可区分非、欧、东亚祖先成分,并报道了塔吉克族、维吾尔族、哈萨克族、柯尔克族等人群的欧亚成分比例。刘海渤等[11]应用27重SNP系统对195名维吾尔族个体进行研究,发现该系统除1例户籍显示维吾尔族的个体被推断为东亚人群,其余均被准确分析为欧亚混合人种。魏丽等[12]应用27重SNP系统对新疆塔吉克族、维吾尔族、锡伯族、哈萨克族、柯尔克族等人群进行研究,发现新疆上述人群的遗传成分呈现在欧洲与东亚祖先之间连续分布。可见,目前已有的报道都着重于27重SNP系统用于新疆人群的研究。
广东省外来流动人员多,因此将27重SNP系统用于特殊案件现场样本人群成分分析的需求非常迫切。为了将27重SNP系统推广用于广东省的法医实践工作,本研究采用该系统对在广东地区收集的欧洲、非洲、南美洲、中亚、西亚、南亚、东亚、东南亚以及新疆维吾尔族、甘肃回族、广西彝族、广西苗族、西藏藏族等13个不同祖先人群的533份样本进行检测,评估了该系统推断上述人群种族来源的效能。
1 材料和方法 1.1 材料根据知情同意原则,收集13个人群共533份无关个体的血FTA卡/口腔FTA卡/口腔拭子/血滤纸,选择HapMap数据库的东亚(CHB)、欧洲(CEU)、非洲(YRI)3个代表人群的分型数据作为参照,人群样本信息见表 1。盲测样本46份,由广州市刑事技术研究所、公安部物证鉴定中心、南方医科大学司法鉴定中心提供。用于种属特异性检测的样本(包括黑猩猩、恒河猴、鸡、鸭、羊、猪、猫、狗、鼠、兔、和鱼等动物血样,)由广州市刑事技术研究所提供。
| 表 1 人群样本信息 Table 1 Information of the world wide population samples |
NanoDrop 2000c分光光度计(美国ThermoFisher Scientific公司)、9700PCR仪和3130xl基因分析仪(美国ABI公司)。MagAttract® DNA Mini M48试剂盒(德国Qiagen),SNaPshot Multiplex试剂盒、GeneScan-120 Liz Size Standard、POP7胶、甲酰胺(美国Life technologies)、核酸外切酶Ⅰ(ExoⅠ, 10 U/μL)和虾碱性磷酸酶(SAP)(美国USB)。
1.2 方法 1.2.1 DNA提取、PCR、毛细管电泳DNA提取MagAttract® DNA Mini M48试剂盒提取DNA,NanoDrop 2000c分光光度计进行DNA定量。以去离子灭菌水调整浓度至5~10 ng/μL。
PCR使用SNaPshot Multiplex试剂盒5 μL反应体系,反应程序95 ℃ 5 min后,94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,共40个循环。加入ExoⅠ(10 U/μL)0.2 μL、SAP(1 U/μL)1 μL、10×SAP buffer 0.3 μL,37 ℃ 60 min,96 ℃ 10 min进行PCR产物纯化。纯化后的产物进行延伸反应,延伸程序为96 ℃ 10 s,50 ℃ 5 s,60 ℃ 30 s,25个循环;延伸产物再次纯化。
毛细管电泳在3100xl基因分析仪,使用GeneScan-120 Liz Size Standard内标、Pop7凝胶,36 cm毛细管进行电泳,Genemapper 3.2软件进行基因分型。
1.2.2 灵敏度试验将10 ng/μL的标准品2800 M进行倍比稀释,获得5、1、0.5、0.25、0.125、0.0625、0.0313、0.0156和0.0078 ng/μL等9种浓度梯度,在相同条件下分别用27重SNP系统扩增和电泳检测,重复3次。
1.2.3 种属特异性试验取黑猩猩、恒河猴、鸡、鸭、羊、猪、猫、狗、鼠、兔和鱼等动物血样,提取DNA,分别用27重SNP系统检测并重复3次。
1.2.4 盲测试验用27重SNP系统对46份样本检测,获得的分型数据用Structure软件进行聚类分析并结合PI软件计算的MP值判断样本来源。通过对比已知的样本信息,验证该系统种族推断的准确性。
1.2.5 人群数据分析根据研究人群获得的SNP分型结果,采用Genepop 4.2软件计算Fst值和Hardy-Weinberg平衡[13];STRUCTURE 2.3.4软件进行人群遗传成分分析[14-15];Distruct1.1软件绘制人群的聚类结果图[16];Past3.0软件进行主成分分析[17];Mega 6.0软件绘制系统发生树(www.megasoftware.net);Forensic Intelligence(FI version 1.0, available online:http://github.com/jiang11989/FI)计算群体匹配概率(MP)值。
2 结果 2.1 灵敏度试验3次重复实验结果均显示DNA模板≥0.125 ng时,27个SNP基因座均检出;DNA模板≤0.0625 ng,部分等位基因峰值过低或缺失。
2.2 种属特异性试验3次重复实验结果均显示猩猩和恒河猴分别检出20和6个位点。除了鱼和阴性对照,其余动物样本在4号位点上均检出4T分型,其他26个位点未见特异性产物(图 1)。
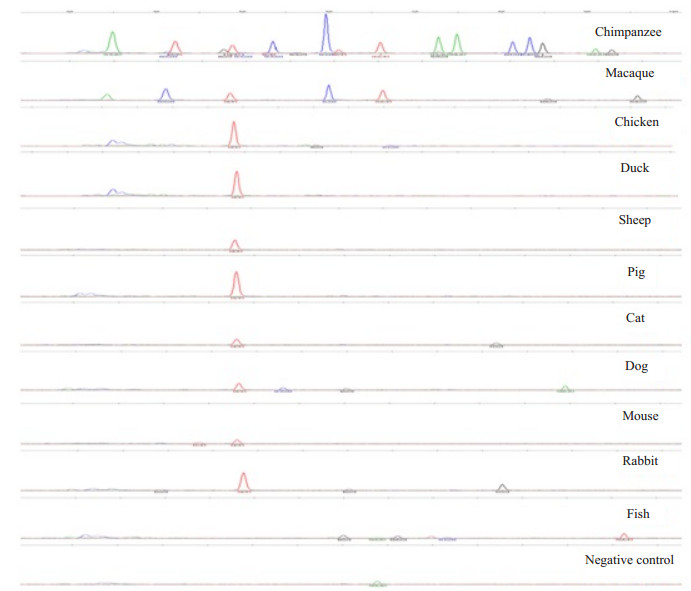
|
图 1 种属特异性电泳图谱 Figure 1 Profiles of electrophoresis for species-specific studies. |
46份盲测样本基于MP值推断的祖先来源类型结果与STRUCTURE软件进行祖先成分分析的结果一致,且所有样本人群类型结果均与样本信息完全一致(表 2)。盲测样本种族推断准确率达到100%。
| 表 2 盲测样本祖先来源分析结果 Table 2 Ancestral origin identification of samples for blind test |
27个SNPs位点信息以及根据16个人群基因分型结果计算获得的Fst值见表 3。27个位点在16个人群中的基因型分布均符合Hardy-Weinberg平衡(经Bonferroni校正,P>0.00001)。
| 表 3 27个SNPs位点信息及根据16个人群计算获得的Fst值 Table 3 Information of the 27 AIMs (Fst based on 16 population) |
用FI软件计算每个人群祖先成分的匹配概率,推断出其最可能的祖先起源。根据MP值,AFR和YRI等人群所有个体被归入非洲人群,EUR和CEU等人群个体均被判别为欧洲人群,SEA、GSH、GXM、GXY、XZZ、CHB和CHG等人群所有个体均被归入东亚人群。AMR、WAS、CAS、SAS、CUX等混合人群大多数样本归类到混合人群中(表 4)。
| 表 4 16个人群个体被归类到4种祖先人群的比例 Table 4 Proportions of individuals from16 populations assigned to 4 ancestry groups |
根据16个人群两两之间的Fst值绘制系统发生树(图 2)。AFR和YRI作为非洲人群代表自成一支,其余14个人群归为另外一支。东亚人群GXY、CHB、GXM、CHG、XZZ、SEA和GSH聚集一支,CUX与其他东亚人群遗传距离较远;CAS、AMR、EUR、CEU、WAS和SAS归为一支,其中EUR、CEU、WAS和SAS聚集。
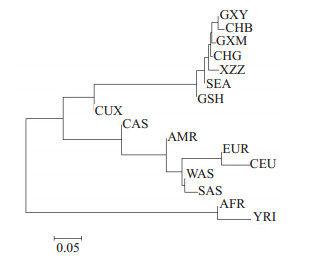
|
图 2 16个人群基于配对Fst构建的系统发生树 Figure 2 Phylogenetic tree constructed based on the pairwise Fst for 16 populations. |
主成分分析结果(图 3)显示,主成分1和主成分2合计占91.33%,16个人群的散点形成了4个大的聚类。左上为欧洲人群,左下为非洲人群,右侧为东亚人群,中亚CAS和维族CUX在东亚和欧洲人群之间。SAS,WAS,AMR与欧洲代表人群CEU和EUR聚在一起。
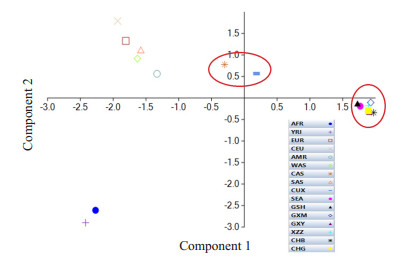
|
图 3 16个人群基于27个祖先信息位点的主成分分析结果 Figure 3 Principal component analysis plot based on 27 AIMs loci for 16 world wide populations. |
16个人群的843份分型数据通过STRUCTURE(K=3)进行人群结构分析,显示上述人群样本主要聚成4类:非洲人群、欧洲人群、欧洲-东亚混合人群、东亚人群(图 4)。
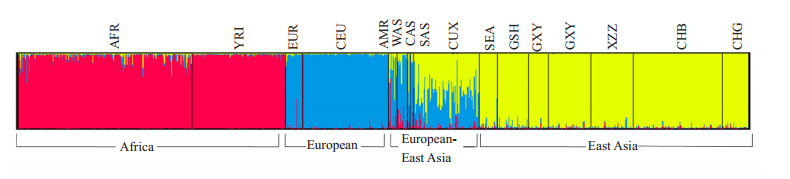
|
图 4 27个AIMs在16个人群的STRUCTURE聚类分析结果 Figure 4 Clustering analysis of 16 populations by STRUCTURE for 27 AIMs (assuming K=3). |
16个人群中非洲(AFR、YRI)、欧洲(EUR、CEU)和东亚(SEA、GSH、GXM、GXY、XZZ、CHB和CHG)均根据其祖先主成分大于0.9正确地聚类。两个中亚人群CUX和CAS则表现出明显的欧亚混合成分(分别为43.3%/53.9%和50.8%/43.2%),西亚(WAS)和南亚(SAS)的欧洲成分大于80%。南美洲(AMR)人群较为特殊,非、欧、东亚3种成分分别为14.8%、66.4%和18.8%(表 5)。
| 表 5 16个世界人群的27个AIMs在K=3条件下聚类分析结果 Table 5 Structure analysis of 16 worldwide populations using the 27 AIMs at K=3 |
27重SNP系统包含8个非洲AIMs位点、9个欧洲AIMs位点和10个东亚AIMs位点。Fst>0.40的位点被认为具有人群特异性[10],本研究显示上述非洲位点、欧洲位点和东亚位点的Fst平均值分别为0.718,0.407和0.535,该系统对16个人群有较好的聚类分析能力。
本研究表明该系统的灵敏度达0.125 ng,表明该系统可用于微量检材。种属特异性试验结果显示,除了鱼和阴性对照,几乎所有的动物样本均在4号位点(rs10496971)检出4T分型,提示4号位点的种属特异性不理想。其他位点的种属特异性都较好,可避免动物样本误导侦查方向。
本研究应用FI软件计算每个人群祖先成分的匹配概率,推断出其最可能的祖先起源,结果显示欧洲、非洲、东亚人群的推断准确率均为100%,混合人群中各群体所占比例反映出相应研究人群中的祖先来源组成。46份盲测样本经MP值和祖先成分分析获得的祖先来源结果,均与样本信息一致,证实该系统可准确分析上述样本的人群成分。
本研究中16个人群的系统发生树、主成分分析及人群结构分析均显示该系统能对非洲、欧洲、东亚、欧亚混合人群分别聚类和区分。本研究中1例样本的非洲、欧洲和东亚成分分别为64.4%、21.0%和14.6%,祖先成分与匹配概率分析均将其推测为非洲人群,而样本信息记录的国籍为哥伦比亚。据Bushnell [18]的报道,86%的哥伦比亚人是白种人和印第安-拉丁民族的混血人群,10%为非洲人群。曾祥培等[5]用其构建的23AIMs位点对94份哥伦比亚人祖先来源进行推断,其中4份被归为非裔美国人。另1例样本的欧洲和东亚成分分别为55.7%和43.6%,本系统检测结果将其归为欧-东亚混合人群,样本信息记录国籍为柬埔寨。而同为柬埔寨的另一个样本则表现出明显的东亚成分(98.4%)。在本例中,structure结果与匹配概率结果是一致的,因此出现该结果的原因可能与样本来源有关。
本研究用27重SNP系统获得新疆维吾尔族人的欧亚成分比例为43%/54%。新疆地处亚欧大陆中部,维吾尔族人群具有东西方人群混合的外貌特征,全基因组分析结果显示其为典型的欧洲/东亚混合祖先,两者的成分非常接近[19],本研究结果与之一致。本研究中同处于中亚的乌兹别克斯坦人也表现出欧亚混合特征,匹配概率分析将其全部归为欧-亚混合人群,欧亚成分比例为51%/43%,较之于新疆维吾尔族有着更高的欧洲成分。本研究中西亚和南亚个体被归入混合人群,但以欧洲成分为主(约为80%),高于中亚人群的欧洲成分。
本研究显示该系统将东南亚人群归为东亚人种,但其东亚祖先成分约为94%,低于广东汉族、北京汉族、西藏藏族、广西彝族和苗族的东亚祖先成分(约98%)。甘肃回族显示欧洲成分稍高,东亚祖先成分(96%)也稍低于上述国内人群。该系统无法细分汉族与彝族、回族、苗族和藏族等少数民族群体,这是由于该系统遗传标记筛选时针对的是洲际人群,未选择针对亚人群区分的位点。Li等[20]筛选了74-SNP panel,可区分更近地理位置的人群,比如区分东亚人群和东南亚人群。因此,筛选深度区分的祖先信息位点以及适应于中国群体亚群分析的生物地理祖先信息遗传标记、构建可为毛细管电泳平台检测的AIM-SNPs复合扩增系统是后续研究的方向。
综上,27重SNP系统可对非洲、欧洲、东亚人群、欧洲/东亚混合人群有较好的推断能力,可用于日益增多的涉外案件中个体群体来源的推断。本研究样本除包括非洲、欧洲和东亚等代表人群外,还包括西亚、中亚、南亚、东南亚、南美洲和中国五个少数民族的人群样本,覆盖35个国家和地区,其中部分国家和地区27个AIMs人群数据和祖先成分分析为首次报道。所获得的SNPs多态性信息,为医学遗传学相关研究和法医鉴定实践提供了重要的人群基础数据。
| [1] | Phillips C, Parson W, Lundsberg B, et al. Building a forensic ancestry panel from the ground up: The EUROFORGEN Global AIM-SNP set[J]. Forensic Sci Int Genet, 2014, 11 : 13-25. DOI: 10.1016/j.fsigen.2014.02.012. |
| [2] | Halder I, Shriver M, Thomas M, et al. A panel of ancestry informative markers for estimating individual biogeographical ancestry and admixture from four continents: Utility and applications[J]. Hum Mutat, 2008, 29 (5): 648-58. DOI: 10.1002/humu.20695. |
| [3] | Kidd JR, Friedlaender FR, Speed WC, et al. Analyses of a set of 128 ancestry informative single-nucleotide polymorphisms in a global set of 119 population samples[J]. Investig Genet, 2011, 2 (1): 1-13. DOI: 10.1186/2041-2223-2-1. |
| [4] | Themudo GE, Mogensen HS, Borsting C, et al. Frequencies of HID-ion ampliseq ancestry panel markers among greenlanders[J]. Forensic Sci Int Genet, 2016, 24 : 60-4. DOI: 10.1016/j.fsigen.2016.06.001. |
| [5] | Zeng XP, Chakraborty R, King JL, et al. Selection of highly informative SNP markers for population affiliation of major US populations[J]. Int J Legal Med, 2016, 130 (2): 341-52. DOI: 10.1007/s00414-015-1297-9. |
| [6] | Fondevila M, Phillips C, Santos C, et al. Revision of the SNPforID 34-plex forensic ancestry test: Assay enhancements, standard reference sample genotypes and extended population studies[J]. Forensic Sci Int Genet, 2013, 7 (1): 63-74. DOI: 10.1016/j.fsigen.2012.06.007. |
| [7] | Zeng XP, Warshauer DH, King JL, et al. Empirical testing of a 23-AIMs panel of SNPs for ancestry evaluations in four major US populations[J]. Int J Legal Med, 2016, 130 (4): 891-6. DOI: 10.1007/s00414-016-1333-4. |
| [8] | De La Puente M, Santos C, Fondevila M, et al. The global AIMs Nano set: a 31-plex SNaPshot assay of ancestry-informative SNPs[J]. Forensic Sci Int Genet, 2016, 22 : 81-8. DOI: 10.1016/j.fsigen.2016.01.015. |
| [9] | Santos C, Fondevila M, Ballard D, et al. Forensic ancestry analysis with two capillary electrophoresis ancestry informative marker (AIM) panels: Results of a collaborative EDNAP exercise[J]. Forensic Sci Int Genet, 2015, 19 : 56-67. DOI: 10.1016/j.fsigen.2015.06.004. |
| [10] | Wei YL, Wei L, Zhao L, et al. A single-tube 27-plex SNP assay for estimating individual ancestry and admixture from three continents[J]. Int J Legal Med, 2016, 130 (1): 27-37. DOI: 10.1007/s00414-015-1183-5. |
| [11] | 刘海渤, 孙启凡, 陈健刚, 等. 27重SNP种族推断体系准确性验证研究[J]. 中国法医学杂志, 2015, 30 (6): 563-6. |
| [12] | 魏丽, 魏以梁, 江丽, 等. 27-plex SNPs复合扩增检测体系构建与应用评价[J]. 中国法医学杂志, 2016, 31 (1): 13-7. |
| [13] | Rousset F. Genepop'007: a complete re-implementation of the genepop software for Windows and linux[J]. Mol Ecol Resour, 2008, 8 (1): 103-6. DOI: 10.1111/j.1471-8286.2007.01931.x. |
| [14] | Pritchard JP. Inference of population structure using multilocus genotype data[J]. Genetics, 2000, 2 (155): 945-59. |
| [15] | Falush D, Stephens M, Pritchard JK. Inference of population structure using multilocus genotype data: linked loci and correlated allele frequencies[J]. Genetics, 2003, 164 (4): 1567-87. |
| [16] | Rosenberg N. Distruct:a program for the graphical display of population structure[J]. Mol Ecol Notes, 2004, 4 (1): 137-8. |
| [17] | Hammer Ø, Harper D, Ryan PP. Paleontological statistics software package for education and data analysis[Z], 2001. |
| [18] | Bushnell D H R. Colombia: a country study[M]. Federal Research Division, Library of Congress, Washingtion D.C: 2010. |
| [19] | S. Xu L J. A genome-wide analysis of admixture in Uyghurs and a high-density admixture map for disease-gene discovery[J]. Genet, 2008, 83 (3): 322-36. |
| [20] | Li CX, Pakstis AJ, Jiang L, et al. A panel of 74 AISNPs: Improved ancestry inference within Eastern Asia[J]. Forensic Sci Int Genet, 2016, 23 : 101-10. DOI: 10.1016/j.fsigen.2016.04.002. |
 2017, Vol. 37
2017, Vol. 37

