2. 广东省东莞市康华医院,广东 东莞 523000
2. Kanghua Hospital of Dongguan, Dongguan 523000, China
死亡相关蛋白激酶(DAPK)基因是Adikimchi于1995年在基因组中扫描启动细胞凋亡的基因和抑癌基因时发现的,是一种钙调蛋白调节的丝氨酸/苏氨酸蛋白激酶,在凋亡途径中起重要作用[1-2]。研究发现在大部分恶性肿瘤中,如头颈部肿瘤、乳腺癌、非小细胞肺癌、胃癌等[3-6],DAPK基因表达水平低或不表达可能与其启动子区CpG岛甲基化有关,从而导致肿瘤发生发展。目前国内外关于急性白血病患者DAPK基因甲基化相关研究较少,而且研究结果各家报道不一[7-11],甲基化阳性率结果相差较大。为研究DAPK基因与急性白血病的关系,我们采用MSP和RT-PCR方法检测DAPK基因在急性白血病患者中的甲基化状态及其mRNA的表达,研究DAPK基因甲基化及基因表达的临床意义,为白血病基因靶向治疗提供证据。
1 资料和方法 1.1 一般资料骨髓标本均来广西医科大学第一附属医院血液科住院收治初诊的患者。诊断均符合急性白血病FAB分型诊断标准和EGIL免疫分型诊断标准。共115例,男61例,女54例,急性髓系白血病(AML)60例,急性淋巴细胞白血病(ALL)55例。AML患者中位数年龄35(15~68)岁;按FAB分型,AML-M1型5例;AML-M2型26例,AML-M3型14例,AML-M4型6例,AML-M5型6例,AML-M6型3例。ALL患者中位数年龄36(15~65)岁;按FAB分型,L1型23例,L2型29例,L3型3例;免疫分型分为B细胞型46例,T细胞型9例。全部患者在初次治疗前留取骨髓单个核细胞,其中93例随访观察1年。17例正常人骨髓作为对照。
1.2 仪器与试剂Ficoll淋巴细胞分离液、TE液(上海生化试剂厂);冰乙酸、氯仿、异丙醇、无水乙醇为国产分析纯;DNA碱基修饰试剂盒(CpGenomeTMDNA Modification Kit,chemicon, S7820);总RNA提取试剂TRIzol、逆转录试剂盒、Ex Taq DNA聚合酶、hot start Taq DNA聚合酶、DNA Marker以及溴化乙锭(EB)、6 × loading buffer(TaKaRa);PCR所涉及引物均由上海生物工程公司合成。试验所用仪器有:低温高速离心机(Sigma)、紫外分光光度仪(日本HITACHI)PCR仪(英国Cambridge)、凝胶扫描仪(美国Kodak)。
1.3 方法 1.3.1 骨髓单个核细胞的提取无菌抽取病人的骨髓4 mL,加入预冷PBS液,充分混匀后加到Ficoll淋巴细胞分离液上方,离心后吸出单个核细胞层分装。
1.3.2 DNA提取及修饰、DAPK基因的甲基化特异PCR(MSP)检测运用酚/氯仿方法常规提取细胞DNA,运用DNA碱基修饰试剂盒对DNA进行修饰纯化,产物进行MSP实验。DAPK基因启动子上下游引物按参考文献[1]设计。甲基化上游引物序列MSF:5'-GGATAGTCGGATCGAGTTAACGTC-3';下游引物序列MSR:5'-CCCTCCCAAACGCCGA-3',目的片段大小为98 bp;非甲基化上游引物序列UMF:5'-GGAGGATAGTTGGATTGAGTTAATGTT-3';下游引物序列UMR 5'-CAAATCCCTCCCAAACACCAA-3',目的片段大小为106 bp。扩增产物在2%的琼脂糖凝胶上电泳40 min,凝胶扫描仪成像。结果判定:甲基化特异引物扩增出目的条带为98 bp,而非甲基化特异引物无条带扩出,称为完全甲基化;非甲基化特异引物扩增出目的条带为106 bp,而甲基化特异引物无条带扩出,称为非甲基化;两对引物均扩增出目的条带,称为部分甲基化。完全甲基化和部分甲基化均称为甲基化。
1.3.3 RNA提取及DAPK mRNA的RT-PCR检测细胞按TRIzoL试剂说明书提取细胞RNA,逆转录参照逆转录酶说明书。反应引物设计与合成如下,并经GenBank验证。DAPK基因的上游引物序列:5;ATCCTAGACGTGGTCCGGTAT-3'下游引物序列:5'-GTTCTCGCAGCCTGGGTA-3',目的片段大小是402 bp;看家基因GAPDH上游引物序列5'-CGGGAAACTGTGGCGTGAT-3',下游引物序列5'-TGGCAACTGTGAGGAGGG-3'目的片段大小是553 bp。PCR条件:预变性,95 ℃,5 min;变性,94 ℃,40 s;复性62 ℃,30 s;延伸72 ℃,30 s;循环30次。产物在2%琼脂糖凝胶电泳上电泳40 min,紫外灯下观察,凝胶扫描仪成像分析,以DAPK及GAPDH两基因扩增产物的光密度比值表示DAPK基因mRNA的表达水平,采用quantityone软件进行半定量分析。
1.4 统计学方法采用SPSS17.0统计软件包进行分析,率的比较采用χ2检验,生存期比较用log-rank检验。相关性采用Spearman等级相关分析。计量资料呈非正态分布,Kruskal-Wallis H检验统计分析2组间和3组间的差异。P<0.05为差异有统计学意义。多组间χ2检验的两两比较P<0.0125为差异有统计学意义。
2 结果 2.1 DAPK基因MSP的检测DAPK基因在17例正常人骨髓细胞中未检测到启动子甲基化。AML患者骨髓细胞中甲基化阳性率为5%(3/60),ALL患者骨髓细胞中甲基化阳性率为29.1%(16/55),3组之间有显著差异(χ2=16.797,P=0.000)。用χ2分割法进一步两两比较,ALL组的DAPK启动子甲基化阳性率高于AML组和正常组(χ2=12.075,6.358,P<0.0125)。在ALL患者中,患者的年龄,性别,外周血白细胞数,骨髓原始细胞比例,FAB分型,免疫分型与DAPK基因甲基化均无关(P均>0.05),DAPK启动子甲基化阳性组和阴性组的生存期经log-rank检验比较无差异(χ2=0.501,P=0.479,图 1)。
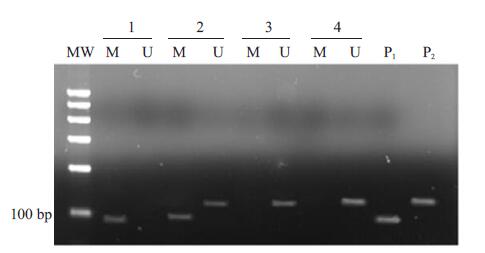
|
图 1 AL患者和正常人DAPK基因的MS-PCR Figure 1 Methylation-specific PCR of DAPK gene methylation in acute leukemia patients and normal subjects. MW: DNA marker; M: Methylated sequence-specific PCR for DAPK; U: Unmethylated sequence-specific PCR for DAPK; P1: Methylated positive control (SssI-treated DNA); P2: Unmethylated positive control (SssI-untreated DNA); Lanes 1-3: Results in acute leukemia patients; Lane4: Results of a normal subject. |
DAPK mRNA在正常人的表达阳性率是76.5%(13/17),AML组阳性率为61.7%(37/60),ALL组阳性率为47.3%(26/55),3组之间无统计学差异(χ2=5.435,P=0.066)。而3组DAPK mRNA的表达水平经Kruskal-Wallis H检验有显著差异(χ2=15.834,P=0.000),以正常组最高,为0.3144(0.2768~0.3356),AML组的表达水平次之,为0.2047(0.0687~0.3847)。ALL组最低,为0.1345(0.0876~0.3526)。且无论是AML组还是ALL组,患者的年龄,性别,外周血白细胞数,骨髓原始细胞比例,FAB分型,免疫分型与DAPK基因表达均无关(P>0.05,图 2)。
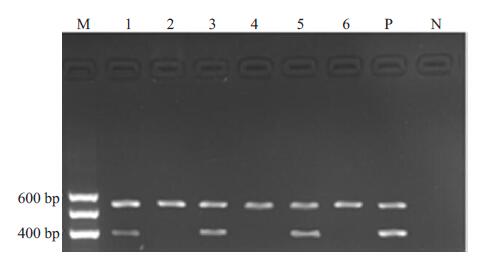
|
图 2 AL患者和正常人DAPK mRNA的表达 Figure 2 RT-PCR analysis of DAPK mRNA expression in acute leukemia patients and normal subjects. M: DNA marker; Lane 1: An ALL patient with positive expression; Lane 2: An ALL patient with negative expression; Lane 3: An AML patient with positive expression; Lane 4:An AML patient with negative expression; Lane 5: A normal subject with positive expression; Lane 6: A normal subject with negative expression; P: Positive control; N: H2O. |
将ALL患者中DAPK mRNA表达水平与正常人其表达水平的中位数进行比较,界定ALL患者表达水平减少50%以上视为表达下调,完全不表达为缺失。经spearman分析ALL患者骨髓细胞DAPK mRNA表达与其启动子甲基化呈显著负相关(rs=-0.347,P<0.05,表 1)。
| 表 1 ALL患者DAPK mRNA表达与其启动子甲基化的关系 Table 1 Correlation of DAPK gene methylation with its mRNAexpression in ALL patients (n, %) |
DNA甲基化修饰异常在急性白血病发生过程中是一个早期的普遍现象,是其重要发病机制之一。特别是抑癌基因的CpG岛甲基化问题近年来愈来愈受到重视。DNA的甲基化是指在DNA甲基转移酶的作用下,哺乳动物基因组DNA的胞嘧啶脱氧核苷酸(C)5'位发生甲基化修饰反应。甲基化改变只影响DNA的结构,而DNA的序列并不发生变化。DAPK基因广泛参与多种途径介导的细胞凋亡,被认为是肿瘤的抑癌基因[1-2]。近来研究发现它在多种肿瘤中出现DNA甲基化,最终导致表达下降甚至沉默,肿瘤细胞无节制的生长。
目前DAPK基因启动子甲基化与ALL的发生发展是否相关仍存在争议。Roman-Gomez等[9]研究95例ALL患者,DAPK基因启动子甲基化为13%,这与Takeuchi等[10]报道相似。本研究运用MSP方法发现,29.1%(16/55)的ALL患者出现DAPK基因甲基化,且ALL患者的临床特征及预后与甲基化均无关。而在ALL患者中均未检测到DAPK基因启动子甲基化发生,是否与预后有关还需更多病例证实[7]。
本研究发现在AML患者中DAPK基因甲基化率为5%(3/60),而国内外关于AML患者DAPK基因甲基化阳性率各家报道不一:0%(0/60)[8],0.4%(1/246)[11]。不同结果的是:22.9%(30/131)[12]、61%(17/28)[13]、17%(7/45)[7]。运用MSP技术检测白血病患者DAPK基因甲基化,对区分急性髓系白血病与其他白血病类型,其敏感性、特异性和准确性分别为59.1%、100%和82.7%[14]。我们推论检测DAPK基因甲基化在急性髓系白血病造成结果相差较大的原因是否与人种、地域及实验条件差异有关,还需要进一步扩大样本量来证实。
我们的实验结果显示,AML与ALL患者相比,AML患者DAPK mRNA表达水平增高,但是却低于正常人。且AL患者的临床特征及预后与DAPK基因表达无关。应用含有406个基因的cDNA芯片分别研究了15例AML患者和5例健康供者的骨髓细胞基因表达谱,结果发现只有在白细胞>100×109/L的患者中DAPK显著高表达[15]。但我们的实验并未得出类似结果。在ALL患者中,相关分析显示DAPK mRNA表达与其启动子甲基化呈显著负相关(rs=-0.347,P<0.05),提示DAPK基因表达水平低或不表达可能与其启动子区CpG岛甲基化有关,这结果这与大部分实体瘤的研究报道一致。
综上所述,DAPK作为一种抑癌基因,其启动子甲基化、表达的机制及其如何被调节、它与急性白血病发生发展间的确切关系等还需要更深入的研究。
| [1] | Katzenellenbogen RA, Baylin SB, Herman JG. Hypermethylation of the DAP-kinase CpG island is a common alteration in B-cell malignancies[J]. Blood, 1999, 93 (12): 4347-53. |
| [2] | Chen CH, Wang WJ, Kuo JC, et al. Bidirectional signals transduced by DAPK-ERK interaction promote the apoptotic effect of DAPK[J]. EMBO J, 2005, 24 (2): 294-304. DOI: 10.1038/sj.emboj.7600510. |
| [3] | Paluszczak J, Misiak P, Wierzbicka M, et al. Frequent hypermethylation of DAPK, RARbeta, MGMT, RASSF1A and FHIT in laryngeal squamous cell carcinomas and adjacent normal mucosa[J]. Oral Oncol, 2011, 47 (2): 104-7. DOI: 10.1016/j.oraloncology.2010.11.006. |
| [4] | Van der Auwera I, Bovie C, Svensson C, et al. Quantitative methylation profiling in tumor and matched morphologically normal tissues from breast cancer patients[J]. BMC Cancer, 2010, 10 : 97. DOI: 10.1186/1471-2407-10-97. |
| [5] | Li Y, Zhu M, Zhang X, et al. Clinical significance of DAPK promoter hypermethylation in lung cancer: a meta-analysis[J]. Drug Des Devel Ther, 2015, 9 : 1785-96. |
| [6] | Qu Y, Dang S, Hou P. Gene methylation in gastric cancer[J]. Clin Chim Acta, 2013, 424 : 53-65. DOI: 10.1016/j.cca.2013.05.002. |
| [7] | Bodoor K, Haddad Y, Alkhateeb A, et al. DNA hypermethylation of cell cycle (p15 and p16) and apoptotic (p14, p53, DAPK and TMS1) genes in peripheral blood of leukemia patients[J]. Asian Pac J Cancer Prev, 2014, 15 (1): 75-84. DOI: 10.7314/APJCP.2014.15.1.75. |
| [8] | Ng HY, Wan TS, So CC, et al. Epigenetic inactivation of DAPK1, p14ARF, mir-34a and-34b/c in acute promyelocytic leukaemia[J]. J Clin Pathol, 2014, 67 (7): 626-31. DOI: 10.1136/jclinpath-2014-202276. |
| [9] | Roman-Gomez J, Jimenez-Velasco A, Castillejo JA, et al. Promoter hypermethylation of cancer-related genes: a strong Independent prognostic factor in acute lymphoblastic leukemia[J]. Blood, 2004, 104 (8): 2492-8. DOI: 10.1182/blood-2004-03-0954. |
| [10] | Takeuchi S, Matsushita M, Zimmermann M, et al. Clinical significance of aberrant DNA methylation in childhood acute lymphoblastic leukemia[J]. Leuk Res, 2011, 35 (10): 1345-9. DOI: 10.1016/j.leukres.2011.04.015. |
| [11] | Claus R, Hackanson B, Poetsch AR, et al. Quantitative analyses of DAPK1 methylation in AML and MDS[J]. Int J Cancer, 2012, 131 (2): E138-42. DOI: 10.1002/ijc.26429. |
| [12] | Voso MT, Scardocci A, Guidi F, et al. Aberrant methylation of DAP-kinase in therapy-related acute myeloid leukemia and myelodysplastic syndromes[J]. Blood, 2004, 103 (2): 698-700. DOI: 10.1182/blood-2003-07-2249. |
| [13] | Ekmekci CG, Gutirrez MI, Siraj AK, et al. Aberrant methylation of multiple tumor suppressor genes in acute myeloid leukemia[J]. Am J Hematol, 2004, 77 (3): 233-40. DOI: 10.1002/ajh.v77:3. |
| [14] | Ye SS, Liu XJ, Mao BY, et al. Value of DAPK gene methylation patterns in the diagnosis of leukemia[J]. Zhongguo Shi Yan Xue Ye Xue Za Zhi, 2016, 24 (3): 687-92. |
| [15] | Larramendy ML, Niini T, Elonen E, et al. Overexpression of translocation-associated fusion genes of FGFRI, MYC, NPMI, and DEK, but absence of the translocations in acute myeloid leukemia. A microarray analysis[J]. Haematologica, 2002, 87 (6): 569-77. |
 2017, Vol. 37
2017, Vol. 37
