Brugada 综合征(Brugada syndrome,BrS)是一种常染色体显性遗传性致心律失常疾病,典型心电图表现为右胸导联(V1~V3)ST段抬高≥2 mm与T 波倒置,而心脏结构正常,易罹患多形室性心动过速、心室颤动和心脏性猝死[1-3]。BrS猝死患者人数每年占猝死总数的4%~12%,占无器质性心脏病猝死总数的20%[4]。其病因认为与基因突变有关。迄今为止,22个基因已经被证实与Brugada综合征有关[4-5],但只有30%~35%的病人确定有致病突变基因[5],遗传背景包括基因突变导致钠离子、钙离子、钾离子等通道功能障碍;其中编码心脏钠离子通道α 亚基的SCN5A 突变占20%~30%[4, 6-8]。SCN5A基因突变可通过延迟Na+通道激活、加快失活、延缓复活或者减少通道细胞膜表达水平损坏Na+通道,明显减少内向Na+电流[9-10]。所有的这些机制都影响心脏Na+通道的功能,内向Na+电流减少会影响心脏动作电位的去极化和复极化。然而,BrS表型潜在的病理生理机制尚存争议[2]。为进一步提供BrS临床诊断和遗传背景机制而进行了本次研究。
先前的研究,首次在中国人无症状BrS患者中发现了SCN5A基因一个新的碱基变异:第1712位密码子的第1个碱基的G→T改变,导致相应编码的甘氨酸(G)被半胱氨酸(C)所取代[11]。为进一步研究中国人BrSSCN5A基因新突变G1712C细胞电生理机制,本研究通过应用大引物PCR定点诱变法,构建携带有基因突变的G1712C 的pRc/CMV-hH1 的表达载体,采用lipo3000脂质转染法建立稳定表达pGFP-IRES-hβ1质粒的人胚肾组织细胞(HEK)293 细胞系,并用G418 进行筛选鉴定。而后,分别做野生型的pRc/CMV-hH1(hH1)和携带有基因突变G1712C 的pRc/CMV-hH1(mhH1)的瞬时转染表达。应用全细胞膜片钳技术,分析瞬时表达的钠通道蛋白的功能。
1 材料和方法 1.1 载体与质粒重构含有正常人全长心脏电压门控钠离子通道蛋白a亚基单位基因SCN5A的cDNA片段的真核细胞表达质粒pRc/CMV-hH1 由美国Mr. Alfred L.George(Director of Vanderbilt University Division of GeneticMedicine,Nashville)赠送。菌种为Promega 公司的Bacterial Strain JM 109。对含有相关SCN5A 基因(G1712C)突变片段采用定点诱变PCR进行扩增,Age I和Sse 8387 I进行质粒酶切鉴定法构建质粒,DNA序列测定来鉴定定点诱变是否成功。
1.2 细胞培养与质粒转染HEK 293培养在含10%胎儿牛血清(FBS)和1%青霉素/链霉素Opti-Mem培养基中,细胞均置于含95%的空气和5% CO2 37 ℃恒温箱中。采用HEK293细胞作为质粒转染的细胞系,首先将Alfred L.George 教授(美国范德比尔特大学)赠送的含重组心脏钠通道β亚单位cDNA 的表达载体pGFP-IRES-hβ1 转染HEK293 细胞,形成稳定表达hβ1的细胞系,再分别瞬时转染野生型、突变型两种质粒,使用lipo3000(Invitrogen,L3000-015,USA)脂质体作为转染媒介,G418 压力筛选,绿色荧光蛋白鉴定。细胞转染后于37 ℃恒温箱中孵育48 h,然后再取生长良好的细胞分别进行膜片钳检测。
1.3 共聚焦免疫荧光显微分析 1.3.1 pcDNA3.1-GFP-SCN5A突变体质粒的构建将SCN5A突变体基因通过BamH I和EcoR I酶切位点插入到pcDNA3.1-GFP的载体中,通过酶切及测序鉴定载体构建。
1.3.2 质粒转染通过脂质体Lipo3000分别将1 μg的pcDNA3.1-GFP 和pcDNA3.1-GFP-SCN5A 突变体质粒转染到处于对数生长期HEK293细胞中,转染36 h后再荧光显微镜下观察转染效率。
1.3.3 细胞染色及拍照转染了上述质粒的细胞用4%多聚甲醛固定15 min,然后用0.5% Txiton处理通透细胞5 min,之后用DAPI对细胞核进行染色5 min。然后再激光共聚焦显微镜(leica TCS SP8)下观察绿色荧光蛋白和细胞核的分布情况。
1.4 电生理检测方法在显微镜(IX71,Olympus)下操纵微电极操纵仪(MP285,Sutter Instruments)将记录电极接用微电极拉制仪(P97,Sutter Instruments)将毛细玻璃管(BF150-86-10,Sutter Instruments)3 步拉制成记录电极。电极入水电阻为2~3MΩ。细胞外液成分(mmol/L):140 NaCl,4 KCl,1 MgCl2,2CaCl2,5 D-Glucosemonohydrate,10 HEPES,(pH=7.4,NaOH调节)。细胞内液成分(mmol/L):145 CsCl,0.1 CaCl2,2 MgCl2,10NaCl,0.5 Na2-GTP,2 Mg-ATP,1.1 EGTA,10 HEPES,(pH 7.2,CsOH调节)。在倒置显微镜下接触到细胞,给予负压抽吸,形成GGΩ封接。形成GGΩ封接后进行快速电容补偿,然后继续给予负压,吸破细胞膜,形成全细胞记录模式。然后进行慢速电容的补偿并记录膜电容及串联电阻。不给予漏电补偿。当全细胞记录的电流稳定后开始记录。所有电生理实验均在室温下进行。实验数据由EPC-10 放大器(HEKA)进行采集并储存于PatchMaster(HEKA)软件中,实验结果由PatchMaster以及IGOR Pro 6.0软件分析。
1.5 钠离子通道动力学研究的刺激方案 1.5.1 IV-曲线首先将细胞的膜电位维持在-90 mV,然后以10 mV的阶跃间隔,将细胞膜电位从-120 mV阶跃至100 mV获得。经由GRAPHpad 6.0软件绘制电流-电压曲线。
1.5.2 INa 稳态激活曲线先将细胞的膜电位维持在-90 mV,然后阶跃至-120 mV 持续200 ms,然后以5 mV 的阶跃间隔,将细胞膜电位阶跃至-80 mV 至100 mV以检测通道的激活状态。稳态激活曲线将IV-曲线换成膜电导后得到,用Boltzmann方程:
G/GMax=1/{l+exp[(V1/2-V)/KI}
进行拟合。公式中G为在膜电压v 时的膜电导,GMax为最大膜电导,V1/2为半激活电压,K为斜率因子。G=I/(V-Erev),I为膜电压V时的电流值,Erev为通过IV-曲线测得的反转电位。
1.5.3 INa稳态失活-曲线采用先将细胞的膜电位维持在-90 mV,然后以5 mV的阶跃间隔,将细胞膜电位从-120 mV阶跃至40 mV,持续1000 ms,接着将细胞膜电位阶跃至0 mV,持续50 ms用以检测钠电流。稳态失活曲线是以测试电压下记录的电流峰值与最大电流峰值的比值对条件脉冲的电压作图,再用Boltzmann方程:
I/Imax=1/{l+exp[(V- V1/2)/K]}
对曲线进行拟合。式中I为测试脉冲引出的峰值电流,Imax为测试脉冲引出的最大峰值电流,v为条件脉冲电压,V1/2为半失活电压,K为斜率因子(表 1)。
| 表 1 曲线方程参数 Table 1 Curve equation parameters |
应用SPSS 20.0软件包,计量资料以均数±标准差表示,对两组数据进行t检验,对多组数据用单因素方差分析,再行Student-Newman-Keuls检验,P<0.05为差异有统计学意义。
2 结果 2.1 载体质粒重构结果DNA 测序表明,在预期位点已经发生了突变,SCN5A基因在第1712位密码子第1个碱基的G→T改变,相应编码的氨基酸由甘氨酸(G)突变为半胱氨酸(C),成功实现定点诱变。
2.2 细胞转染结果细胞孵育48 h后,在G418培养基中生长良好,在荧光显微镜下可见绿色荧光。瞬时转染WT和G1712C的细胞生长贴壁良好,形态清楚,在微电极下钳制较为容易。
2.3 膜片钳试验结果 2.3.1 IV-曲线维持电位(EH):-90 mV,指令电位(EC)从-120 mV阶跃至100 mV,阶跃间隔为10 mV,钳制时间(TC):50 ms,经由IGOR-Pro软件绘制IV-曲线。野生型hH1 INa 阈电位为-50 mV,此后随EC增加,内向INa峰值逐渐增大,至EC-20 mV 时,INa峰值达最大值,此电位即峰值电位(Peak potential)。随后INa峰值逐渐减小,至EC + 50 mV 时,INa 峰值为0,此电位即反转电位(Reversal potential)。反转电位后INa变为外向电流。INa 2 ms 左右达峰值,10 ms 左右从峰值达稳态。EC从-60 mV 逐渐上升,钠电流也渐变大,在-20 mV 时完全激活。激活电压在-60 mV 到-50 mV,而它的反转电位在50 mV 左右,钠通道动力学特性与正常心肌细胞钠通道动力学特性类似。而突变型G1712C未能记录到钠电流(图 1)。
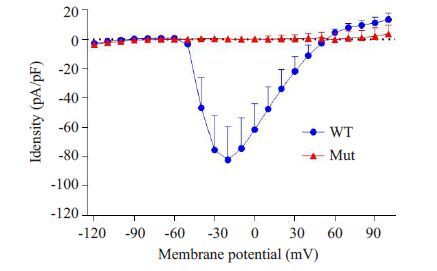
|
图 1 野生型(WT,n=7)和G1712C突变型(Mut,n=10)电流密度-电压曲线(IV-曲线) Figure 1 Current voltage plots of the INa density (IV-curve).No current was recorded in the MUT group. |
维持电位(EH):-90 mV,阶跃至-120 mV 持续200 ms,然后以5 mV的阶跃间隔,将细胞膜电位-120 mV阶跃至-80 mV,TC:50 ms;再阶跃至100 mV TC:50 ms。可见当阶跃至-50 mV 钠通道开始激活,随着条件脉冲EC向正值方向移动,钠电流渐变大,当EC至-20 mV时完全激活(图 2)。
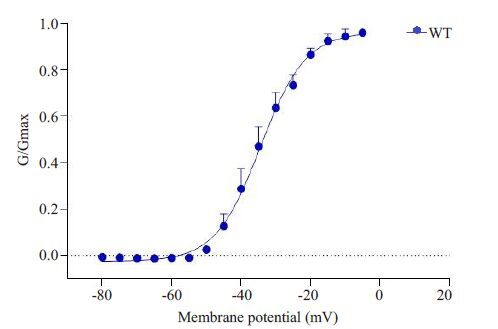
|
图 2 野生型(WT,n=7)钠通道稳态激活曲线 Figure 2 Wild-type sodium channel steady-state activationcurve (current-voltage plot). |
当条件脉冲EC向正值方向移动时,钠通道逐渐失活。条件脉冲EC达-60 mV基本失活,在-90 mV至-70 mV,钠通道基本失活。在电压-120 mV到-90 mV 时膜处于超极化状态,这时钠通道完全备用状态,没有失活现象。当电压处于-90 mV 至-70 mV时,给予小的膜电位,通道即可完全失活。当膜去极化电压至-50 mV时,钠通道将完全失活(图 3)。
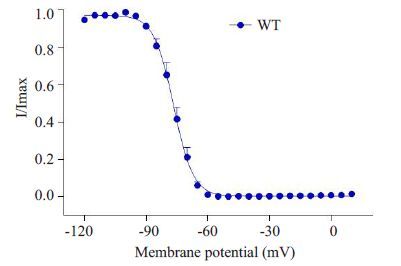
|
图 3 野生型(WT,n=7)钠通道稳态失活曲线 Figure 3 Wild-type sodium channel steady-stateinactivation curve (current voltage plot). |
本次发现的G1712C位于Na+通道蛋白α 亚单位的DⅣ区S5与S6之间,即P-loop上(图 4)。
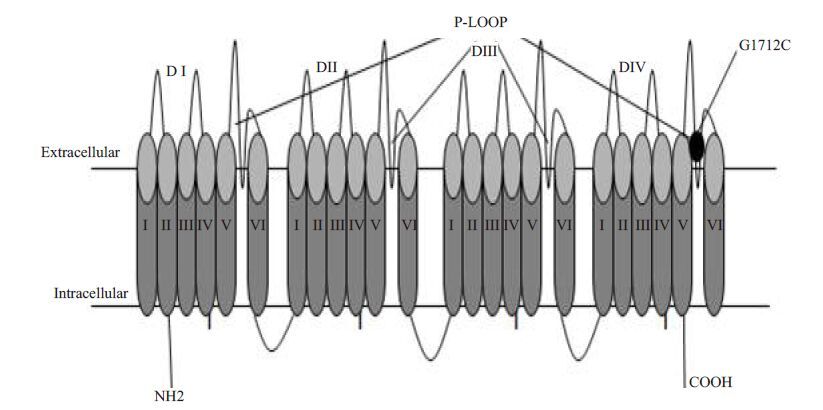
|
图 4 G1712C在心脏电压依赖Na+通道蛋白α 亚基单位突变位置:位于Ⅳ区S5与S6的P-loop环上 Figure 4 G1712C mutation in the alpha subunit of the voltage dependent sodium channel. The circle denotes theposition of the mutation. |
在激光共聚焦显微镜下,看到GFP蛋白在细胞核和细胞质中都有分布,在细胞核中居多。但GFP-SCN5A 突变体(G1712C)主要在细胞质的外周(细胞膜)中分布,突变位点成功定位到了细胞膜上。
3 讨论前期我们从一名表现为1型BrS心电图征男性患者中,已经证实G1712C为SCN5A 基因新突变位点。本次电生理分析表明,G1712C突变导致氨基酸的改变,对心脏电压依赖钠通道蛋白α亚基的关键结构P环产生了影响,导致钠通道未能正常表达,或导致细胞上正常钠通道数量大量减少,未能记录出钠电流。已有大量研究表明,BrS SCN5A基因突变会导致钠离子通道功能损坏[12-17]。目前本研究的结果初步表明,SCN5A新突变位点G1712C错义突变导致Nav1.5通道功能的损坏,可能是该家系发病的病因。
G1712C突变引起跨膜节段DⅣS5与S6之间孔道中性氨基酸负性改变,明显影响了钠通道细胞膜的表达和门控动力学功能。Nav1.5通道的中心传导孔隙由S5与S6 之间的氨基酸链形成,对离子具有选择性功能。已有许多研究探讨了错义突变对电流密度减少的影响机制。机制之一就是由特定的基因突变,影响了离子传导孔道阻碍了Na+扩散,导致钠电流明显减少[18-20]。另一种机制是突变通道无法通过高效的控制系统表达正常数量的细胞膜和细胞器,如R1432G突变[21],其免疫荧光定位主要位于细胞质,影响了内质网的表达而未能记录到电流。与同是位于S5与S6之间的R1432G突变不同的是,G1712C 突变主要位于细胞膜,这可能是G1712C未能记录到电流的机制。
本研究欲通过比较野生型和突变型钠通道IV-曲线及钠通道稳态激活曲线,反映突变型钠通道的表达是否会改变INa峰值的阈电位、峰电位、快速激活时间与速度以及反转电位。稳态失活曲线用来研究突变型钠通道是否会改变钠通道灭活的速度、时间等作用于通道灭活过程,通过比较这些钠通道的动力学特性,来反映出突变型钠通道的表达在BrS 发病机理中所起的作用。而在我们应用膜片钳软件分析SCN5A 基因突变型G1712C钠通道的研究中未能记录到钠电流。但在野生型钠通道的HEK293 细胞中记录到钠电流,且与正常钠通道的特征相符合。与同是在DⅣS5 与S6 之间的D1690N突变[19]以及与其他区域、节段的突变[22-25]所致的钠通道稳态激活和失活电流减少不一样,本研究的突变结果是导致钠通道电流的缺失,故推测此突变所致的钠通道电流的缺失可能是BrS发病机制。
本次的研究结果初次表明,SCN5A基因G1712C突变,影响了心脏钠离子通道的关键结构,导致了功能缺失。我们下一步的研究计划将进行构建多种点突变来探索可能机制。
| [1] | Brugada P, Brugada J. Right bundle branch block, persistent ST segment elevation and sudden cardiac death:a distinct clinical and electrocardiographic syndrome[J]. J Am Coll Cardiol, 1992, 20 (6): 1391-6. DOI: 10.1016/0735-1097(92)90253-J. |
| [2] | Berne P, Brugada J. Brugada syndrome 2012[J]. Circ J, 2012, 76 (7): 1563-71. DOI: 10.1253/circj.CJ-12-0717. |
| [3] | Mizusawa Y, Wilde AM. Brugada syndrome[J]. Circ Arrhythm Electrophysiol, 2012, 5 (3): 606-16. DOI: 10.1161/CIRCEP.111.964577. |
| [4] | Antzelevitch C, Brugada P, Borggrefe M, et al. Brugada syndrome - Report of the second consensus conference - Endorsed by the Heart Rhythm Society and the European Heart Rhythm Association[J]. Circulation, 2005, 111 (5): 659-70. DOI: 10.1161/01.CIR.0000152479.54298.51. |
| [5] | Juang JJ, Horie M. Genetics of brugada syndrome[J]. J Arrhythm, 2016, 32 (5): 418-25. DOI: 10.1016/j.joa.2016.07.012. |
| [6] | Wang Q, Ohno S, Ding WG, et al. Gain-of-Function KCNH2 mutations in patients with brugada syndrome[J]. J Cardiovasc Electrophysiol, 2014, 25 (5): 522-30. DOI: 10.1111/jce.12361. |
| [7] | Kapplinger JD, Tester DJ, Alders MA, et al. An international compendium of mutations in the SCN5A-encoded cardiac Sodium Channel in patients referred for Brugada syndrome genetic testing[J]. Heart Rhythm, 2010, 7 (1): 33-46. DOI: 10.1016/j.hrthm.2009.09.069. |
| [8] | Le Scouarnec S, Karakachoff M, Gourraud JB, et al. Testing the burden of rare variation in arrhythmia-susceptibility genes provides new insights into molecular diagnosis for Brugada syndrome[J]. Hum Mol Genet, 2015, 24 (10): 2757-63. DOI: 10.1093/hmg/ddv036. |
| [9] | Makiyama T, Akao M, Haruna Y, et al. Mutation analysis of the glycerol-3 phosphate dehydrogenase-1 like (GPD1L) gene in Japanese patients with Brugada syndrome[J]. Circ J, 2008, 72 (10): 1705-6. DOI: 10.1253/circj.CJ-08-0508. |
| [10] | Kattygnarath D, Maugenre S, Neyroud NA, et al. MOG1 A new susceptibility gene for brugada syndrome[J]. Circ Cardiovasc Genet, 2011, 4 (3): 261-8. DOI: 10.1161/CIRCGENETICS.110.959130. |
| [11] | 阮发晖, 孟素荣, 陈哲明, 等. Brugada综合征患者SCN5A基因突变检测[J]. 心脏杂志, 2007, 19 (2): 218-21. |
| [12] | Wang L, Meng X, Yuchi Z, et al. De Novo mutation in the SCN5A gene associated with brugada syndrome[J]. Cell Physiol Biochem, 2015, 36 (6): 2250-62. DOI: 10.1159/000430189. |
| [13] | Turker I, Makiyama T, Vatta M, et al. A novel SCN5A mutation associated with drug induced brugada type ECG[J]. PLoS One, 2016, 11 (8): e161872. |
| [14] | Garcia-Molina E, Sabater-Molina M, Munoz CA, et al. An R1632C variant in the SCN5A gene causing Brugada syndrome[J]. Mol Med Rep, 2016, 13 (6): 4677-80. |
| [15] | Ortiz-Bonnin B, Rinne S, Moss R, et al. Electrophysiological characterization of a large set of novel variants in the SCN5A-gene: identification of novel LQTS3 and BrS mutations[J]. Pflugers Arch, 2016, 468 (8): 1375-87. DOI: 10.1007/s00424-016-1844-3. |
| [16] | Kinoshita K, Takahashi H, Hata Y, et al. SCN5A(K817E), a novel Brugada syndrome-associated mutation that alters the activation gating of Na(V)1.5 Channel[J]. Heart Rhythm, 2016, 13 (5): 1113-20. DOI: 10.1016/j.hrthm.2016.01.008. |
| [17] | Zhu JF, Du LL, Tian Y, et al. Novel heterozygous mutation c.4282G>T in the SCN5A gene in a family with Brugada syndrome[J]. Exp Ther Med, 2015, 9 (5): 1639-45. |
| [18] | Amin AS, Verkerk AO, Bhuiyan ZA, et al. Novel brugada syndrome-causing mutation in ion-conducting pore of cardiac Na+ Channel does not affect ion selectivity properties[J]. Acta Physiol Scand, 2005, 185 (4): 291-301. DOI: 10.1111/j.1365-201X.2005.01496.x. |
| [19] | Zeng Z, Xie Q, Huang Y, et al. D1690N Sodium voltage-gated channel subunit 5 mutation reduced Sodium current density and is associated with brugada syndrome[J]. Mol Med Rep, 2016, 13 (6): 5216-22. |
| [20] | Calloe K, Refaat MM, Grubb S, et al. Characterization and mechanisms of action of novel Na(V)1.5 channel mutations associated with brugada syndrome[J]. Circ Arrhythm Electrophysiol, 2013, 6 (1): 177-84. DOI: 10.1161/CIRCEP.112.974220. |
| [21] | Baroudi G, Pouliot V, Denjoy I, et al. Novel mechanism for brugada SyndromeDefective surface localization of an SCN5A mutant (R1432G)[J]. Circ Res, 2001, 88 (12): E78-33. DOI: 10.1161/hh1201.093270. |
| [22] | Park DS, Cerrone M, Morley G, et al. Genetically engineered SCN5A mutant pig hearts exhibit conduction defects and arrhythmias[J]. J Clin Invest, 2015, 125 (1): 403-12. DOI: 10.1172/JCI76919. |
| [23] | Zheng J, Zhou F, Su T, et al. The biophysical characterization of the first SCN5A mutation R1512W identified in Chinese sudden unexplained nocturnal death syndrome[J]. Medicine(Baltimore), 2016, 95 (23): e3836. |
| [24] | Tan BY, Yong RY, Barajas-Martinez H, et al. A brugada syndrome proband with compound heterozygote SCN5A mutations identified from a Chinese family in Singapore[J]. Europace, 2016, 18 (6): 897-904. DOI: 10.1093/europace/euv058. |
| [25] | Uzi?b?o-?yczkowska B, Gielerak G, Siedlecki P, et al. Genetic diversity of SCN5A gene and its possible association with the concealed form of brugada syndrome development in polish group of patients[J]. Biomed Res Int, 2014, 2014 : 462609. |
 2017, Vol. 37
2017, Vol. 37

