肺炎克雷伯菌(Klebsiella pneumoniae)是一类存在于人体上呼吸道和肠道的正常菌群,可引起呼吸道、泌尿道、伤口等部位的感染[1]。目前已成为社区获得性感染和医院内感染的重要病原菌之一[2-3]。肺炎克雷伯菌有极强的生物膜形成能力。而生物膜形成能力被证明与肺炎克雷伯菌的致病性与耐药性相关[4-6],荚膜多糖(capsular polysaccharide, CPS)与黏附素(Ⅰ型菌毛与Ⅲ型菌毛)参与肺炎克雷伯菌生物膜的形成[7-9]。荚膜多糖被认为是肺炎克雷伯菌重要的毒力因子,可使细菌逃避吞噬细胞、血清补体和抗菌药物的杀菌效应,同时也是生物膜形成胞外结构的最重要成分[10]。magA(mucoviscosity-asscociated gene A)为K1血清型肺炎克雷伯菌荚膜多糖的结构基因[11]。细菌生物膜形成过程是一个多基因调控的过程,Fur [12]、CRP [13]、OxyR [14]等调控子已被证明参与细菌生物膜形成的调控。密度感应系统(quorum-sensing system, QS系统)是一种细胞密度依赖性的细菌细胞间信息传递系统,在多个细菌中均发现密度感应系统参与细菌生物膜的形成[15]。
通过STM分析发现,肺炎克雷伯菌NTUH-K2044 KP1_2626基因(KbvR基因)突变能够影响细菌生物膜的形成。生物信息学分析发现KbvR基因为LuxR家族的一类转录调控因子。LuxR家族调控蛋白是LuxR/I群体感应信号系统中的枢纽蛋白,调控许多其他蛋白的表达,从而影响整个群体感应过程[16]。虽然通过大规模筛选技术证明KbvR调控子可能参与肺炎克雷伯菌生物膜形成调控,但其调控机制仍不清楚。本研究通过敲除肺炎克雷伯菌密度感应系统LuxR家族中KbvR基因,分析该基因在肺炎克雷伯菌生长、生物膜形成及荚膜形成过程中的作用。
1 材料和方法 1.1 主要实验材料和试剂肺炎克雷伯菌NTUH-K2044来自于军事医学科学院微生物流行病研究所,大肠杆菌DH5α及自杀质粒pKO3-km,pGEM-T easy质粒为本室保存。ExTaq酶、Pfu酶、NcoⅠ酶切、SalⅠ酶切、NotⅠ酶切为Promega公司产品;DNA提取试剂盒、切胶回收试剂盒、质粒提取试剂盒、RNA提取试剂盒及RNA纯化试剂盒为Qiagen公司产品。反转录试剂盒为Invitrogen公司产品。T4 DNA连接酶、DNAmarker为大连宝生物公司产品。结晶紫为sigma公司产品。蛋白胨、酵母粉为OXOID公司产品;NaCl、琼脂粉购自北京拜尔迪生物技术有限公司;卡那霉素购自晶美公司,氨苄青霉素钠盐(Amp)、硫酸庆大霉素(Cn)和5%绵羊血平板购自武汉天源生物技术有限公司。本研究所用引物见表 1。
| 表 1 本研究用于扩增相关基因的引物 Table 1 Primers used in this study |
利用融合PCR的方法获得肺炎克雷伯菌KbvR基因上下侧翼序列的一个1669 bp的基因片段。克隆至温度敏感性自杀载体pKO3-Km上获得重组pKO3-Km-KbvR突变盒质粒,电转至肺炎克雷伯菌野生株(WT株)NTUH-K2044中获得KbvR基因缺失突变株(ΔKbvR株)[17]。然后将包含KbvR基因编码区、启动子结合区及转录终止区的1876 bp片段克隆至pGEM-T-easy载体上转化入KbvR基因缺失突变株获得回补株(C-KbvR株)[18]。提取野生株、ΔKbvR、C-KbvR RNA并逆转录为cDNA,以16sRNA为对照,检测不同菌株KbvR基因表达情况。
1.2.2 细菌生长曲线的检测将肺炎克雷伯菌WT株、ΔKbvR株、C-KbvR株接种至LB培养基,37 ℃下200 r/min培养过夜,调整A600值约1.2后100倍稀释接种到15 mL新鲜LB培养基中,37 ℃下200 r/min连续培养,每隔0.5~1 h取菌液测A600值,分析不同菌株生长速度的变化。
1.2.3 结晶紫染色定量检测KbvR在细菌生物膜形成中的作用过夜培养的肺炎克雷伯菌WT株、ΔKbvR株、C-KbvR株分别以1:100的比例转接至含3 mL新鲜LB液体培养基的试管中,室温静置培养48 h,观测不同菌株在液体培养基表面生物膜形成的情况。然后将试管中的菌液转移至另一干净的小试管中,吹打混匀,测定不同菌株的A600。而带有生物膜的原试管用3 mL去离子水洗去游离细菌后加入3 mL 0.1%的结晶紫溶液,染色30 min后弃去结晶紫染液,用去离子水润洗5次后每管加入3 mL的乙醇,间隔振荡3 min溶解试管壁上结晶紫染液后检测溶解液的A570值,每个样品重复3次,按照100×A570/A600计算每种菌株生物膜相对形成量的均值与标准差,采用t检验进行数据统计分析。
1.2.4 拉丝实验与离心试验检测KbvR对细菌荚膜形成的影响将WT株、ΔKbvR株、C-KbvR株分别接种于5%绵羊血平板,培养箱中37 ℃静置培养16 h,用200 µL灭菌枪头分别拉伸3种细菌菌落,当拉伸长度>5 mm时,判定为超黏性阳性[19-20]。同时挑取单个WT株、ΔKbvR株、C-KbvR株菌落分别接种于3 mL LB培养液中,37 ℃培养过夜,调整A600至1.0,1:50稀释接种于15 mL培养至A600 1.4。各取1 mL菌液,10 000×g离心观察细菌沉淀情况[20-21]。
RT-PCR检测KbvR对细菌荚膜结构基因的影响将WT株、ΔKbvR菌株37 ℃ 200 r/min培养至平台期,稀释至1.2后按1:100的比例接入LB培养基中,37 ℃下200 r/min培养至对数中期,取菌液各500 µL于无菌EP管内4 ℃ 5000×g离心10 min,弃上清用PBS洗菌1次,加入1 mL RNA protect混匀,裂解细菌,通过RNeasy ®Mini kit试剂盒提取细菌RNA,RNase-Free DNase Set去DNA污染,SuperSriptⅢ First-strand synthesis system逆转录成cDNA,以cDNA为模板,16sRNA为内参基因,1%琼脂糖凝胶电泳定量分析肺炎克雷伯菌KbvR敲除株与野生株比较荚膜结构基因magA的表达情况。
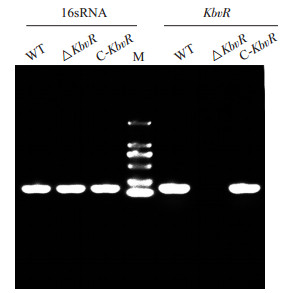
2 结果 2.1 肺炎克雷伯菌KbvR基因缺失突变株与回补株的构建通过同源重组及pKO3-Km所携带的SacB反筛基因成功的获得了肺炎克雷伯菌KbvR基因的缺失突变株。如图 1中所示,16sRNA对照在WT株、ΔKbvR株、C-KbvR株中均能检测到,KbvR基因特异引物能够在WT株中扩增到相应条带,而突变株中无KbvR表达,说明突变株中KbvR基因被成功敲除。回补株能够扩增出与野生株同样大小的条带,说明KbvR基因回补至缺失突变株中,成功构建了肺炎克雷伯菌KbvR基因回补株。
|
图 1 RT-PCR鉴定肺炎克雷伯菌KbvR突变株与回补株的电泳图 Figure 1 RT-PCR identification of Klebsiella pneumoniae KbvR deletion mutant and complementation strains. |
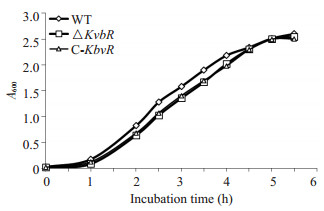
通过绘制不同菌株生长曲线,ΔKbvR株与WT株及回补株间的生长速度无明显差异(图 2),说明敲除KbvR基因不影响细菌生长。
|
图 2 不同菌株生长曲线 Figure 2 Growth of Klebsiella pneumoniae NTUH-K2044 (WT), ΔKbvR, and C-KbvR strains. |
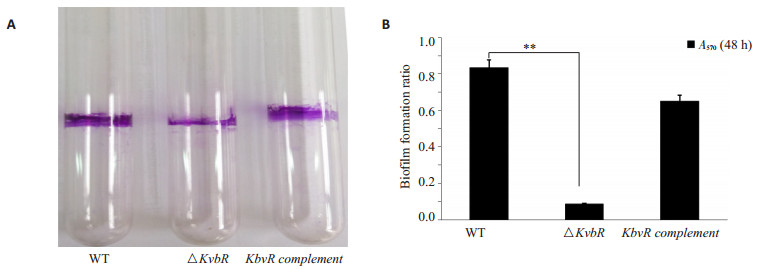
将肺炎克雷伯菌WT株、ΔKbvR株、C-KbvR株静置培养48 h后发现与WT株相比ΔKbvR株液体培养基表面生物膜形成能力明显下降(图 3A),而C-KbvR株能够形成明显的生物膜,说明回补株生物膜表型能恢复到野生株的状态,因此排除KbvR基因缺失突变的极性效应,即KbvR基因缺失突变为非极性基因突变,KbvR基因与肺炎克雷伯菌生物膜形成具有一一对应关系。结晶紫染色定量检测发现ΔKbvR株的生物膜相对形成量明显低于WT株(图 3B),说明KbvR基因正调控肺炎克雷伯菌生物膜的形成。
|
图 3 KbvR基因对肺炎克雷伯菌生物膜形成的影响 Figure 3 Effect of KbvR on Klebsiella pneumoniae biofilm formation. A: A crystal violet staining of biofilm; B: Quantitative detection of crystal violet staining. **P < 0.01. |
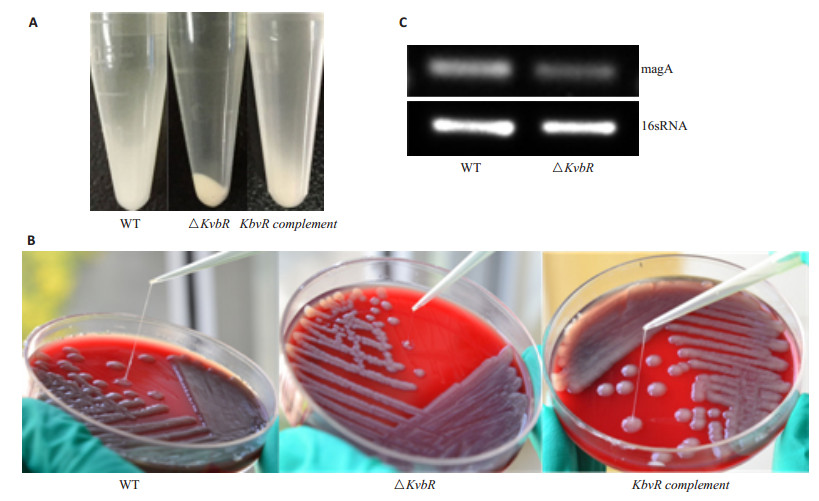
拉丝实验可见3种菌株拉伸长度均>5 mm,但与WT株及C-KbvR株相比较,ΔKbvR株的拉伸长度最短,说明KbvR基因敲除后细菌超黏性下降(图 4B)。WT株、C-KbvR株细菌离心后呈云雾状聚集于Ep管下端,ΔKbvR株细菌沉于Ep管底部(图 4A)。拉丝实验与离心试验说明ΔKbvR基因敲除后,荚膜形成能力下降。RT-PCR结果也证明了KbvR基因正调控荚膜的形成。敲除株与野生株相比较,荚膜形成结构基因magA表达量降低(图 4C)。
|
图 4 KbvR基因对肺炎克雷伯菌荚膜合成的影响 Figure 4 Effect of KbvR on Klebsiella pneumoniae capsular synthesis. A: Centrifugal test; B: String test; C: RT-PCR. |
密度感应系统(QS系统)可以调控细菌基因表达,使得细菌适应环境、产生毒力因子。QS系统在细菌生物膜形成过程中起着重要调控作用。生物膜中的细菌间可以通过阈感分子即QS系统产生的信号分子感应周围环境的变化,从而调节菌群的密度与代谢[22]。典型的QS系统由LuxI基因编码的酶催化合成的酰基-高丝氨酸内酯(acylhomoserine lactone, AHL)类信号分子与LuxR转录调控蛋白组成。但最近的研究发现许多细菌虽然拥有典型的QS系统LuxR蛋白但无相应LuxIAHL合成酶,这些蛋白称为LuxR孤儿蛋白[23]。在铜绿假单胞菌中此类LuxR孤儿蛋白PsoR能够参与众多调控通路的调控[24]。
目前肺炎克雷伯菌已成为社区获得性感染和医院内感染的重要病原菌之一。肺炎克雷伯菌生物膜形成能力与细菌的致病性和耐药性密切相关。多种转录调控因子参与肺炎克雷伯菌生物膜形成的调控[25]。肺炎克雷伯菌中无典型的LuxI/LuxR密度系统,但生物信息学分析证明在肺炎克雷伯菌中存在6个典型的LuxR蛋白,包括已知的三型菌毛合成调控因子MrkI [26]及调控因子KvhA [27]。KP1-2626(KbvR基因)为具有HTH结构的LuxR超家族一员,与大肠杆菌科的HTH转录调控因子家族同源。KbvR为LuxR孤儿蛋白,其在肺炎克雷伯菌生长与致病中的作用不清楚,本研究通过构建KbvR基因的缺失突变株及回补株分析KbvR基因在肺炎克雷伯菌生长、生物膜形成、荚膜形成中的作用。细菌生长曲线实验证明KbvR基因不影响细菌生长速度,结晶紫定量实验发现KbvR基因敲除菌株生物膜形成能力明显下降,回补株C-KbvR的生物膜形成能力可以部分恢复到野生株,排除基因缺失突变的极性效应,说明KbvR正调控生物膜的形成。目前已经知道的能够影响细菌生物膜形成能力的因素主要包括荚膜、细菌黏附结构及LPS。荚膜为生物膜形成胞外结构的最重要成分。通过拉丝实验和离心实验发现KbvR基因敲除株的超黏性下降。肺炎克雷伯菌的超粘性与细菌的荚膜相关,说明KbvR敲除后细菌荚膜形成能力下降。定量PCR实验发现KbvR基因敲除株的荚膜合成基因magA表达下降, 证明KbvR基因正调控荚膜形成,说明KbvR可能通过影响荚膜的形成而影响生物膜的形成。其具体调控靶标及精细调控机制有待下一步研究。
致谢: 感谢军事医学科学院微生物流行病研究所,湖北医药学院基础研究所和李蓓导师在我的研究生生活学习中的大力支持。| [1] | Qu TT, Zhou JC, Jiang Y, et al. Clinical and microbiological characteristics of Klebsiella pneumoniae liver abscess in East China[J]. BMC Infect Dis,2015, 15 : 161. DOI: 10.1186/s12879-015-0899-7. |
| [2] | Ko WC, Paterson DL, Sagnimeni AJ, et al. Community-acquired Klebsiella pneumoniae bacteremia:global differences in clinical patterns[J]. Emerg Infect Dis,2002, 8 (2) : 160-6. DOI: 10.3201/eid0802.010025. |
| [3] | Gupta A. Hospital-acquired infections in the neonatal intensive care unit——Klebsiella pneumoniae[J]. Semin Perinatol,2002, 26 (5) : 340-5. DOI: 10.1053/sper.2002.36267. |
| [4] | Laverty G, Gorman SP, Gilmore BF. Biomolecular mechanisms of pseudomonas aeruginosa and escherichia coli biofilm formation[J]. Pathogens,2014, 3 (3) : 596-632. DOI: 10.3390/pathogens3030596. |
| [5] | Donlan RM, Costerton JW. Biofilms:survival mechanisms of clinically relevant microorganisms[J]. Clin Microbiol Rev,2002, 15 (2) : 167-93. DOI: 10.1128/CMR.15.2.167-193.2002. |
| [6] | Davies D. Understanding biofilm resistance to antibacterial agents[J]. Nat Rev Drug Discov,2003, 2 (2) : 114-22. DOI: 10.1038/nrd1008. |
| [7] | Shon AS, Bajwa RP, Russo TA. Hypervirulent (hypermucoviscous) Klebsiella pneumoniae:a new and dangerous breed[J]. Virulence,2013, 4 (2) : 107-18. DOI: 10.4161/viru.22718. |
| [8] | Struve C, Bojer M, Krogfelt KA. Identification of a conserved chromosomal region encoding Klebsiella pneumoniae type 1 and type 3 fimbriae and assessment of the role of fimbriae in pathogenicity[J]. Infect Immun,2009, 77 (11) : 5016-24. DOI: 10.1128/IAI.00585-09. |
| [9] | Boddicker JD, Anderson RA, Jagnow J, et al. Signature-tagged mutagenesis of Klebsiella pneumoniae to identify genes that influence biofilm formation on extracellular matrix material[J]. Infect Immun,2006, 74 (8) : 4590-7. DOI: 10.1128/IAI.00129-06. |
| [10] | Li B, Zhao YL, Liu CT, et al. Molecular pathogenesis of Klebsiella pneumoniae[J]. Future Microbiol,2014, 9 (9) : 1071-81. DOI: 10.2217/fmb.14.48. |
| [11] | Fang FC, Sandler N, Libby SJ. Liver abscess caused by magA +Klebsiella pneumoniae in North America[J]. J Clin Microbiol,2005, 43 (2) : 991-2. DOI: 10.1128/JCM.43.2.991-992.2005. |
| [12] | Lin CT, Wu CC, Chen YS, et al. Fur regulation of the capsular polysaccharide biosynthesis and iron-acquisition systems in Klebsiella pneumoniae CG43[J]. Microbiology,2011, 157 (Pt 2) : 419-29. |
| [13] | Lin CT, Chen YC, Jinn TR, et al. Role of the cAMP-dependent Carbon catabolite repression in capsular polysaccharide biosynthesis in Klebsiella pneumoniae[J]. PLoS One,2013, 8 (2) : e54430. DOI: 10.1371/journal.pone.0054430. |
| [14] | Hennequin C, Forestier C. oxyR, a LysR-type regulator involved in klebsiella pneumoniae mucosal and abiotic colonization[J]. Infect Immun,2009, 77 (12) : 5449-57. DOI: 10.1128/IAI.00837-09. |
| [15] | De Araujo C, Balestrino D, Roth L, et al. Quorum sensing affects biofilm formation through lipopolysaccharide synthesis in Klebsiella pneumoniae[J]. Res Microbiol,2010, 161 (7) : 595-603. DOI: 10.1016/j.resmic.2010.05.014. |
| [16] | Fuqua WC, Winans SC, Greenberg EP. Quorum sensing in bacteria:the LuxR-LuxI family of cell density-responsive transcriptional regulators[J]. J Bacteriol,1994, 176 (2) : 269-75. |
| [17] | 郑艳, 刘喜朋, 刘建华. 外源载体高效转化肺炎克雷伯菌的新途径[J]. 微生物学报,2007, 47 (4) : 721-4. |
| [18] | 王景, 穆媛媛, 黎庶, 等. 革兰阴性菌基因敲除载体的构建及其应用[J]. 第三军医大学学报,2009, 31 (23) : 2299-301. |
| [19] | Fang CT, Chuang YP, Shun CT, et al. A novel virulence gene in Klebsiella pneumoniae strains causing primary liver abscess and septic metastatic complications[J]. J Exp Med,2004, 199 (5) : 697-705. DOI: 10.1084/jem.20030857. |
| [20] | Hsu CR, Lin TL, Chen YC, et al. The role of Klebsiella pneumoniae rmpA in capsular polysaccharide synthesis and virulence revisited[J]. Microbiology,2011, 157 (Pt 12) : 3446-57. |
| [21] | Lai YC, Peng HL, Chang HY. RmpA2, an activator of capsule biosynthesis in Klebsiella pneumoniae CG43, regulates K2 cps gene expression at the transcriptional level[J]. J Bacteriol,2003, 185 (3) : 788-800. DOI: 10.1128/JB.185.3.788-800.2003. |
| [22] | Atkinson S, Williams P. Quorum sensing and social networking in the microbial world[J]. J R Soc Interface,2009, 6 (40) : 959-78. DOI: 10.1098/rsif.2009.0203. |
| [23] | Subramoni S, Venturi V. LuxR-Family'solos':bachelor sensors/regulators of signalling molecules[J]. Microbiology,2009, 155 (Pt 5) : 1377-85. |
| [24] | Latifi A, Foglino M, Tanaka K, et al. A hierarchical quorum-sensing cascade in Pseudomonas aeruginosa links the transcriptional activators LasR and RhIR (VsmR) to expression of the stationary-phase sigma factor RpoS[J]. Mol Microbiol,1996, 21 (6) : 1137-46. DOI: 10.1046/j.1365-2958.1996.00063.x. |
| [25] | Wu MC, Lin TL, Hsieh PF, et al. Isolation of genes involved in biofilm formation of a Klebsiella pneumoniae strain causing pyogenic liver abscess[J]. PLoS One,2011, 6 (8) : e23500. DOI: 10.1371/journal.pone.0023500. |
| [26] | Wilksch JJ, Yang J, Clements A, et al. MrkH, a novel c-di-GMPdependent transcriptional activator, controls Klebsiella pneumoniae biofilm formation by regulating type 3 fimbriae expression[J]. PLoS Pathog,2011, 7 (8) : e1002204. DOI: 10.1371/journal.ppat.1002204. |
| [27] | Lin CT, Peng HL. Regulation of the homologous Two-Component systems KvgAS and KvhAS in klebsiella pneumoniae CG43[J]. J Biochem,2006, 140 (5) : 639-48. DOI: 10.1093/jb/mvj196. |
 2016, Vol. 36
2016, Vol. 36
