2. 广东省第二人民医院消化内科,广东 广州510317 ;
3.广东省妇幼保健院新生儿科,广东 广州 510010
2. Department of Gastroenterology, Guangdong Second Provincial People's Hospital, Guangzhou 510317, China ;
Department of Neonatology, Guangdong Women and Children's Hospital, Guangzhou 510010, China
生物节律是一种内生的、自我维持的节律,运作周期为24 h,以维持睡眠-清醒周期、行为、代谢、激素分泌、细胞周期等生物行为以适当的节奏在体内运转[1]。大量临床病例和实验数据表明,哺乳动物体内生物节律的扰乱,生物节律关键基因表达异常可导致肿瘤发生[2]。NPAS2(neuronal PAS domain protein 2)又名MOP4,是调控生物基因节律的重要基因之一,并参与调节细胞生长、分化等过程[3]。研究证实NPAS2在除睾丸之外的人体所有周围组织中广泛表达,在中枢神经系统主要分布于视神经交叉核中的生物节律中心[4-5]。NPAS2的表达与多种节律相关疾病相关,如高血压、代谢综合征、心脑血管疾病等[6]。最新研究表明NPAS2在肿瘤组织中存在异常表达。NPAS2高表达与乳腺癌患者预后相关,可促进患者5年生存率,NPAS2可作为判断乳腺癌患者预后的指标之一[7];Rana等人发现NPAS2基因位点rs2305160单核苷酸多态性与人类慢性淋巴细胞白血病密切相关[8]。目前国内外关于NPAS2在结直肠癌中的作用和意义研究较少,本研究拟通过研究生物节律基因NPAS2在结直肠癌及癌旁组织中的表达,分析NPAS2表达对结直肠癌患者生存率的影响,探讨NPAS2这一生物节律基因在预测结直肠癌患者预后中的价值。
1 材料和方法 1.1 标本收集结直肠癌新鲜标本:收集广东省第二人民医院普外科2015年1月~2015年8月结直肠癌术后新鲜标本40例,及其对应距肿块6cm外切缘的癌旁组织(均经病理证实)。标本置于液氮中保存备用。结直肠癌及癌旁组织蜡块:调取广东省第二人民医院病理科2005年2月~2010年8月结直肠癌及相应癌旁组织蜡块120例(均经病理证实)。其中男性79例,女性41例;年龄43~79岁,中位年龄约为57.5岁;按分化程度分级:高、中分化57例,低分化63例;浸润至黏膜及黏膜下层45例,浸润深度至肌层及浆膜75例;36例患者肿瘤大于5 cm,84例小于或等于5 cm。74例淋巴结转移,23例有远处转移;此外TNMⅠ期及Ⅱ期68例,Ⅲ期及Ⅳ期52例。患者术前均未进行放化疗。标本均经10%中性福尔马林固定和常规石蜡包埋,4μm厚连续切片,烤干后备用。
随访资料收集:对以上120例结直肠癌患者进行电话、邮件随访,截止到2015年3月,共收集到78例有效随访病例,按免疫组化染色强度和范围打分(方法如下),以中位数为界,将其分为NPAS2高表达组及低表达组各39例。所有随访病例总体生存率为58.9%。
1.2 试剂和方法 1.2.1 试剂兔抗人NPAS2多克隆抗体购自美国Santa cruz公司;二抗SABC试剂盒及DAB显色试剂盒购自北京中杉金桥生物科技有限公司。总RNA提取试剂RNAiso plus及RT-qPCR sybr green I试剂盒购自日本TAKARA公司;异丙醇,氯仿,无水乙醇购自广州化学试剂有限公司。NPAS2及GAPDH引物由Invitrogen公司合成;RT-qPCR使用美国ABI 7500型荧光定量qPCR仪。
1.2.2 方法RNA提取:从液氮罐中取出组织标本,低温下碾碎,后加入1 mL RNAiso,充分混匀。每个样品加入200μL氯仿,用力震荡后,静置5 min,12 000 r/min 4℃离心10 min;吸取上清至另一EP管中,并加入等体积异丙醇,轻混后静置15 min;12 000 r/min 4℃离心10 min,肉眼可见沉淀即为RNA。1 mL 75%乙醇清洗RNA,12 000 r/min 4℃离心10 min,弃上清,空气干燥RNA,后加20μL DEPC水溶解后,检测RNA浓度。
逆转录:按照Takara逆转录试剂盒说明配置逆转录体系,将RNA逆转成cDNA。
RT-qPCR:按照Takara RT-qPCR sybr green I试剂盒说明书配置体系。引物如下:forward:ACACCCT TTCAAGACCTTGCC,reverse:AGGTTCGTCAACT ATGCACATTT;GAPDH forward:GGAGCGAGATC CCTCCAAAAT,reverse:GGCTGTTGTCATACTTC TCATGG。反应程序设置为,94℃2 min;94℃30 s,60℃30 s,72℃30 s,共40个循环;72℃5min。每个样品设置3个复孔。反应结束后由软件计算得出定量结果。
免疫组织化学SABC法:石蜡切片常规切片烤干后,脱蜡至水,SABC法按照中杉金桥SABC试剂盒说明书操作;NPAS2一抗浓度为:NPAS2:1:200;以PBS作为阴性对照。
1.3 结果判断 1.3.1 RT-qPCR结果判断读取每个样品模板的Ct值(每个反应管内荧光信号达到设定的阈值所经历的循环数),计算△Ct=Ct(目的基因)-Ct(内参基因),△Ct值越小,表示原始RNA样品中目的基因mRNA表达越高[9]。
1.3.2 免疫组织化学结果判断NPAS2蛋白在结直肠癌及癌旁组织中主要定位于细胞浆中,以细胞浆中呈清晰棕黄色颗粒为阳性表达;采用半定量积分法判断阳性结果[10]。(1)按染色强度评分:无阳性着色为0分,浅黄色为1分,棕黄色为2分,棕褐色为3分;(2)高倍镜(×400)下随机对每张切片取10个视野,每个视野计数100个细胞,计算阳性细胞占同类细胞的百分数:无阳性细胞为0分,阳性细胞数< 25%为1分,25%~50%为2分,> 50%为3分。两项评分相乘,< 2记为阴性,> 2记为阳性。
1.4 统计学方法采用SPSS 20.0及Graphpad prism 6.02软件进行统计学分析。配对t检验分析RT-qPCR结果;免疫组织化学结果采用χ2分析,Spearman等级相关性分析指标间相关性;生存分析采用Kaplan-Meier生存分析法;P≤0.05为具有显著统计学差异。
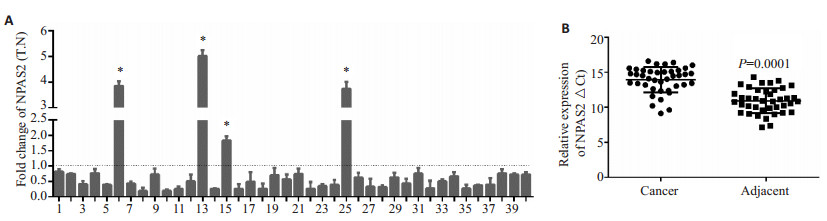
2 结果 2.1 RT-qPCR检测结直肠癌组织及癌旁组织中NPAS2 mRNA的表达采用实时荧光定量PCR检测40例结直肠癌及相应癌旁组织中NPAS2 mRNA表达,结果发现:40对结直肠癌组织中,只有4对癌组织NPAS2 mRNA表达倍数高于对应的癌旁组织(图 1A,P < 0.05),结直肠癌组织中NPAS2 mRNA表达△Ct值高于对应的癌旁组织(图 1B,P < 0.01),这些结果表明:结直肠癌组织中NPAS2 mRNA表达显著低于相应的癌旁组织。
|
图 1 结直肠癌组织及癌旁组织中NPAS2 mRNA的表达 Figure 1 Expression of NPAS2 mRNA in fresh colorectal cancer and paired adjacent tissues. A: T/N; B:△Ct; *: T/N>1. |
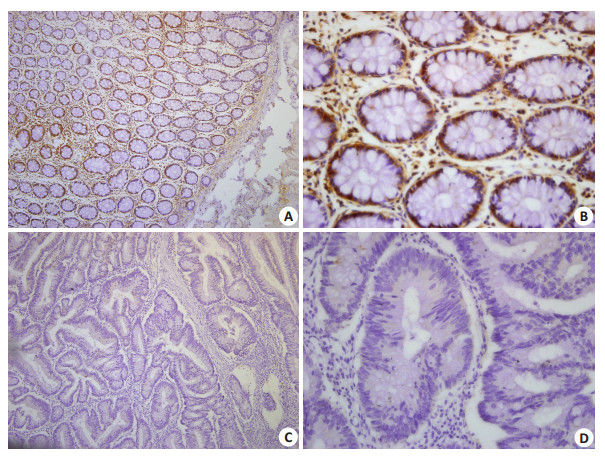
为了进一步说明结直肠癌和癌旁组织NPAS2蛋白水平的表达差异,我们采用了免疫组织化学SABC法分别检测二者在120例肿瘤及癌旁组织中的表达。结果显示:NPAS2表达于细胞浆中,阳性细胞呈黄色、棕黄色颗粒状(图 2)。NPAS2在结直肠癌中表达阳性率为19.2%(23/120),而对应癌旁组织阳性率为62.5%(75/ 120),差异具有显著统计学意义(P < 0.05)。
|
图 2 结直肠癌及癌旁组织中NPAS2蛋白的表达 Figure 2 Expression of NPAS2 in paraffin-embedded CRC and paired adjacent tissues (Immunohistochemistry with SABC method). A: Adjacent tissue (Original magnification:×100); B: Adjacent tissue (×400); C: CRC tissue (×100); D: CRC tissue (×400). |
根据免疫组织化学结果及卡方分析的方法,分析NPAS2的表达与结直肠癌临床病理学参数的关系,结果表明:结直肠癌组织中NPAS2的表达与肿瘤大小,淋巴结转移及TNM分期密切相关(P < 0.05,表 1),提示NPAS2可能是结直肠癌恶性进程的重要基因。
| 表 1 NPAS2表达与结直肠癌临床病理学参数的相关性 Table 1 Clinicopathologic characteristics and NPAS2 expression in CRC patients |
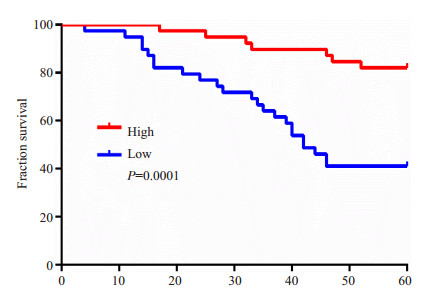
采用Kaplan-Meier生存分析法对NPAS2的表达水平与结直肠癌患者预后关系进行比较。结果表明,NPAS2高表达组患者5年生存率显著高于低表达组,两组差异明显(图 3,P=0.0001);NPAS2的表达与患者生存率呈正相关,NPAS2高表达组患者生存率为82.05%(32/39),显著高于NPAS2低表达组的35.89%(14/39)(P < 0.01)。
|
图 3 NPAS2表达水平与结直肠癌患者生存曲线比较 Figure 3 Survival analysis of colorectal cancer patients with different NPAS2 expression levels. |
结直肠癌的发生发展与遗传、环境及肠道菌群等多种综合因素有关,但具体的机制仍有待进一步研究[11]。目前研究表明,生物节律改变及生物节律相关基因功能或表达异常是结直肠癌发生发展的重要原因之一[12-14]。生物节律基因CRY2在化疗抵抗的结直肠癌标本中高表达,并且与患者预后密切相关,敲除CRY2可增强结直肠癌细胞对奥沙利铂的敏感性[15]。Oshima等[16]研究发现,Clock和Bmal1在结直肠癌组织中高表达,而PER1,PER3低表达,其中Bmal1和PER1在结直肠癌中的异常表达与肿瘤的肝转移密切相关,可作为预测结直肠癌肝转移的指标。
NPAS2是目前人类发现的最大的生物节律基因,属于转录因子bHLH-PAS家族[17]。人NPAS2基因定位于人染色体2p11.2-2q13上。NPAS2多肽链由622个氨基酸组成,N端存在一个bHLH和两个PAS结构域,介导蛋白质间相互作用,促进蛋白二聚体的形成;C端由核受体连接区构成,调节NPAS2与相关核受体的结合[18]。研究表明,NPAS2不仅参与调节生物节律,而且与肿瘤的发生发展密切相关[1-2]。Hoffman等[19]干扰NPAS2表达后,发现细胞周期及DNA损失修复相关基因表达受到抑制,并且NPAS2表达与乳腺癌、非霍奇金淋巴瘤相关。NPAS2基因中的两个SNP位点,rs1053096和rs2305160的多态性与肝细胞癌患者的生存率相关性显著[20]。近年来,有研究报道NPAS2在肿瘤生长抑制等方面也中发挥重要作用,可能是一种新的抑癌基因[21-22]。
NPAS2在结直肠癌中的作用和机制研究较少。Xue等人干扰结直肠癌细胞系DLD1中NPAS2的表达后,可显著促进细胞的增殖、浸润及划痕愈合能力,并且在结直肠癌组织中NPAS2低表达,与肿瘤大小、远处转移及TNM分期密切相关[23]。国内外尚没有NPAS2表达与结直肠癌患者生存率相关性的分析报道。本实验中,我们采用RT-qPCR的方法检测40对结直肠癌及配对癌旁组织中NPAS2 mRNA的表达,发现NPAS2 mRNA在结直肠癌组织中表达显著低于相应癌旁组织;免疫组织化学结果显示,在蛋白水平上,NPAS2在肿瘤中的表达也低于配对的癌旁组织。这些结果提示,结直肠癌组织中,NPAS2存在低表达现象。统计学分析表明,结直肠癌组织中NPAS2低表达与肿瘤大小、淋巴结转移及TNM分期密切相关,这与文献报道基本吻合;此外Kaplan-Meier生存分析表明,NPAS2高表达结直肠患者的5年生存率显著高于低表达患者。这些结果表明,结直肠癌中NPAS2可能起抑癌基因的作用,并且NPAS2可作为判断结直肠癌患者预后及生存期的指标。
生物节律相关基因在肿瘤中的作用及机制,以生物节律基因为靶点设计肿瘤治疗方法,是当前肿瘤研究领域的热点之一[2]。本研究通过实验表明NPAS2在结直肠癌中的作用和意义,为临床了解生物节律与结直肠癌相关性及设计结直肠癌治疗方案提供有力的理论参考。
| [1] |
Masri S, Kinouchi K, Sassone-Corsi PC, et al. Circadian clocks, epigenetics, and cancer[J].
Curr Opin Oncol,2015, 27 (1) : 50-6.
DOI: 10.1097/CCO.0000000000000153. ( 0) 0)
|
| [2] |
Fu L, Lee CC. The circadian clock: pacemaker and tumour suppressor[J].
Nat Rev Cancer,2003, 3 (5) : 350-61.
DOI: 10.1038/nrc1072. ( 0) 0)
|
| [3] |
Franken P, Dudley CA, Estill SJ, et al. NPAS2 as a transcriptional regulator of non-rapid eye movement sleep: genotype and sex interactions[J].
Proc Natl Acad Sci USA,2006, 103 (18) : 7118-23.
DOI: 10.1073/pnas.0602006103. ( 0) 0)
|
| [4] |
Wu X, Wiater MF, Ritter S. NPAS2 deletion impairs responses to restricted feeding but not to metabolic challenges[J].
Physiol Behav,2010, 99 (4) : 466-71.
DOI: 10.1016/j.physbeh.2009.12.010. ( 0) 0)
|
| [5] |
Reick M, Garcia JA, Dudley C, et al. NPAS2: an analog of clock operative in the mammalian forebrain[J].
Science,2001, 293 (5529) : 506-9.
DOI: 10.1126/science.1060699. ( 0) 0)
|
| [6] |
Englund A, Kovanen L, Saarikoski ST, et al. NPAS2 and PER2 are linked to risk factors of the metabolic syndrome[J].
J Circadian Rhythms,2009, 7 : 5.
DOI: 10.1186/1740-3391-7-5. ( 0) 0)
|
| [7] |
Yi C, Mu L, De La Longrais IA, et al. The circadian gene NPAS2 is a novel prognostic biomarker for breast cancer[J].
Breast Cancer Res Treat,2010, 120 (3) : 663-9.
DOI: 10.1007/s10549-009-0484-0. ( 0) 0)
|
| [8] |
Rana S, Shahid A, Ullah H, et al. Lack of association of the NPAS2 gene Ala394Thr polymorphism (rs2305160: G > A) with risk of chronic lymphocytic leukemia[J].
Asian Pac J Cancer Prev,2014, 15 (17) : 7169-74.
DOI: 10.7314/APJCP.2014.15.17.7169. ( 0) 0)
|
| [9] |
张磊, 孙雪竹, 孙景洲. SDF-1/CXCR4及VEGF在结直肠腺癌中的表达及其意义[J].
中国免疫学杂志,2015 (5) : 674-7.
( 0) 0)
|
| [10] |
Kawasaki H, Altieri DC, Lu CD, et al. Inhibition of apoptosis by survivin predicts shorter survival rates in colorectal cancer[J].
Cancer Res,1998, 58 (22) : 5071-4.
( 0) 0)
|
| [11] |
Paschos KA, Canovas D, Bird NC. The role of cell adhesion molecules in the progression of colorectal cancer and the development of liver metastasis[J].
Cell Signal,2009, 21 (5) : 665-74.
DOI: 10.1016/j.cellsig.2009.01.006. ( 0) 0)
|
| [12] |
Karantanos T, Theodoropoulos G, Gazouli M, et al. Association of the clock genes polymorphisms with colorectal cancer susceptibility[J].
J Surg Oncol,2013, 108 (8) : 563-7.
DOI: 10.1002/jso.23434. ( 0) 0)
|
| [13] |
Mazzoccoli G, Vinciguerra M, Papa G, et al. Circadian clock circuitry in colorectal cancer[J].
World J Gastroenterol,2014, 20 (15) : 4197-207.
DOI: 10.3748/wjg.v20.i15.4197. ( 0) 0)
|
| [14] |
Karantanos T, Theodoropoulos G, Pektasides D, et al. Clock genes: their role in colorectal cancer[J].
World J Gastroenterol,2014, 20 (8) : 1986-92.
DOI: 10.3748/wjg.v20.i8.1986. ( 0) 0)
|
| [15] |
Fang L, Yang Z, Zhou J, et al. Circadian clock gene CRY2 degradation is involved in chemoresistance of colorectal cancer[J].
Mol Cancer Ther,2015, 14 (6) : 1476-87.
DOI: 10.1158/1535-7163.MCT-15-0030. ( 0) 0)
|
| [16] |
Oshima T, Takenoshita S, Akaike M, et al. Expression of circadian genes correlates with liver metastasis and outcomes in colorectal cancer[J].
Oncol Rep,2011, 25 (5) : 1439-46.
( 0) 0)
|
| [17] |
Yi CH, Zheng T, Leaderer D, et al. Cancer-related transcriptional targets of the circadian gene NPAS2 identified by genome-wide ChIP-on-chip analysis[J].
Cancer Lett,2009, 284 (2) : 149-56.
DOI: 10.1016/j.canlet.2009.04.017. ( 0) 0)
|
| [18] |
Zhou YD, Barnard M, Tian H, et al. Molecular characterization of two mammalian bHLH-PAS domain proteins selectively expressed in the central nervous system[J].
Proc Natl Acad Sci USA,1997, 94 (2) : 713-8.
DOI: 10.1073/pnas.94.2.713. ( 0) 0)
|
| [19] |
Zhu Y, Leaderer D, Guss C, et al. Ala394Thr polymorphism in the clock gene NPAS2: a circadian modifier for the risk of nonHodgkin's lymphoma[J].
Int J Cancer,2007, 120 (2) : 432-5.
DOI: 10.1002/(ISSN)1097-0215. ( 0) 0)
|
| [20] |
Yuan P, Wang S, Zhou F, et al. Functional polymorphisms in the NPAS2 gene are associated with overall survival in transcatheter arterial chemoembolization-treated hepatocellular carcinoma patients[J].
Cancer Sci,2014, 105 (7) : 825-32.
DOI: 10.1111/cas.2014.105.issue-7. ( 0) 0)
|
| [21] |
Bertolucci C, Cavallari N, Colognesi I, et al. Evidence for an overlapping role of CLOCK and NPAS2 transcription factors in liver circadian oscillators[J].
Mol Cell Biol,2008, 28 (9) : 3070-5.
DOI: 10.1128/MCB.01931-07. ( 0) 0)
|
| [22] |
Wang F, Zou YF. Further studies based on better design are needed to explore the association of NPAS2 gene polymorphisms with breast cancer[J].
Breast Cancer Res Treat,2011, 127 (2) : 565-8.
DOI: 10.1007/s10549-010-1341-x. ( 0) 0)
|
| [23] |
Xue X, Liu F, Han Y, et al. Silencing NPAS2 promotes cell growth and invasion in DLD-1 cells and correlated with poor prognosis of colorectal cancer[J].
Biochem Biophys Res Commun,2014, 450 (2) : 1058-62.
DOI: 10.1016/j.bbrc.2014.06.104. ( 0) 0)
|
 2016, Vol. 36
2016, Vol. 36